冷水鱼肠道乳酸菌的遗传差异分析
2017-12-06,,,,
,,, ,
(石河子大学食品学院,新疆石河子 832000)
冷水鱼肠道乳酸菌的遗传差异分析
黄丽丽,罗宝龙,倪永清,张艳,周红*
(石河子大学食品学院,新疆石河子 832000)
为分析新疆额尔齐斯河流域及黑龙江流域冷水鱼肠道中乳酸菌遗传差异,为乳酸菌资源的开发奠定基础。本实验利用MRS、Elliker、M17培养基对冷水鱼肠道中低温乳酸菌进行分离鉴定,并测定其最适生长温度。根据16S rRNA基因序列初步确定低温乳酸菌的系统发育关系,并利用rep-PCR指纹图谱技术进一步区分高度同源性菌株。从冷水鱼的肠道中分离得到134 株低温乳酸菌,其最适生长温度在15~24 ℃之间。16S rRNA测序结果表明,这些菌株分别隶属于Lactobacillus、Lactococcus、Enterococcus、Streptococcus、Leuconostoc、Weissella、Carnobacterium7个属,19 个种,其中Lactobacillus为优势菌。Rep-PCR指纹图谱分析表明同一属的乳酸菌在种水平及同一种的不同菌株之间存在不同程度的遗传差异。
冷水鱼肠道,低温乳酸菌,指纹图谱
乳酸菌广泛存在于自然界中,且种类繁多,生存于不同区域、不同生境的乳酸菌也各有所异。据报道在乳制品、腌制泡菜、发酵肉制品及人类和动物肠道内都存在着各种各样的乳酸菌[1-4],包括乳杆菌、明串珠菌、魏斯氏和肠球菌等在内,目前发现的乳酸菌约有41 个属,320 个种[5]。乳酸菌被认为是重要的微生物资源,其功能特征一直都是科学界关注的热点[6]。现有研究显示,以乳酸菌为代表的益生菌代谢产生细菌素等活性物质[7-8],能安全、有效地抑制食品中腐败菌及病原菌的生长[9-11],以保持食品的营养价值[1,10,12],可作为天然的生物防腐剂直接应用于食品工业中[2,13]。乳酸菌通常耐酸且嗜酸,可在较低的pH环境下生长,能耐受脊椎动物和鱼类胃肠道的酸性环境[5]。因而,益生乳酸菌也常添加于动物饲料中作为一种活性微生态制剂,不仅能通过拮抗病原菌以维持水产动物肠道微生态平衡,促进动物的健康生长,而且能提高动物的免疫力和生产性能[14]。
许多外界的微生物(如鱼类所生活的水体环境中的微生物)通过鱼类摄食而定植于鱼肠道内,从而构成了复杂的肠道微生态系统[15]。冷水鱼常年生活在水温较低(15~20 ℃)的环境中,其肠道内形成了独特的低温环境,为低温菌提供了良好的生存保障。低温环境下的乳酸菌因生长温度相对较低,被称为低温乳酸菌。低温乳酸菌是一类特殊益生资源,其在0 ℃也可以生长[16]。目前国外有关低温乳酸菌的研究主要集中在泡菜、低温冷藏海鲜产品和肉制品[17-19]中,而国内关于低温乳酸菌的研究并不多见。近年来不少学者研究发现在低温发酵的泡菜(sikhae和kimchi)中,乳酸菌是主要的优势微生物,其生长代谢的生物活性物质,能抑制食源性腐败菌的生长,进而维持泡菜风味以延长食品的保质期[17]。Matamoro等利用从海产品中分离所得的低温乳酸菌进行抑菌实验,发现其对李斯特菌、葡萄球菌、假单胞菌、沙雷菌等病原菌有不同程度的抑制作用,这对于海产品在低温条件下贮藏具有重大意义[19]。近来发现,冷冻肉制品在真空或微氧的低温下包装后,体系中的微生物会慢慢地向CO2耐受型群系发展,占主导地位的群体是乳酸菌(主要包括乳杆菌[20]和明串珠菌[21]),乳酸菌的增长会延长冷藏肉制品货架期。
低温乳酸菌在提高食品风味及食品生物防腐保鲜方面极具潜力,有着更为广泛的应用前景,但其并未得到充分的开发和研究。本研究主要是以生长在新疆额尔齐斯河流域和黑龙江流域的野生冷水鱼为实验对象,从冷水鱼肠道中分离筛选低温乳酸菌,对低温乳酸菌的遗传差异进行研究,为低温乳酸菌的应用提供科学依据。
1 材料和方法
1.1材料与仪器
样品 2014 年7月采集新疆阿勒泰地区额尔齐斯河流域(水温16~20 ℃,47°00′~49°10′ N,85°31′~90°31′ E)冷水鱼:东方欧鳊Abramisbramaorientalis(OB),高白鲑Coregonuspeled(GB),河鲈Percafluviatilis(HL),乔尔泰Esoxlucius(QE),丁鲑Tincaeus(DG),及同纬度黑龙江流域(水温13~18 ℃43°06′~48°17′ N,129°10′~137°53′ E)冷水鱼:鳜鱼Sinipercachuatsi(GY),黑斑狗鱼Esoxreicherti(HB),大马哈鱼Oncorhynchusketa(MH),细鳞鱼Brachymystaxlenok(XL),红尾鱼Rasboraborapetensis(HW),每种鱼2~3 条,体重1.5~2.8 kg,年龄约3 年,标记后置于4 ℃冰箱内冷藏,12 h内运回实验室;琼脂糖 美国BBI公司;MRS、Elliker、M17培养基 海博生物技术有限公司。
PCR仪 德国Biometra公司;凝胶成像仪 BioRad公司。
1.2实验方法
1.2.1 样品前处理 首先用75% 的酒精对冷水鱼样品进行漂洗,然后在超净工作台上解剖(解剖器具经过灭菌处理)冷水鱼样品,完整地取出其肠道,并用75%酒精漂洗肠道外表面,待酒精晾干后,获取肠道内容物及粘膜,并置于离心管中,4 ℃条件下保存备用。
1.2.2 菌株的分离 取1 g鱼肠道内容物和粘膜样品加入100 mL无菌生理盐水中,16 ℃恒温摇床上振荡24 h,使肠道样品充分散。取100 μL 菌悬液置于900 μL无菌水中,混匀后进行梯度的稀释,然后选取10-3~10-7等梯度的悬液100 μL均匀涂布于MRS、Elliker、M17培养基上,16 ℃条件下培养3~5 d。
1.2.3 菌株DNA提取 根据Juste等人改良后的CTAB[22]法提取单菌落DNA,经0.8%琼脂糖凝胶电泳检测后置于-20 ℃保存。
1.3菌株最适生长温度的测定
初筛菌株按2%的量接种于MRS液体培养基中,分别置于4、10、15、18、21、24、30、37 ℃条件下培养24 h,随后在600 nm 测定其OD 值。
1.416SrRNA基因PCR扩增及其系统发育分析
采用细菌16S rRNA基因通用引物27F(5′-AGAGTTTGATCCTGGCTCA-3′)和1492R(5′-GGTTACCTTGTTACGACTT-3′)进行PCR扩增,反应体系:2×Taq MasterMix 12.5 μL,引物各0.5 μL(0.4 μM/mL),菌株DNA 2 μL,补充ddH2O至25 μL。扩增条件:95 ℃预变性5 min,95 ℃变性1 min,56 ℃退火1 min;72 ℃延伸1 min 30 s,30个循环,最终72 ℃延伸7 min。PCR产物经凝胶电泳检测后,送上海生工公司测序,将测序结果提交到GenBank数据库中进行序列同源性对比(BLAST),得到同源性高的菌株序列。用CLUSTAL X 1.83软件将序列排列对齐,邻接法neighbor-joining method计算进化距离,进化树分支稳定性分析采用p-distences和Kimura-2parameter双参数法,并用Bootstrap检验1000 次。
1.5rep-PCR指纹图谱分析
使用引物(GTG)5(5′-GTGGTGGTGGTGGTG-3′)进行PCR扩增。扩增条件:95 ℃预变性5 min,95 ℃变性30 s,40 ℃退火30 s,65 ℃延伸8 min,循环30 次,最后65 ℃延伸16 min。扩增产物在1.4%琼脂糖凝胶、80 V下电泳2 h 50 min后置于凝胶成像系统下拍照,用GelCompar Ⅱ软件依据UPGMA进行聚类分析。
2 结果与分析
2.1低温乳酸菌的筛选
从冷水鱼肠道中共分离得到纯培养物206株,根据其菌落大小、形态、颜色等特征以及革兰氏染色和过氧化氢酶实验进行初筛,初步确定134株疑似乳酸菌。如表1部分低温乳酸菌的最适生长温度测定结果显示,其生长温度范围在4~37 ℃之间,最适生长温度为15~24 ℃,属于低温菌范畴。
2.2菌种鉴定及其系统发育
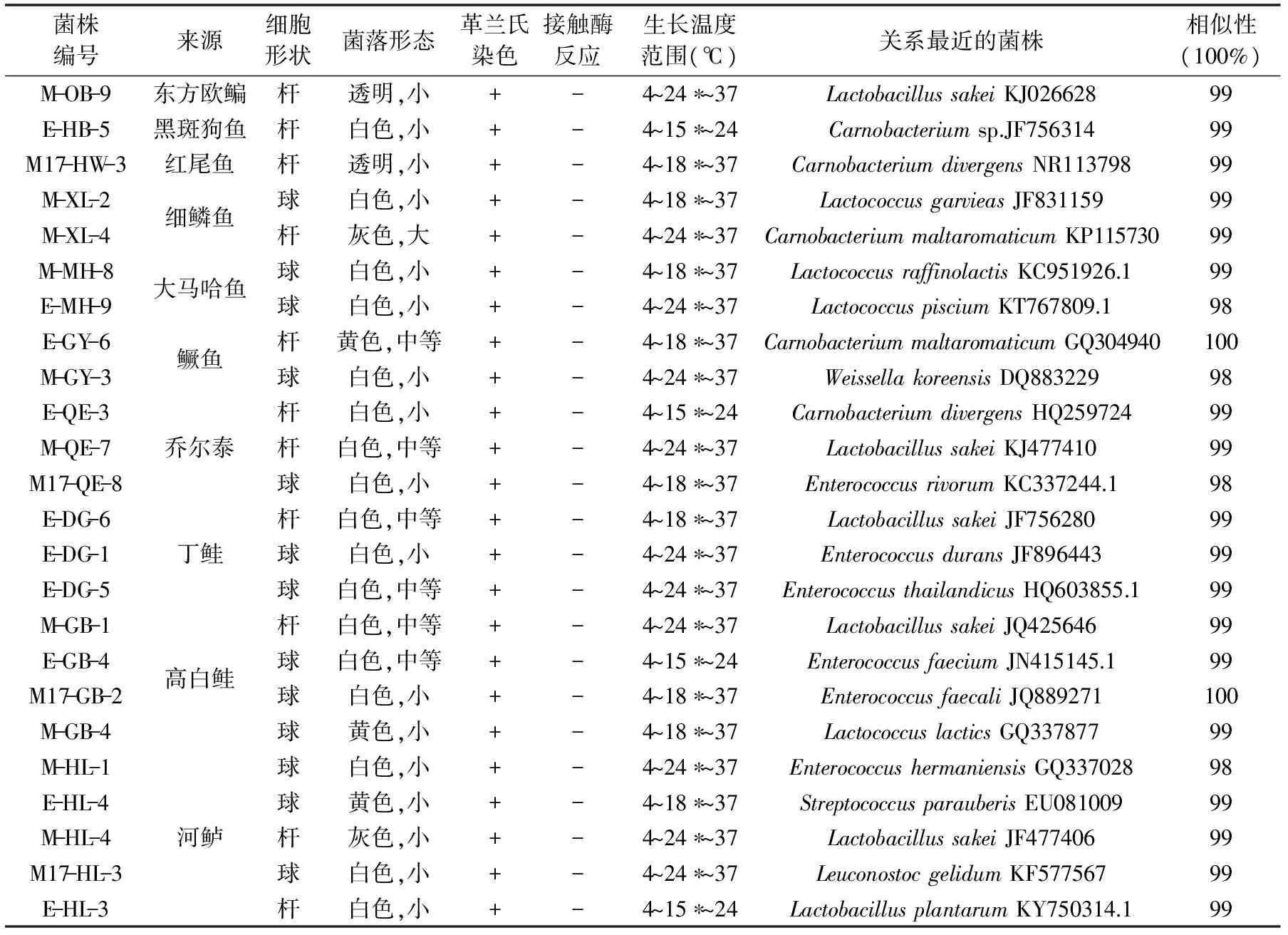
表1 从冷水鱼肠道中筛选的部分低温乳酸菌的特征
注:*表示最适生长温度。将测得的菌株序列与GenBank数据库中已知的16S rRNA基因序列进行同源性比对,被归属到种水平菌株的序列相似度均在98%以上(表1)。
134 株低温乳酸菌,隶属于Lactobacillus、Lactococcus、Enterococcus、Streptococcus、Leuconostoc、Weissella、Carnobacterium7个属。基于16S rRNA基因序列构建系统发育树(图1)。Enterococcus包括菌株E-DG-5,E-DG-1,E-GB-4,M-HL-1,M17-GB-8,M17-GB-2,M-GB-3,分别与已知种Enterococcusthailandicus,Enterococcusduurans,Enterococcusfaecium,Enterococcushermanniensis,Enterococcusrivorum与Enterococcusfaecalis构成小分支。M-XL-4,E-GY-4,E-GY-6与Carnobacteriummaltaromaticum的序列同源性为99%,M17-HW-3,E-QE-3,E-HB-5,M17-OB-2划为一个分支,隶属于Carnobacteriumdivergens。M-GB-4,M-XL-3,M-XL-2,M-MH-8,E-MH-9均属于Lactococcus属,分别与已知种Lactococcuslactis,Lactococcusgarvieas,Lactococcusraffinolactis,Lactococcuspiscium的16SrRNA基因序列相似性达到99%。E-DG-6,M-DG-4,M17-QE-1,M-GB-1,M-QE-7,M-HL-4,M-OB-9与Lactobacillussakei的相似度达到99%。M-GY-5,M-GY-3均属于Weissella属,E-HL-4,M17-HL-3两株菌分别隶属于Leuconostoc,Streptococcus2个不同的属,形成了单独的分支。
2.3基于rep-PCR低温乳酸菌的指纹图谱分析
图2可知,乳杆菌被划分为9 个群:群Ⅰ~Ⅸ以及一些小分支。Lactobacillussakei主要在Ⅰ、Ⅱ、Ⅴ、Ⅵ、Ⅶ群中,共33 株,占Lactobacillus的61%,Lactobacillusplantarum在Ⅲ、Ⅳ、Ⅷ、Ⅸ群中,占29%。如图3所示,Carnobacterium被分成6 个群:群Ⅰ~Ⅵ。其中C.maltaromaticum最多,主要集中在Ⅱ、Ⅲ、Ⅵ群,占Carnobacterium的54%。由图4可知,肠球菌中共25 株,分为5 个群。Enterococcusfaecium12株,分布在Ⅰ~Ⅴ群中,占Enterococcus的48%,而Enterococcusfaecalis有6株,其余Enterococcus种比例较少。图5中,Lactococcus、Leuconostoc、Weissella、Streptococcus的聚类图分为Ⅰ、Ⅱ群以及2个小分支和4个单独的分支。Lactococcus主要分布在Ⅰ群的2 个小分支中,Lactococcusgarvieas有5 株,占Lactococcus的50%,Leuconostocgelidum主要在Ⅱ群,而Weissella、Streptococcus各有两株。
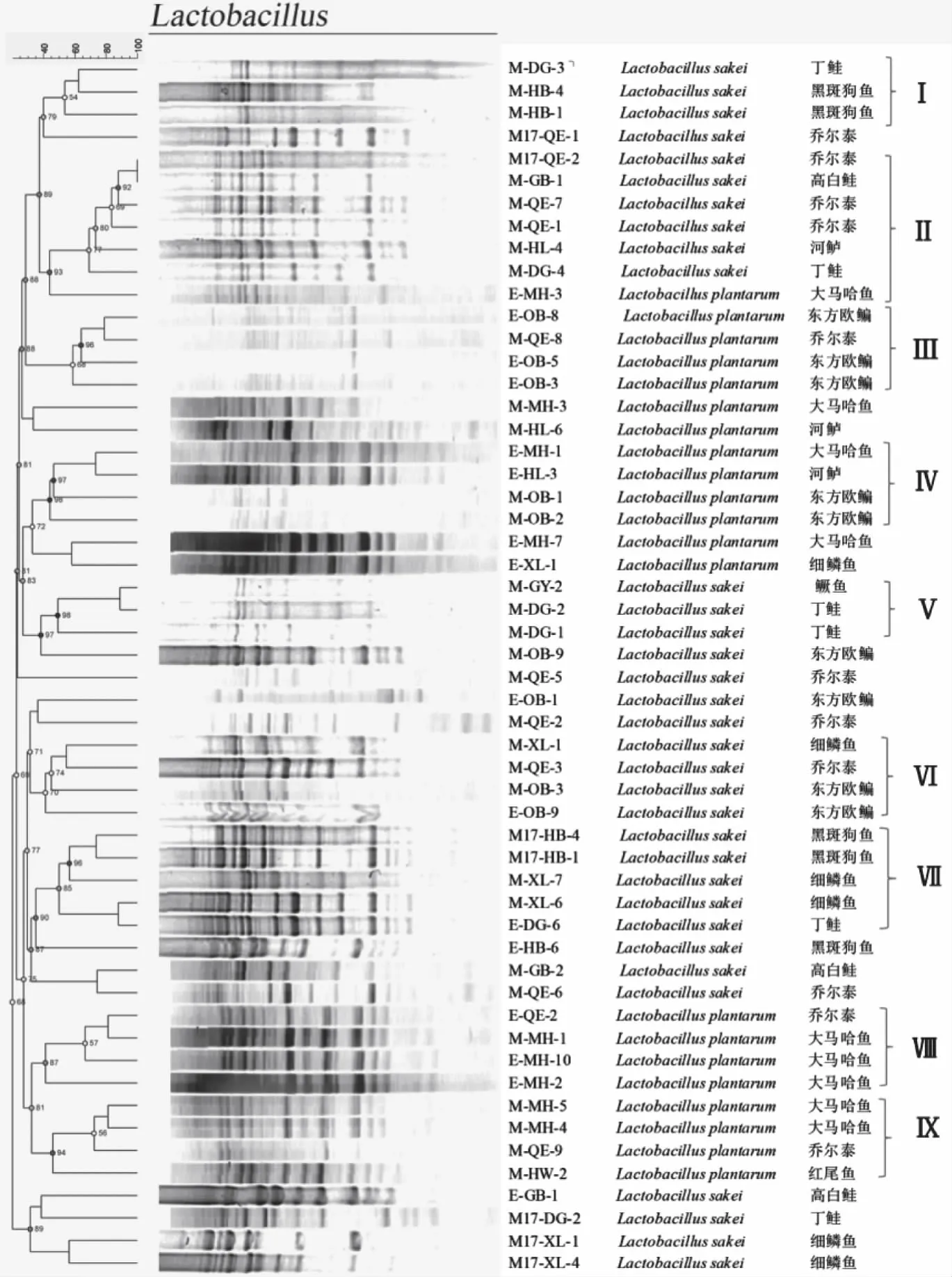
图2 Lactobacillus指纹图谱聚类分析图
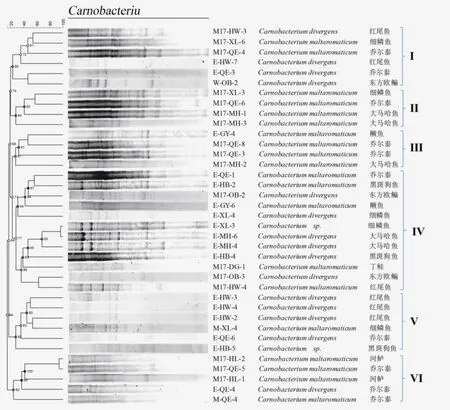
图3 Carnobacterium指纹图谱聚类分析图

图4 Enterococcus指纹图谱聚类分析图
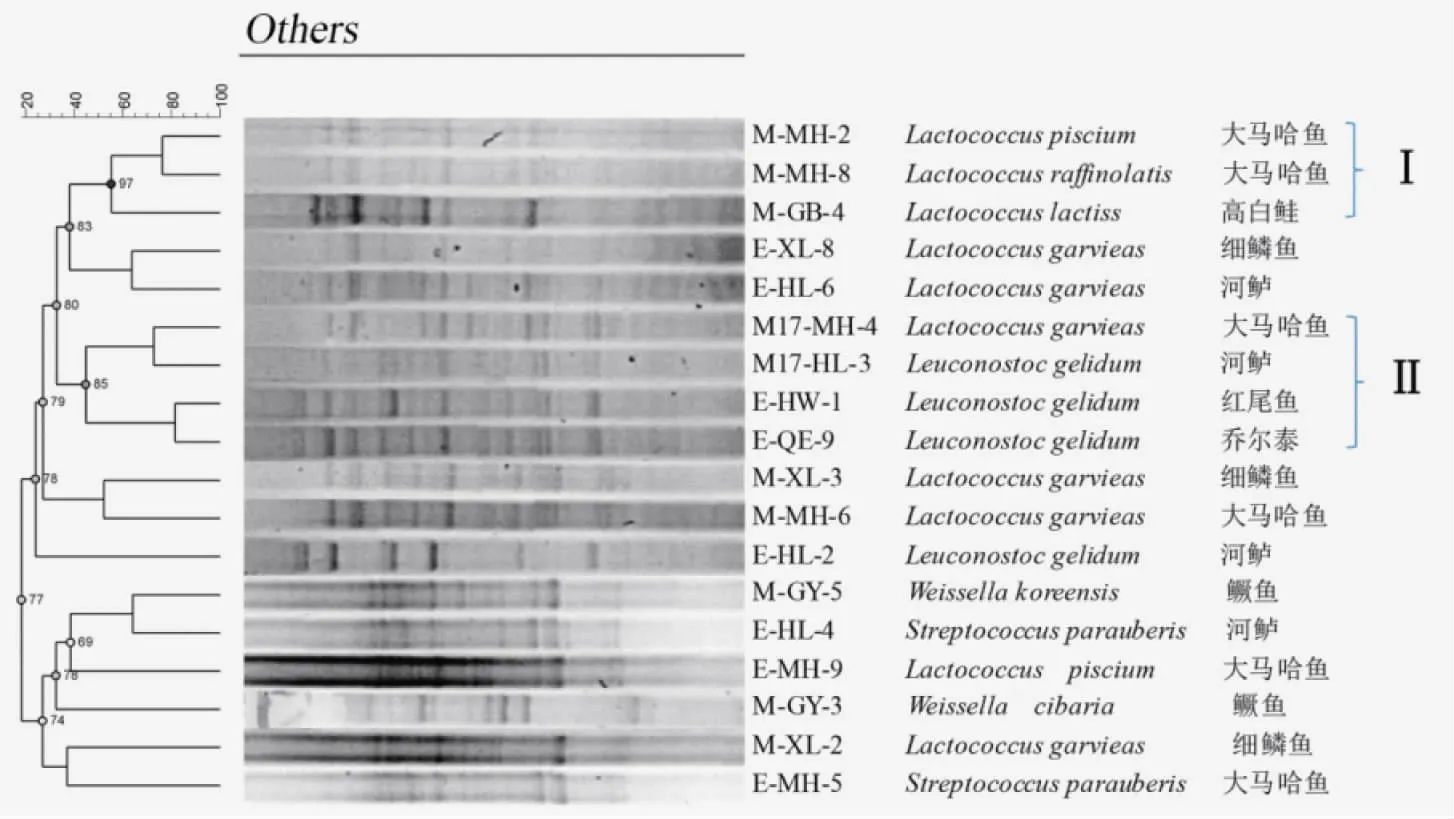
图5 Lactococcus、Leuconostoc、Weissella、Streptococcus指纹图谱聚类分析图
通过凝胶电泳检测,Rep-PCR指纹图谱的条带在500~5000 bp之间。尽管指纹图谱产生的条带带型较多,但是不同的乳酸菌带型有所不同,进而能够比较分析不同菌株在种水平上的差异。其中,Lactobacillusplantarum的条带以6~12为主,从同一类冷水鱼肠道中分离出Lactobacillussakei的带型相似,图2 Ⅶ群中黑斑狗鱼和细鳞鱼聚类在一起,两者都是鲑形目,但细鳞鱼肠道中Lactobacillusplantarum带型与Lactobacillussakei的带型相差较大,被聚类到不同的群中。图3 Ⅲ群中大马哈鱼和鳜鱼是不同种类的鱼,这两种鱼肠道中Carnobacteriummaltaromaticum带型不同。图4 Ⅲ群,大马哈鱼和高白鲑是来自不同流域的冷水鱼,从其肠道中筛选的Enterococcusfaecium也存在差异。
乳酸菌广泛存在于不同的生境中,遗传多样性极其丰富。本实验中依据低温乳酸菌的rep-PCR指纹图谱发现,同一种乳酸菌的指纹图谱存在一些特征性的条带,如此可以将不同种类的乳酸菌鉴别出来。从冷水鱼肠道中乳酸菌多样性方面揭示了鱼肠道中存在着不同类群的细菌,不同种类、不同流域的冷水鱼肠道中乳酸菌多样性存在差异,这可能是由于鱼的生活环境及习性等因素影响,乳酸菌形成不同的生态位,造就了这种差异。
2.4鱼肠道内低温乳酸菌的多样性分析
样品的Chao1指数和Simpson、Shannon指数用来评估物种丰度和多样性。由表2可知,大马哈鱼和细鳞鱼肠道中乳酸菌的多样性较复杂,大马哈鱼肠道中乳酸菌种类最为丰富,鉴定出9种乳酸菌,细鳞鱼和黑斑狗鱼肠道中次之,鉴定出8种乳酸菌,乔尔泰和丁鲑肠道中只鉴定出3种乳酸菌。图6所示,同种类的冷水鱼肠道乳酸菌多样性比较相似,红尾鱼和东方欧鳊,河鲈和鳜鱼,细鳞鱼和黑斑狗鱼各自聚在一个分支,但白斑狗鱼,丁鲑,高白鲑和大马哈鱼例外,这可能表明它们肠道中乳酸菌的多样性受众多因素的影响。10种样品中共鉴定出乳酸菌19个种:Lactobacillussakei、Lb.plantarum、Lactococcuslactis、L.garvieas、L.raffinolactis、L.piscium、Leuconostocgelidum、Strepotococcusparauberis、Weissellakoreensis、W.cibaria、Enterococcusthailandicus、E.duurans、E.faecium、E.hermanniensis、E.rivorum、E.faecalis、Carnobacteriummaltaromaticum、C.divergens。在样品中L.Sakei丰富度最高,其次是Lb.plantarum和C.maltaromaticum,它们普遍存在于在大多数冷水鱼肠道中。
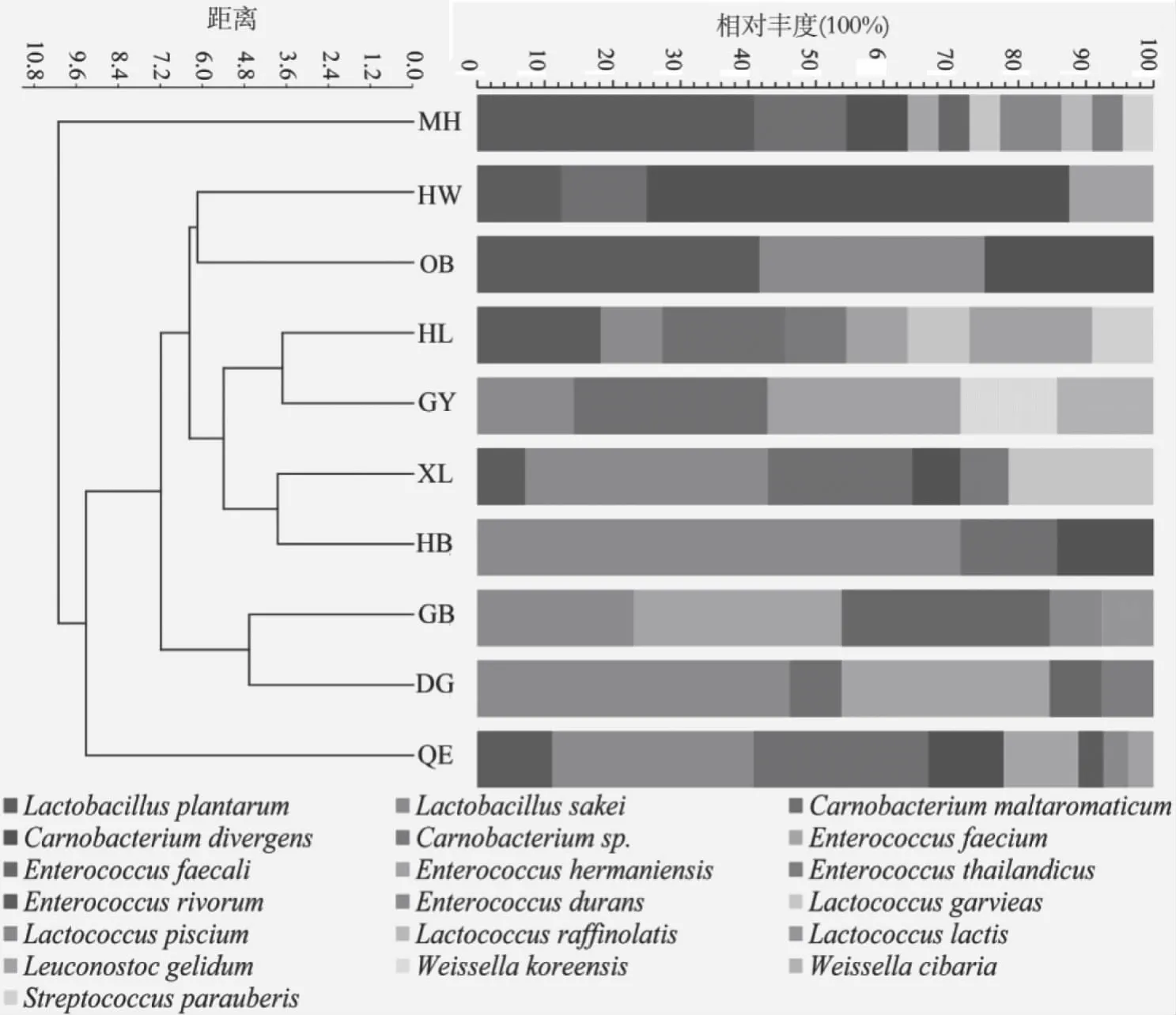
图6 样品菌种聚类分析图
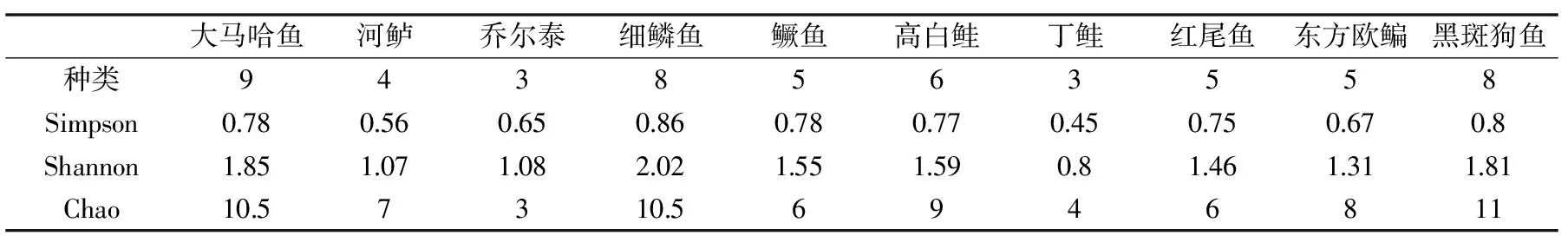
表2 样品乳酸菌的多样性
3 结论
刚孵化鱼仔的胃肠道一般是无菌的,随着鱼类的生长发育,通过摄食或接触环境逐渐形成了一个由好氧菌和厌氧菌组成的菌群,是一个十分复杂而又处于动态平衡的微生态系统[15,23-24]。我国新疆额尔齐斯河流域、黑龙江流域处于45 °N以上的中高纬度地区,属于寒温带气候,其高海拔、高纬度、低气温的特点使其湖泊、河流中栖居着丰富的冷水鱼,为乳酸菌提供了理想的栖息环境。本文采用3 种不同培养基从10 种冷水鱼肠道中分离出了134 株低温乳酸菌。通过16S rRNA基因序列分析构建系统发育树,结果显示所有乳酸菌被划为7 个属,如Lactobacillus、Lactococcus、Enterococcus、Streptococcus、Leuconostoc、Weissella、Carnobacterium,其中Lactobacillus为优势属。本实验中依据低温乳酸菌的rep-PCR指纹图谱发现,同一种乳酸菌的指纹图谱存在一些特征性的条带,如此可以将不同种类的乳酸菌鉴别出来。从冷水鱼肠道中乳酸菌多样性方面揭示了鱼肠道中存在着不同类群的细菌,不同种类、不同流域的冷水鱼肠道中乳酸菌多样性存在差异,这可能是由于鱼的生活环境及习性等因素影响,乳酸菌形成不同的生态位,造就了这种差异。在指纹图谱聚类中,尽管某些种群中存在一定的交叉情况,但主要种群的菌株都能聚在一起,不同种的乳酸菌株条带有所不同,表现出了差异性。实验结果发现不同种类、不同流域的冷水鱼,其肠道乳酸菌的指纹图谱存在差异。由于rep-PCR指纹图谱技术在实验过程中对DNA、PCR体系及PCR仪等要求严格,可重复性差,近年来许多研究应用分子生态学方法鉴定低温乳酸菌,如高通量测序技术(NGS)能够更加快速、准确的分析和鉴定菌群[25-26],提高了对样品中微生物菌群分析的深度和广度,特别是一些不可培养的微生物。在后续的实验中,我们将结合多种技术进一步探究低温乳酸菌的开发和利用。
[1]Abbasiliasi S,Tan J S,Ibrahim T A T. Isolation of Pediococcus acidilactici Kp10 with ability to secrete bacteriocin-like inhibitory substance from milk products for applications in food industry[J].BioMed Central Microbiology,2012,12:260.
[2]Gao Y R,Jia S R,Gao Q,et al. A novel bacteriocin with a broad inhibitory spectrum produced byLactobacillussake C2,isolated from traditional Chinese fermented cabbage[J].Food Control,2010,21:76-81.
[3]Aymerich M T,Garriga M,Monfort J M,et al. Bacteriocin-producinglactobacilliin Spanish style fermented sausages:Characterization of bacteriocins[J].Food Microbiol,2000,17(1):33-45.
[4]Solís G,Gavilan C G,Fernández N,et al. Establishment and development of lactic acid bacteria and bifidobacteria microbiota in breast-milk and the infant gut[J].Anaerobe,2010,16(3):16307-16310.
[5]郭兴华,凌代文. 乳酸细菌现代研究实验技术[M].北京,科学出版社,2013:3-10.
[6]Cherl L. Lactic acid fermented foods and their benefits in Asia[J]. Food control,1997,8(5/6):259-269.
[7]Roginski H,Fuquay J W,Fox P F. Encyclopedia of Dairy Sciences[M]. Boulevard:Elsevier,2003.
[8]Varsha K,Nampoothiri K. Appraisal of lactic acid bacteria as protective cultures[J]. Food Control,2016,69:61-64.
[9]Pilar C M,Samuel A,Karola B,et al. Current Applications and Future Trends of Lactic Acid Bacteria and their Bacteriocins for the Bio-preservation of Aquatic Food Products[J]. Food Bioprocess Technol,2008,1:43-63.
[10]KunzesS A,Anila K,Null S,et al. Probiotic characterization of lactic acid bacteria isolated from fermented foods and beverage ofLadakh[J]. LWT-Food Science & Technology,2016,66:428-435.
[11]Bao Y,Zhang Y C,Zhang Y,et al. Screening of potential probiotic properties ofLactobacillusfermentum isolated from traditional food products[J].Food Control,2010,21(5):695-701.
[12]MerabtirI R,Bekhouche F,Chuat V,et al. A large diversity of lactic acid bacteria species is involved in the fermentation of wheat used for the manufacture of lemzeiet[J]. Europe Food Research Technology,2015,241:137-149.
[13]Varshak K,Nampoothirok K M. Appraisal of lactic acid bacteria as protective cultures[J].Food Control,2016,69:61-64.
[14]郭本恒,刘振民.益生菌[M].北京:化学工业出版社,2015:362-364.
[15]Kendalld C,Estherr A,Wlinn M,et al. Intestinal microbiota in fishes:What’s known and what’s not[J].Molecular Ecology,2014,23(8),1891-1898.
[16]杨洪岩,王小芬,崔宗均,等. 低温环境中乳酸菌的开发利用[J].微生物学报,2008,48(1):132-137.
[17]Cheigh H S,Park KY. Biochemical,microbiological,and nutritional aspects of kimchi(Korean fermented vege
Table products)[J]. Crit Rev Food Sci Nutr,1994,34(2):175-203.
[18]Matamoros S,Pilet M F,Gigout F,et al. Selection and evaluation of seafood-borne psychrophilic lactic acid bacteria as inhibitors of pathogenic and spoilage bacteria[J]. Food Microbiology,2009,26(6):638-644.
[19]Castellano P,Vignolo G. Inhibition of Listeria innocua and Brochothrix thermosphacta in vacuum-packaged meat by addition of bacteriocinogenicLactobacilluscurvatus CRL705 and its bacteriocins[J].Letters Applied Microbiology,2006,43(2):194-199.
[20]Dykes G A,Von H A. Taxonomic status of atypicalLactobacillussake andLactobacilluscurvatus strains associated with vacuum packaged meat spoilage[J]. Curr Microbiol,1994,28(4):197-200.
[21]Dykes G A,Cloete T E,Von H A. Identification ofLeuconostocspecies associated with the spoilage of vacuum packaged Vienna sausage by DNA-DNA hybridization[J]. Food Microbiol,1994,11(4):271-274.
[22]Juste A,Thommab P H J,Livevens B. Recent advances in molecular techniques to study microbial communities in food-associated matrices and processes[J].Food Microbiology,2008,25(6):745-761.
[23]Sou M,David K N and Ulrich S,et al. Diet strongly influences the gut microbiota of surgeonfishes[J].Molecular Ecology,2015,24(3):656-672.
[24]Wang A R,Chao R,Ringø E,et al. Progress in fish gastrointestinal microbiota research[J]. Reviews in Aquaculture,2017,0:1-15.
[25]Xing M X,Hou Z H,Yuan J B,et al. Taxonomic and functional metagenomic profiling of gastrointestinal tract microbiome of the farmed adult turbot(Scophthalmusmaximus)[J]. FEMS Microbiol Ecol,2013,86(3):432-443.
[26]Laura B,Joan L R,Ave T K,et al. Gut Microbiota Dynamics during Dietary Shift in Eastern African Cichlid Fishes[J]. Plos One,2015,10(5):1-23.
Geneticheterogeneityoflacticacidbacteriaisolatedfromcold-waterfishintestine
HUANGLi-li,LUOBao-long,NIYong-qing,ZHANGYan,ZHOUHong*
(Food College of Shihezi University,Shihezi 832000,China)
The purpose of this study was to analyze the genetic and species heterogeneity of lactic acid bacteria(LAB)from the intestinal tract of cold-water fishes in the Eerqisi river of Xinjiang and Heilong River,which lay the foundation for exploitation resources of lactic acid bacteria. Psychrophilic LAB isolated from cold-water fishes intestinal tract by using MRS,Elliker,M17 culture medium,and measured its optimal growth temperature. According to 16S rRNA gene sequences preliminarily to determine the phylogenetic relationship of psychrophilic LAB,those highly homologous strains was based on rep-PCR fingerprint to further distinguish. It was obtained that 134 psychrophilic LAB from the gastrointestinal tract of cold-water fishes,and the optimum growth temperature was 15~24 ℃. The sequencing results indicated that the selected psychrophilic LAB were divided into seven genus:Lactobacillus,Lactococcus,Enterococcus,Streptococcus,Leuconostoc,Weissella,Carnobacterium7 genus,19 species,respectively,and the dominant bacteria was Lactobacillus. Rep-PCR fingerprint showed that there was a different degree of genetic difference,not only in the same genus of lactic acid bacteria of different species level,but also among various strains of a specie.
cold-water fish intestine flora;psychrophilic lactic acid bacteria;fingerprints
2017-04-05
黄丽丽(1990-),女,硕士研究生,研究方向:食品生物技术,E-mail:huangli00821@sina.com。
*
周红(1969-),女,本科,高级实验师,主要从事微生物等方面的研究,E-mail:zhh_food@shzu.cn.com。
国家自然基金项目(31360001)。
TS201.3
A
1002-0306(2017)22-0082-08
10.13386/j.issn1002-0306.2017.22.017
