烟台市区河流入海口微生物群落的分析
2017-11-21李文军赵彦宏
蒲 洋, 李文军, 王 凯, 赵彦宏, 秦 松
烟台市区河流入海口微生物群落的分析
蒲 洋1, 李文军2, 王 凯3, 赵彦宏1, 秦 松2
(1. 鲁东大学农学院, 山东烟台 264025; 2. 中国科学院烟台海岸带研究所, 山东烟台 264003; 3. 上海海洋大学, 上海 200090)
2015年4月在烟台市区4条主要河流(辛安河、逛荡河、鱼鸟河和夹河)入海口处采集12个样品, 利用PCR-DGGE方法分析了此区域的细菌群落组成和优势菌群。通过统计学手段对DGGE图谱进行分析, 结果表明4条河流入海口细菌群落丰富度都较高, 12个取样点扩增的DGGE条带数都在30以上, 样品的Shannon指数均高于3.3, 个别甚至可达3.61。其中, 辛安河和逛荡河的Shannon指数平均值均高于鱼鸟河和夹河, 夹河多样性最低; 而且UPGMA聚类分析结果显示地理位置越接近, 其细菌组成的相似度越高。在DGGE电泳条带中选取14条主要条带进行扩增和序列测定, 所得到的序列进行了系统进化分析发现4条河流入海口的优势菌群主要为变形杆菌门(Proteobacteria)和拟杆菌门(Bacteroidetes)。这与近年来关于山东近岸海域细菌多样性的研究结果相符合, 为研究和保护烟台市区河口处环境提供科学依据。
烟台; 河流入海口; PCR-DGGE; 微生物多样性分析
海洋浮游细菌对海洋生态系统稳定有着十分重要的作用[1], 其活动规律也受到近岸海域和海洋环境的影响, 通过调查研究近岸河口细菌的基本生态状况, 可以初步反映环境变化及海洋健康状态, 对于进一步评价河道水质恢复状况非常必要[2]。
变性梯度凝胶电泳(Denatured Gradient Gel Electrophoresis,DGGE)由Lerman等发明, 刚开始主要被用来检测DNA片段中的点突变, 1993 年开始, Muyzer等[3]、Earing等[4]和Zhang等[5]将DGGE应用于微生物群落结构研究中。利用针对16S rRNA的PCR-DGGE方法, 对各种环境内的细菌群落结构组成进行分子生态学研究。相比于传统先培养再分析鉴定的方法, 它拥有快速、简便、突变检出率高等优点, 并且对不可培养的微生物也可起到分离鉴定的效果, 不但改变了繁琐的基于传统培养方法的程序, 而且大大加深和扩展了对种群时空变化情况的理解和认识[6-9]。随着这种技术的发展和优化, 其被广泛用于微生物分子生态学研究的各个领域, 包括细菌、蓝细菌、古菌群落的生物多样性, 目前已经发展成为研究细菌特定环境群落结构主要分子生物学方法之一[10-11]。
之前的相关报道对山东近岸海域、黄海北部、黄海西北部和黄海冷水团的水体和沉积物中细菌群落结构及多样性有过相关调查研究, 确定了山东半岛沿岸相关海域中的优势群落的优势菌群主要为变形杆菌门(Proteobacteoriate)、拟杆菌门(Bacteroidetescidal)、蓝细菌门(Cyanobacteria)和浮霉菌门(Planctomycetes)等[2, 12-15]。但是对于地处山东半岛东北部的黄海沿岸河流入海口这一受人类活动影响极大的区域相关的研究还未涉及到。烟台, 地处山东半岛东北部, 是山东省重要的海滨旅游城市。烟台4个市辖区(芝罘区、莱山区、福山区、牟平区)是烟台经济发展的中心区域, 人口密集,生产活动频繁, 故而生态环境所面临的压力也较大。本次研究选取2015年4月的辛安河、逛荡河、鱼鸟河和夹河的河流入海口为研究区域。这4条河均位于4个市辖区内, 流入黄海。其中, 辛安河及鱼鸟河位于牟平区和莱山区之间, 逛荡河位于莱山区, 源于凤凰山水库, 夹河位于福山区, 流域面积较大。选取这4条河的河流入海口水域样品作为研究对象, 利用PCR-DGGE检测技术, 可以在较大的深度和广度上对烟台近岸河流入海口海域春季的细菌群落组成进行全面分析, 同时能够初步了解其河道生态系统的概况[16], 同时可以与近岸相关研究进行比较, 因此进行细菌群落结构和多样性研究具有重要意义, 能够为进一步积累和丰富特定近岸海域的生态资料, 研究和保护烟台市区河口处的近岸环境资源提供科学依据。
1 材料与方法
1.1 材料获取及处理
研究材料为2015年4月份所采集的烟台4条河流入海口区域水样, 取样地点分别为辛安河(121°32′15″E, 37°26′30″N)、逛荡河(121°28′2″E, 37°28′14″N)、夹河(121°17′52″E, 37°34′36″N)、鱼鸟河(121°34′25″E, 37°26′25″N)的河流入海口区域。每条河流入海口设3个取样点, 均在表层30 cm处取样, 所采水样于冰盒中密封。所取水样用25 μm筛绢过滤, 再用0.22 μm的滤膜抽滤, 保留抽滤后的滤膜进行后续研究。
1.2 实验方法
1.2.1 样品细菌宏DNA获取
采用FastDNATMSPIN Kit试剂盒, 按操作说明提取样品基因组DNA。
1.2.2 细菌16S rDNA片段的PCR扩增
以样品基因组DNA为模板, 采用细菌通用引物GC-338F和518R扩增样品16S rDNA高变区序列(表1)。
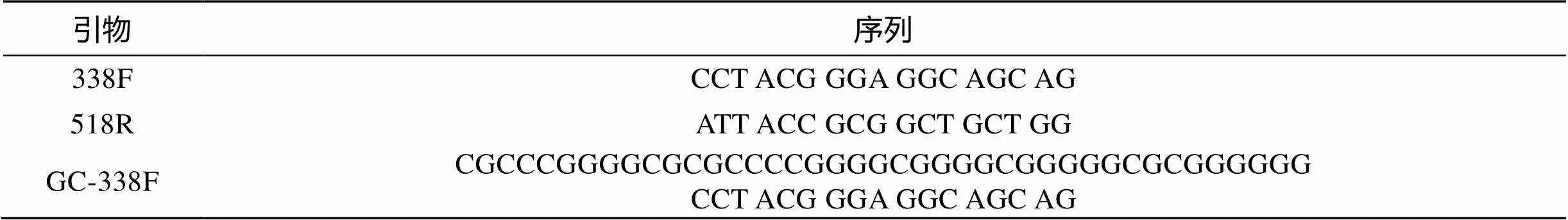
表1 PCR所用引物序列
PCR扩增体系(50 μL)为: 10× PCR buffer 5 μL; dNTP Mixture(2.5 mmol/L)3.2 μL;(5 U/μL)0.4 μL; 上游引物GC-338F(20 μmol/L)1 μL; 下游引物518R(20 μmol/L)1 μL; 模板DNA 1μL; 补ddH2O至50 μL。PCR扩增程序为: 94℃预变性5 min; 94℃变性1 min, 55℃复性45 s, 72℃延伸1 min, 30个循环; 最终72℃延伸10 min。PCR产物采用OMEGA公司DNA Gel Extraction Kit纯化回收。
1.2.3 PCR产物的DGGE分析
取10 μL 纯化回收产物进行DGGE分析。使用变性梯度为35%~55%、含量为7%的聚丙烯酰胺凝胶在1×TAE缓冲液中150 V, 60℃下电泳5 h, 完毕后, 采用银染法进行染色。
1.2.4 DGGE图谱中优势条带的回收与测序
选取具有代表性的DGGE条带(14条), 切下并回收目的条带。以2 μL回收产物作为模板, 338F/518R作为引物再次进行PCR扩增。将新扩增的DNA片段回收并纯化后, 连接到Pmd18-T载体上, 并转化至DH5α感受态细胞中, 筛选阳性克隆, 每个回收条带选取3个克隆进行序列测定。然后登录NCBI (http: www.ncbi.nlm.nih.gov blast), 在GeneBank数据库中用BLAST进行检索和同源性比对[17-19]。
1.2.5 图谱统计分析
DGGE图谱采用Quantity one软件对每个样品的电泳条带数目、条带密度进行数字化分析, 香农指数()、丰度()和均衡指数()等指标被用来比较不同样品的多样性情况[19-20]。其算法如下:
= –∑PlnP= –∑(N/) ln (N/)
/max/ln
其中,P为样品中单一条带的强度在该样品所有条带总强度中所占的比率,为DGGE图谱单一泳道上所有条带的丰度,N为第条带的丰度;是某样品中所有条带数目总和。
测序结果采用DNAstar和Cluster软件进行序列分析, 根据序列比对结果, 下载最相似的菌株序列作为系统发育树的参考序列。然后采用MEGA5.0软件中的邻接法(N-J法)构建系统发育树。
2 结果与分析
2.1 细菌16S rDNA的PCR扩增
以提取的过滤膜总DNA为模板, 以GC-338F和518R为引物扩增16S rDNA序列, 得到目的DNA片段。其中, 1、2、3为辛安河入海口水样, 4、5、6为逛荡河入海口水样, 7、8、9为鱼鸟河入海口水样, 10、11、12为夹河入海口水样。
如图1所示, 目的片段大小为180 bp, 扩增特异性很好, 获得的目的条带可进一步用于DGGE分析。
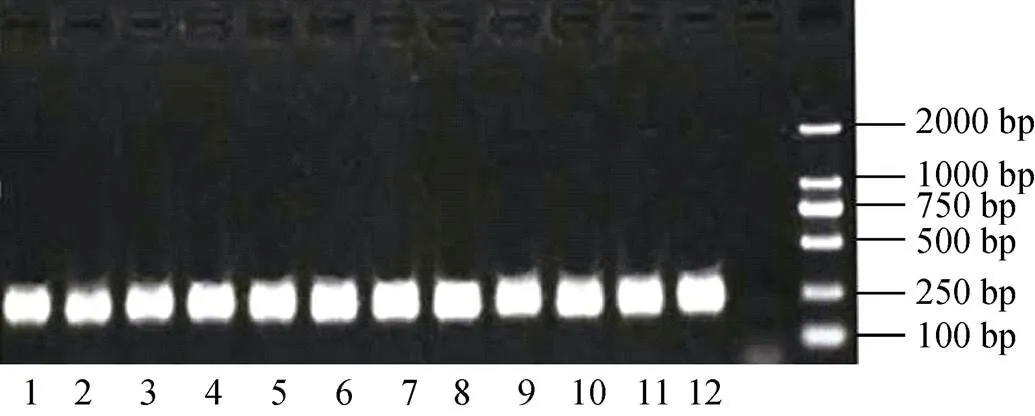
图1 12个样品的16S rDNA基因PCR电泳图谱
2.2 PCR产物的变性梯度凝胶电泳(DGGE)图谱及相似度分析
各采样点细菌组成的DGGE图谱如图2, 可以看出在本次所研究的4个地点的12个样品中, 共扩增出64条电泳条带, 每条泳道的条带数目较多(图2A), 均在30以上, 表明所调查地点的细菌多样性很高。同时, 个别条带(如#5, #29, #32等)在12个泳道中均有出现, 说明#5、#29和#32所代表的种群在所有样品中是普遍存在的, 而条带的亮度代表了该种群在环境中的数量, 亮度越高, 数量越多, 意味着#29和#32应该是所有样品的优势种群。而有些条带(如#16、#36、#63、#64)只出现在夹河入海口样品(10、11、12)中, 说明该条带所代表的种群可能是该取样点的特有种群。
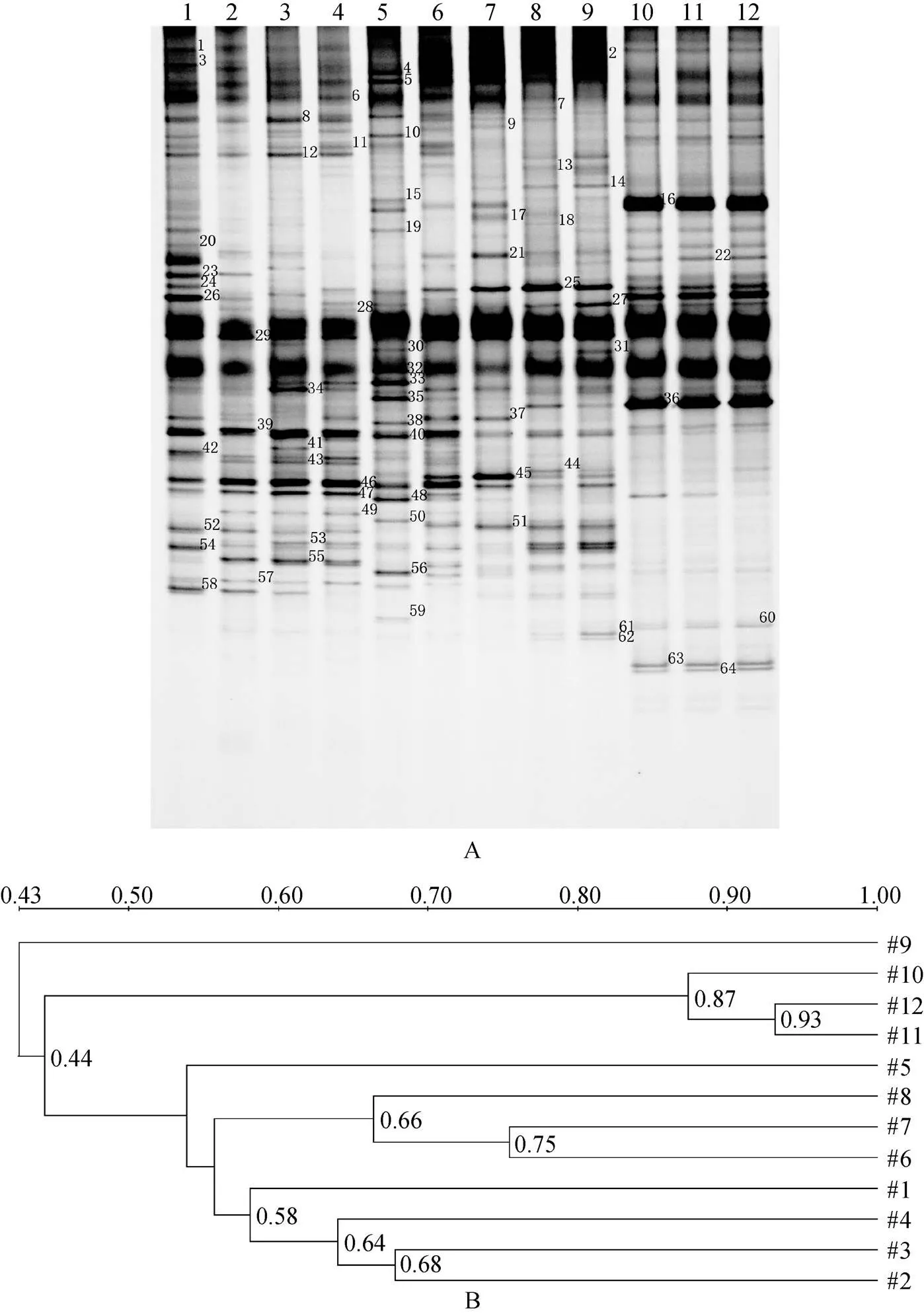
图2 12个样品的DGGE图谱及聚类分析
香农指数(Shannon index)的高低反映了一个环境中物种多样性的高低, Shannon指数越高, 表明该环境中物种越丰富。由表2看出, 本次实验12个样品的Shannon指数均高于3.3, 甚至个别可达3.61, 表明样品整体的多样性较高。其中, 辛安河样品(1~3)和逛荡河样品(4~6)的香浓指数平均值相同, 而且均高于鱼鸟河样品(7~9)和夹河样品(10~12), 夹河多样性最低。
通过UPGMA聚类分析图(图2B)和主成分分析图(PCA, 图3)可以清楚地看出, 同一河流入海口的3个样品间的微生物组成相似度很高; PCA分析图显示了PC1的贡献度为33.7%, PC2的贡献度为24%, 并且没有样品能够聚集在一起, 说明了取样点之间的细菌组成具有一定的差异。并且, 辛安河(样品1~3)、逛荡河(样品4~6)及鱼鸟河(样品7~9)由于地理位置相距较近, 所以细菌组成相似度较高; 辛安河(样品1~3)和鱼鸟河(样品7~9)相距更近, 因此微生物组成相似度更高一些, 而夹河入海口(样品10~12)位于烟台西北方, 故而微生物组成与其他3处相差较大。
共对14条代表性的DGGE凝胶条带进行切割回收, 这14条条带包括: 4个地点共有条带(#29, #40), 辛安河、逛荡河及鱼鸟河共有条带(#46), 夹河独有条带(#16, #36, #63), 鱼鸟河独有条带(#25), 辛安河独有条带(#23), 逛荡河独有条带(#38)以及其他亮度明显的条带。代表性条带按照上述相同程序再次进行PCR扩增并进行序列测定, 测序结果的比对分析见表3。采用MEGA5软件, Neighbor-joining法构建的系统发育树见图4。
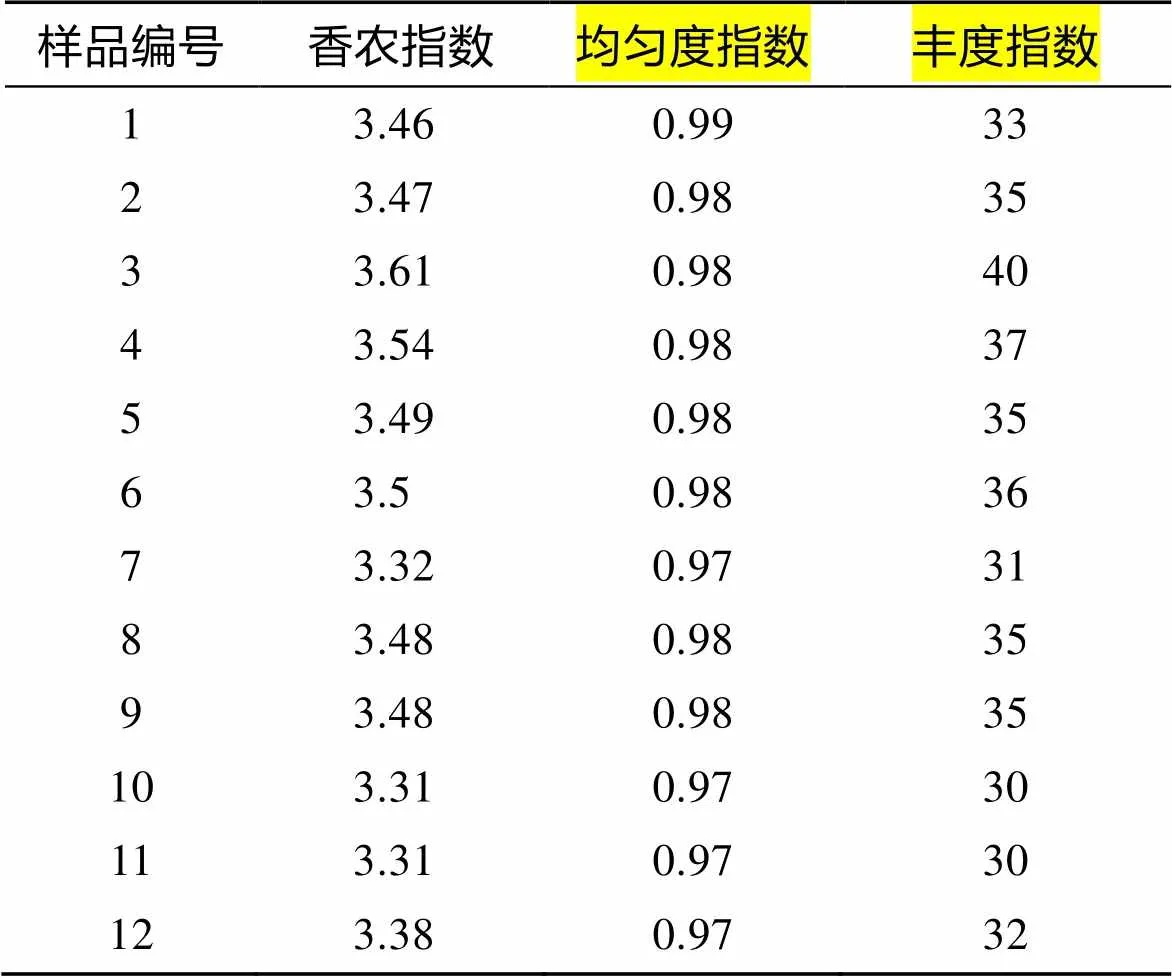
表2 12个样品之间的香农指数
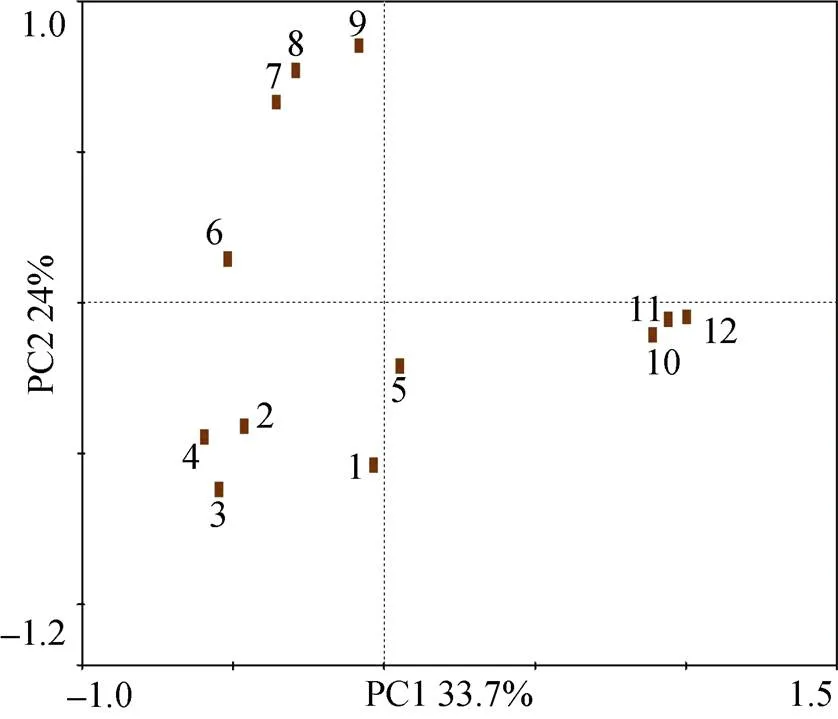
图3 12个不同样品间的主成分分析图
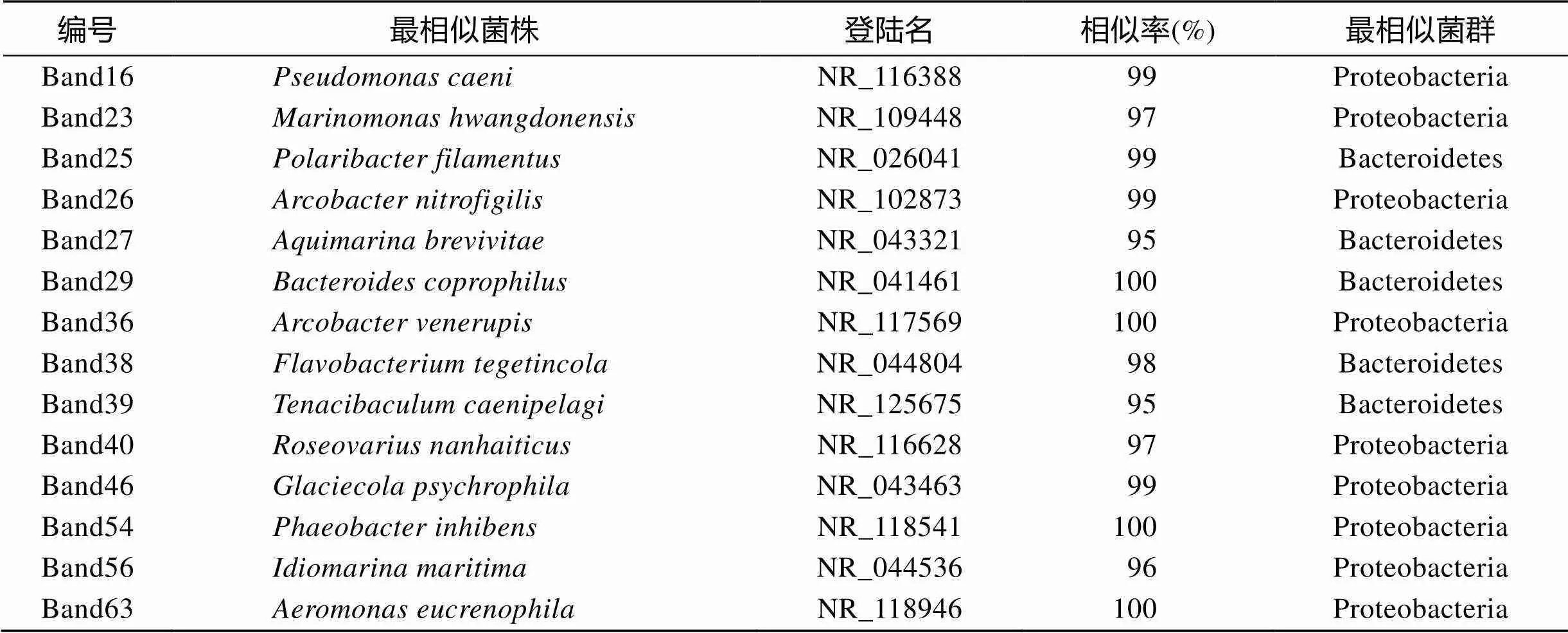
表3 DGGE代表性条带的比对结果
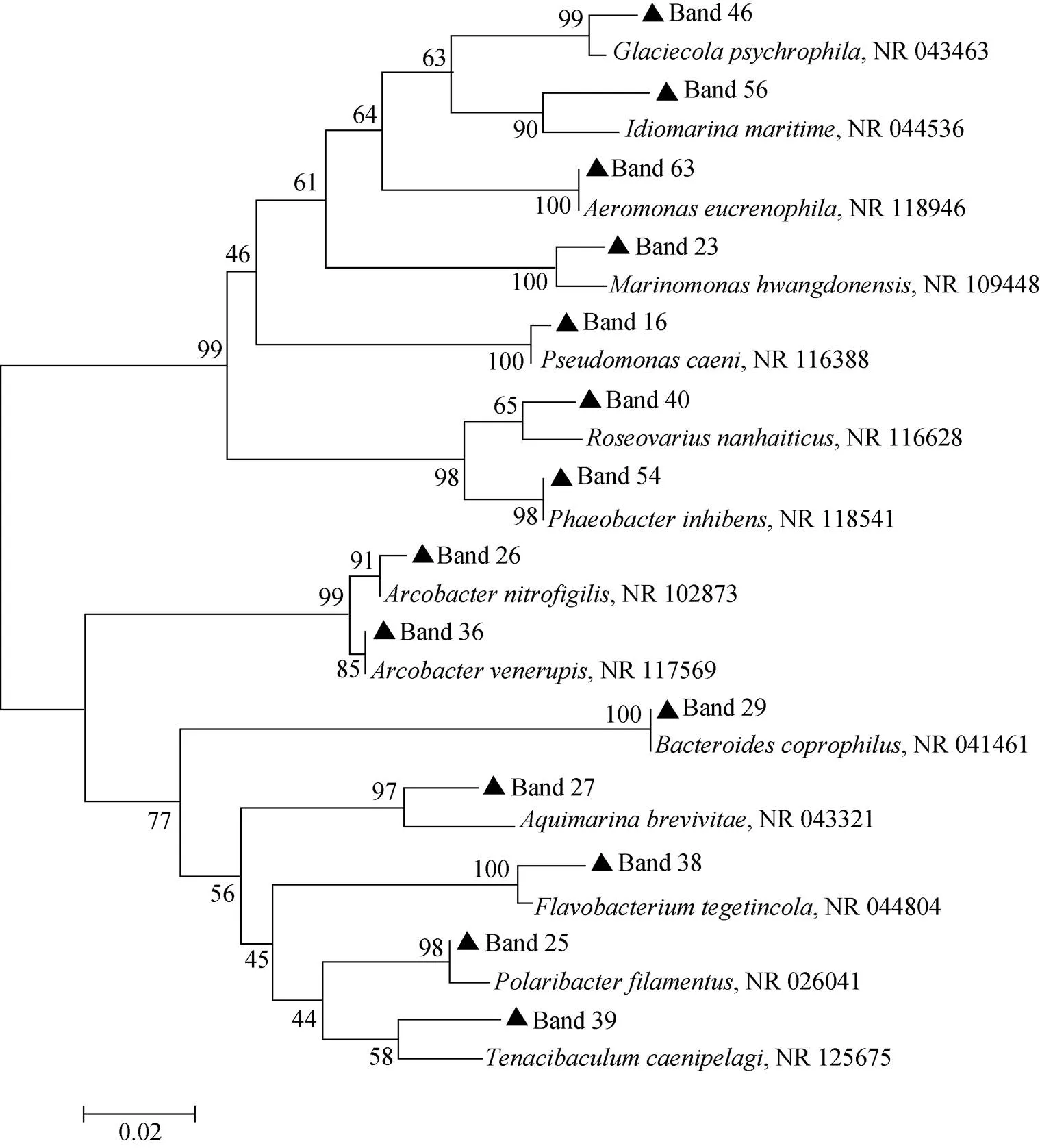
图4 4条河流入海口细菌16S rDNA序列系统发育图谱
主要代表性条带进行序列鉴定并通过GeneBank 数据库比对分析表明: 全部测序序列的16S rDNA与数据库中序列的相似性在95%~100%之间, 相似性较高可以认为条带代表菌株与GeneBank中的菌株为同种菌株[21]。可看出, 4个河流入海口的共同优势菌群为变形菌门(Proteobaceria)的玫瑰变色菌属()和拟杆菌门(Bacteroidetes)的拟杆菌属(); 此外, 辛安河、逛荡河及鱼鸟河3个河流入海口的优势菌群还有变形菌门的居冰菌属(); 夹河入海口优势种群还包括变形菌门的假单胞菌属()、弓形菌属()以及气单胞菌属(); 鱼鸟河入海口的优势种群还包括拟杆菌门的极地杆菌属(), 辛安河入海口的优势种还包括变形菌门的海单胞菌属(); 逛荡河入海口的优势种群包括拟杆菌门的黄杆菌属()。
3 讨论与结论
本文采用PCR-DGGE方法对烟台河流入海口进行微生物群落的分析鉴定, 实验结果表明烟台市4大河流(辛安河、逛荡河、鱼鸟河及夹河)入海口微生物丰富度及多样性都很高, 这可能是由于春天气温回暖, 水体中微生物繁殖速度加快, 再加上地处入海口这样的咸淡水交汇区, 营养物质较为丰富, 所以微生物多样性较高。还有更重要的原因就是, 本次实验所选的地点都处于人类活动较为频繁的区域, 这也可能会造成水中微生物的大量繁殖。
另外, 序列分析表明, 4个河流入海口的细菌类群分布表现出了明显的地理空间分布上的相关性, 优势菌群主要为变形菌门(Proteobcteria)和拟杆菌门(Bacteroidetes), 此结论与近年来国内有关黄海海域微生物多样性的研究结果部分一致[2, 14-15, 22-23]。变形菌门(Proteobacteria)的主要类群为γ-Proteobacteria变形菌纲, 此外还包含α-Proteobacteria变形菌纲、ε-Proteobacteria变形菌纲。鱼鸟河入海口的优势种群还包括拟杆菌门的极地杆菌属(), 逛荡河入海口的优势种群也有拟杆菌门的黄杆菌属()。γ-Proteobacteria变形菌纲适应性强, 分布范围广, 在4个河流入海口均有发现。α-Proteobacteria变形菌纲在陆地土壤环境中占优势, 有可能是河流将泥沙带入入海口造成的[21]。其中夹河入海口优势种群变形菌门的气单胞菌属()和逛荡河入海口的优势种群拟杆菌门的黄杆菌属()为兼性厌氧菌, 常见于低氧水体[2]。可能由于此两处河流含有丰富且组成更为复杂的有机质, 微生物的分解导致水体含氧量下降[14]。
各水域的细菌种群多样性不同, 与所处的生态环境息息相关, 如果生态环境改变, 细菌的菌群结构也会发生变化[22, 24]。进一步考察季节等因素如何影响入海口处细菌多样性和群落结构变化, 对进一步了解烟台市区河流入海口环境状况, 改善河口海域水体质量, 维持生态系统稳定性及开发利用此生态环境的微生物资源具有重要意义。
[1] 臧红梅, 樊景凤, 王斌, 等. 海洋微生物多样性的研究进展[J]. 海洋环境科学, 2006, 25(3): 96-100.Zang Hongmei, Fan Jingfeng, Wang Bin, et al. Research progress on marine microbial diversity[J]. Marine Environmental Science, 2006, 25(3): 96-100.
[2] 刘敏, 朱开玲, 李洪波, 等. 应用PCR-DGGE技术分析黄海冷水团海域的细菌群落组成[J]. 环境科学, 2008, 29(4): 1082-1091. Liu Min, Zhu Kailing, Li Hongbo, et al. Bacterial community composition of the Yellow Sea cold water mass studied by PCR-denaturing gradient gel electrophoresis[J]. Environmental Science, 2008, 29(4): 1082- 1091.
[3] Muyzer G, De waal E C, Uitterlinden A G. Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction- amplified genes coding for 16S rRNA[J]. Applied and Environmental Microbiology, 1993, 59(3): 695-700.
[4] Earing J E, Durig A C, Gellin G L, et al. Bacterial colonization of the equine gut; Comparison of mare and foal pairs by PCR-DGGE[J]. Advances in Microbiology, 2012, 2(2): 79-86.
[5] Zhang Yi, Wang Gaochan, Wu Yupeng, et al. PCR-DGGE analysis of earth-worm gut bacteria diversity in stress of0157: H7[J]. Advances in Bioscience and Biotechnolog, 2013, 4(3): 437-441.
[6] Yu Zhongtang, Morrison M. Comparisons of different hypervariable regions of rrs genes for use in fingerprinting of microbial communities by PCR-Denaturing gradient gel electrophoresis[J]. Applied and Environmental Microbiology, 2004, 70(8): 4800-4806.
[7] 邢德峰, 任南琪. 应用DGGE研究微生物群落时的常见问题分析[J]. 微生物学报, 2006, 46(2): 331-335. Xing Defeng, Ren Nanqi. Common problems in the analyses of microbial community by denaturing gradient gel Electrophoresis (DGGE)[J]. Acta Microbiologica Sinica, 2006, 46(2): 331-335.
[8] Muyzer G, Smalla K. Application of denaturing gradient gel electrophoresis (DGGE) and temperature gradient gelelectrophoresis (TGGE) in microbial ecology[J]. Antonie Van Leeuwenhoek, 1998, 73(1): 127-141.
[9] Watanabe T, Asakawa S, Nakamura A, et al. DGGE method for analyzing 16S rDNA of methanogenic archaeal community in paddy field soil[J]. FEMS Microbiology Letters, 2004, 232(2): 153-163.
[10] 马悦欣, Holmstrm C, Webb J, 等. 变性梯度凝胶电泳(DGGE)在微生物生态学中的应用[J]. 生态学报, 2003, 23(8): 1561-1569. Ma Yuexin, Holmstrm C, Webb J, et al. Application of denaturing gradient gel electrophoresis (DGGE) in microbial ecology[J]. Acta Ecologica Sinica, 2003, 23: 1561-1569.
[11] 周琳, 张杰. 群落分析中的16S rRNA及其基因16S rDNA优化扩增[J]. 微生物学报, 2010, 50(1): 7-14. Zhou Lin, Zhang Jie. 16S rRNA gene in community structure analysis and the optimized amplification[J]. Acta Microbiologica Sinica, 2010, 50(1): 7-14.
[12] 肖慧, 张喆, 张艳, 等. 春秋季山东南部近岸浮游细菌生态分布[J]. 中国海洋大学学报, 2009, 39(4): 652- 656. Xiao Hui, Zhang Zhe, Zhang Yan, et al. Distribution characteristics of bacterioplankton in spring and autumnin costal waters of South Shandong[J]. Periodical of Ocean University of China, 2009, 39(4): 652-656.
[13] 白洁, 李海艳, 赵阳国.黄海北部不同站位海洋细菌群落分布特征[J]. 微生物学报, 2009, 49(3): 343-350.Bai Jie, Li Haiyan, Zhao Yangguo. Bacterial distribution at different stations in the Northern Yellow Sea[J]. Acta Microbiologica Sinica, 2009, 49(3): 343-350.
[14] 张健, 赵阳国, 李海艳, 等. 黄海西北近岸沉积物中细菌群落空间分布特征[J]. 海洋学报, 2010, 32(2): 118-127. Zhang Jian, Zhao Yangguo, Li Haiyan, et al. Temporal and spatial distribution characterization of bacterial community in sediments from the inshore of the northwest Huanghai Sea[J]. Acta Oceanologica Sinica, 2010, 32(2): 118-127.
[15] 李海艳. 黄海西北部真细菌群落结构及多样性研究[D]. 青岛: 中国海洋大学, 2009. Li Haiyan. Study of the eubacterial community structure and diversity in the northwest of Yellow sea[D]. Qing Dao: Ocean University of China, 2009.
[16] 周燕飞, 彭三妹, 王博林, 等.不同污水中细菌多样性DGGE检测[J]. 中国公共卫生, 2015, 31(8): 1075-1079. ZhouYanfei, Peng Sanmei, Wang Bolin, et al. Detection of bacterial diversity in clean and contaminated surface water with DGGE[J]. Chinese Journal of Public Health, 2015, 31(8): 1075-1079.
[17] 王雅苹, 史晓翀, 于少兰, 等. 青岛潮间带沉积物可培养厌氧细菌多样性的研究[J]. 海洋科学. 2015, 39(3): 92-99. Wang Yaping, Shi Xiaochong, Yu Shaolan, et al. Diversity of culturable anaerobic bacteria isolated from intertidal sediments of Qingdao[J]. Marine Sciences, 2015, 39(3): 92-99.
[18] 王恩辉, 张晓黎, 张莹, 等. 山东半岛北岸不同生境潮间带浮游细菌多样性研究[J]. 海洋科学. 2016, 40(6): 8-16. Wang Enhui, Zhang Xiaoli, Zhang Ying, et al. Bacterioplankton diversity in different littoral habitats along the coast of the northern Shandong peninsula[J]. Marine Sciences, 2016, 40(6): 8-16.
[19] 赵兴青, 杨柳燕, 陈灿, 等. PCR-DGGE技术用于湖泊沉积物中微生物群落结构多样性研究[J]. 生态学报, 2006, 26(11): 3610-3616. Zhao Xingqing, Yang Liuyan, Chen Can, et al. Study on the microbial diversity in lake sediments by the method of PCR-DGGE[J]. Acta Ecologica Sinica, 2006, 26(11): 3610-3616.
[20] Ravenschlag K, Sahm K, Peruthaler J, et al. High bacterial diversity in permanently cold marine sediments[J]. Applied and Environmental Microbiology, 1999, 65(9): 3982-3989.
[21] 张喆. 山东近岸海域浮游细菌、病毒生态学调查及沉积物细菌多样性研究[D]. 青岛: 中国海洋大学, 2008. Zhang Zhe. Distribution characteristics of bacterioplankton and virioplankton and bacterial diversity in sediments in coastal line of Shandong[D]. Qing Dao: Ocean University of China, 2008.
[22] 张艳. 山东近岸海域水体细菌多样性研究[D]. 青岛: 中国海洋大学, 2010. Zhang Yan. Diversity of bacterial communities in coastal areas of Shandong Province[D]. Qing Dao: Ocean University of China, 2010.
[23] 付新华, 刘国宁, 何健龙, 等. 山东省渤海海洋保护区典型海域表层海水微生物群落多样性分析[J]. 海洋科学, 2017, 41(1): 39-47. Fu Xinhua, Liu Guoning, He Jianlong, et al. Analysis of microbial community diversity in the Bohai Sea marine protected areas of the Shandong Province[J]. Marine Sciences, 2017, 41(1): 39-47.
[24] 姜彩虹, 张美玲, 陶琰洁, 等. 上海市内不同水质的河道春季浮游细菌群落结构分析[J]. 微生物学通报, 2009, 36(4): 522-527.Jiang Caihong, Zhang Meiling, Tao Yanjie, et al. Bacterial community structure in four different rivers of Shanghai in Spring[J]. Microbiology, 2009, 36(4): 522-527.
Analysis of microorganism community in river estuaries of Yantai
PU Yang1, LI Wen-jun2, WANG Kai3, ZHAO Yan-hong1, QIN Song2
(1. School of Agriculture, Ludong University, Yantai 264025, China; 2. Yantai Institute of Coastal Zone Research, Chinese Academy of Sciences, Yantai 264003, China; 3. Shanghai Ocean University, Shanghai 200090, China)
A total of 12 water samples were collected from four river estuaries of Yantai city (Xin,an River, Guangdang River, Yuniao River, and Jiahe River, respectively) in April 2015. The structure of the bacterial community and the dominant groups were examined by PCR-Denatured Gradient Gel Electrophoresis based on 16S rRNA PCR amplification(PCR-DGGE). Results of the analysis demonstrated the presence of high diversities and enrichment in the four river estuaries. Each of the 12 samples showed more than 30 bands, and the Shannon bacterial diversity index varied from 3.3 to 3.61. The Shannon bacterial diversity index was higher in Xinan River and Guangdang River, lower in Yuniao River, and the lowest in Jiahe River. The cluster analysis, by UPGMA analysis, showed that the closer the geographical position was, the higher was the similarity of the bacteria. A total of 14 major DGGE bands were cloned and sequenced. Phylogenetic analysis of the 14 samples showed that Proteobacteria and Bacteroidetes were dominant in the four river estuaries. These results were highly consistent with those of a previous study on the diversity of bacterial communities in the coastal areas of Shandong province. This study could be useful for protecting the environment in the river estuaries of Yantai.
Yantai; river estuaries; PCR-DGGE; microorganism diversity analysis
(本文编辑: 张培新)
P6040
A
1000-3096(2017)07-0009-07
10.11759/hykx20170112002
2017-01-12;
2017-05-17
鲁东大学科研启动基金(ly2014041); 山西省煤基重点科技攻关项目(FT2014-01)
[Ludong University Research Initiation Funds, No.ly2014041; Key Project of Coal-based Science and Technology in Shanxi Province, No. FT2014-01]
蒲洋(1981-), 男, 河北衡水人, 讲师, 主要从事分子藻类学的研究, 电话: 13884664603, E-mail: c5h12o6@sohu.com
Jan.12, 2017
