龙眼中性转化酶基因(DlNI)的克隆及分析
2017-11-16廖玲燕韩冬梅吴振先
帅 良,廖玲燕,韩冬梅,吴振先
(1.华南农业大学 园艺学院,广东 广州 510642;2.贺州学院 食品与生物工程学院/食品科学与工程技术研究院,广西 贺州 542899;3.广东省农业科学院 果树研究所,广东 广州 510640)
龙眼中性转化酶基因(DlNI)的克隆及分析
帅 良1,2,廖玲燕2,韩冬梅3*,吴振先1*
(1.华南农业大学 园艺学院,广东 广州 510642;2.贺州学院 食品与生物工程学院/食品科学与工程技术研究院,广西 贺州 542899;3.广东省农业科学院 果树研究所,广东 广州 510640)
【目的】中性转化酶是果实蔗糖代谢关键酶之一,克隆中性转化酶基因对于揭示龙眼果实糖代谢具有重要的意义。【方法】以‘石硖’龙眼试材,采用RT-PCR结合RACE技术成功克隆了3个龙眼中性转化酶基因全长cDNA序列。【结果】3个龙眼中性转化酶基因全长cDNA序列分别命名为DlNI-1、DlNI-2和DlNI-3,NCBI登录号为KP769773、KP769774和KP769775,其中DlNI-1全长2090 bp,编码589个氨基酸,比对结果发现与木薯(82 %)的中性转化酶基因的氨基酸序列同源性最高;DlNI-2全长2558 bp,编码709个氨基酸,比对结果发现与克莱门柚(80 %)的中性转化酶基因的氨基酸序列同源性最高;DlNI-3全长2444 bp,编码805个氨基酸,比对结果发现与克莱门柚(80 %)的中性转化酶基因的氨基酸序列同源性最高。3个龙眼中性转化酶基因的氨基酸序列都具有中性转化酶的12个高度保守的结构域,并且都具有2个催化残基,推测其蛋白都属于α类,且都定位于细胞器中。3个基因在不同组织中表达量具有较大差异,其中DlNI-1和DlNI-2在叶片中都具有较高的表达量,而DlNI-3在果皮组织中具有最高的表达量。【结论】成功克隆了3个龙眼中性转化酶基因,为后期深入研究中性转化酶基因结构和功能奠定基础。
龙眼;中性转化酶;基因克隆;表达
1 材料与方法
1.1 供试材料
试验材料为“石硖”龙眼(DimocarpuslonganLour.‘Shixia’),采自广东省农业科学院果树研究所种质资源圃。组织特异性表达样品分别使用“石硖”的根、茎、叶、花、幼果、成熟果实的果皮和果肉。其中根、茎、叶、幼果的采摘时间为龙眼果实谢花后30 d。
本实验中所用的Amp、X-gal、IPTG、氯化钠、氯化钙、四硼酸钠、硼酸、磷酸钠、LB培养基等试剂购自上海生物工程有限公司;RNAOUT试剂盒购自北京华越洋生物科技有限公司;琼脂糖、大肠杆菌感受态细胞、TaKaRa PCR Amplification kit、PMD 20-T Vector kit、TaKaRa 3’-Full RACE Core Set Ver.2.0和TaKaRa Gel DNA Purification Kit Ver2.0等购自TaKaRa公司;M-MLV逆转录酶购自美国Life公司;SMARTTM RACE cDNA Amplification Kit试剂盒购自Clontech公司;荧光定量试剂盒购自德国Roche公司。
1.2 试验方法
1.2.1 RNA的提取及cDNA的合成 龙眼不同组织总RNA的提取采用RNAOUT试剂盒,严格按照试剂盒说明书操作要求进行。去基因组DNA后,以总RNA为模板合成cDNA,反转录采用M-MLV (Life),引物采用Oligo(dT)15Primer (TaKaRa)。cDNA的合成反应体系按照说明书要求进行。反应体系于PCR仪中进行如下反应程序:37 ℃ 80 min,70 ℃ 15 min。得到的cDNA于-20 ℃冰箱中备用。
1.2.2 龙眼DlNI基因的中间片段克隆 依据GenBank上已经报道的其它物种的中性转化酶基因序列,设计简并引物P1和P2(表1)。以cDNA为模板进行PCR扩增,用1 %琼脂糖凝胶电泳检测扩增片段,切胶,回收,连接转化,测序。通过去载体分析所扩增的片段。
国家水资源监控能力建设。项目建设全面推进。积极开展三大监控体系建设,完成中央、流域、省级水资源监控管理三级信息平台和水资源管理业务三级通用软件开发;加强建设管理,完成制度体系建设;搭建技术框架,各地编制技术方案通过审查。
1.2.3 龙眼DlNI基因的3’-RACE克隆和5’-RACE克隆 根据所得到的片段序列,利用Primer Premier 5.0软件,参照TaKaRa 3’-Full RACE Core Set Ver.2.0试剂盒和Clontech SMARTTMRACE cDNA Amplification Kit要求设计3’-RACE(P3、P4、P5、P6、P7、P8)和5’-RACE(P9、P10、P11)引物(表1),严格按照说明书进行后续PCR扩增,目的基因片段回收、连接、转化、鉴定、测序及结果分析同上。
1.2.4 龙眼DlNI基因全长的获得及ORF验证 将上述获得的3’RACE和5’RACE结果序列和片段序列采用ContigExpress软件进行拼接,获得DlNI基因全长cDNA序列,通过使用NCBI的Blast和SIB的翻译工具(Translate tool)(http://web.expasy.org/translate/)进行序列分析,找出基因全长序列的开放阅读框(ORF),设计ORF序列引物(P12和P13、P14和P15、P16和P17),对ORF序列进行克隆,检测,以确定所拼接的序列为正确的基因序列(表1)。
1.2.5 龙眼DlNI基因的生物信息学分析 使用NCBI上的ORF Finder(http://www.ncbi.nlm.nih.gov/gorf/gorf.html)工具查找基因的ORF;使用SIB(Swiss Institute of Bioinformatics)的(Translate tool)(http://web.expasy.org/translate/)对ORF进行翻译;蛋白质分子量及理化性质分析采用ProtParam tool(http://web.expasy.org/protparam/)工具分析;使用NCBI上的Blastn和Blastx对序列进行比对分析;同源性及序列多重比较分析使用Clustal X 1.83软件并用GeneDoc软件进行美化编辑;运用MEGA 5软件中的Neighbor-Joining(邻位相连法,NJ)法构建系统进化树并进行编辑。

表1 龙眼中性转化酶基因克隆与表达分析中使用的引物
1.2.6 Real-time定量RT-PCR(RT-qPCR) 使用BatchPrimer3在线引物设计工具,进行RT-qPCR的引物序列设计(P18和P19、P20和P21、P22和P23、P24和P25、P26和P27)。RT-qPCR使用荧光染料为SYBR Green I Master(Roche),仪器为Roche Lightcycler®480,使用384孔模块。10 μl扩增体系:cDNA模板1 μl,5 μM的上游引物和下游引物各0.5 μl,SYBR Green I Master(Roche)5 μl,ddH2O 3 μl。使用DlActin和DlEF-1a基因作为双内参进行RT-qPCR分析[19-20]。并用相对定量软件对PCR结果的熔解曲线和标准曲线进行分析、定量。相对表达量计算采用2-△△CT法,由Roche Lightcycler®480软件系统自动分析得出结果。
1.3 数据处理与作图
使用Microsoft Office 2013软件进行数据处理和分析,使用Origin 8.5作图,并使用Adobe Illustrator CS6软件进行图形美化和编辑。
2 结果与分析
2.1 龙眼总RNA的提取
如图1所示,图中28S和18S两条rRNA条带完整清晰,且没有明显的拖带现象,28S条带比18S条带要亮,说明RNA完整性较好,未发生降解。经测定提取的龙眼各组织混合总RNA的OD260/OD280比值均在1.8 ~ 2.0,OD260/OD230比值均高于2.0,说明提取的龙眼总RNA的质量较高,纯度和完整性较好,可以用于基因克隆和荧光定量表达等后续分子生物学相关试验。

图1 龙眼总RNA的电泳图Fig.1 Electrophoresis pattern of total RNA of longan
2.2 龙眼糖代谢相关基因的全长扩增
以龙眼不同组织混合cDNA为模板,使用P1和P2引物进行PCR扩增,得到一条长度为400 bp左右的片段(图2A),切胶,回收,连接,转化后送测序。测序结果比对分析发现,本次扩增成功获得了3条长度为363 bp的龙眼NI基因序列,分别命名为DlNI-1,DlNI-2和DlNI-3。
根据测序得到的DlNI-1、DlNI-2、DlNI-3基因片段序列,设计3’RACE(P3、P4、P5、P6、P7、P8)引物(表1),按照TaKaRa 3’-Full RACE Core Set Ver.2.0试剂盒内的PCR条件进行巢式PCR扩增,电泳检测后均得到与目的片段相符的特异性片段,DlNI-1、DlNI-2和DlNI-3的3’RACE第二轮扩增产物约600、700和600 bp(图2B),这些片段大小基本与预期相符合,切胶,回收,连接,转化后送测序,测序结果分析表明,所获得的3’末端序列与扩增的片段序列有相应的高度重复的片段,说明各基因的3’末端序列扩增成功。
根据测序得到的糖代谢相关基因片段DlNI-1、DlNI-2、DlNI-3的序列,设计5’RACE(P9、P10、P11)引物(表1),按照Clontech SMARTTMRACE cDNAAmplification Kit试剂盒内PCR条件进行扩增,电泳检测后均得到相应的与目的片段相符的特异性片段,DlNI-1、DlNI-2和DlNI-3的5’RACE扩增产物约1500、1700和1700 bp左右(图2C),这些片段大小基本与预期相符合,切胶,回收,连接,转化后送测序,测序结果分析表明,所获得的5’末端序列与扩增的片段序列有相应的高度重复的片段,说明各基因的5’末端序列扩增成功。

M:DL2000;A:中间片段扩增结果;B:3’端片段扩增结果;C:5’端片段扩增结果;D:ORF序列扩增结果M: DL2000; A: Intermediate fragment; B: 3’ RACE result; C: 5’ RACE result; D: PCR product of ORF图2 龙眼DlNI-1、DlNI-2和DlNI-3基因序列扩增Fig.2 PCR product of DlNI-1, DlNI-2 and DlNI-3 gene from longan
根据扩增的糖代谢相关基因片段以及3’RACE和5’RACE结果,使用ContigExpress软件对片段序列进行拼接,分别获得各基因全长cDNA序列,再使用NCBI上的ORF Finder对序列ORF进行查找,根据ORF序列设计ORF全长(P12和P13、P14和P15、P16和P17)引物(表1),验证拼接全长序列的准确性,使用LATaq酶进行PCR扩增,经电泳检测后均发现与预测片段大小相一致的条带,DlNI-1、DlNI-2和DlNI-3的ORF扩增产物约1900、2300和2100 bp左右(图2D),切胶,回收,连接,转化后送测序,测序结果去载体后经Blast比对分析发现,各基因条带的ORF序列都正确,结果表明成功获得了DlNI-1、DlNI-2和DlNI-3基因全长cDNA序列。
2.3 龙眼DlNI-1、DlNI-2和DlNI-3基因的序列分析
图3显示,DlNI-1基因序列全长2090 bp,包括一个长度为96 bp的5’UTR和224 bp的3’UTR,ORF长度为1770 bp,编码589个氨基酸,蛋白质的分子质量为66.40 kDa,等电点为6.18,蛋白质的不稳定系数为47.87,脂溶指数为89.61,其疏水性平均值为﹣0.187,表明该蛋白为亲水不稳定脂溶蛋白,NCBI的登录号为KP769773,经Blastn比对后发现与甜橙(XM_006471320.1和XM_006471319.1)和克莱门柚(XM_006424241.1)的NI基因核苷酸序列同源性最高,都高达89 %,BlastX比对结果显示,与木薯(XP_007024490.1)、蓖麻(XP_002529075.1)和甜橙(XP_006471382.1)的NI基因推导的氨基酸序列同源性最高,分别为82 %,81 %和76 %。
DlNI-2基因序列全长2558 bp,包括一个长度为113 bp的5’UTR和315 bp的3’UTR,ORF长度为2130 bp,编码709个氨基酸,蛋白质的分子质量为79.91 kDa,等电点为7.92,蛋白质的不稳定系数为38.03,脂溶指数为85.64,其疏水性平均值为﹣0.114,表明该蛋白为亲水稳定脂溶蛋白,NCBI的登录号为KP769774,经Blastn比对后发现与甜橙(XM_006472173.1)和克莱门柚(XM_006433502.1)的NI基因核苷酸序列同源性最高,都高达87 %,BlastX比对结果显示,与克莱门柚(XP_006433565.1)和甜橙(XP_006472236.1、KDO81624.1)的NI基因推导的氨基酸序列同源性最高,都高达80 %。
DlNI-3基因序列全长2444 bp,包括一个长度为259 bp的5’UTR和148 bp的3’UTR,ORF长度为2037 bp,编码678个氨基酸,蛋白质的分子质量为76.09 kDa,等电点为5.77,蛋白质的不稳定系数为35.94,脂溶指数为90.28,其疏水性平均值为-0.205,表明该蛋白为亲水稳定脂溶蛋白,NCBI的登录号为KP769775,经Blastn比对后发现与甜橙(XM_006488730.1)和克莱门柚(XM_006419242.1)的NI基因核苷酸序列同源性最高,都高达84 %,BlastX比对结果显示,与克莱门柚(XP_006419305.1)和甜橙(XP_006488793.1)的NI基因推导的氨基酸序列同源性最高,都高达80 %。

下划线表示保守区;箭头表示催化残基Underline indicate the motifs; Arrows show the catalytic residues图3 眼DlNI-1、DlNI-2和DlNI-3基因氨基酸序列比对Fig.3 Alignment of DlNI-1,DlNI-2 and DlNI-3 amino acid sequences
2.4 龙眼DlNI-1、DlNI-2和DlNI-3基因的多序列比对及进化树分析
龙眼DlNI-1、DlNI-2和DlNI-3基因推导的氨基酸序列比对结果表明,DlNI-1的氨基酸序列与DlNI-2和DlNI-3的氨基酸序列一致性为74 %和65 %,DlNI-2的氨基酸序列与DlNI-3的氨基酸序列一致性为68 %,同时这3个基因的氨基酸序列都具有中性转化酶的12个高度保守的结构域,并且都具有两个催化残基[21]。
为进一步分析龙眼中性转化酶氨基酸序列的进化关系,本试验从拟南芥中挑选At1g22650、At1g35580、At1g56560、At1g72000、At3g05820、AT3G06500、At4g09510、AT4G34860和At5g22510共9个中性转化酶氨基酸序列[22],从水稻中挑选BAH00142、BAH00419、BAG89564和BAG95698 4个中性转化酶氨基酸序列[21, 23],和龙眼DlNI-1、DlNI-2和DlNI-3基因的氨基酸序列构建进化树。
由进化树结果可知,中性转化酶可以明显的分为α和β两大类[13, 21],其中α类的中性转化酶主要定位于细胞器中,而β类的中性转化酶则定位于非细胞器中,而α类又可以分为两大类,一类定位于线粒体中,另一类则在定位于质体中[23]。本试验克隆的3个龙眼中性转化酶蛋白推测都属于α类,即都定位于细胞器中,其中龙眼DlNI-1与拟南芥At5g22510的进化关系较近,可能定位于质体中,而DlNI-2和DlNI-3分别与At1g56560和AT3G06500进化关系较近,可能定位于线粒体中(图4)。
2.5 龙眼DlNI-1、DlNI-2和DlNI-3基因的不同组织特异性表达分析
龙眼不同组织中DlNI-1、DlNI-2和DlNI-3基因的表达量相差较大,DlNI-1基因在叶片中具有最大的表达量,其次为花、幼果、根、果皮和果肉,表达量最小的为茎中(图5 A);DlNI-2基因在叶片中具有最大的表达量,其次为花、果皮、幼果、根和茎,表达量最小的为果肉中(图5 B);DlNI-3基因在果皮中具有最大的表达量,其次为叶片、根、花和幼果中,而在果肉中表达量较低,在茎中几乎不表达(图5 C)。由此可知DlNI-1和DlNI-2基因可能在调节叶片组织中蔗糖含量的高低起作用,而DlNI-3基因在果皮组织中起调节果皮蔗糖含量。
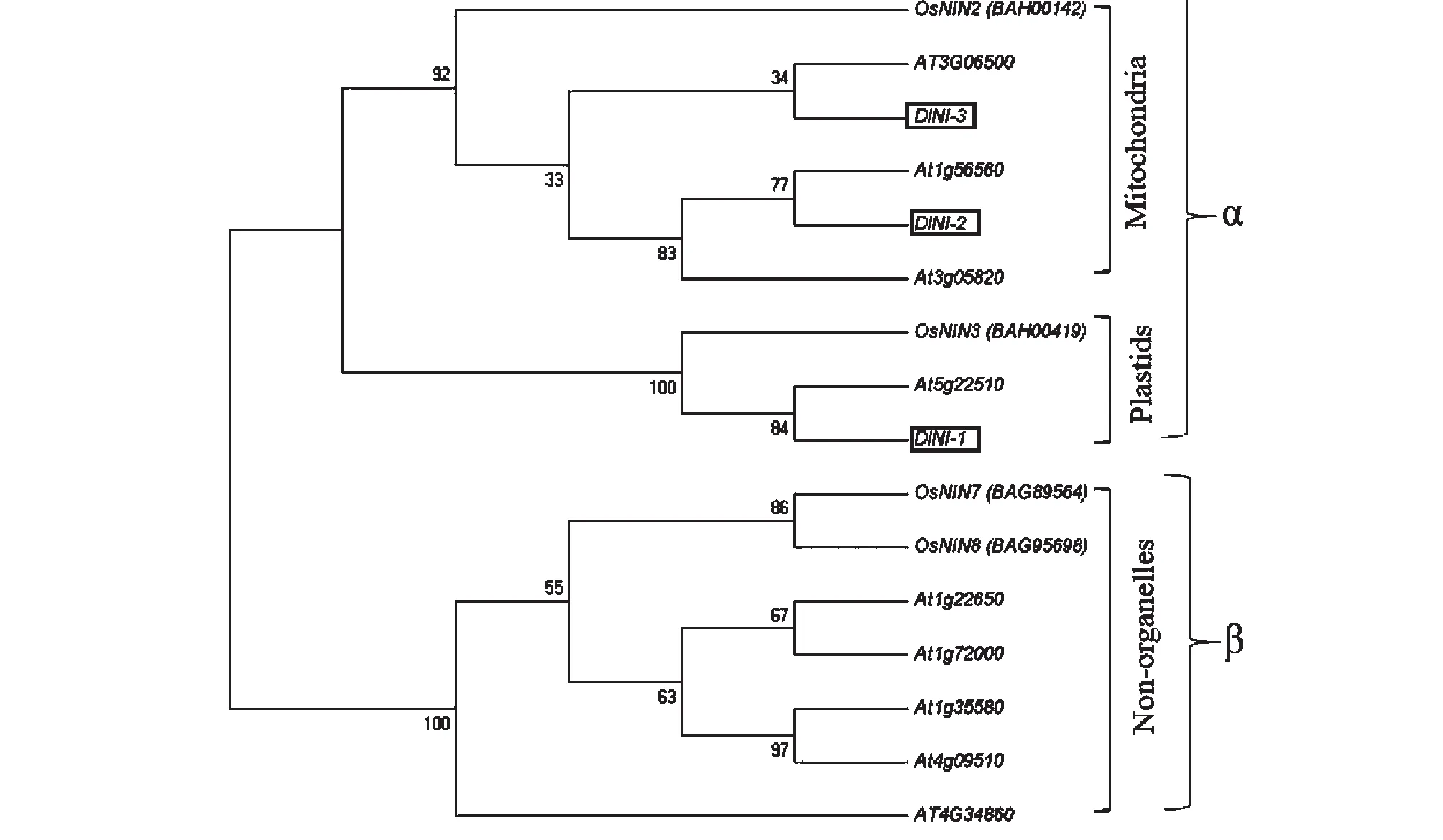
图4 龙眼DlNI-1、DlNI-2和DlNI-3系统进化树分析Fig.4 Phylogenetic analysis of DlNI-1, DlNI-2 and DlNI-3 and other plant NI amino acid sequences

图5 龙眼DlNI-1(A)、DlNI-2(B)和DlNI-3(C)基因在不同组织中的表达Fig.5 DlNI-1 (A), DlNI-2 (B) and DlNI-3 (C) gene expression in different tissues of longan
3 讨论与结论
中性转化酶最基本的功能是分解蔗糖成为葡萄糖和果糖,相比于在酸性转化酶上的研究成果,目前对中性转化酶的功能了解还较少,主要原因是因为中性转化酶蛋白具有不稳定性和低活性的特点[5, 24]。随着近年来对其研究的深入,发现中性转化酶在影响拟南芥侧根生长,百脉根花粉形成及开花的过程中起主要作用[25-26]。通过RT-PCR结合RACE技术成功克隆了3个龙眼中性转化酶基因全长cDNA序列,分别命名为DlNI-1、DlNI-2和DlNI-3,NCBI登录号为KP769773、KP769774和KP769775。其中DlNI-1全长2090 bp,编码589个氨基酸,氨基酸序列比对结果发现与木薯和蓖麻的中性转化酶基因的氨基酸序列同源性最高;DlNI-2全长2558 bp,编码709个氨基酸,氨基酸序列比对结果发现与甜橙和克莱门柚的中性转化酶基因的氨基酸序列同源性最高;DlNI-3全长2444 bp,编码805个氨基酸,氨基酸序列比对结果发现与甜橙和克莱门柚的中性转化酶基因的氨基酸序列同源性最高。同源性比对发现,3个中性转化酶基因都具有12个中性/碱性转化酶所特有的保守域,这些保守域具体功能还不是很清楚[21, 24],进化树结果表明,所有的中性转化酶基因可以分类α和β两大类[13, 21],其中α类的中性转化酶主要定位于细胞器中,而β类的中性转化酶则在非细胞器中,其中α类又可以分为两大类,一类在定位于线粒体中,而另一类则定位于质体中[23]。分析发现龙眼DlNI-1可能定位于质体中,而DlNI-2和DlNI-3分别可能定位于线粒体中。不同组织特异性表达结果显示,DlNI-1、DlNI-2和DlNI-3 3个基因在不同组织中表达量具有较大差异,其中DlNI-1和DlNI-2在叶片中都具有较高的表达量,这与在番茄[14]和大豆[27]中对中性转化酶的研究结果一致。DlNI-1和DlNI-2在花中的表达量也较高,可能是由于植物进入生殖生长时期,库器官生长发育,不断催化蔗糖水解为葡萄糖和果糖,对能量物质需求过大所导致。而DlNI-3在果皮组织中具有最高的表达量,其次为根、叶片中,而在茎中几乎不表达,由此可知DlNI-3在龙眼根生长中不起作用,而在调节龙眼果皮中碳水化合物含量多少起主要作用。
[1]Wang L, Li X R, Lian H, et al. Evidence That High Activity of Vacuolar Invertase Is Required for Cotton Fiber and Arabidopsis Root Elongation through Osmotic Dependent and Independent Pathways, Respectively[J]. Plant Physiology, 2010, 154(2): 744-756.
[2]Sturm A. Invertases. Primary structures, functions, and roles in plant development and sucrose partitioning[J]. Plant Physiol, 1999, 121(1): 1-8.
[3]Jin Y, Ni D A, Ruan Y L. Posttranslational elevation of cell wall invertase activity by silencing its inhibitor in tomato delays leaf senescence and increases seed weight and fruit hexose level[J]. Plant Cell, 2009, 21(7): 2072-2089.
[4]Sturm A, Tang G Q. The sucrose-cleaving enzymes of plants are crucial for development, growth and carbon partitioning[J]. Trends Plant Sci, 1999, 4(10): 401-407.
[5]Roitsch T, González M. Function and regulation of plant invertases: sweet sensations[J]. Trends in Plant Science, 2004, 9(12): 606-613.
[6]Winter H S H. Regulation of sucrose metabolism in higher plants: localization and regulation of activity of key enzymes[J]. Critical Reviews in Biochemistry and Molecular Biology, 2000,35(4): 253.
[7]Barratt D H, Derbyshire P, Findlay K, et al. Normal growth of Arabidopsis requires cytosolic invertase but not sucrose synthase[J]. Proc Natl Acad Sci U S A, 2009,106(31):13124-13129.
[8]Welham T, Pike J, Horst I, et al. A cytosolic invertase is required for normal growth and cell development in the model legume, Lotus japonicus[J]. Journal of Experimental Botany, 2009, 60(12): 3353-3365.
[9]王 菲. 枳中性转化酶基因PtrNINV克隆及其功能分析[D]. 华中农业大学, 2013.
[10]Gallagher J A, Pollock C J. Isolation and characterization of a cDNA clone fromLoliumtemulentumL. encoding for a sucrose hydrolytic enzyme which shows alkaline/neutral invertase activity[J]. Journal of Experimental Botany, 1998, 322(49): 789-795.
[11]Vargas W A, Pontis H G, Salerno G L. Differential expression of alkaline and neutral invertases in response to environmental stresses: characterization of an alkaline isoform as a stress-response enzyme in wheat leaves[J]. Planta, 2007, 226(6): 1535-1545.
[12]刘术金,李旖璠,唐朝荣. 橡胶树2个胶乳转化酶基因的原核表达[J]. 热带作物学报,2010(7):1091-1097.
[13]Bocock P N, Morse A M, Dervinis C, et al. Evolution and diversity of invertase genes in Populus trichocarpa[J]. Planta, 2008, 227(3): 565-576.
[14]高媛媛,杨郁文,张保龙,等. 番茄中性/碱性蔗糖转化酶基因的电子克隆、分析及表达载体的构建[J]. 江苏农业科学,2009(6):36-38.
[15]牛俊奇,王爱勤,黄静丽,等. 甘蔗中性/碱性转化酶基因SoNIN1的克隆和表达分析[J]. 作物学报, 2014(2):253-263.
[16]姜立智,林长发, 梁宗锁,等. 水稻蔗糖转化酶基因的克隆及其功能的初步探讨[J]. 复旦学报(自然科学版),2003(4):588-592.
[17]Yang Z, Wang T, Wang H, et al. Patterns of enzyme activities and gene expressions in sucrose metabolism in relation to sugar accumulation and composition in the aril ofLitchichinensisSonn.[J]. Journal of Plant Physiology, 2013, 170(8): 731-740.
[18]帅 良, 钱盼红, 刘文浩, 等. 不同龙眼品种果实成熟时糖含量及其特征研究[J]. 热带作物学报, 2016(5):915-921.
[19]帅 良,李 静,韩冬梅,等. 龙眼己糖激酶基因的克隆及原核表达[J]. 华南农业大学学报, 2015(3):91-97.
[20]帅 良,薛晓清,牛佳佳,等. 龙眼果实发育过程中果糖激酶活性及其基因表达分析[J]. 华南农业大学学报,2015(5):99-104.
[21]Ji X, Van den Ende W, Van Laere A, et al. Structure, Evolution, and Expression of the Two Invertase Gene Families of Rice[J]. Journal of Molecular Evolution, 2005,60(5): 615-634.
[22]Vargas W, Cumino A, Salerno G L. Cyanobacterial alkaline/neutral invertases. Origin of sucrose hydrolysis in the plant cytosol?[J]. Planta, 2003, 216(6): 951-960.
[23]Murayama S, Handa H. Genes for alkaline/neutral invertase in rice: alkaline/neutral invertases are located in plant mitochondria and also in plastids[J]. Planta, 2007, 225(5): 1193-1203.
[24]Ruan Y L, Llewellyn D J. Suppression of Sucrose Synthase Gene Expression Represses Cotton Fiber Cell Initiation, Elongation, and Seed Development[J]. THE PLANT CELL ONLINE, 2003, 15(4): 952-964.
[25]俞 锞, 李志邈, 万红建, 等. 高等植物蔗糖转化酶功能的研究进展[J]. 安徽农业科学, 2013(33):12815-12818.
[26]Qi X, Wu Z, Li J, et al. AtCYT-INV1, a neutral invertase, is involved in osmotic stress-induced inhibition on lateral root growth in Arabidopsis[J]. Plant Molecular Biology, 2007, 64(5):575-587.
[27]王 彬,徐志华,侯金锋,等. 大豆细胞质转化酶基因GmCInv的克隆与植物表达载体构建[J]. 大豆科学,2014(2):168-172.
CloningandSequenceAnalysisofNeutralInvertaseGene(DlNI)fromLonganFruit
SHUAI Liang1,2, LIAO Ling-yan2, HAN Dong-mei3*, WU Zhen-xian1*
(1.College of Horticulture, South China Agricultural University, Guangdong Guangzhou 510642, China; 2.College of Food and Biological Engineering/Institute of Food Science and Engineering Technology, Hezhou University, Guangxi Hezhou 542899, China; 3.Institute of Fruit Tree Research, Guangdong Academy of Agricultural Sciences, Guangdong Guangzhou 510640, China)
【Objective】Neutral invertase was a key enzyme of fruit sucrose metabolism, and cloning neutral invertase gene sequences had a significance for sugar metabolism. 【Method】TakenDimocarpuslongan‘Shixia’ cultivar as tested materials, the whole lengths of its three neutral invertase gene cDNA were cloned and sequenced by RT-PCR and RACE. 【Result】Three neutral invertase gene cDNAs named asDlNI-1,DlNI-2 andDlNI-3, the NCBI numbers were KP769773, KP769774 and KP769775, of which theDlNI-1 gene consisted of 2090 bp encoding a polypeptide of 589 amino acids, which had the highest homology with theManihotesculentacrantz(82 %) through the NCBI blastx; TheDlNI-2 gene consists of 2558 bp encoding a polypeptide of 709 amino acids, which had the highest homology with theCitrusclementina(80 %) through the NCBI blastx; TheDlNI-3 gene consists of 2444 bp encoding a polypeptide of 805 amino acids, which had the highest homology with theCitrusclementine(80 %) through the NCBI blastx. Amino acids of three longan neutral invertase gene sequence had 12 highly conservative domain structure and two catalytic residues, it was concluded that the three neutral invertase protein belonged to α class and located in organelles. The expression level of three neutral invertase genes had bigger difference in different tissues, theDlNI-1 andDlNI-2 had the highest expression level in leaves, but theDlNI-3 had the highest expression in pericarp. 【Conclusion】Three longan neutral invertase gene were cloned, which would provide references for the investigation of the relation between structure and function of neutral invertase gene.
Dimocarpuslongan; Neutral invertase; Gene cloning; Expression
1001-4829(2017)10-2202-08
10.16213/j.cnki.scjas.2017.10.008
2016-07-20
国家现代农业产业技术体系(荔枝龙眼CARS-33-14)资助;广东省扬帆计划项目(2014YT02H013)
帅 良(1986-),男,江西新干人,博士,研究方向:农产品贮藏保鲜,E-mail:shuailiang1212@163.com;*为通讯作者:韩冬梅,E-mail: handm2009@qq.com;吴振先,E-mail:litchi2008@126.com。
S667.2
A
(责任编辑 陈 虹)
