用高基元微卫星标记分析中国糜子遗传多样性
2017-11-11王瑞云刘笑瑜王海岗陆平刘敏轩陈凌乔治军
王瑞云,刘笑瑜,王海岗,陆平,刘敏轩,陈凌,乔治军
用高基元微卫星标记分析中国糜子遗传多样性
王瑞云1,2,刘笑瑜1,王海岗2,陆平3,刘敏轩3,陈凌2,乔治军2
(1山西农业大学农学院,山西太谷030801;2山西省农业科学院农作物品种资源研究所/农业部黄土高原作物基因资源与种质创制重点实验室/杂粮种质资源发掘与遗传改良山西省重点实验室,太原030031;3中国农业科学院作物科学研究所,北京100081)
开发高基元(4—6)碱基重复微卫星标记,分析种质资源遗传多样性,为糜子遗传和进化研究提供理论基础。用隶属函数、主成分分析和聚类分析综合评价糜子资源表型多样性,用前期糜子转录组测序获得高基元SSR引物对地理来源差异大的糜子材料进行PCR扩增检测其多态性,用PowerMarker 3.25计算遗传多样性参数,用PopGen 1.32计算Nei’s遗传距离,用MEGA 5.0进行聚类分析,用Structure 2.2鉴定遗传类群。96份糜子资源株高和穗长变异最丰富,多样性指数分别为2.08和1.91。PCR扩增发现,占56.29%的85对引物具多态性,其中四、五和六碱基重复引物分别为71对(83.53%)、10对(11.76%)和4对(4.7%)。85个标记扩增产物大小分布为100—450 bp,PIC值平均为0.51,Rp值为1.00—5.75,平均为3.15。四、五和六碱基重复SSR的平均Rp值分别为3.15、2.8和4.0。基于Rp值分析SSR的分布频次,发现85个标记分布区间为0—1、1—2、2—3、3—4、4—5和5—6,分别包含1(1.18%)、15(17.65%)、31(36.47%)、20(23.53%)、12(14.12%)和6(7.06%)个标记,60%(51个)的标记分布在区间2—3和3—4。用85个SSR扩增96份糜子资源,共检测到232个等位变异,每个位点检测到等位变异2—3个,平均2.7294个;62个位点产生3个变异,23个位点产生2个变异;多样性指数为0.2842—1.0633,平均为0.7708;PIC值为0.0400—0.7281,平均为0.4723。不同生态区糜子种质间的遗传距离为0.0093—0.5052(平均为0.1798),遗传一致度为0.6034—0.9907(平均为0.8485)。基于UPGMA将96个糜子基因型聚为4个群组,第一群组主要属于北方春糜子区;第二群主要属于东北春糜子区;第三群组主要属于华北夏糜子区;第四群组主要属于黄土高原春夏糜子区。遗传结构分析将96份试材划分为4个类群,分别代表黄土高原、华北、东北和北方基因库。UPGMA聚类分析和遗传结构分析结果基本一致,均与地理起源相关。在糜子中构建了85个四、五和六碱基重复微卫星标记,这些高基元SSR的引物分辨率(Rp)高,对不同基因型分辨能力强,PCR扩增多态性好;用其评估中国糜子资源的遗传差异发现,黄土高原春夏糜子区和北方春糜子区资源遗传多样性最丰富。
糜子;高基元SSR;遗传多样性;聚类分析;遗传结构
0 引言
【研究意义】糜子(L.)又叫黍稷,为旱地农业重要经济作物。糜子在亚洲中部和东部、印度、非洲、中东、东欧(俄罗斯和乌克兰)和北美均有分布[1]。糜子脱壳后称黄米,糯质黄米可用于蒸糕、炸油糕、包粽子和酿酒,粳质黄米多用于制作凉粉、煎饼、黄米酸饭和炒米[2]。糜子在欧美主要用于饲鸟和禽畜育肥。糜子作为C4植物,蒸腾速率低,在干旱、高温等恶劣条件下快速成熟,是干旱、半干旱地区的稳产作物。中国是糜子起源地,资源丰富,明确不同种质的遗传背景,准确评估糜子遗传多样性,有利于糜子作物的合理开发和高效利用。【前人研究进展】分子标记是评估作物遗传差异和鉴定群体间亲缘关系远近的有效手段。由于糜子是异源四倍体,基因组复杂,序列信息缺乏,分子标记的开发相对欠缺。1994—2006年相继开发的适用于糜子多样性分析的分子标记(RAPD、AFLP、RFLP、ISSR等)非常有限[3-6],尤其是变异丰富、多态性高、重复性好、检测能力强[7]的SSR标记更少。2008—2014年,HU等[8]和RAJPUT等[9]分别开发了46个和254个来自小麦、水稻、燕麦和柳枝稷的糜子非特异性微卫星标记,然而种间差异的存在影响遗传多样性评估准确性。2010年,CHO等[10]首次开发了糜子特异性微卫星标记25个,随后利用上述标记评估糜子遗传资源差异的研究渐次进行。2011年,HUNT等[11]利用其中16对引物分析了欧亚大陆的98份糜子资源,检测到78个等位基因,每个位点检测到等位变异为2—15个(平均4.9个),基因多样性指数和多态性信息含量(polymorphism information content,PIC)分别为0.391和0.360;2015年,连帅等[12]利用5对SSR引物分析来源于5个糜子生态区的40份资源的多样性,检测到15个等位变异,平均为3个,遗传多样性指数和PIC值分别为0.76和0.48;董俊丽等[13]利用其中19对引物检测96份糜子种质,发现112个等位变异,基因多样性指数和PIC值分别为0.4097和0.392。2016年,刘笑瑜等[14]选用其中6对SSR引物分析中国不同省份的40份糜子资源,发现20个等位变异,遗传多样性指数和PIC值分别为0.5426和0.3403;2017年,王瑞云等[15]用15对引物,评估不同生态区132份糜子的遗传多样性,共检测到107个等位变异,每个位点鉴定到的2—14个(平均7个),基因多样性指数和PIC值分别为0.5298和0.4864。近年来,随着高通量测序技术的广泛运用,全基因组SSR标记批量挖掘取得长足进展。2016年,RAJPUT等[16]用100个SSR(80个来自柳枝稷、7个来自水稻、6个为糜子、5个来自小麦、2个来自燕麦)评估了欧亚和北美等25个国家的90份糜子资源,检测到1 287个等位变异(长度为40—1 500 bp),每个位点检测到等位变异1—29个(平均4.9个),每份材料检测到等位变异41—317。同年,LIU等[17]和连帅等[18]用糜子高通量测序开发的130条SSR引物分析了280份国内外的糜子栽培品种和野生资源。【本研究切入点】糜子基因组序列未知,可供遗传分析的SSR标记数量有限,且以往研究所用标记多为低基元(二、三核苷酸)序列重复。【拟解决的关键问题】基于RNA-Seq手段筛选到一批高基元(四、五、六核苷酸)序列重复SSR,并用以评估糜子遗传多样性,以期为糜子连锁图谱构建及进化研究提供更多分子检测工具,批量筛选高基元微卫星标记,明确引物分辨率特征。
1 材料与方法
1.1 供试材料
试验材料包括96份种质资源,来源于国家资源库的地方品种(有国家种质资源库统一编号)76份、笔者实验室征集的地方品种(标注为农家种,无国家种质资源库统一编号)18份和育成品种2份(附表1),分布于6个糜子生态栽培区(表1)。

表1 糜子资源生态区分布
1.2 表型性状测量
96份糜子资源分别于2015年5月12日和2016年5月14日种植在山西农业大学农作站,分5行点播,每行10株,株距5 cm,行距8 cm,小区间距50 cm。参照王星玉等[19]的方法,成熟期测量表型性状(株高、穗长、分蘖数、主茎节数、千粒重、叶长、叶宽)。
1.3 基因组DNA提取
取15—20 d糜子幼苗(1株)叶片约0.3 g,用改良CTAB法[20]提取基因组DNA。利用1%琼脂糖凝胶电泳检测DNA质量,用紫外微量核酸仪(NanoDropND-1000,Eppendorf)测定DNA纯度和浓度。
1.4 SSR分析
用地理来源差异显著的8份糜子材料对前期抗旱和抗盐相关转录组测序获得的151个高基元SSR引物进行初步筛选(附表2和附表3)。85个标记可以扩增出清晰度高且重复性好的多态性片段(附表4,由上海生工生物工程技术服务有限公司合成),用于评估96份糜子资源遗传多样性。
PCR反应体系(20 μL)包括10×buffer(Mg2+含量25 mmol·L-1)2 μL、10 mmol·L-1dNTP 1.8 μL,5 U·L-1Taq聚合酶0.4 μL、1 mmol·L-1前后引物各0.6 μL、ddH2O 13.6 μL和30 ng·μL-1DNA模板1 μL。PCR扩增在LifeEco基因扩增仪(TC-96/G/H(b)c,杭州博日科技有限公司)上进行,程序为94℃5 min;94℃45 s,不同Tm退火50 s,72℃1 min,38个循环;72℃10 min。用8%聚丙烯酰胺凝胶电泳检测PCR扩增产物,硝酸银染色显影。等位变异长度用50 bp DNA Marker(北京庄盟国际生物基因科技有限公司)比对条带。
1.5 数据分析
用SAS软件对各性状进行主成分和相关性分析。利用模糊隶属函数法计算各性状的隶属函数值以进行综合评价及Shannon-wiener遗传多样性指数评价,方法参考董俊丽等[13]。
凝胶条带表示标记(>50 bp)大小,相邻条带大小相差25 bp。SSR标记如果在至少2份材料扩增出不同的DNA条带,则具有多态性。DNA条带代表等位变异,相同条带表示相同等位变异。引物分辨率(resolving power,Rp)计算按照Prevost等[21]方法,Rp = ∑,其中,1-(2×︱0.5 -︱),为某个等位基因信息量,为某个等位基因在6份材料中出现的频率。用PowerMarker 3.25[22]计算每对引物的多样性参数,包括等位基因数(Na)、等位基因频率、基因多样性指数(H)、多态性信息含量指数(PIC)等。用PopGen1.32[23]进行群体间聚类分析。用MEGA 5.0[24]构建Neighbour-Joining聚类图。用Structure 2.2[25]分析群体遗传结构。
2 结果
2.1 糜子表型遗传多样性分析
对96份糜子资源的7个表型性状进行了表型多样性分析(表2),7项指标变异系数为18.21%—42.19%,分蘖数最大,主茎节数和株高次之,叶长最小,说明糜子不同种质分蘖数相差较大。糜子7项指标遗传多样性指数为1.78—2.08,株高最大,说明株高变异丰富。
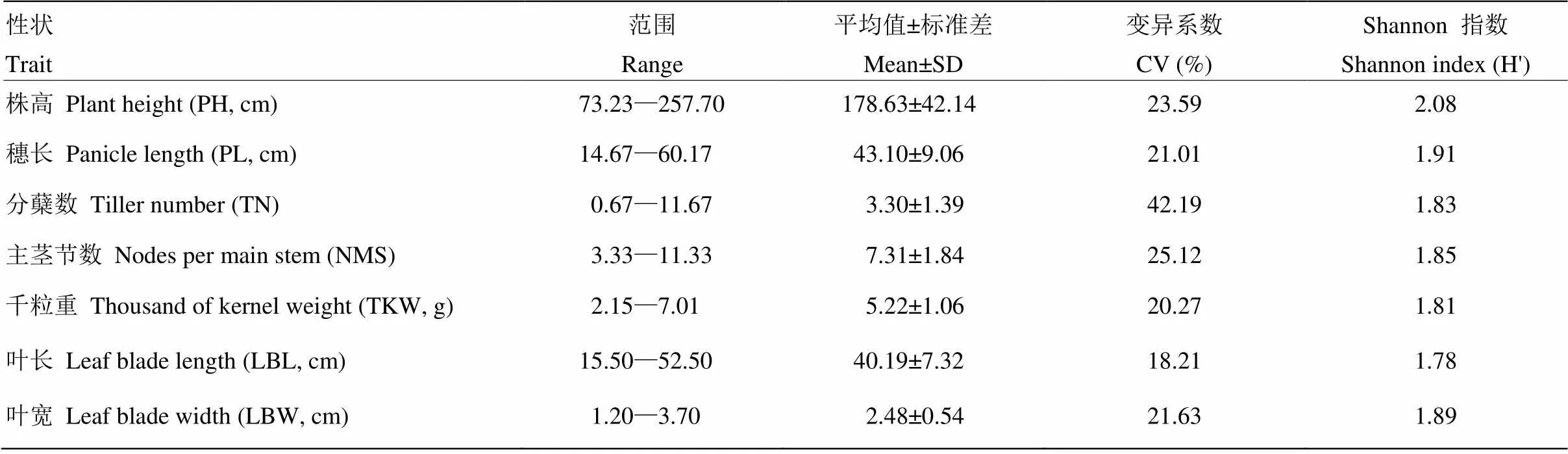
表2 糜子表型性状多样性分析
2.2 糜子表型性状综合分析
对96份糜子资源的表型性状进行相关性分析(表3)。21对相关性中,13对(61.9%)呈正相关,8对呈负相关,7对(33.3%)极显著相关,1对(千粒重和分蘖数)显著相关。其中,株高与主茎节数相关系数最大(0.601),其次为叶长与叶宽(0.561),说明株高越高,主茎节数越多,叶长越长,叶宽也越大。分蘖数和主茎节数极显著负相关(-0.216),说明主茎节数越少,分蘖数越多。
对96份糜子资源进行主成分分析(表4)。结果表明,前5个主成分累积贡献率为91.51%,包含表型特征多数信息,可用以综合分析糜子资源。其中,第一主成分贡献率33.29%,株高载荷值最大,第一主成分值大的资源,株高较高。第二主成分和第五主成分均为分蘖数载荷值最大,累积贡献率29.36%,说明第二和第五主成分值大的资源分蘖数多;同时第五主成分穗长和千粒重均为负值,且绝对值较大,育种中选择第五主成分小的种质,分蘖数少,穗长和千粒重大。第三主成分千粒重、株高和穗长载荷值均较大,为产量相关性状,在育种中可选择第三主成分值大的种质。第四主成分千粒重、叶长和叶宽贡献率均较大,株高、穗长为负值,绝对值较大,说明第四主成分值高的种质,千粒重大、叶片长而宽,但植株低、穗短,可用于矮杆、高产育种的选育。
分析糜子资源表型性状隶属值分布(图1)。株高主要集中在4—9级,占材料的84.38%;穗长主要集中在5—8级,占材料的77.08%;分蘖数主要集中在2—4级,占材料的84.38%;主茎节数主要集中在3—8级,占材料的82.29%;千粒重主要集中在5、6、8和10级,占材料的68.75%,叶长主要集中在6—10级,占材料的80.12%,叶宽主要集中在4—8级,占材料的76.04%。综合评价各试验材料隶属函数平均值,发现甘肃省的榆中黄糜(00002787)的值最高(0.738),综合评价最好;山西孝义黄糜子值最低(0.231),综合评价最低。
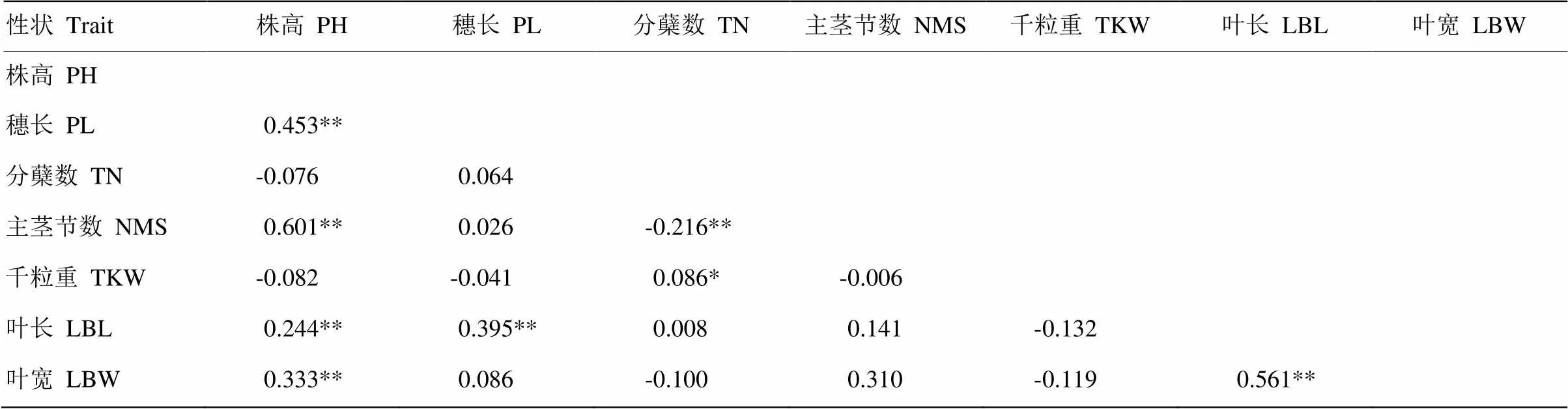
表3 糜子表型性状间的相关性分析
*和** 分别表示在0.05和0.01水平差异显著
* and ** indicate significant difference at the probability level of 0.05 and 0.01, respectively
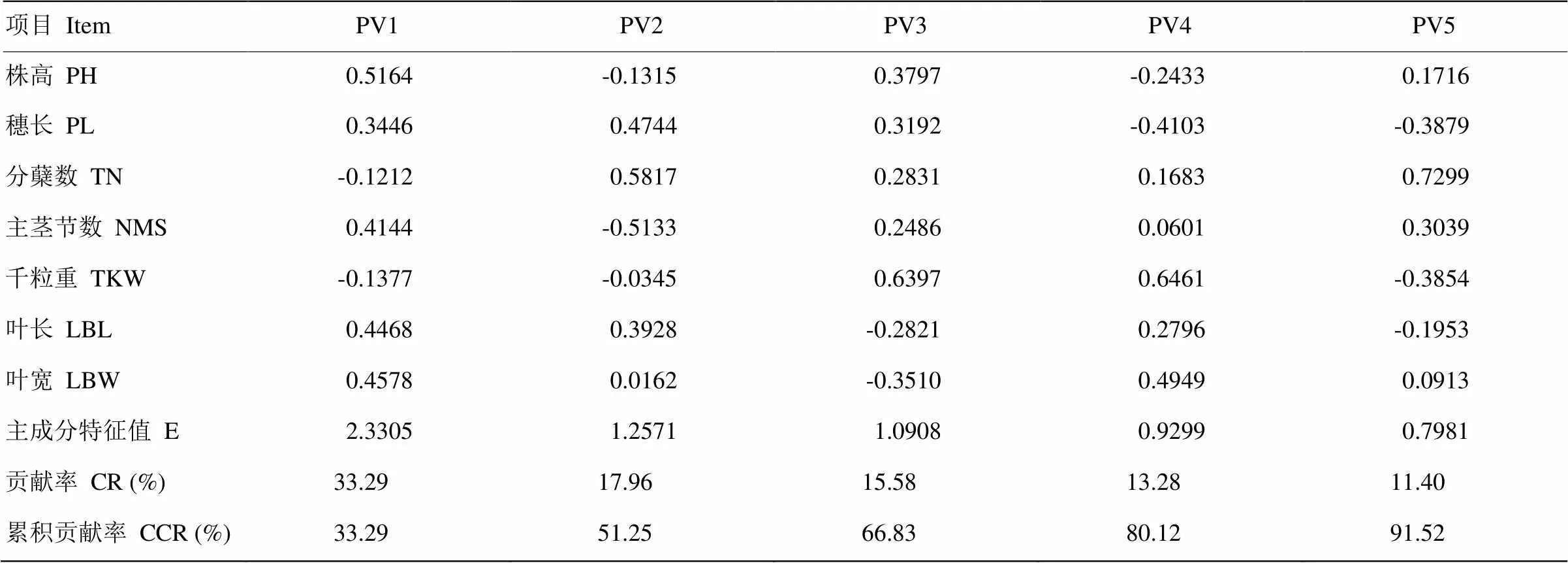
表4 表型性状5个主成分的特征向量、主成分特征值、贡献率及累积贡献率
2.3 高基元SSR引物筛选
山西农业大学农学院糜子分子育种课题组前期基于RNA-Seq筛选到151对高基元SSR引物,用这些引物扩增6份糜子材料。发现25对扩增不出DNA条带,126对(83.4%)有扩增条带。126对引物中,41对扩增的条带呈单态性,85对(附表4)呈多态性,占151对引物的56.3%。其中四、五和六碱基重复引物分别为71对(83.5%)、10对(11.8%)和4对(4.7%)。

图1 糜子资源表型性状隶属值分布
引物分辨率(Rp值)是衡量引物对不同基因型辨别能力的指标。85对高基元SSR的引物特征(等位基因大小和Rp值)见附表5和图2。从附表5可以看出,85个SSR的等位基因为100—450 bp;85个SSR的Rp值为1.00—5.75(平均3.15),标记RYW42的Rp值最小,标记RYW47的Rp值最大;四、五和六碱基重复SSR的平均Rp值分别为3.15、2.8和4.0,六碱基重复SSR数量最少(4个),但Rp值较高,可能与其重复单元差异较大有关。基于Rp值分析85个SSR的分布频次(图2),发现频次分布区间为0—1、1—2、2—3、3—4、4—5和5—6,分别包含1(1.18%)、15(17.65%)、31(36.47%)、20(23.53%)、12(14.12%)和6(7.06%)个标记,60%(51个)的标记分布在区间2—3和3—4。

图2 85个糜子多态性SSR标记的Rp值
2.4 基于高基元SSR分析糜子资源遗传多样性
用85对SSR引物扩增96份试材,分析糜子资源遗传多样性(附表6)。从附表6可以看出,85个标记在96份试材中共检出232个等位变异,每个位点检测到等位变异2—3个,平均为2.7294个;62个位点产生3个变异,23个SSR位点产生和2个变异。85个位点多样性指数为0.2842(RYW75)—1.0633(RYW37),平均为0.7708。85个位点PIC值为0.1133(RYW46)—0.7281(RYW8),平均为0.4723。
分析不同生态区糜子资源的遗传多样性(表5),结果发现,北方春糜子区PIC值、多样性指数及观测等位基因数等遗传多样性衡量指标最高,黄土高原春夏糜子区次之,南方秋冬糜子区均最低,说明北方春糜子区和黄土高原春夏糜子区遗传多样性比较丰富。
2.5 不同生态区糜子种质间遗传相似性分析
利用PopGen 1.32计算不同生态区糜子种质间的遗传相似性(表6)。结果表明,遗传距离为0.0093—0.5052(平均0.1798)。遗传一致度为0.6034—0.9907(平均0.8485)。南方秋冬糜子区与西北春夏糜子区遗传距离最大,遗传一致度最低,这与2个生态区地理分布较远、气候环境差别大、亲缘关系较远有关;北方春糜子区和黄土高原春夏糜子区遗传距离最小,遗传一致度最高,这与2个生态区相邻、气候差异小、亲缘关系较近有关。说明遗传距离越大,亲缘关系越远,遗传相似性越低。

表5 不同生态区糜子的遗传多样性分析

表6 不同生态区糜子资源的遗传距离与遗传一致度
Nei氏遗传一致度(对角线以上)和遗传距离(对角线以下)Nei’s genetic identity (above diagonal) and genetic distance (below diagonal)
2.6 基于UPGMA的糜子资源聚类分析
基于UPGMA对不同生态区糜子进行聚类(图3),发现6个生态区资源在遗传距离0.1827处划分为4个类群(类群Ⅰ、Ⅱ、Ⅲ和Ⅳ)。类群Ⅰ将南方秋冬糜子区归为一类,试材来自海南。类群Ⅱ将西北春夏糜子区归为一类,试材来自新疆。类群Ⅲ将北方春糜子区和黄土高原春夏糜子区归为一类,试材来自青海、甘肃、内蒙古、山西、陕西和宁夏。类群Ⅳ将东北春糜子区和华北夏糜子区归为一类,试材来自黑龙江、吉林、辽宁、河北、山东、江苏和河南。
基于UPGMA对96个糜子基因型进行聚类(图4),发现96份试材归为4个群组,第一群组30份,包括山西2份、陕西3份、宁夏4份、内蒙古3份、青海7份,甘肃9份、新疆2份,大部分基因型属于北方春糜子区和黄土高原春夏糜子区;第二群组20份,其中内蒙古3份、山西6份、黑龙江5份、吉林3份及陕西、辽宁和江苏各1份,主要属于东北春糜子区;第三群组11份,包括河北9份、山东2份,属于华北夏糜子区;第四群组35份,包括青海6份、甘肃1份、内蒙古4份、山西12份、陕西4份、宁夏6份、海南和河南各1份,主要属于黄土高原春夏糜子区和北方春糜子区。各群组分布与地理来源基本相符。

图3 基于遗传距离的不同生态区糜子资源聚类图
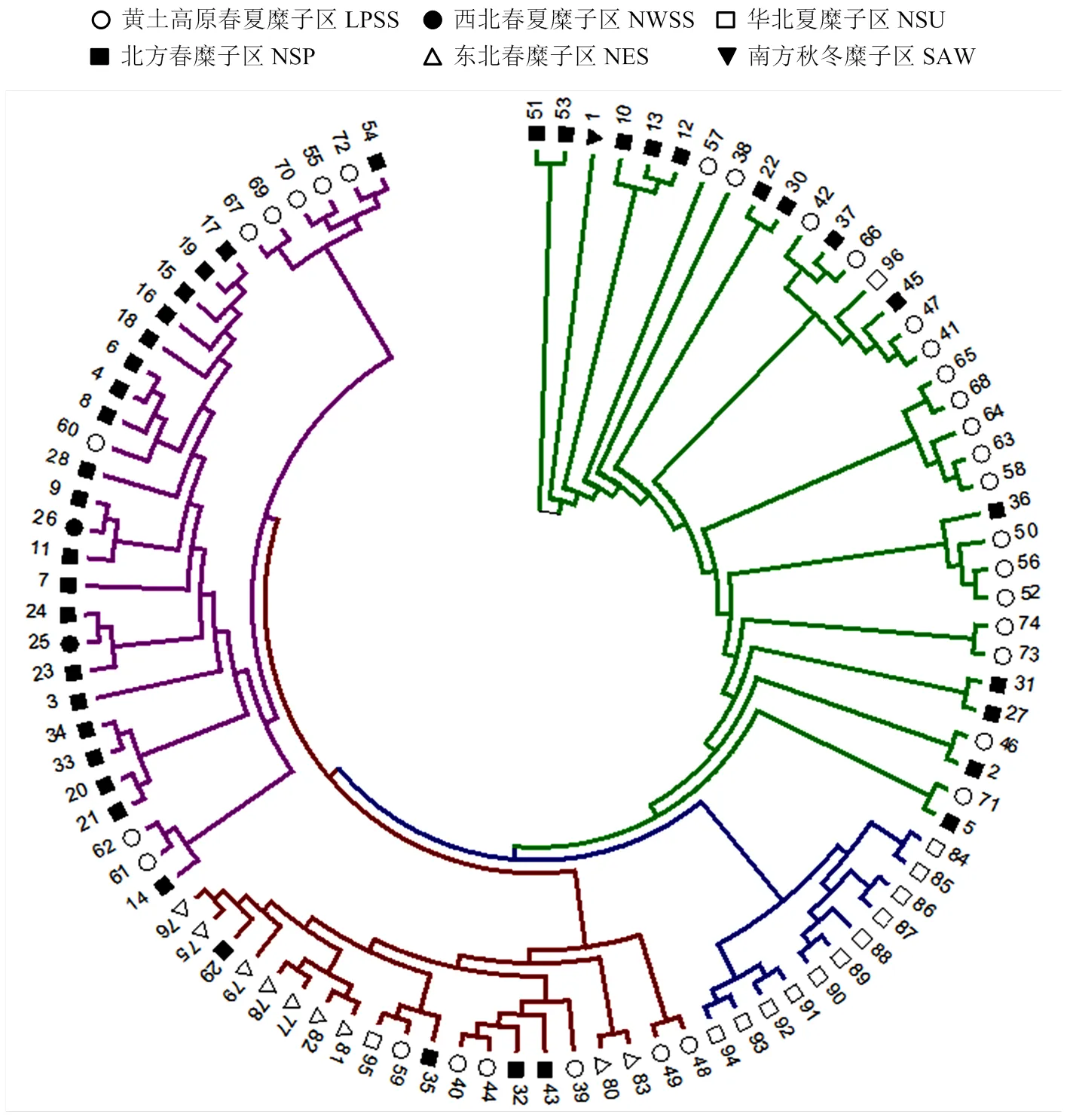
图4 基于遗传距离的96份糜子种质资源聚类图
2.7 基于模型的群体结构分析
对96份试材进行基于模型的群体结构分析(图5)。从图5可以看出,等位变异频率特征数(遗传群体数)在=2、=4和=9处峰值明显,基于=4和=9峰值基本相同,且越大且值越小模拟结果越可靠,仅对=2和=4进行分析,分别将试材划分为2和4个群组(图6)。由图6可知,=2时,划分为2个类群,类群Ⅰ为红色(60份),主要来自于北方春糜子区和黄土高原春夏糜子区;类群Ⅱ为绿色(36份),主要来自于北方春糜子区和黄土高原春夏糜子区;=4时,划分为4个类群,类群Ⅰ为红色(19份),代表黄土高原基因库,多样性指数和PIC值均最高;类群Ⅱ为绿色(22份),代表东北和华北基因库;类群Ⅲ为蓝色(20份),代表黄土高原基因库和北方基因库;类群Ⅳ为黄色(35份),代表黄土高原基因库和北方基因库。

Delta K根据Evanno等[26]的方法计算得到,针对基因库数目(K)建模

横坐标的数字代表糜子材料序号 Numbers in the horizontal axis represent serial number of accession
=2红色类群分化为红、绿、蓝和黄色(=4),14份山西糜子中1份(7.14%)为红色,蓝色和黄色分别为10份(71.43%)和3份(21.43%)。10份青海糜子中1份(10%)属于蓝色,9份(90%)属于黄色。
=2绿色类群半数以上材料没分化,其余分化为红色和蓝色(=4),6份山西材料为红色,8份河北材料中,绿色和红色分别为7份(87.5%)和1份(12.5%)。
分析=2和=4遗传结构图各分类群的遗传多样性参数(表7),结果发现,=2时,红色类群和绿色类群分别占62.5%和37.5%;就多样性指数和PIC值而言,绿色类群(分别为0.7726和0.4845)高于红色类群(分别为0.7499和0.4379)。绿色类群资源主要来自于河北、宁夏和山西,地区间相距较远,气候条件差异大,资源间遗传多样性丰富;红色类群糜子主要来自于山西、甘肃和内蒙古,这些地区彼此接壤,气候相似,资源间亲缘关系近,遗传一致性程度高。=4时,黄色类群资源最多(53.03%);红色类群的PIC值和多样性指数均为最大(分别为0.4868和0.7627)。
3 讨论
3.1 高基元糜子特异性SSR是分析糜子遗传多样性的有效标记
基于SSR引物初步筛选,983个种间SSR中,209个(38%)扩增不出条带[9];本研究用PCR扩增151个标记,发现25个(16.56%)无结果,这些SSR可能受到基序插入、缺失、点突变、侧翼重复缺失等因素影响,引起DNA电泳条带不出现[27]。
用983个水稻等种间SSR扩增118份糜子资源,46个标记(4.7%)扩增出多态性片段[8]。用548个柳枝稷SSR扩增8份糜子材料,254个标记(46.4%)扩增片段具多态性[9]。本研究用151个SSR扩增6份糜子材料,85个标记(56.3%)扩增出多态性片段,明显高于上述研究结果,这可能与引物来自糜子基因组有关,糜子特异性标记筛选效果优于种间标记。

表7 遗传结构图中各分类群的多样性统计
转录组测序是开发SSR标记的有效手段,用糜子高通量测序获得的500对SSR引物扩增8份糜子材料,162个标记(32.4%)扩增出多态性片段[17],低于本研究结果,可能与本研究引物为高基元有关。本研究首次利用RNA-Seq开发了一批高基元糜子微卫星引物,为糜子育种提供了有效分子检测工具。
Rp值(引物分辨率)反映SSR多态性,直接关联标记信息[21],Rp值越高,遗传多样性越丰富。以往研究发现254和100个糜子非特异性SSR(6个为糜子细菌人工染色体克隆除外)的Rp值分别为2.71和5.43[9,16],与本研究结果(3.15)基本一致;同时发现,糜子分子标记的Rp值和苎麻[28]的研究结果(3.22)类似,低于密花石斛(6.15)、柽麻(6.59)和穿心莲(10.8)[29-31]。Rp值与糜子种特异性/非特异性引物的相关性大小有待于进一步研究。本研究首次构建的一批高基元SSR,不仅丰富了糜子作物DNA标记,而且也为糜子连锁图谱构建和基因组功能研究提供了理论依据。
高基元SSR多态性低于低基元多态性,小麦中已有报道[32],糜子中也检测到类似结果。用低基元微卫星标记分析糜子遗传多样性,192、132和118份资源的PIC值分别为0.4855[18]、0.5298[15]和0.793[8],本研究高基元SSR检测结果为0.4723,明显低于上述结果。
迄今,糜子中可用的高级基元SSR极少,仅包括1个糜子种特异性SSR[10,13]和3个种间SSR[9]。其中,糜子特异性标记EF117731由CHO等[10]开发,为四碱基重复“(AGTC)4”。CHO等[10]用该标记检测50份糜子材料,发现期望杂合度为0.04,PIC值为0.04;董俊丽等[13]用该标记检测评估96份糜子的遗传差异,发现基因多样性指数为0.2112,PIC值为0.2035;与上述研究类似,本研究也筛选到一个四碱基重复引物“(AGTT)6”,遗传多样性分析发现期望杂合度(0.4575)、基因多样性指数(0.6500)和PIC值(0.3226)均高于上述研究结果,这可能与本研究所选试材地理来源差异大、资源丰富有关。
3.2 中国糜子资源的遗传多样性和群体结构
用RAPD标记分析5个黍属植物遗传多样性,发现糜子多态性高,且划分类群与地理来源一致[3]。用RFLP标记比较12份栽培和野生糜子资源,发现UPGMA聚类结果与地理来源无关[5]。上述相互冲突的结果可能与材料来源及检测标记不同有关。本研究用85个高基元SSR扩增96份试材,发现多样性指数(0.7708)和PIC值(0.4723)均高于RAPD和低基元SSR的标记鉴定结果(最高分别为0.725和0.3920)[3,10-15,17-18],这可能与本研究试材来源广泛(6个生态区)、多样性丰富有关,也可能与高基元引物有关。
基于遗传结构欧亚大陆糜子资源划分群组与地理来源相符[11],基于遗传距离美国糜子资源划分群组与地理来源、系谱发生及农艺特征相符[16],基于遗传结构和遗传距离中国糜子资源聚类群组与地理来源相关[13,15]。本研究基于UPGMA将6个生态区及96个基因型均划分为4个主要类群,分别属于北方、黄土高原、东北和华北糜子区;遗传结构分析将96个基因型划归4个主要的基因库:北方、黄土高原、东北和华北基因库。遗传距离和遗传结构聚类结果基本一致,均与地理起源相关,表明不同生态区内遗传变异广泛,糜子不同群体间遗传关系复杂[17]。本研究西北基因库和南方基因库资源选材偏少、青藏高原基因库资源缺乏,未来研究需丰富材料来源,有利于准确评价中国糜子资源的遗传多样性。
4 结论
在糜子中开发了85个四、五和六碱基重复微卫星标记,分析这些SSR的引物分辨率(Rp),发现信息量大、多态性高。运用该高基元分子检测系统评估中国糜子资源的遗传差异,发现聚类群组与地理起源相关,其中北方春糜子区和黄土高原春夏糜子区遗传多样性最丰富。
[1] MOTUZAITE-MATUZEVICIUTE G, STAFF R A, HUNT H V, LIU X Y, JONES M K. The early chronology of broomcorn millet() in Europe., 2013, 87: 1073-1085.
[2] 柴岩, 冯佰利, 王宏岩. 中国黄米食品. 杨凌: 西北农林科技大学出版社, 2012.
CHAI Y, FENG B L, WANG H Y.. Yangling: Northwest A&F University Press, 2012. (in Chinese)
[3] M’RIBU H K, HILU K W. Detection of interspecific and intraspecific variation inmillets through random amplified polymorphic DNA., 1994, 88: 412-416.
[4] LÁGLER R, GYULAI G, HUMPHREYS M, SZABÓZ, HORVÁTH L, BITTSÁNSZKY A, KISS J, HOLLY L, HESZKY L. Morphological and molecular analysis of common millet () cultivars compared to a DNA sample from the 15th century (Hungary)., 2005, 146: 77-85.
[5] KARAM D, WESTRA P, NISSEN S J, WARD S M, FIGUEIREDO J E F. Genetic diversity among proso millet () biotypes assessed by AFLP technique., 2004, 22: 167-174.
[6] KARAM D, WESTRA P, NISSEN S J, WARD S M, FIGUEIREDO J E F. Assessment of silver-stained AFLP markers for studying DNA polymorphism in proso millet (L.)., 2006, 29: 609-615.
[7] VAN INGHELANDT D, MELCHINGER A E, LEBRETON C, STICH B. Population structure and genetic diversity in a commercial maize breeding program assessed with SSR and SNP markers., 2010, 120: 1289-1299.
[8] HU X Y, WANG J F, LU P, ZHANG H S. Assessment of genetic diversity in broomcorn millet (L) using SSR markers., 2009, 36(8): 491-500.
[9] RAJPUT S G, TAMMY P H, DIPAK K S. Development and characterization of SSR markers in proso millet based on switchgrass genomics., 2014, 5(1): 175-186.
[10] CHO Y I, CHUNG J W, LEE G A, MA K H, DIXIT A, GWAG J G, PARK Y J. Development and characterization of twenty-five new polymorphic microsatellite markers in proso millet (L.)., 2010, 32: 267-273.
[11] HUNT H V, CAMPANA M G, LAWES M C, PARK Y J, BOWER M A, HOWE C J, JONES M K. Genetic diversity and phylogeography of broomcorn millet (L) across Eurasia., 2011, 22: 4756-4771.
[12] 连帅, 王瑞云, 马跃敏, 刘笑瑜, 季煦. 不同生态区糜子种质资源的遗传多样性分析. 山西农业大学学报(自然科学版), 2015, 35(3): 225-231.
LIAN S, WANG R Y, MA Y M, LIU X Y, JI X. Genetic diversity of broomcorn millet (L.) germplasms of different ecotype zone of China., 2015, 35(3): 225-231. (in Chinese)
[13] 董俊丽, 王海岗, 陈凌, 王君杰, 曹晓宁, 王纶, 乔治军. 糜子骨干种质遗传多样性和遗传结构分析. 中国农业科学, 2015, 48(16): 3121-3131.
DONG J L, WANG H G, CHEN L, WANG J J, CAO X N, WANG L, QIAO Z J. Analysis of genetic diversity and structure of proso millet core germplasm., 2015, 48(16): 3121-3131. (in Chinese)
[14] 刘笑瑜, 王瑞云, 刘敏轩, 邱岩岩, 季煦, 连帅, 乔治军, 王纶, 王海岗. 利用SSR标记分析40份糜子资源的遗传多样性. 分子植物育种, 2016, 14(6): 1631-1636.
LIU X Y, WANG R Y, LIU M X, QIU Y Y, JI X, LIAN S, QIAO Z J, WANG L, WANG H Z. Genetic diversities among 40 parts of broomcorn millet revealed by SSR markers., 2016, 14(6): 1631-1636. (in Chinese)
[15] 王瑞云, 季煦, 陆平, 刘敏轩, 许月, 王纶, 王海岗, 乔治军. 利用荧光SSR分析中国糜子遗传多样性. 作物学报, 2017, 43(4): 530-548.
Wang R Y, Ji X, Lu P, Liu M X, XU Y, Wang L, Wang H G, Qiao Z J. Analysis of genetic diversity in common millet () using fluorescent SSR in China., 2017, 43(4): 530-548. (in Chinese)
[16] RAJPUT S G, SANTRA D K. Evaluation of genetic diversity of proso millet () germplasm available in the USA using SSR markers., 2016, 56: 1-9.
[17] LIU M X, XU Y, HE J H, ZHANG S, WANG Y Y, LU P. Genetic diversity and population structure of broomcorn millet (L.) cultivars and landraces in China based on microsatellite markers., 2016, 17(3): E370.
[18] 连帅, 陆平, 乔治军, 张琦, 张茜, 刘敏轩, 王瑞云. 利用SSR分子标记研究国内外黍稷地方品种和野生资源的遗传多样性. 中国农业科学, 2016, 49(17): 3264-3275.
LIAN S, Lu P, QIAO Z J, ZHANG Q, ZHANG Q, LIU M X, WANG R Y. Genetic diversity in broomcorn millet (L.) from China and abroad by using SSR markers., 2016, 49(17): 3264-3275. (in Chinese)
[19] 王星玉, 王纶. 黍稷种质资源描述规范与数据标准. 北京: 中国农业出版社, 2006.
WANG X Y, WANG L.L.. Beijing: China Agriculture Press, 2006. (in Chinese)
[20] EDWARDS K, JOHNSTONE C, THOMPSON C. A simple and rapid method for the preparation of plant genomic DNA for PCR analysis.1991, 19: 1349.
[21] PREVOST A, WILKINSON M J. A new system of comparing PCR primers applied to ISSR fingerprinting of potato cultivars., 1999, 98(1): 107-112.
[22] LIU K, MUSE S V. PowerMarker: integrated analysis environment for genetic marker data., 2005, 21: 2128-2129.
[23] YEH F C, BOYLE T J. Population genetic analysis of co-dominant and dominant markers and quantitative traits., 1997, 129: 157.
[24] TAMURA K, PETERSON D, PETERSON N, STECHER G, NEI M, KUMAR S. MEGA5: Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance and maximum parsimony methods., 2011, 665: 2731-2739.
[25] FALUSH D, STEPHENS M, PRITCHARD J K. Inference of population structure using multilocus genotype data: linked loci and correlated allele frequencies., 2003, 164: 1567-1587.
[26] Evanno G, Regnaut S, Goudet J. Detecting the number of cluster of individuals using the software structure: a simulation study., 2005, 14: 2611-2620.
[27] YU J, LA ROTA M, KANTETY R, SORRELLS M. EST derived SSR markers for comparative mapping in wheat and rice., 2004, 271(6): 742-751.
[28] SATYA P, KARAN M, JANA S, MITRA S, SHARMA A, KARMAKAR P G, RAY D P. Start codon targeted (SCoT) polymorphism reveals genetic diversity in wild and domesticated populations of ramie (L. Gaudich.), a premium textile fiber producing species., 2015, 3: 62-70.
[29] SATYA P, BANERJEE R, KARAN M, MUKHOPADHYAY E, CHAUDHARY B, BERA A, MARUTHI R T, SARKAR S K. Insight into genetic relation and diversity of cultivated and semi-domesticated under-utilizedspecies gained using start codon targeted (SCoT) markers., 2016, 66: 24-32.
[30] BHATTACHARYA P, KUMARIA S, KUMAR S, TANDON P. Start Codon Targeted (SCoT) marker reveals genetic diversity ofLindl., an endangered medicinal orchid species., 2013, 529: 21-26.
[31] TIWARI G, SINGH R, SINGH N, CHOUDHURY D R, PALIWAL R, KUMAR A, GUPTA V. Study of arbitrarily amplified (RAPD and ISSR) and gene targeted (SCoT and CBDP) markers for genetic diversity and population structure in Kalmegh [(Burm. f.) Nees]., 2016, 86: 1-11.
[32] DREISIGACKER S, ZHANG P, WARBURTON M L, VAN GINKEL M, HOISINGTON D, BOHN M, MELCHINGER A E. SSR and pedigree analyses of genetic diversity among CIMMYT wheat lines targeted to different megaenvironments., 2004, 44(2): 381-388.
(责任编辑 李莉,岳梅)
附表1 96份糜子试材明细表
Table S1 The detail of 96 accessions of common millet material in this experiment

编号Serial number统一编号Unicode名称Name原产地Origin来源Accession donor备注Remark 100006653金黍Jinshu海南省琼海市Qionghai, HainanICS, CAAS地方品种Landrace 200007435黑硬糜Heiyingmi青海省乐都县Ledu, QinghaiICS, CAAS地方品种Landrace 300007445褐红糜Hehongmi青海省乐都县Ledu, QinghaiICS, CAAS地方品种Landrace 400007463白皮糜Baipimi青海省平安县Ping’an, QinghaiICS, CAAS地方品种Landrace 500007469灰麻糜Huimami青海省循化县Xunhua, QinghaiICS, CAAS地方品种Landrace 600007478白圪塔糜Baigedami青海省湟中县Huangzhong, QinghaiICS, CAAS地方品种Landrace 700007491二白糜Erbaimi青海省湟中县Huangzhong, QinghaiICS, CAAS地方品种Landrace 800007503褐红糜Hehongmi青海省西宁市Xining, QinghaiICS, CAAS地方品种Landrace 900007468灰糜子Huimizi青海省循化县Xunhua, QinghaiICS, CAAS地方品种Landrace 1000007473灰糜子Huimizi青海省循化县Xunhua, QinghaiICS, CAAS地方品种Landrace 1100007450白鸽子蛋Baigezidan青海省民和县Minhe, QinghaiICS, CAAS地方品种Landrace 1200007504白老糜Bailaomi青海省西宁市Xining, QinghaiICS, CAAS地方品种Landrace 1300007471青扫帚糜Qingsaozhoumi青海省循化县Xunhua, QinghaiICS, CAAS地方品种Landrace 1400007443黄硬糜Huangyingmi青海省乐都县Ledu, QinghaiICS, CAAS地方品种Landrace 1500002783榆中小黄糜Yuzhongxiaohuangmi甘肃省榆中县Yuzhong, GansuICS, CAAS地方品种Landrace 1600000711靖远中堡青糜Jingyuanzhongpuqingmi甘肃省靖远县Jingyuan, GansuICS, CAAS地方品种Landrace 17红糜子Hongmizi甘肃省靖远县Jingyuan, Gansu永新乡卧中村Wozhong, Yongxin农家种Accession from farmer 1800002787榆中黄糜Yuzhonghuangmi甘肃省榆中县Yuzhong, GansuICS, CAAS地方品种Landrace 1900007320金积小黄糜Jinjixiaohuangmi甘肃省GansuICS, CAAS地方品种Landrace 2000007333黄糜子Huangmizi甘肃省GansuICS, CAAS地方品种Landrace 2100002776皋兰鸡蛋青Gaolanjidanqing甘肃省皋兰县Gaolan, GansuICS, CAAS地方品种Landrace 2200002780白银五砚黄糜Baiyinwuguanhuangmi甘肃省兰州市Lanzhou, GansuICS, CAAS地方品种Landrace 2300000718华池红软糜子Huachihongruanmizi甘肃省华池县Huachi, GansuICS, CAAS地方品种Landrace 2400002695永昌糜子Yongchangmizi甘肃省永昌县Yongchang, GansuICS, CAAS地方品种Landrace 2500000750白糜子Baimizi新疆沙湾县Shawan, XinjiangICS, CAAS地方品种Landrace 2600007345疏勒附糜Shulefumi新疆沙湾县Shawan, XinjiangICS, CAAS地方品种Landrace 2700000635达旗黄杆大白黍Daqihuanggandabaimi内蒙古伊盟达旗Yimengdaqi, Inner MongoliaICS, CAAS地方品种Landrace 2800002487伊盟大黄糜Yimengdahuangmi内蒙古伊盟达旗Yimengdaqi, Inner MongoliaICS, CAAS地方品种Landrace 2900002250临河黄糜子Linhehuangmizi内蒙古巴盟临河Linhe, Bameng, Inner MongoliaICS, CAAS地方品种Landrace 30伊糜5号Yimi 5内蒙古伊盟达旗Yimengdaqi, Inner MongoliaICSAR, SAAS育成品种Bred variety 3100004268小红黍Xiaohongshu内蒙古Inner MongoliaICS, CAAS地方品种Landrace 3200007224东胜野糜Dongshengyemi内蒙古伊盟东胜Dongsheng, Yimeng, Inner MongoliaICS, CAAS地方品种Landrace 3300007225异交野糜内蒙古伊盟东胜Dongsheng, Yimeng, Inner MongoliaICS, CAAS地方品种Landrace 3400007238大红糜子Yijiaoyemi内蒙古巴盟Bameng, Inner MongoliaICS, CAAS地方品种Landrace 3500007275糜子Mizi内蒙古Inner MongoliaICS, CAAS地方品种Landrace 36黄糜子Huangmizi内蒙古固阳县Guyang, Inner Mongolia固阳县Guyang农家种Accession from farmer 续附表1 Continued table S1 编号Serial number统一编号Unicode名称Name原产地Origin来源Accession donor备注Remark 3700000956大青黍Daqingshu山西灵丘县Lingqiu, ShanxiICS, CAAS地方品种Landrace 38红黍子Hongshuzi山西省阳泉市平定县Pingding, Yangquan, Shanxi冠山镇南坳村Nan’ao, Guanshan农家种Accession from farmer 39黍子Shuzi山西省阳泉市平定县Pingding, Yangquan, Shanxi冠山镇西沟村Xigou, Guanshan农家种Accession from farmer 40黍子Shuzi山西省阳泉市平定县Pingding, Yangquan, Shanxi张庄镇宁艾村Ning’ai, Zhangzhuang农家种Accession from farmer 41白糜子Baimizi山西省临汾市乡宁县Xiangning, Linfen, Shanxi昌宁镇韩村Han, Changning农家种Accession from farmer 42白糜子Baimizi山西省长治市长子县Zhangzi, Changzhi, Shanxi色头镇色头村Setou, Setou农家种Accession from farmer 4300000992大白黍Dabaishu山西省大同市左云县Zuoyun, Datong, ShanxiICS, CAAS地方品种Landrace 44白糜子Baimizi山西省阳泉市平定县Pingding, Yangquan, Shanxi冠山镇卫垴村Weinao, Guanshan农家种Accession from farmer 4500000985紫罗带Ziluodai山西省大同市Datong, ShanxiICS, CAAS地方品种Landrace 46青黍子Qingshuzi山西省吕梁市汾阳市Fenyang, Lüliang, Shanxi杨家庄镇南偏城村Nanpiancheng, Yangjiazhuang农家种Accession from farmer 47黄糜子Huangmizi山西省孝义市Xiaoyi, Shanxi下堡镇Xiapu农家种Accession from farmer 48黄糜子Huangmizi山西省吕梁市Lüliang, Shanxi柱濮镇Zhupu农家种Accession from farmer 49白糜子Baimizi山西省太原市阳曲县Yangqu, Taiyuan, Shanxi泥屯镇东青善村Dongqingshan, Nitun农家种Accession from farmer 50红糜子Hongmizi山西省阳泉市盂县Yuxian, Yangquan, Shanxi北下庄乡Beixiazhuang农家种Accession from farmer 51黄黍子Huangshuzi山西省忻州市偏关县Pianguan, Xinzhou, Shanxi窑头乡寺埝堡村Sinianpu, Yaotou农家种Accession from farmer 5200001514黄硬黍Huangyingshu山西省临汾市翼城县Yicheng, Linfen, ShanxiICS, CAAS地方品种Landrace 53雁黍7号Yanshu 7山西省大同市Datong, ShanxiICSAR, SAAS育成品种Bred variety 54大红黍Dahongshu山西省忻州市定襄县Dingxiang, Xinzhou, Shanxi师家岗村Shijiagang地方品种Landrace 55黍子Shuzi山西省晋中市榆社县Yushe, Jinzhong, Shanxi榆社县Yushe农家种Accession from farmer 56红糜子Hongmizi山西省阳泉市平定县Pingding, Yangquan, Shanxi冠山镇甘井村Ganjing, Guanshan农家种Accession from farmer 5700005272黄糜子Huangmizi陕西省定边县Dingbian, ShaanxiICS, CAAS地方品种Landrace 5800005423紫穗糜Zisuimi陕西省延安市Yan’an, ShaanxiICS, CAAS地方品种Landrace 59红糜子Hongmizi陕西省榆林市Yulin, Shaanxi榆林市Yulin农家种Accession from farmer 6000001635二瓦灰Erwahui陕西省榆林市Yulin, ShaanxiICS, CAAS地方品种Landrace 6100004821糯糜子(黍)Nuomizi (Shu)陕西省ShaanxiICS, CAAS地方品种Landrace 6200004824糯糜(黍)Nuomi (Shu)陕西省ShaanxiICS, CAAS地方品种Landrace 6300003806长糜Changmi陕西省志丹县Zhidan, ShaanxiICS, CAAS地方品种Landrace 64红黍子Hongshuzi陕西省渭南市合阳县Heyang, Weinan, Shaanxi路井镇高原寨村Gaoyuanzhai, Lujing农家种Accession from farmer 续附表1 Continued table S1 编号Serial number统一编号Unicode名称Name原产地Origin来源Accession donor备注Remark 6500002620古城红糜子Guchenghongmizi宁夏固原市Guyuan, NingxiaICS, CAAS地方品种Landrace 6600002518平罗紫秆Pingluozigan宁夏平罗县Pingluo, NingxiaICS, CAAS地方品种Landrace 6700006781鼓鼓头-2 Gugutou-2宁夏固原市Guyuan, NingxiaICS, CAAS地方品种Landrace 680000678360天小黑糜60-dayXiaoheimi宁夏固原市Guyuan, NingxiaICS, CAAS地方品种Landrace 6900006790泾源小青Jingyuanxiaoqing宁夏固原市泾源县Jingyuan, Guyuan, NingxiaICS, CAAS地方品种Landrace 7000007287中卫大黄Zhongweidahuang宁夏NingxiaICS, CAAS地方品种Landrace 7100007306五太保灰糜Wutaibaohuimi宁夏NingxiaICS, CAAS地方品种Landrace 7200007308九十天糜90-day mi宁夏NingxiaICS, CAAS地方品种Landrace 7300007309黄糜Huangmi宁夏NingxiaICS, CAAS地方品种Landrace 7400007297贺兰大红Helandahong宁夏NingxiaICS, CAAS地方品种Landrace 7500000081黄糜子Huangmizi黑龙江省富锦市Fujin, HeilongjiangICS, CAAS地方品种Landrace 7600000100白糜子Baimizi黑龙江省桦川县Huachuan, Heilongjiang ICS, CAAS地方品种Landrace 7700000012白糜子Baimizi黑龙江省德都县Dedu, HeilongjiangICS, CAAS地方品种Landrace 7800000143黑糜子Heimizi黑龙江省密山市Mishan, HeilongjiangICS, CAAS地方品种Landrace 7900000177红糜子Hongmizi黑龙江省宁安市Ning’an, HeilongjiangICS, CAAS地方品种Landrace 8000000411白糜子Baimizi吉林省吉安市Ji’an, JilinICS, CAAS地方品种Landrace 8100002044红糜子Hongmizi吉林省延吉市Yanji, JilinICS, CAAS地方品种Landrace 8200002045糜子Mizi吉林省延吉市Yanji, JilinICS, CAAS地方品种Landrace 8300000413昌图红糜子Changtuhongmizi辽宁省昌图县Changtu, LiaoningICS, CAAS地方品种Landrace 84白糜子Baimizi河北省辛集市Xinji, Hebei旧城镇耿庄村Gengzhuang, Jiucheng农家种Accession from farmer 85黑黍子Heishuzi河北省石家庄市赞皇县Zanhuang, Shijiazhuang, Hebei嶂石岩乡三六沟村Sanliugou, Zhangshiyan农家种Accession from farmer 8600000763黍子Shuzi河北省宽城县Kuancheng, HebeiICS, CAAS地方品种Landrace 8700007076红糜子Hongmizi河北省宽城县Kuancheng, HebeiICS, CAAS地方品种Landrace 8800007096黍子(糜)Shuzi (mi)河北省宽城县Kuancheng, HebeiICS, CAAS地方品种Landrace 8900007100小黄糜子Xiaohuangmizi河北省赤城县Chixheng, HebeiICS, CAAS地方品种Landrace 9000007118糜子Mizi河北省阜平县Fuping, HebeiICS, CAAS地方品种Landrace 9100007198陈庄白黍Chenzhuangbaishu河北省完县Wanxian, HebeiICS, CAAS地方品种Landrace 9200007214褐粒黍Helishu河北省南宫市Nabgong, HebeiICS, CAAS地方品种Landrace 9300007343白糜Baimi山东ShandongICS, CAAS地方品种Landrace 9400004409黑黍子Heishuzi山东ShandongICS, CAAS地方品种Landrace 9500004202狸黍子Lishuzi江苏淮阴Huaiyin, JiangsuICS, CAAS地方品种Landrace 96红黍子Hongshuzi河南省安阳市Anyang, HenanICS, CAAS地方品种Landrace
ICS、CAAS:中国农业科学院作物科学研究所;ICSAR、SAAS:山西省农业科学院高寒区作物研究所
ICS, CAAS: Institute of Crop Science, Chinese Academy of Agricultural Sciences; ICSAR, SAAS: Institute of Crop Science in Alpine Region, Shanxi Academy of Agricultural Sciences
附表2 筛选抗盐相关引物的糜子材料
Table S2 SSR primers based on salt resistance RNA-Seq for primary screen

编号Serial number统一编号Unicode名称Name来源Origin 100000956大青黍Daqingshu山西省灵丘县Lingqiu, Shandong 200000100白糜子Baimizi黑龙江省桦川县Huashuan, Heilongjiang 300002518平罗紫秆Pingluozigan青海省平罗县Pingluo, Qinghai 400004189散尾儿Sanweier江苏省Jiangsu 500006653金黍Jinshu海南省琼海县Qionghai, Hainan 600007345疏勒附糜Shulefumi新疆沙湾县Shawan, Xinjing
附表3 筛选抗旱相关引物的糜子材料
Table S3 SSR primers based on drought resistance RNA-Seq for primary screen

编号Serial number统一编号Unicode名称Name来源Origin 100000104鹅头Etou黑龙江省桦川县Huachuan, Heilongjiang 200006653金黍Jinshu海南省琼海市Qionghai, Hainan 300007287中卫大黄Zhongweidahuang宁夏Ningxia 400007343白糜Baimi山东省Shandong 500007435黑硬糜Heiyingmi青海省乐都县Ledu, Qinghai 600007358大黄糜Dahuangmi山西省Shanxi
附表4 85个SSR引物特性
Table S4 Characteristics of 85 SSR primers

标记Marker正向引物F-Sequence反向引物R-Sequence重复基序Repeated motif 序列Sequence (5′-3′)退火温度Tm (℃)序列Sequence (5′-3′)退火温度Tm (℃) RYW1TAACGCTTCACCTTCAGACC55.25′ TGAGATGGAGTTGGCTGATG56.2(TCATCT)6 RYW2TTAGGGCTCTCCTGCATCC57.45′ CAGCGAGTTCACCGTCAAG57(CGAAGC)5 RYW3GGAGGCGTGACAATAAAAC52.85′ GGCGTGAGGTGTTGTTTTT55.7(CTGCAA)5 RYW4AATCCACAACGCACACGAC56.95′ ATTTGCTCCTCTCGTCGGT56.9(GTGCCG)5 RYW5GACGATGCTCTTGACCTTGT54.75′ CACCGTGAAATGTCTCTGCT55.6(CCTTT)5 RYW6AGCCGATTTGCTGTGGAGT57.85′ CTGCCTCCGATGAGTTGGT56.4(ACACC)5 RYW7TCCACTCATCCATTGCTCGT58.45′ GATGGATTCAAAGGGACGCT58.9(CGCGC)5 RYW8GGGTCAGAGAATACACAGCG55.3GTAGGGAAGGAGAAGTGGGT56(AATAG)5 RYW9GGACCCTTCCCTCACAGATT57.6TCCAGTTGCTCTTGCCGTT58.9(CTAG)6 RYW10TGGATTGGGTGGTGGTAT53AAGGACGGCAGCACAAAT56(CGAG)5 RYW11TGCTCGTCTTCTCGCTTCG59.7AGTAGTCCTCCACCGCCATCT60.1(GGTA)5 RYW12ACCATCCCAGCACAAACCA58.9TGCCTGAAGGAGAAGAGCG58.6(AGCT)5 RYW13CTCCTGCTCTGCTTCGTTT55.3CTTCCAGCAGACCTTCACTC54.4(CGGC)5 RYW14CGCACAACGACCACAAGAG57.8ATACACCAGAGGAGCACGC55.5(GGCC)5 RYW15ACCGTGAAAAGAAATGCTG53.4GTCCTGGATGTCGTCAACT51.9(ACAA)5 RYW16ATCTCCTCCGCCTTCTAACCC61.4TGGCAATGGTCGTACAAACT56.3(GAGC)5 RYW17TCAGCTACTTCGAACGGC54.2GGATCATGCGATACATTTGG56.1(TTTC)5 RYW18CTCCCTCTTTGTCCTCGTT53.8GCTGCCTCTTCGCTATCTT54.8(AGTT)6 RYW19GAATGATAGGTCCGCAAGG55.3CAGCCTTTGTTCAGTTGTCTC54.9(TTAT)5 续附表4 Continued table S4 标记Marker正向引物F-Sequence反向引物R-Sequence重复基序Repeated motif 序列Sequence (5′-3′)退火温度Tm (℃)序列Sequence (5′-3′)退火温度Tm (℃) RYW20ACCTCTTGCCGCACACTAC55.6TTCTACATCCCCGAACCAC55.2(TTGG)6 RYW21CCCTCCTACTGCTCCCTTT55.8ATTACTCGTTCTCGCCTCG55.3(CGGA)6 RYW22AAGCCAACACATTTGAGGT52.4CGTGATAACTGAATACGAGCC55.3(GGAA)5(GCA)5 RYW23AGGAACAGCAGAGAGAGGG53.7CAGAACACCACGAAACACC53.6(GGAA)5 RYW24TTTCTTCGGCTCGTGTGAC56.5CGTCGCTCTTGTTCACTCC56(TGGA)5 RYW25AATCGCTCCACCGCTAATC57.7GACATCCCGAAACCCTAACC58.3(ATAC)5 RYW26TAAGGGTGGCGTTGGATAG56AACCCAACAGGTCCTCCAT56.1(AGGA)6 RYW27GCTGCTGGTTTCATTTTCC55.5CCAAGATTGATTCGGTTCG56.1(GGAA)5 RYW28CCAAGGCTGAGCAGAAAGAT57ACAAGGTGAAACCCGAAGC57.3(AGGC)5 RYW29CTTGATTTCTCACGCACCG57.1TGTCCAGCAGTAGTCGTTCCT57.2(GCAG)5 RYW30TAGCCTTCTTTGCCACCACT57.5GCCCGTGATGATATTCGAC55.7(TTTC)5 RYW31ACCCAGAGTCCAGAGAAGC53.4GATGTCCTCCTCCTTCTCC52.6(AGCG)5 RYW32CAGGTTATGGGAGGACGAG55GGTGCTACGGTTACAGGGT54.7(ATCTT)5 RYW33CGATTCTACACCGACGAGG56.1TGTAGGGTTCCATTCATCTCC56.4(CCATC)5 RYW34TCCCCCGATTAGGAAAGAT56.1CTGGTGAGGTGATGAAGCC55.4(CGATT)5 RYW35ATTAGCATCCCCCTCCAC54.1ATCCGCTTTCCCAACCAC57.7(CGTGC)5(GGA)6 RYW36TATTGTCCTTCCGCTCCC55.4ATGACTACTCTCCCCCCCT54.6(GGCTT)5 RYW37CATTCCGTTCCTTGTCTTCC56.9CAGTCTCACTCCTGCGATGT55.3(GCGAT)5 RYW38TCCCTGAAGGAGAAGAGCG57.5CACCATCCCAGCACAAACC58.9(AGCT)5 RYW39GTTGGGCGAGGTCAATCTG58.3TAGGGAGCCGAAGCAGAAG58(TCCT)5 RYW40TGCTCTTCGGCTCTTCTCC57.7ATCAGCTCATCGTGACCCC57.5(CAGC)6 RYW41GCAAACACTTGGCGGATAG56.5TGGAATGCCCAGGAGAAAT57.5(GATG)6 RYW42AGACACCCTGGGCAACATC57.4CTGGACTGGGCTTCGTTCT57.3(GGCT)5 RYW43GGAGATGCTTGCTTGGTTG56.2CAGGAATCGCAAGGAACAG56.2(GGAG)5 RYW44TAAAGCAGGTCGGCAAGAG56.4TCGGGTTTCTCCAGCATAAT57.3(CAAT)5 RYW45TCGCTGCTCAACATCAACC57.3TGGATCGGGCTATGTTGC57.1(TGGC)5 RYW46TAGCGGATTCACGAACGAG56.9AGACGCTAAAAAGCCCGAC57.3(CCTC)6 RYW47TTGTTTTTGCTGCTGCCTC57.2TGCTGGACTTCTTTTTGCC56.1(GCCT)5 RYW48TTTCTGAACTCACGCCACC56.3GGCAGCAACTAACCGTGTG57.1(GAAG)5 RYW49GCTAAATCCGCTGATGAGGT57TGTATGTTGCTCCAGCCTTG56.9(TATC)6 RYW50CAAGGCAGATAGGGCAAGT55.1TCGTCTGCTGCTGGTTTGT57(GGAG)5 RYW51TATCGCCGCACCTTACAAC57TGAGCCTGCTTCCATCTTG56.6(CTGC)5 RYW52AGTAGTCCTCCACCGCCAT56.5CTCTTCCTCGTTCTCGGCT56.8(TACC)5 RYW53ATGCCTCCGATGTAGATGC55.4GCCGCCTTCTCTTCATTCT56.8(GAGG)5 RYW54GCACTTGCTCCTGCTTCTC55.5GACCTTGCCGATGTTGTTG56.7(CCTC)5 RYW55CTGGTGGTGGTAGTTAGCG53.7TTATGCCACCCACCGTAGC59.1(TAGC)5 RYW56TCGTTTTCTTCCCTGCTTG56.1TTCGGCAGAACCTGTCGT56.6(AGAA)5 RYW57GGATTGCGGCTAAGTGTTG56.5CCCGCCTTTTTTTTACTACC56.6(TGTA)5 RYW58AGCCAGACCGAGAGTTTCC56.4AGCCACCTCACCTATGTTCC56.3(TTCT)5 续附表4 Continued table S4 标记Marker正向引物F-Sequence反向引物R-Sequence重复基序Repeated motif 序列Sequence (5′-3′)退火温度Tm (℃)序列Sequence (5′-3′)退火温度Tm (℃) RYW59CAGCAAAGCAACCTTCACC56.3TCGGAGAGAGGGTGAGAGAT55.7(GGCA)5 RYW60GGGAGATGCTTGCTTGGTT57.4GCTTTCTTTCCTTAGCCGAT56.1(GGAA)6 RYW61ACTGCTGCTTGCTCCTTGT55.5ACATCCCGAAACCCTAACC56.2(ATAC)5 RYW62GTTTAGAGAGCAGGAGGCG55AGCCCTGTCCACCCTAATC56.1(GCTC)5 RYW63TTTCTCTGCCACCATTTTC53.1CTGGTGCCTTTGCGATTAT56.1(GAGT)5 RYW64CCAGCGACTGACGAAACTT55.8AACATCAGGAACCACACGC55.9(ACCG)6 RYW65TAGCGTCGTCAAGAAGCACT56ACAGAGGTGACGAAACGATG55.4(GCCG)5 RYW66AGGATGTTGGCGGTGTTC55.8TTGGTGTGGGAGTATGCG55.1(GGTT)5 RYW67GAAGGAAACGCACCAGAGT54.7TTGGGTTTGTGCTTGGAGT55.9(TGCG)5 RYW68AGAGAACAATCCACACCGC55.2ATGTGGAGGTGGCATTCTG55.8(AGGG)5 RYW69AGCCGTTGGAAGAGGATT54.2GCTTGCTTTTTTGAGGGG55.7(GGAA)5 RYW70AGGTTAGGGATTGGGGGAT57.2CCTTTTTTTCCAGTCTCACA52.7(CGAG)5 RYW71CATCAACTTCATCTGCCCC55.6TGCTCTTGTGGTCGTTGTG55.3(GGCC)5 RYW72GGCACCTCCATTCATTCAC55.3CCAGATTTTCAAACCCCAT54.6(ACGC)5 RYW73TTATTTTGAATGCTCCCCC55.2TGGTCAGTCTTGAAGTCGC53.5(ACAA)5 RYW74AACGGATGTTCAGAAGCAG53TGGTTCCCAGCACCTAAAT55.2(GCAG)5 RYW75GTTCTCGTAACTGAAGGCG53.1GCCAGTAGGCTCTCTGAACA55.1(TTAT)5 RYW76TCCCTGTTTTTCTTCTCCG55.1GAATCTTCACGGAATCAGG52.2(GGAT)5 RYW77CAGCAAGCAGACAGAGCAG54.6ACAAAGCAGAGAGGGGAAG54(ATAC)5 RYW78ATGGTTGGATGTCTCTCGC55GCGTGAGCCTTTTTCTTTT55(ATCG)5 RYW79TCAGCAGACAACCCAAAAG53.9CGATTGTTGAGGGAAGTGA53.5(ATCA)5 RYW80ACCCAGAGTCCAGAGAAGC53.4GATGTCCTCCTCCTTCTCC52.6(AGCG)5 RYW81TCAGATTTTACAGCCGCAT54TCTTGGTTCTTCCCACTTG52.8(GTTT)6 RYW82TGCTGGTTGCTGGTAGGT53.9GTGCCGAAGAAGATGGTG53.7(AGCG)5 RYW83TTGTAGAGCAGGAAGGAGAT51.4TAGACGAAGGACACGAAAC50.3(GAAA)5 RYW84AACCCACCCATCCATTCCT58.5TAAGTCGTCGTCGGCAGAGT58.2(GAGC)5 RYW85GATGGATGGATTGCGTTTC55.7AACGGATTGTCTCCATTGC55.3(TCAC)5
附表5 85个糜子SSR标记的特性
Table S5 Characteristics of 85 common millet SSR markers with polymorphism

引物名称Marker name观察等位基因大小Observed allele size in common millet (bp)分辨率(Rp值)Resolving power (Rp) value RYW1100; 300; 4503.5 RYW2100; 150; 200; 250; 4503 RYW3100; 150; 350; 4004.25 RYW4100; 150; 200; 2505.25 RYW5300; 350 2.75 RYW6100; 150; 400; 450 3.75 RYW7150; 200; 3004 RYW8350; 4002.25 续附表5 Continued table S5 引物名称Marker name观察等位基因大小Observed allele size in common millet (bp)分辨率(Rp值)Resolving power (Rp) value RYW9150; 350; 4004.5 RYW10150; 250; 4004.5 RYW11150; 350; 4003.5 RYW12150; 200; 2504.5 RYW13100; 200; 250; 3505.25 RYW14400; 4503 RYW15100; 150; 2003 RYW16100; 150; 250; 300; 3504.5 RYW17150; 300; 3504.25 RYW18100; 350; 4003.5 RYW19100; 150 3 RYW20150; 350; 4003.5 RYW21150; 200; 300; 3505.5 RYW22100; 200; 300; 4005.5 RYW23100; 150; 200; 400; 4504.5 RYW24100; 150 3 RYW25100; 150; 4004.5 RYW261001.5 RYW27250; 4002.25 RYW28100; 200; 3003.75 RYW29100; 200; 3503.75 RYW30100; 200; 3504 RYW31100; 200; 300; 4005.5 RYW32100; 150; 200; 4002.75 RYW33150; 250; 350; 400; 4502.75 RYW34150; 250; 3501.5 RYW35100; 150; 200; 3503 RYW36100; 200; 350; 4502 RYW37150; 200; 250; 3003.25 RYW38200; 300; 3502.25 RYW39100; 150; 350; 4002.5 RYW40150; 350; 4002 RYW41150; 300; 4004 RYW42100; 250 1 RYW43250; 350; 4503.75 RYW44100; 1501.75 RYW45100; 2502.25 RYW46100; 150; 2503.25 RYW47100; 200; 300; 4005.75 RYW48100; 250; 4003.75 续附表5 Continued table S5 引物名称Marker name观察等位基因大小Observed allele size in common millet (bp)分辨率(Rp值)Resolving power (Rp) value RYW49200; 2503 RYW502001.5 RYW51100; 200; 4504 RYW52100; 250; 3002.5 RYW53100; 150; 250; 3002.75 RYW54150; 200; 3501.5 RYW55150; 250; 350; 400; 4503.25 RYW56200; 350; 4004.25 RYW57200; 300; 3503 RYW58100; 150; 200; 3503 RYW59150; 250; 3501.5 RYW60150; 250; 3502.25 RYW61250; 300; 3501.5 RYW62200; 250; 350; 4004.25 RYW63100; 150; 2002.5 RYW64100; 250; 4002.25 RYW65150; 250; 350; 400; 4502.5 RYW66150; 200; 250; 3002 RYW67100; 250; 3003 RYW682001.5 RYW69100; 2002.25 RYW70150; 3001.25 RYW71100; 150; 250; 4503.25 RYW72100; 200; 3502 RYW73100; 150; 3502.5 RYW74200; 3502.25 RYW75100; 150; 4002 RYW76100; 150; 350; 4002.75 RYW77100; 150; 200; 4003.5 RYW78100; 150; 200; 3503 RYW79100; 150; 4503 RYW80100; 300; 350; 4004 RYW81100; 250; 3503.25 RYW82200; 300; 3504.25 RYW83100; 150; 250; 3505 RYW84250; 4502 RYW85150; 200; 3502.75 平均Mean3.15
附表6 85对引物检测的遗传参数
Table S6 Genetic parameters of the 85 polymorphic SSR markers used in the study

位点Locus观测等位基因Na有效等位基因Ne多样性指数I观测杂合度Ho期望杂合度HeNei’s期望杂合度Nei多态性信息含量PIC RYW132.12570.87810.55210.52960.43020.6318 RYW231.96230.77790.61460.49040.46730.4824 RYW331.93310.72930.53120.48270.44590.5882 RYW432.21860.88670.65620.54930.44490.5751 RYW532.32290.93080.66670.56950.55570.5616 RYW632.16260.84360.64580.53760.43350.5612 RYW731.87090.77170.43750.46550.39060.6343 RYW832.60231.01170.43750.61570.57190.7281 RYW932.02550.76570.60420.50630.47530.5744 RYW1031.95670.74770.48960.48890.41220.5665 RYW1132.05260.80980.59380.51280.49860.5690 RYW1232.54690.99980.51040.60740.55930.6393 RYW1332.12060.82630.65620.52840.51530.6185 RYW1421.84320.65000.70830.45750.44720.3278 RYW1521.99910.69290.97920.49980.49980.0400 RYW1632.00430.73450.59380.50110.48050.5650 RYW1731.98560.72970.51040.49640.47970.6176 RYW1821.84320.65000.70830.45750.36530.3226 RYW1931.91460.69420.57290.47770.46820.5311 RYW2021.99460.69180.71880.49860.40380.4527 RYW2121.98620.68970.79170.49650.48600.3548 RYW2221.54630.53830.37500.35330.35370.4328 RYW2321.98940.69050.65620.49730.49320.4926 RYW2421.89140.66420.55210.47130.38050.5727 RYW2522.00000.69310.68750.50000.43970.5052 RYW2621.93210.67550.75000.48240.48350.3504 RYW2721.81110.64010.46880.44790.35870.5371 RYW2831.68840.66980.43750.40770.41330.6196 RYW2931.66280.72170.34380.39860.40440.5866 RYW3032.37920.95540.39580.57970.48060.6522 RYW3132.47510.97400.66670.59600.45440.5971 RYW3231.97850.83390.47920.49460.37480.5296 RYW3332.01050.76190.72920.50260.40570.3819 RYW3432.06150.76250.80210.51490.41950.3360 RYW3532.18930.88010.71880.54320.44590.4105 RYW3621.95230.68090.69790.48780.48770.4993 续附表6 Continued table S6 位点Locus观测等位基因Na有效等位基因Ne多样性指数I观测杂合度Ho期望杂合度HeNei’s期望杂合度Nei多态性信息含量PIC RYW3832.23150.90720.75000.55190.53290.4922 RYW3921.71670.60820.57290.41750.35140.3980 RYW4032.33580.92550.52080.57190.54500.6771 RYW4132.61971.01660.70830.61830.58260.4453 RYW4232.35370.92910.83330.57510.56250.3423 RYW4332.25300.88310.63540.55620.53400.5465 RYW4431.64040.61720.50000.39040.41260.4052 RYW4521.78860.63280.65620.44090.44200.3494 RYW4632.01160.71980.93750.50290.50180.1133 RYW4721.92460.67340.80210.48040.47380.2760 RYW4832.08200.77950.82290.51970.51710.3105 RYW4932.20820.86070.71880.54710.46570.4356 RYW5031.78550.67100.48960.43990.31800.4702 RYW5131.85340.72790.62500.46040.39610.4686 RYW3732.81531.06330.65620.64480.60270.4765 RYW5231.94780.70330.72920.48660.48410.3576 RYW5332.76761.05290.60420.63870.59900.6038 RYW5432.10530.83020.72920.52500.41280.5198 RYW5532.11350.82470.71880.52690.42400.4679 RYW5632.64561.03540.44790.62200.56010.5826 RYW5732.56711.00730.56250.61050.57070.6416 RYW5832.26020.90070.80210.55760.55670.5219 RYW5932.42050.97720.78120.58690.54740.5060 RYW6032.06290.76280.54170.51520.43780.5862 RYW6131.93920.81800.50000.48430.37750.5448 RYW6221.98620.68970.81250.49650.49060.3020 RYW6321.86330.65600.66670.46330.46070.5868 RYW6432.00890.74950.75000.50220.42200.3502 RYW6532.04230.80720.65620.51040.49280.4388 RYW6632.14750.82430.86460.53430.52360.2360 RYW6732.06520.77550.78120.51580.50710.4080 RYW6832.14650.84690.57290.53410.43080.5809 RYW6932.37650.93780.69790.57920.47990.5282 RYW7021.90020.66670.77080.47370.47740.2909 续附表6 Continued table S6 位点Locus观测等位基因Na有效等位基因Ne多样性指数I观测杂合度Ho期望杂合度HeNei’s期望杂合度Nei多态性信息含量PIC RYW7121.90870.66900.65620.47610.39560.5107 RYW7231.66580.68970.44790.39970.42960.4642 RYW7332.06940.84580.53120.51680.49980.5117 RYW7431.89610.68900.75000.47260.37790.3592 RYW7531.14660.28420.11460.12790.12460.1879 RYW7632.08390.79080.54170.52010.39500.6074 RYW7721.99910.69290.91670.49980.49890.1522 RYW7821.61330.56800.40620.38020.28490.5219 RYW7921.71670.60820.57290.41750.35040.5911 RYW8021.91680.67130.72920.47830.46430.3803 RYW8132.04160.74380.85420.51020.50120.2501 RYW8231.99420.77790.69790.49850.41840.4476 RYW8331.36520.52690.23960.26750.24220.4215 RYW8432.00110.71720.76040.50030.49720.4262 RYW8531.95520.70530.67710.48860.48320.6097 平均 Mean2.72942.04000.77080.63360.49980.45510.4723 标准差SD0.44170.28740.13470.15330.07620.07800.1339
Evaluation of Genetic Diversity of Common Millet () Germplasm Available in China using High Motif Nucleotide Repeat SSR Markers
Wang RuiYun1,2, LIU XiaoYu1, Wang HaiGang2, Lu Ping3, Liu MinXuan3, CHEN Ling2, Qiao ZhiJun2
(1College of Agriculture, Shanxi Agricultural University, Taigu 030801, Shanxi;2Institute of Crop Germplasm Resources, Shanxi Academy of Agricultural Sciences/Key Laboratory of Crop Gene Resources and Germplasm Enhancement on Loess Plateau, Ministry of Agriculture/Shanxi Key Laboratory of Genetic Resources and Genetic Improvement of Minor Crops, Taiyuan 030031;3Institute of Crop Science, Chinese Academy of Agricultural Sciences, Beijing 100081)
Theobjective of this study to develop SSRs with high motif (tetra-, penta- and hexa-) nucleotide repeat and use them to evaluate the genetic diversity of common millet germplasms, and to provide significant implications for future linkage maps construction and evolution research of common millet.Comprehensive evaluation of phenotypic diversity was carried out based on membership function, principal component analysis (PCA) and cluster analysis. SSR primers developed in author’s laboratory by high-throughput sequencing were used to identify polymorphisms in different common millet accessions from various geographic origins. PowerMarker 3.25 and PopGen 1.32 were used to calculate the measures of genetic diversity and Nei’s genetic distance, respectively. Mega 5.0 and Structure 2.2 were used to carry out cluster analysis and identify genetic groups.A set of 96 common millet accessions were used and seven phenotypic traits were investigated. Results showed that the genetic variation of tiller numbers and plant height were the most abundant. A total of 85 pairs of amplified primers (56.29%) showed high polymorphism among the 96 genotypes. A total of 71 tetra-nucleotide repeat SSRs were detected, accounted for 83.53%. Meanwhile, 10 (11.76%) penta- and 4 (4.7%) hexa- nucleotide repeat SSRs were identified, respectively. The size of 85 SSRs ranged from 100 to 450 bp with an average value of polymorphism information content (PIC) of 0.51. Resolving power (Rp) value varied from 1.00 to 5.75 (mean = 3.15).Theaverage Rp value of tetra-, penta- and hexa- nucleotide repeat SSRs was 3.15, 2.8 and 4.0, respectively. Evaluating the distribution frequency of 85 SSRs based on Rp, it was observed that their ranges were 0-1, 1-2, 2-3, 3-4, 4-5 and 5-6, with 1 (1.18%), 15 (17.65%), 31 (36.47%), 20 (23.53%), 12 (14.12%) and 6 (7.06%) markers at each interval, respectively. Fifty-one markers accounting for 60% distributed at intervals of 2-3 and 3-4. A total of 232 alleles were amplified among 96 accessions by the 85 SSR markers. 2-3 alleles were generated by each locus, with an average of 2.7294 alleles. Sixty-two markers produced 3 alleles, and 23 markers produced 2 alleles. The range of gene diversity was 0.2842-1.0633, with an average of 0.7708. The range of PIC was 0.0400-0.7281, with an average of 0.4723.Genetic distance and genetic identity of common millet resources with different ecotopes were 0.0093-0.5052 (average=0.1798) and 0.6034-0.9907 (average=0.8485). Cluster analysis based on unweighted pair group method of mathematical averages (UPGMA) separated the 96 accessions into four groups (Northeast spring-sowing, Northern spring-sowing, Northern summer-sowing and Loess Plateau spring & summer-sowing ecotopes). A genetic structure assay indicated a close correlation between geographical region and genetic diversity.The present work developed a set of 85 tetra-, penta- and hexa- nucleotide repeat SSRs in common millet. These high motif microsatellite markers with high Rp value can distinguish different genotypes, generate high polymorphisms in polymerase chain reaction amplification. Based on the above molecular markers, the genetic diversity of Chinese common millet germplasms was assessed and those accessions from Northern spring-sowing and Loess Plateau spring & summer-sowing ecotopes are highly and genetically diverse.
common millet (); high motif SSR; genetic diversity; clustering analysis; population structure
2017-05-17;接受日期:2017-06-12
国家自然科学基金(31271791)、山西省回国留学人员科研资助项目(2016-066)、国家现代农业产业技术体系建设专项(CARS-06-13.5-A16)、山西省重点研发计划(一般项目)(农业)项目(201603D221003-5)
王瑞云,Tel:15234420135;E-mail:wry925@126.com。刘笑瑜,E-mail:251719093@qq.com。王瑞云和刘笑瑜为同等贡献作者。通信作者王瑞云。通信作者乔治军,Tel:0351-7065530;E-mail:nkypzs@126.com
