版纳微型猪近交系FANK1基因CDS克隆、qPCR表达及功能生物信息学分析
2017-11-10霍晋华
霍晋华,张 霞,王 配*
(1.华中农业大学生命科学技术学院,武汉 430070;2.云南农业大学动物科学技术学院,昆明 650201)
版纳微型猪近交系FANK1基因CDS克隆、qPCR表达及功能生物信息学分析
霍晋华1,张 霞2,王 配2*
(1.华中农业大学生命科学技术学院,武汉 430070;2.云南农业大学动物科学技术学院,昆明 650201)
【目的】获得猪FANK1基因CDS序列、组织表达和蛋白质功能信息。【方法】以GenBank下载的猪及近缘物种的FANK1 mRNA序列为参考序列,设计特异引物从版纳微型猪近交系(BMI)睾丸组织中扩增FANK1基因完全编码序列及部分侧翼序列。应用实时荧光定量PCR技术检测BMI 15个重要组织的FANK1 mRNA转录表达水平,并对FANK1翻译的蛋白质序列进行多种功能生物信息学分析,预测FANK1蛋白质的功能,最后构建FANK1多物种氨基酸系统进化树。【结果】获得了BMI FANK1编码区序列长1041 bp,编码346个氨基酸,序列已提交GenBank,基因登录号为KU705617和KU705618,对应的氨基酸登录号为AOC89035和AOC89036。基因组结构分析表明FANK1基因定位于猪14号染色体,有11个外显子和10个内含子。多组织相对荧光定量表达分析表明FANK1基因在睾丸、尿道球腺和精囊腺中呈高表达水平;在其他组织中呈中低表达水平。功能生物信息学分析表明FANK1蛋白质含有2种保守结构域FN3和ANK,无跨膜螺旋结构,无N端信号肽序列,二级结构以无规卷曲为主,N末端和C末端均亲水,有4类功能活性位点。系统进化分析表明,FANK1基因在进化中高度保守,且与牛、羊的亲缘关系最近,符合物种的系统分类学。【结论】成功克隆了版纳微型猪近交系FANK1基因的CDS序列并进行了多种组织表达分析,为后续研究FANK1基因在小型猪精细胞分裂及调节信号通路方面的作用及功能奠定基础。
纤连蛋白3(FN3);锚蛋白重复(ANK);版纳微型猪近交系;组织表达;生物信息学
据报道非细胞凋亡蛋白FANK1基因在睾丸中特异性表达[1-3],通过多物种比对,同源性较高,是高度保守的基因[4]。FANK1由FN3和ANK两种保守结构域组成[5]。有研究证明,FANK1是一种非细胞凋亡蛋白,它是通过激活AP-1(activator protein 1)信号通路来抑制细胞凋亡的[4,6],而且FN3和ANK都有增强AP-1通路活性的作用[7]。Wang H.等通过酵母双杂交的方法,发现FANK1与Jab1(Jun活化结构域结合蛋白1)可以相互作用[7]。研究发现,Jab1是AP-1信号通路的共同激活蛋白[8]。通过构建敲除FANK1基因的小鼠模型,发现敲除FANK1基因的小鼠细胞凋亡数目高于正常小鼠[9]。通过RT-PCR和Western Blot证明,FANK1基因在精细胞减数分裂过程中以及不同日龄的小鼠睾丸中的表达量不同[9]。
版纳微型猪近交系(Bannamini-pig inbred line,BMI)是全球第一个培育成功的大型哺乳动物近交系,因其基因高度纯合、遗传背景清楚,生理学、疾病发生机理和解剖学等方面与人类极为相似,从而可为新药临床前安全性评价、猪基因组计划中的功能基因研究、人类疾病动物模型制作和人类异种器官移植等众多领域提供实验材料和器官供体,具有重要的研究应用价值[10-13]。但是在正常繁育BMI的37年来,有多个亚系断代,前期研究发现是部分公猪不育造成的,这些不育公猪已成为阻碍其群体进一步扩大的障碍,故版纳微型猪近交系雄性不育方面的基础研究是亟待开展的重要研究方向。鉴于FANK1基因在雄性哺乳动物精子生成和睾丸功能中的重要作用,本研究通过克隆版纳微型猪近交系FANK1基因,确定其序列特征;通过mRNA的多组织表达谱确定其发挥功能的重要组织;通过对蛋白质进行功能生物信息学分析预测其功能,为今后进一步深入研究FANK1基因在BMI公猪繁殖力方面的功能奠定基础。
1 材料和方法
1.1 材料
屠宰BMI成年公猪,取睾丸和附睾(性腺),前列腺、精囊腺和尿道球腺(副性腺),5种雄性生殖重要相关组织;取心脏、肝脏、脾脏、肺脏、肾脏、胃脏、脑、肌肉、十二指肠(小肠)、结肠(大肠)等常规组织;共15种组织样品。
1.2 方法
1.2.1 总RNA提取和第一链cDNA合成
参照说明书利用柱式RNA提取试剂盒(TaKaRa:9767)分别提取各组织总RNA,用核酸蛋白测定仪检测所提RNA的浓度及纯度,用琼脂糖凝胶电泳检测所提RNA的完整性,合格样品按反转录试剂盒(TaKaRa:6210A)操作说明书反转录为第一链cDNA保存备用。
1.2.2 引物设计及合成
根据 GenBank下载的牛(NM_001003904)、猫(XM_006938270)、羊(XM_012108577)、人(NM_145235)、马(XM_001490071)等物种的 FANK1 mRNA序列,结合猪表达序列标签EST序列(CV865262和CX063842),再参照GenBank下载的预测的猪FANK1 mRNA 序列(XM_001924803、XM_005671566、XM_005671567、XM_005671568),利用 Oligo 7 软件设计F1/R1特异引物(F1:CGGCCTGGGAGCAGCTGAC,R1:AATGTCTGGCCTCAGGAATGC)来扩增 FANK1基因全长CDS序列及部分5'和3'侧翼序列,设计F2/R2特异引物(F2:AACGACCCGAGAGCCCAT,R2:AACGACCCGAGAGCCCAT)扩增 97bp,并以 GAPDH基因为内参(F:CCTTCATTGACCTCCACTACATGGT,R:CCACAACATACGTAGCACCAGCATC)扩增183bp,利用荧光定量法分析各组织的表达水平。
1.2.3 RT-PCR分离FANK1基因
PCR反应总体系25μL,以睾丸cDNA为模板,采用TaKaRa Ex TaqE体系,琼脂糖凝胶电泳确定扩增的FANK1基因片段与引物设计时预期的片段大小吻合,胶回收后送公司测序。
1.2.4 FANK1基因多组织表达分析
以50 ng/μL起始浓度的睾丸cDNA为模板,5倍倍比连续稀释获得7个标准品,以F2/R2为引物,制作标准曲线。以各组织cDNA为模板,以GAPDH为内参进行多组织qPCR表达水平检测。
1.2.5 序列确定和数据分析
利用Lasergene软件中的SeqMan程序进行组装、拼接、并人工校对,确定FANK1基因编码区序列并提交GenBank数据库,同时推导出其编码的氨基酸序列。计算15个组织的mRNA表达水平。最后进行FANK1蛋白质的功能生物信息学分析,并构建多物种FANK1氨基酸序列系统进化树。
2 结果与分析
2.1 FANK1基因RT-PCR扩增结果
以BMI睾丸组织cDNA为模板,扩增的FANK1基因长度为1152 bp,见图1。
2.2 FANK1基因序列及其编码的氨基酸序列
利用F1/R1扩增产物测序,结果使用DNAStar软件中的SeqMan程序进行组装,获得BMI FANK1全长编码序列1041 bp,因为BMI FANK1基因的CDS第939位碱基C/T杂合,是第313个氨基酸的第三位,编码的氨基酸不变,均为甘氨酸。获得两条mRNA GenBank登录号,分别为:KU705617和KU705618,其对应的氨基酸登录号分别为:AOC89035和AOC 89036。编码346个AA,含有2种保守结构域FN3、ANK,见图2。
2.3 FANK1基因组结构分析
为了获得BMI FANK1基因座的基因组DNA,在ensembl数据库中进行搜索,发现该基因位于猪14号染色体NC_010456.4的146827125-146928694位置,其全长101569 bp,包含11个外显子和10个内含子,见图3。
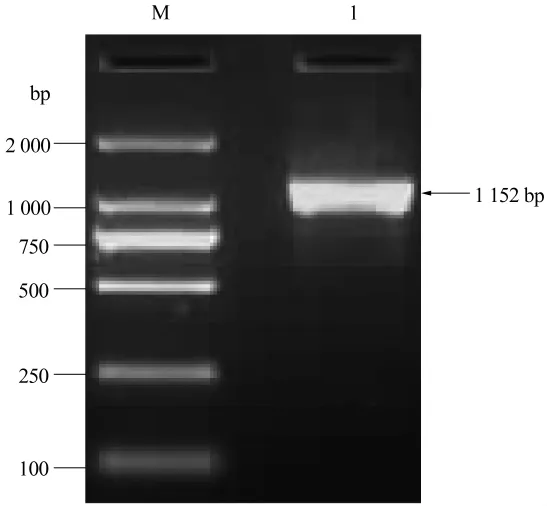
图1 FANK1基因反转录PCR扩增产物Figure 1 The RT-PCR product of FANK1 gene
2.4 多组织荧光定量表达分析
以GAPDH为内参基因,实时荧光定量检测FANK1基因15种组织的mRNA表达量,用2-ΔΔCt法分析各组织中的qPCR相对表达水平绘制出柱状图,结果表明FANK1基因mRNA在睾丸、尿道球腺、精囊腺、脑、脾中高表达(由高到低);在结肠、十二指肠、前列腺、附睾中中度表达(由高到低);在肺、肾、肝、心、胃中低表达(由高到低);在肌肉中几乎不表达,见图4。
2.5 FNAK1蛋白质一级结构信息
利用ProtParam tool程序获悉BMI FANK1蛋白质的一级结构和部分理化性质,结果见表1。
2.6 FANK1蛋白质二级结构
利用SOPMA程序预测的BMI FANK1蛋白质二级结构为:33.53%的无规则卷曲(含116个氨基酸),30.64%的α-螺旋(含106个氨基酸),24.86%的延伸链结构(含86个氨基酸),10.98%的β-转角(含38个氨基酸)。
2.7 FANK1蛋白质功能域
利用SMART程序预测BMI FANK1蛋白质功能域,发现其在9-94 AA、109-139 AA、143-172 AA、176-205 AA、209-238 AA、243-273 AA、334-343 AA是蛋白功能域,见图5。
2.8 FANK1蛋白质的疏水性
利用ProtScale程序预测BMI FANK1蛋白质的疏水、亲水性,结果表明其N末端和C末端均亲水,且第147位置有最大疏水值2.000,第337位置有最小疏水值-3.022。
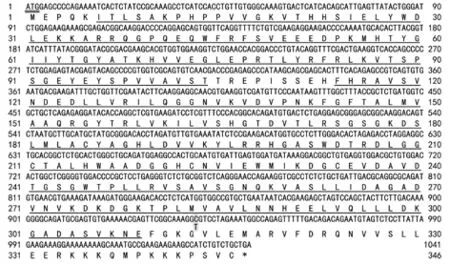
图2 FANK1基因CDS核酸序列与对应的氨基酸序列Figure 2 The coding sequence and corresponding amino acids sequence of FANK1 gene

图3 BMI FANK1基因组、mRNA和蛋白结构Figure 3 Genomic organization,mRNA and protein domain structure of BMI FANK1 gene
2.9 FANK1蛋白质功能位点分析
利用Prosite程序预测BMI FANK1蛋白质功能位点,发现共含有4类功能活性位点,包括cAMP和cGMP依赖性蛋白激酶、蛋白酶C、酪蛋白激酶II等磷酸化位点以及N-十四酰化位点,见表2。
2.10 FANK1蛋白质信号肽及跨膜结构分析
利用SignalP 4.1程序预测FANK1蛋白质是否含有信号肽,显示不包含N端信号肽序列。利用TMHMM 2.0程序预测FANK1蛋白质的跨膜螺旋,显示不存在跨膜螺旋,是非跨膜蛋白。
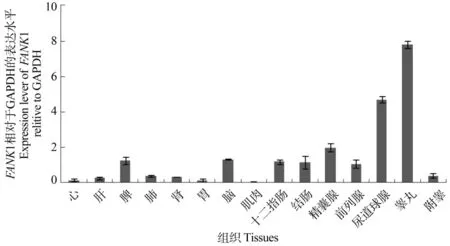
图4 FANK1基因多组织相对表达水平Figure 4 The multi-tissues relative expression lever of FANK1 gene
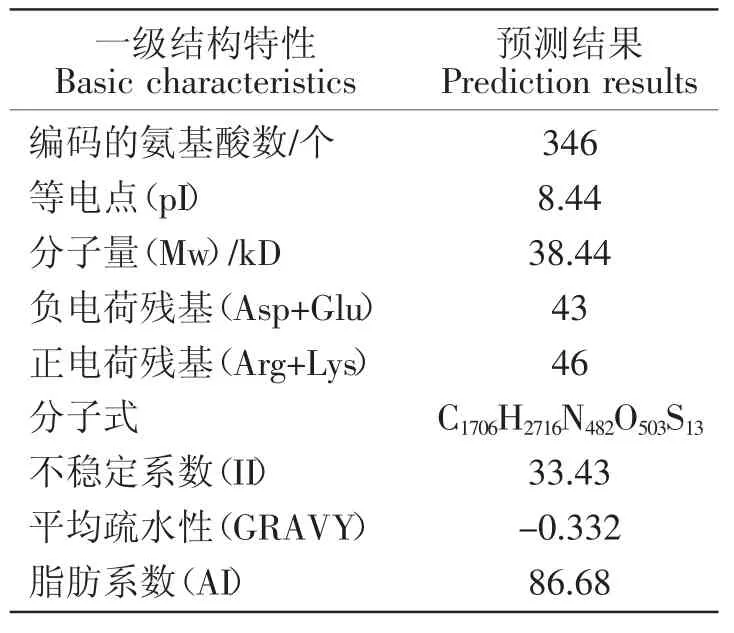
表1 FANK1蛋白质的基本信息Table 1 Primary information of FANK1 protein

图5 FANK1蛋白质功能域Figure 5 Protein function domain of FANK1 protein
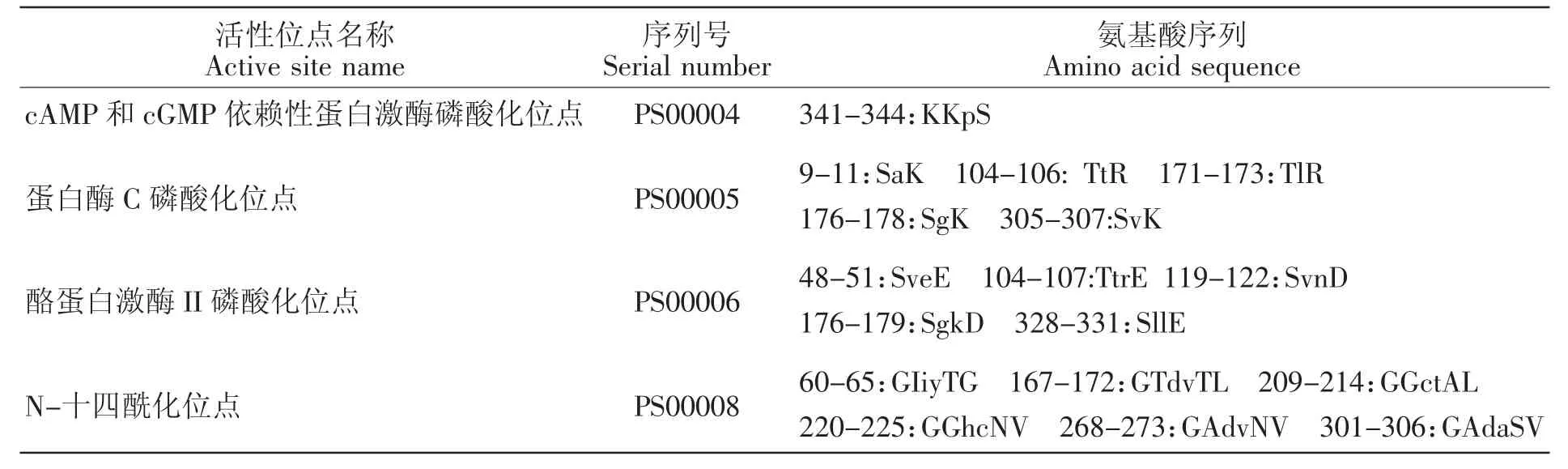
表2 FANK1蛋白质的功能活性位点Table 2 Functional active site of FANK1 protein
2.11 FANK1蛋白质序列多物种系统进化分析
利用MEGA7.0软件将BMI FANK1的蛋白质序列与牛(XP_010818432)、猫(XP_006938332)、羊(XP_004020311)、人(NP_660278)、狗(XP_005637948)等5个物种的蛋白质序列进行比对并计算相似度,相似度分别为:92.5%,92.2%,91.9%,91.6%,91.5%。并构建6个物种的分子系统进化树,见图6。
3 讨论和结论
近年来对于FANK1基因的结构与功能的研究越来越深入,FANK1基因具有DNA结合活性[9],在雄性配子形成的过程中,可能作为转录因子控制分子信号通路[4,7]。到目前为止,FANK1基因已在小鼠[9]以及人[4]等生物中被分离鉴定。本研究首先把从GenBank数据库中获取的人、牛、羊、狗、猫等物种的FANK1 mRNA序列进行比对,设计特异性保守引物,然后以BMI为实验材料进行扩增,获得了预期片段。通过对扩增产物测序并拼接,得到BMI FANK1基因的cDNA序列。
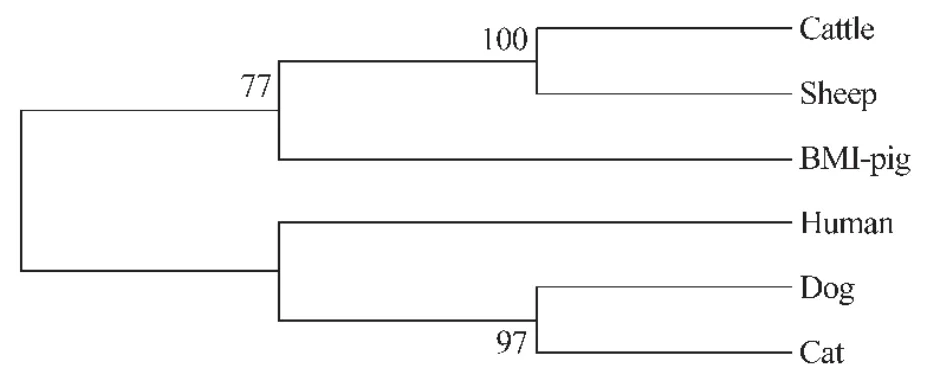
图6 FANK1氨基酸序列进化树Figure 6 The amino acids sequences phylogenetic tree of FANK1
以GAPDH为内参对照校正的实时荧光定量多组织表达谱分析表明BMI FANK1基因在睾丸、尿道球腺、精囊腺中高表达,由此推断该基因主要在猪的性腺和副性腺中发挥功能。据报道小鼠及人FANK1基因主要在睾丸和卵巢中高表达,在肝中几乎不表达[14],而我们研究发现FANK1基因在猪的睾丸和尿道球腺中高表达,在肝中也是低度表达,这可能由于前人未检测尿道球腺样品,也可能与物种差异有关,也可能与其他因素有关。
蛋白质的结构与功能之间存在必然联系[15],功能生物信息学分析显示BMI FANK1蛋白质二级结构中无规则卷曲和α螺旋含量较高,分别为33.53%和30.64%,无规卷曲是构成酶重要活性部位和蛋白质特异功能部位的二级结构元件,而α螺旋是蛋白质二级结构中最常见、最典型、含量也较为丰富的二级结构元件[15]。FANK1蛋白含有FN3结构域和5个重复的ANK结构域[4]。FN3是纤连蛋白中发现的三种类型的内部重复之一,广泛存在于动植物组织中,在细胞内外蛋白质、跨膜细胞因子受体、生长激素受体、酪氨酸磷酸酶受体和黏附分子等中均含有该结构域,在细菌糖基水解酶也有发现[16-17]。ANK即锚蛋白,该结构域可介导蛋白质之间的相互作用,ANK重复可以与其他类型的结构域组合发生作用[18-19],该基因5个ANK结构域衔接,具有转录起始因子、细胞周期调控、细胞骨架、离子转运蛋白、信号转换器等功能[4-5]。BMI FANK1蛋白无N端信号肽序列,表明其属于非分泌蛋白,可直接进入到细胞质基质中发挥其功能。FANK1蛋白无跨膜螺旋表明其属于非跨膜蛋白。化学修饰是蛋白质翻译后加工的重要内容,蛋白质磷酸化可以大大提高酶蛋白的活性,蛋白质酰胺化可防止羧基端被羧肽酶降解,它们是蛋白质化学修饰的两种重要方式,发现FANK1蛋白存在cAMP和cGMP依赖性蛋白激酶磷酸化、蛋白酶C磷酸化、酪蛋白激酶II磷酸化3类磷酸化位点和1类酰胺化位点,为今后深入进行FANK1蛋白的功能研究奠定了基础。
BMI猪、牛、猫、羊、人和狗等6个物种间FANK1蛋白质序列两两间相似性均在91%以上,说明FANK1基因在进化中高度保守。6个物种FANK1蛋白质序列构建的系统进化树表明,牛和羊聚在一起,然后再与猪聚为一大类,牛和羊均属于偶蹄目牛科动物,猪为偶蹄目猪科动物,它们均为哺乳纲动物,猪、牛、羊是重要的家畜,是人类驯化较早的家养动物;猫和狗聚在一起,然后再与人聚为一大类,猫为食肉目猫科,狗为食肉目犬科,猫和狗也是人类驯化较早的家养动物,是人类的伴侣,符合物种的系统分类学。
综上所述,该研究利用RT-PCR方法成功分离了BMI FANK1基因的全长编码区序列,采用qPCR确定了其在BMI 15种组织中的表达情况,并进一步对其编码的蛋白质进行了功能生物信息学分析,研究结果将为深入研究FANK1基因在猪中的功能奠定基础。
[1]EDDY E M,O'BRIEN D A.Gene expression during mammalian meiosis[J].Current Topics in Developmental Biology,1998,37:141-200.
[2]EDDY E M.Male germ cell gene expression[J].Recent Progress in Hormone Research,2002,57:103-128.
[3]HECHT N B.Molecular mechanisms of male germ cell differentiation[J].Bioessays,1998,20(7):555-561.
[4]ZHENG Z,ZHENG H,YAN W.Fank1 is a testis-specific gene encoding a nuclear protein exclusively expressed during the transition from the meiotic to the haploid phase of spermatogenesis[J].Gene Expression Patterns,2007,7(7):777-783.
[5]MOSAVI L K,MINOR D L,PENG Z Y.Consensus-derived structural determinants of the ankyrin repeat motif[J].Proceedings of the National Academy of Sciences,2002,99(25):16029-16034.
[6]SHALINI S,BANSAL M P.Role of selenium in spermatogenesis:differential expression of cjun and cfos in tubular cells of mice testis[J].Molecular and Cellular Biochemistry,2006,292(1-2):27-38.
[7]WANG H,SONG W,HU T,et al.Fank1 interacts with Jab1 and regulates cell apoptosis via the AP-1 pathway[J].Cellular and Molecular Life Sciences,2011,68(12):2129-2139.
[8]CLARET F X,HIBI M,DHUT S,et al.A new group of conserved coactivators that increase the specificity of AP-1 transcription factors[J].Nature,1996,383(6599):453-457.
[9]DONG W W,HUANG H L,YANG W,et al.Testis-specific Fank1 gene in knockdown mice produces oligospermia via apoptosis[J].Asian Journal of Andrology,2014,16(1):124-130.
[10]YU P,ZHANG L,LI S,et al.Screening and analysis of porcine endogenous retrovirus in Chinese Bannaminipig inbred line[J].
Transplant Proceedings,2004,36(8):2485-2487.
[11]ZENG R,ZENG Y Z.Molecular cloning and characterization of SLA-DR genes in the 133-family of the Bannamini-pig inbred line[J].Animal Genetics,2005,36(3):267-269.
[12]HUO J L,WANG P,ZHAO Y,et al.Molecular cloning,mRNA expression and characterization of a novel FAIM1 gene from Chinese Bannamini-pig inbred line (BMI)[J].Journal of Animal and Veterinary Advances,2012,11(8):1080-1086.
[13]WANG P,HUO H L,WANG S Y,et al.Cloning,sequence characterization,and expression patterns of members of the porcine TSSK family[J].Genetics and Molecular Research,2015,14(4):14908-14919.
[14]HWANG K C,PARK S Y,PARK S P,et al.Specific maternal transcripts in bovine oocytes and cleavaged embryos:identification with novel DDRT-PCR methods[J].Molecular Reproduction and Development,2005,71(3):275-283.
[15]王镜岩,朱圣庚,徐长法.生物化学[M].3版.北京:高等教育出版社,2002:207-220.
[16]MARCHLER-BAUER A,LU S,ANDERSON J B,et al.CDD:a Conserved Domain Database for the functional annotation of protein[sJ].Nucleic Acids Res,2011,39:225-229.
[17]MARCHLER-BAUER A,DERBYSHIRE M K,GONZALES N R,et al.CDD:NCBI's conserved domain database[J].Nucleic Acids Res,2015,4:222-226.
[18]MARCHLER-BAUER A,ANDERSON J B,CHITSAZ F,et al.CDD:specific functional annotation with the Conserved Domain Database[J].Nucleic Acids Res,2009,37:205-210.
[19]MARCHLER-BAUER A,BRYANT S H.CD-Search:protein domain annotations on the fly[J].Nucleic Acids Res,2004,32(Web Server issue):W327-W331.
CDS Cloning,qPCR Expression and Functional Bioinformatics Analysis of FANK1 Gene from Banna Mini-pig Inbred Line
HUO Jin-hua1,ZHANG Xia2,WANG Pei2*
(1.Faculty of Life Science and Technology,Huazhong Agricultural University,Wuhan 430070,China;2.Faculty of Animal Science and Technology,Yunnan Agricultural University,Kunming 650201,China)
【Objective】In order to obtain the CDS sequence,tissue expression profile and protein function information of swine FANK1 gene.【Methods】 The FANK1 mRNA sequences of pig and related species from GenBank were used as reference sequences,we designed specific primers and amplified the coding sequences and flanking sequence of FANK1 gene from Bannamini-pig inbred line(BMI)testis tissues.Real-time quatitative PCR was applied to analyse the FANK1 gene expression profiles of 15 important tissues.FANK1 protein sequence was used to carry out functional bioinformatics analysis and construct multiple species phylogenetic tree.【Results】A coding sequence of 1041 bp of BMI FANK1 gene was obtained with GenBank accession number KU705617 and KU705618,which encoded a pro-tein of 346 amino acids,with GenBank accession number AOC89035 and AOC89036.Genomic structural analysis revealed that the FANK1 gene was located on porcine chromosome 14 and consisted of 11 exons and 10 introns.Real-time quantitative PCR in multi-tissues indicated that FANK1 gene expressed highly in the testis,urethral gland and seminal vesicle,middle or low expression in the other tissues.Further functional bioinformatics analysis indicated that FANK1 protein contained two conserved domains FN3 and ANK without transmembrane region and signal peptide sequences.Its secondary structure was predominantly random coils and its N-terminal and C-terminal were hydrophilic.FANK1 protein had four kinds functional active sites.Phylogenetic analysis demonstrated that BMI had the closest relationship with cattle and sheep,which was coincident with classical taxonomy.【Conclusion】We have successfully cloned the FANK1 gene of Bannamini-pig inbred line,and detected this gene expression in 15 pigs'tissues.The results of this article would lay a foundation for further study of the gene functions in the process of spermatocyte division and regulation of signal pathway.
fibronectin type Ⅲ(FN3);ankyrin repeats(ANK);bannamini-pig inbred line(BMI);tissue expression;bioinformatics
Q78
A
1000-2650(2017)03-0408-06
10.16036/j.issn.1000-2650.2017.03.019
2017-02-24
国家自然科学基金项目(31660637;31460580;31660650)。
霍晋华,本科,主要从事生物技术研究,E-mail:jinhuahuo2016@163.com;*责任作者:王配,博士,实验师,主要从事动物分子遗传学研究,E-mail:peipei99999@126.com。
(本文审稿:朱 砺;责任编辑:秦碧雯;英文编辑:刘益平)
