花生杂种分子鉴定和ahFAD2基因分型技术研究进展
2017-11-01袁美王传堂韩锁义任艳尹亮石延茂徐平王效华李双铃郑奕雄
袁美,王传堂,韩锁义,任艳,尹亮,石延茂,徐平,王效华,李双铃,郑奕雄
(1. 山东省花生研究所/山东省花生重点实验室/农业部花生生物学与遗传育种重点实验室,山东 青岛 266100;2. 仲恺农业工程学院农学院,广东 广州 510225;3. 河南省农业科学院经济作物研究所,河南 郑州 450002)
花生杂种分子鉴定和ahFAD2基因分型技术研究进展
袁美1,王传堂1,韩锁义3,任艳1,尹亮1,石延茂1,徐平1,王效华1,李双铃1,郑奕雄2
(1. 山东省花生研究所/山东省花生重点实验室/农业部花生生物学与遗传育种重点实验室,山东 青岛 266100;2. 仲恺农业工程学院农学院,广东 广州 510225;3. 河南省农业科学院经济作物研究所,河南 郑州 450002)
杂种真实性鉴定是花生杂交育种及遗传学研究的重要环节。本研究综述了花生杂种F1真实性鉴定的分子标记方法,详述了控制花生高油酸性状(F435型)的ahFAD2基因的检测方法及其在杂种鉴定和高油酸育种中的应用。
花生;杂种鉴定;高油酸含量;ahFAD2;SNP;AhMITE;SSR
花生是一种严格的自花授粉作物。截至目前,杂交育种依然是花生育种的主要方法。花生杂交过程包括前一天下午的去雄和第二天早晨的授粉。理想情况下,来自杂交花的果应是真正的杂种。但在实际操作中,去雄不彻底、杂交开始前或杂交结束后的摘花不合适等因素都会导致假杂种的产生。花生杂交F1代真实性鉴定常用的方法有形态学鉴定法和分子鉴定法。形态学鉴定法是观察株高、分枝数量及类型、叶片形状及颜色、荚果形状及大小等性状在双亲间的表型差异,依据各相对性状在F1代的显隐性特征进行杂种鉴定,这是花生育种家长期沿用的方法,也是目前应用最多的方法。形态学鉴定法的显著特点是简便、直观,但存在鉴定周期长、标记数量不足的缺点,且有些形态特征易受环境条件的影响,特别是亲缘关系近、性状相似的双亲间的组合很难通过形态学鉴定进行辨别。随着分子生物学技术的发展,各种分子鉴定法应运而生。
随着花生基因组学的发展,各种分子标记技术应用于花生的遗传多样性分析、品种鉴定、遗传图谱构建等方面,其中,SSR分子标记是重复性最好、在花生中应用最广泛的分子标记之一[1-12]。本研究综述了分子标记用于花生杂种鉴定和高油酸性状鉴定的研究进展,以期为花生杂交育种和分子标记辅助高油酸花生的选育提供参考。
1 普通油酸亲本杂交组合的F1鉴定
根据所用的标记种类,花生杂种的分子鉴定法包含种子贮藏蛋白鉴别法、SSR分子标记法、AhMITE分子标记法和SNP标记法等4种(见表1)。因种子贮藏蛋白在花生材料间有限的多态性,仅见1例报道使用该方法[13]。Gomez等[14]于2008年率先报道了利用SSR分子标记结合水平PAGE电泳法鉴定来自8个组合共179个单株的杂合性,真杂种比率介于50%~100%。在随后的10年间,越来越多的报道使用SSR分子标记鉴定F1杂种[15-17,19,21-23,25,27,31-33]。AhMITE(花生微型反向重复转座子标记)因操作性强、多态性高、带型简单、重复性高等特点应用于花生真伪F1杂种的鉴定[20,26,33]。目前花生SNP标记在杂种鉴定上的应用较少。韩燕等[28]利用3亲本间(Tifrunner、四粒红和鲁花14)的1个SNP位点差异开发成CAPS标记,鉴定了来自4个组合的139个F1单株,发现真杂种比率在10.5%~36.7%之间。其他用于杂种鉴定的SNP位点均是基于控制花生高油酸性状的ahFAD2基因[18,24,29,34-36]。
2 高油酸亲本杂交组合的F1鉴定
自从1987 年美国的Norden等[37]筛选出油酸含量高达80%的高油酸花生自然突变体F435后,学者们围绕花生油酸、亚油酸含量的遗传规律及其分子基础、高油酸品种的选育进行了大量研究并取得了显著进展[38-48]。研究表明,花生高油酸性状受两个主效基因ahFAD2A、ahFAD2B控制。与普通油酸花生材料相比,属于F435型的高油酸花生的ahFAD2A基因编码区448 nt处碱基由G向A的转换,导致编码氨基酸由天冬氨酸D变为天冬酰胺N,ahFAD2A酶活性大幅降低;同时ahFAD2B基因编码区442 nt处插入1个A碱基,导致编码蛋白提前终止,从而导致ahFAD2B酶活性降低(图1)。基于上述2个SNP位点,开发了CAPS(Cleaved Amplified Polymorphic Sequence)[40]、PCR产物直接测序法[18,54,56]、TaqMan探针法[49,50]、AS-PCR(Allele-Specific PCR)[51,52]、KASP[30,36]等方法,并用于花生杂交后代F1的真假鉴定、高油酸品种的分子辅助选择和新种质创制(见表1、表2)。而Patel等[53]的研究发现,花生高油酸化学突变体M2-225 和C458 分别是在ahFAD2B基因起始密码子后997 bp和665 bp处存在微型反向重复转座元件插入,导致基因功能丧失。

表1 分子鉴定法在花生杂种F1鉴定上的应用汇总
注:NA表示数据不详。
2.1 CAPS法
酶切扩增多态性序列法(CAPS法)由Chu等[40]最先提出并使用,先分别用ahFAD2A、ahFAD2B的特异引物aF19f/1056r、bF19f/FADR1(引物序列见表2)进行PCR扩增,随后分别用限制性内切酶Hpy99Ⅰ和Hpy188Ⅰ酶切。ahFAD2A基因的PCR产物经Hpy99Ⅰ酶切后,普通油酸型(野生型,448G)可检测出598 bp和228 bp两个片段,而高油酸型(突变型,448A)不能被酶切,只有一个826 bp片段;ahFAD2B基因的PCR产物经Hpy188Ⅰ酶切后,野生型可检测到736、263、171 bp三个片段,而高油酸型(突变型,442A插入)可检测出550、263、213 bp 和171 bp四个片段(图1、表2)。CAPS法被成功地用于高油酸种质的突变类型检测及高油酸杂交组合F1的鉴定。陈静等[55]利用此法对5个高油酸含量花生品种(系)和2个普通油酸含量花生品种的ahFAD2 位点基因型进行了分析,结果发现高油酸品系材料06B16的ahFAD2B基因不是野生型但也不符合441_442insA突变类型这一特殊类型。赵术珍等[34]采用此法对来自3个杂交组合的85粒F1种子进行分子鉴定,其中33粒种子的基因型为AaBb,平均真杂种率为33%。
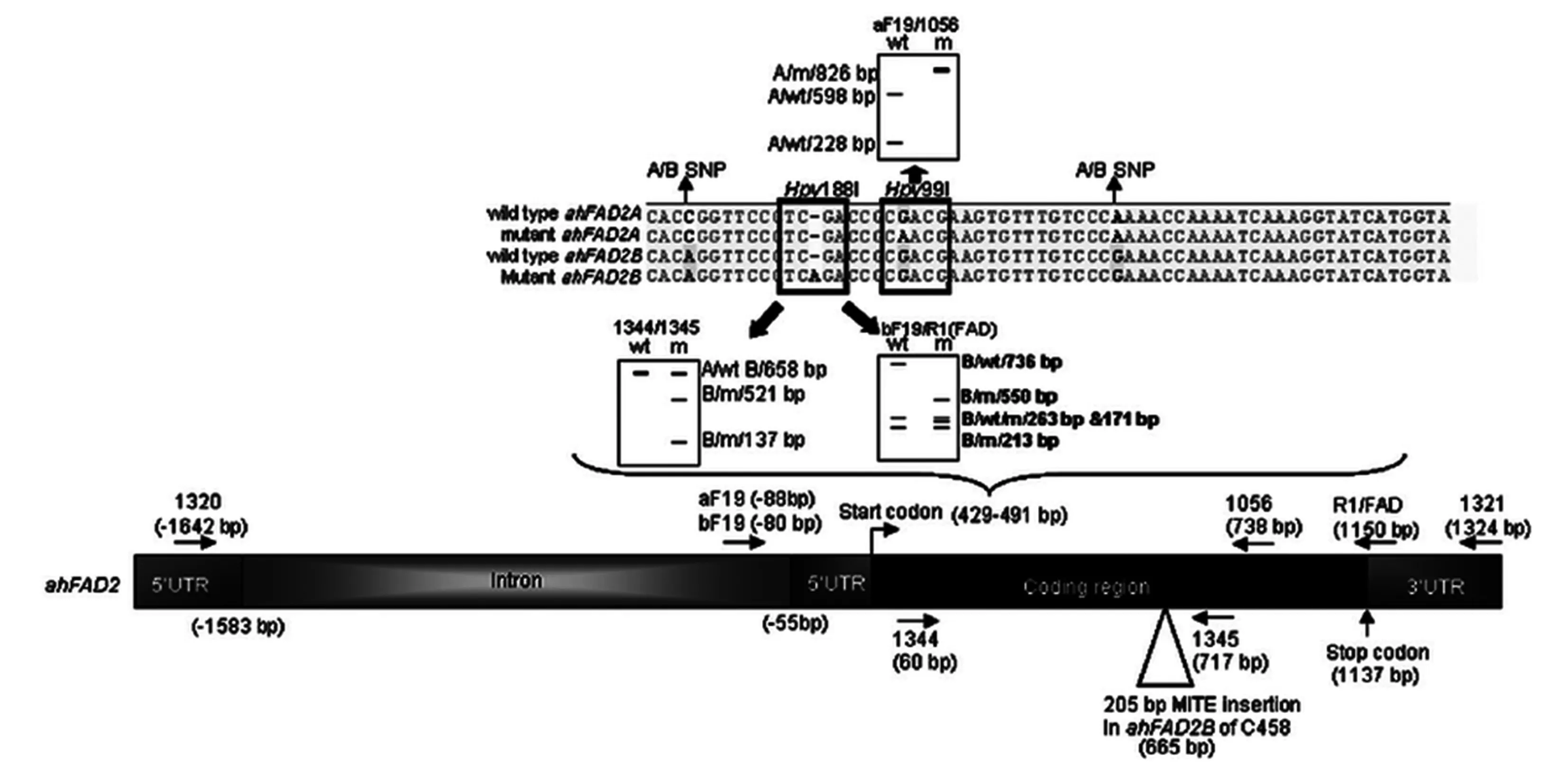
图1ahFAD2基因的结构和ahFAD2A和ahFAD2B的突变位点[40]
2.2 PCR产物直接测序法
依据高油酸突变体的ahFAD2B基因编码区442 nt处插入1个A碱基的特征(ahFAD2B441_442insA),杂交F1真杂种应同时具有ahFAD2B野生型及突变型基因,因此采用bF19/R1引物扩增ahFAD2B基因,F1真杂种ahFAD2B野生型及突变型基因均得到扩增,PCR产物测序峰图上从某处开始显示一连串套峰,伪杂种则否。Wang等[18]应用该种方法对来自4个组合的40个F1种子进行分析,鉴定出了15个真杂种(图2)。雷永等[54]利用花生ahFAD2A特异性PCR引物aF19/R3的PCR扩增产物测序法调查ahFAD2A等位基因在中国花生小核心种质中的分布,发现ahFAD2A突变型(ahFAD2A-m, 448bpA)的存在与其花生种仁中相对较高的油酸含量密切相关。廖伯寿等[56]使用F0.7/R3引物PCR扩增后的产物进行直接测序可同时检测ahFAD2A、ahFAD2B两个位点的基因型。赵术珍等[34]、于明洋等[35]用F0.7/R3引物对大量的回交组合BCF1种子进行鉴定。
2.3 TaqMan探针法
TaqMan探针法是高度特异的定量PCR技术,其核心是利用Taq酶的3′→5′外切核酸酶活性切断探针,产生荧光信号。2010年,Barkley等[49]报道了用TaqMan探针法检测ahFAD2B基因442 bp位置碱基A的缺失插入(442delA与442insA)。随后,报道了ahFAD2A448 bp位置G/A的TaqMan探针检测法[50]。在一个荧光定量PCR反应中,包含一对特异引物以及针对SNP位点的两种不同荧光标记的探针。如对ahFAD2B基因的分型,采用的荧光探针为442delA-VIC和442insA-6FAM,高油酸材料中可检测到两种荧光信号,但蓝色荧光信号非常强(图3a),而普通油酸材料中只有绿色荧光信号(图3b),杂合型材料中两种信号强度相似(图3c)。在ahFAD2A的G/A分型检测中,G448-VIC、448A-6FAM分别为检测ahFAD2A448 bp的G(普通油酸型)和A(高油酸型)探针。在普通油酸材料(基因型Ol1Ol1)中,可检测绿色VIC荧光信号(图3d),高油酸材料(ol1ol1)可检测蓝色6FAM荧光信号(图3e),杂交种(Ol1ol1)可检测到两种荧光信号(图3f)。Mienie等[57]利用此法成功检测了南非商业化花生品种(回交亲本)和500个回交后代群体中ahFAD2基因的基因型。

注:从阴影碱基处开始出现重叠峰。
图2ahFAD2B基因的PCR产物直接测序鉴定花生F1真杂种的峰图[18]

注:a~c分别为ahFAD2B基因分型中的高油酸材料、普通油酸材料和杂合型材料的扩增结果;d~f分别为ahFAD2A基因分型中的普通油酸材料、高油酸材料和杂合型材料的扩增结果。
2.4 AS-PCR法
等位基因特异性PCR(AS-PCR)又称为扩增阻碍突变系统PCR(ARMS-PCR,Amplication refractory mutation system PCR), 其检测SNP的原理是Taq酶进行PCR扩增时,引物3′端最末一个碱基与目标片段的碱基错配时,链延伸反应就会因3′,5′磷酸二酯键形成的障碍而扩增受阻,只有与SNP位点碱基互补的特异PCR引物有扩增产物。Chen等[51]针对ahFAD2A的448 bp位置的G/A设计的特异引物分别为F435WT-R和F435SUB-R,ahFAD2B的442 bp位置的A插入设计引物为F435INS-R。每个PCR反应包含3个引物,F435-F、 F435IC-R和一特异位点引物,F435-F/F435IC-R为一对内对照引物,其扩增产物大小为250 bp,作为PCR反应是否成功的检测;同时F435-F为通用引物,可与任意特异SNP位点引物配对,如F435-F/F435WT-R的扩增片段大小为193 bp,说明448 bp位置为碱基G(野生型),F435-F/F435SUB-R的扩增片段大小为203 bp,则448 bp位置为碱基A(突变型),F435-F/F435INS-R的扩增产物为大小为195 bp或无产物则表明ahFAD2B的442 bp位置有A碱基的插入(突变型)或无插入(野生型)。运用这种方法综合3个PCR反应结果可明确鉴别两种基因型,普通油酸型(Ol1Ol1Ol2Ol2)和高油酸型(ol1ol1ol2ol2)。由于栽培种花生两个亚基因组A和B高度同源,扩增子可来自任意一种拷贝,因此这种方法存在不能区分很多基因型的缺点,如Ol1ol1/Ol2Ol2与ol1ol1/Ol2Ol2、Ol1Ol1/Ol2ol2与Ol1Ol1/ol2ol2。为了解决这些问题,Yu等[52]通过在等位特异引物3′端添加错配碱基提高反应的特异性,针对每个SNP位点设计一条特异引物,组成一套AS-PCR引物(序列见表2),每个PCR反应包含一对通用引物和一个位点特异引物,综合4个反应的结果可准确判断所有的9种基因型(表3)。孟硕等[24]用ahFAD2B442nt A插入的特异引物组合FAD2B-F+FAD2B-A/FAD2-R扩增4个杂交组合的330个后代,230个获得539 bp特异扩增带,平均真杂种率为62.16%。
2.5 KASP法
竞争特异性等位基因PCR法(Kompetitive allele specific PCR ,KASP)是由英国LGC (Laboratory of the Government Chemist 政府化学家实验室)有限公司推出的, 可以在单个反应中检测两个等位基因。在该方法中,使用等位基因特异性引物用热循环仪扩增样品DNA。等位基因特异性引物分别在其5′端与荧光染料HEX和FAM缀合。当FRET盒引物与DNA杂交时,荧光染料和淬灭剂分离,导致相应的荧光的发射,通过读取荧光信号容易检测基因型。KASP反应及其组分描述于http://www.lgcgenomics.com/genotyping/kasp-genotyping-reagents/how-does-kasp-work。徐平丽等[30]设计ahFAD2A的448 bp位置的G/A特异引物分别为kFAD2A-F/A、 kFAD2A-F/G,ahFAD2B的442 bp位置的A插入或缺失的特异引物分别为kFAD2B-F/AI、kFAD2B-F/-D(表2),这些SNP位点特异引物的5′端带有LGC公司的等位基因1或2标签序列。每个PCR反应包含1个通用引物和2个5′端带等位基因1或2标签序列的SNP特异引物,以及公司提供的Master Mix,最后通过检查荧光信号就可判断SNP类型。在检测ahFAD2B442nt A/-InDel上,KASP与TaqMan探针法的一致率高达96.7%;而针对ahFAD2A448nt G/A 差异位点检测一致率仅为 71.7%。Zhao等[36]利用相同的KASP技术基于野生型和突变体ahFAD2A和ahFAD2B基因的序列设计了等位基因特异性引物(表2),可准确区分FAD2A/FAD2B野生型、突变型和杂合型三种基因型。
表2高油酸亲本杂交后代的鉴定方法汇总

方法基因引物名称及序列(5'-3')野生型扩增片段大小(bp)或特征突变型扩增片段大小(bp)或特征文献来源CAPS法ahFAD2AaF19f:GATTACTGATTATTGACTT1056r:CCAACCCAAACCTTTCAGAG598,228826[39]ahFAD2BbF19f:CAGAACCATTAGCTTTGFADR1:CTCTGACTATGCATCAG736,263,171550,263,213和171[40]PCR产物直接测序法ahFAD2BbF19f:CAGAACCATTAGCTTTGFADR1:CTCTGACTATGCATCAG[18]ahFAD2AaF19f:GATTACTGATTATTGACTTR3:CCCTGGTGGATTGTTCA[54]ahFAD2AahFAD2BF0.7:CACTAAGATTGAAGCTCR3:CCCTGGTGGATTGTTCA[56]TaqMan探针法ahFAD2BFAD2Bf:GCCGCCACCACTCCAACFAD2Br:TGGTTTCGGGACAAACACTTC442delA-probe:VIC-ACAGGTTC-CCTCGAC-MGBNFQ442insA-probe:6FAM-ACAGGTTCCCT-CAGAC-MGBNFQ59/6FAM60/VIC[49]ahFAD2AFAD2Af:GCCGCCACCACTCCAACACFAD2Ar:GTTATACCATGATACCTTT-GATTTTGGTTTTGProbe-448A:6FAM-CCTCGACCG-CAACG-MGBNFQProbe-G448:VIC-CCTCGACCGCGACG-MGBNFQ83/VIC83/VIC[50]ahFAD2AqFAD2A-F:GCCGCCACCACTCCAACACCqFAD2A-R:TGAGTGTGATGAAGAGG-GAGTAMRAqFAD2A-pA:TCTCTCGACCG-CAACGAAGTGTCY5-qFAD2A-pG:TTCTCTCGAC-CGCGACGAAGTGTTAMRACY5[30]ahFAD2BqFAD2B-F:GCCGCCACCACTCCAACACAqFAD2B-R:TGAGTGTGATGAAAAGG-GAAFAM-qFAD2B-p0:CCTCGAC-CGCGACGAAGTGTHEX-qFAD2B-pA442:GGTTCCCT-CAGACCGCGACGAHEXFAMAS-PCR法ahFAD2AF435-F:ATCCAAGGCTGCATTCTCACF435SUB-R:TGGGACAAACACTTCGTTF435IC-R:CTCCCTGGTGGATTGTTCAT-GT-203[51]ahFAD2BF435-F:ATCCAAGGCTGCATTCTCACF435INS-R:AACACTTCGTCGCGGTCT-195ahFAD2AFAD2A-F:GATTACTGATTATTGACTT-GCTTTGFAD2A-G:GTTTTGGGACAAACACT-TCTTCFAD2A-A:GTTTTGGGACAAACACT-TCTTTFAD2-R:CTCTGACTATGCATCAGAACT-TGT557[52]ahFAD2BFAD2B-F:CAGAACCATTAGCTTTGTAG-TAGTGFAD2B-C:AACACTTCGTCGCGGTTGFAD2B-A:AACACTTCGTCGCGGTTTFAD2-R:CTCTGACTATGCATCAGAACT-TGT539
续表2

方法基因引物名称及序列(5'-3')野生型扩增片段大小(bp)或特征突变型扩增片段大小(bp)或特征文献来源KASPahFAD2AkFAD2A-F/A:1_tail+TTTGGTTTTGG-GACAAACACTTCGTTkFAD2A-F/G:2_tail+GGTTTTGGGA-CAAACACTTCGTCkFAD2A-R:CCACCACTCCAACACCG-GTTCTHEXFAM[30]ahFAD2BkFAD2B-F/-D:1_tail+ACAAACACT-TCGTCGCGGTCGkFAD2B-F/AI:2_tail+GACAAACACT-TCGTCGCGGTCTkFAD2B-R:CGCCACCACTCCAACACAG-GTTHEXFAMahFAD2AAhFAD2A-Allele-l:GTTTTGGGA-CAAACACTTCGTTAhFAD2A-Allele-2:GTTTTGGGA-CAAACACTTCGTCAhFAD2A-common:CGCCACCACTC-CAACACCAllele-lTail(FAMtail):GAAGGTGAC-CAAGTTCATGCTAllele-lTail(FAMtail):GAAGGTCG-GAGTCAACGGATTHEXFAM[36]ahFAD2BAhFAD2B-Allele-l:CAAACACT-TCGTCGCGGTCTAhFAD2B-Allele-2:CAACACT-TCGTCGCGGTCGAhFAD2B-common:CCGCCACCACTC-CAACACAAllele-lTail(FAMtail):GAAGGTGAC-CAAGTTCATGCTAllele-lTail(FAMtail):GAAGGTCG-GAGTCAACGGATTHEXFAM
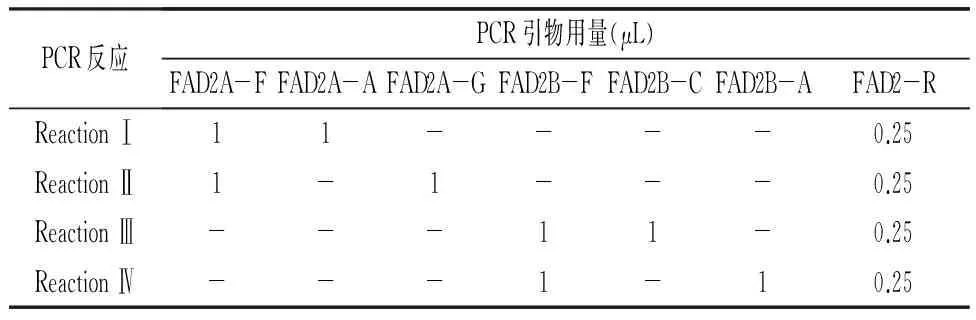
表3 用于ahFAD2A/ahFAD2B基因分型的4个反应的引物组合[52]
注:每个反应的总体积是20 μL,引物原始浓度为10 μmol/L。
3 小结
杂种F1真实性的分子鉴定是构建遗传研究群体的重要环节。从花生杂种真实性分子鉴定的报道来看,获得花生真杂种F1的比率变幅很大,效果好时可达到100%,不理想时得不到真杂种。因此,对构建遗传研究群体的杂交组合F1的分子鉴定显得非常必要。对杂交组合F1的分子鉴定,建议优先选用AhMITE标记。而对以高油酸为目标性状的回交BCF1的分子鉴定,可根据具体情况选择鉴定方法。①CAPS法和TaqMan探针法准确,但限制性内切酶和荧光探针的合成费较昂贵,而且后者还需荧光定量PCR仪等贵重的仪器。②与CAPS法和TaqMan探针法相比,改进的AS-PCR法[52]更经济和简单,不需要昂贵的实验设备,普通的分子实验室就可以开展。③以F0.7/R3为引物的PCR产物测序法(一次PCR反应和一次测序反应,PCR 扩增产物是一个同时包含FAD2A/FAD2a及FAD2B/FAD2b的混合产物)可同时检测2个突变位点,具有经济、快速的优点,不同产物的扩增量的差别必然会在一定程度上干扰读取结果, 因此利用测序法检测FAD2a和FAD2b位点也存在一定的误差, 虽然这种误差很小[35]。④最近发展起来的KASP法基于自己独特的ARM PCR原理,让所有的位点检测最终都使用通用荧光引物扩增,这大大降低了LGC KASP的试剂成本,既有金标准的准确,又降低了使用成本,比TaqMan还具有更好的位点适应性。而且,利用LGC公司推出的SNPline平台可进行高通量的检测,因此具有高通量、高效性和低成本的优点,比较适合大量样品的快速鉴定。
[1] 王传堂. DNA分子标记技术在花生上的应用研究[J]. 中国油料作物学报, 1999, 21(4): 72-76.
[2] 王传堂, 杨新道, 于翔, 等. DNA分子标记技术在花生品种鉴定上的应用研究[J]. 华北农学报, 2006, 21(S2): 110-113.
[3] 任小平, 姜慧芳, 廖伯寿, 等. 花生分子标记的研究进展[J]. 植物遗传资源学报, 2006, 7(3): 368-371.
[4] 苏君伟, 于树涛, 史普想, 等. 花生分子标记的研究进展[J]. 安徽农业科学, 2011, 39(10): 5704-5705,5710.
[5] 赵婷, 王俊宏, 徐国忠, 等. 花生高产优质育种与生物技术应用的研究进展[J]. 热带作物学报, 2011, 32 (11): 2187-2195.
[6] 江建华, 倪皖莉, 肖美华, 等. 花生SSR标记的开发及其应用现状和前景 [J]. 花生学报, 2012, 41 (2): 39-45.
[7] 李洁, 马金娜, 谷献锋, 等. 分子标记技术在花生上的应用研究[J]. 农业科技通讯, 2016(5): 17-22.
[8] 尹亮, 李双铃, 任艳, 等. 42个花生品种的SSR标记指纹图谱构建[J]. 花生学报, 2017, 46 (1): 8-13.
[9] Stalker H T, Weissinger A K, Milla-Lewis S, et al. Genomics: an evolving science in peanut [J]. Peanut Science, 2009, 36(1): 2-10.
[10] Varshney R K, Kudapa H, Roorkiwal M, et al. Advances in genetics and molecular breeding of three legume crops of semi-arid tropics using next-generation sequencing and high-throughput genotyping technologies [J]. J. Biosci., 2012, 37(5): 811-820.
[11] Pandey M K, Monyo E, Ozias-Akins P, et al. Advances inArachisgenomics for peanut improvement [J]. Biotechnology Advances, 2012, 30(3): 639-651.
[12] Guo B Z, Pandey M K, He G H, et al. Recent advances in molecular genetic linkage maps of cultivated peanut [J]. Peanut Science, 2013, 40(2): 95-106.
[13] 吴兰荣, 陈静, 胡文广, 等. 利用贮藏蛋白PAGE电泳鉴定花生远缘杂种的研究[J]. 花生学报, 2003, 32(1): 12-16.
[14] Gomez S M, Denwar N N, Ramasubramanian T, et al. Identification of peanut hybrids using microsatellite markers and horizontal polyacrylamide gel electrophoresis [J]. Peanut Science, 2008, 35(2): 123-129.
[15] 陈静, 胡晓辉, 石运庆, 等. 花生品种间杂种F1代的SSR标记分析[J]. 核农学报, 2009, 23(4): 617-620,625.
[16] 李双铃, 王辉, 任艳, 等. 利用荧光标记SSR技术鉴定花生F1代杂交种[J]. 花生学报, 2009, 38(4): 35-38.
[17] 张平湖, 刘冠明. 花生杂交F1代真假杂种SSR标记鉴定体系的建立[J]. 广东农业科学, 2009(10): 49-51.
[18] Wang C T, Yu S L, Zhang S W, et al. Novel protocol to identify true hybrids in normal oleate×high oleate crosses in peanut [J]. Electronic Journal of Biotechnology, 2010, 13(5): 18-19.
[19] 胡东青, 王秀贞, 唐月异, 等. 利用SSR标记鉴定花生真杂种[J]. 花生学报, 2012, 41(4): 22-25.
[20] 王洁, 李双铃, 王辉, 等. 利用AhMITE1转座子分子标记鉴定花生F1代杂种[J]. 花生学报, 2012,41(2): 8-12.
[21] 洪彦彬, 李少雄, 李杏瑜, 等. 利用SSR快速鉴别花生杂交F1真伪[J]. 分子植物育种, 2012, 10(1): 110-114.
[22] 王瑾, 李玉荣, 张嘉楠, 等. 花生杂交种F1真伪的SSR标记检测[J]. 中国油料作物学报, 2013, 35(增刊): 171-175.
[23] Meng S, Wang X Z, Tang Y Y, et al. Identification of peanut intersectional hybrids with SSR markers [M]// Zhang T C, Nakajima M. Advances in applied biotechnology, lecture notes in electrical engineering 333. Springer-Verlag Berlin Heidellberg, 2015. DOI: 10.1007/978-3-662-46318-5_60.
[24] 孟硕, 李丽, 何美敬, 等. 高油酸花生(ArachishypogaeaL.)杂交后代ahFAD2B基因的分子标记辅助选择[J]. 植物遗传资源学报, 2015, 16(1): 142-146.
[25] 王辉, Khera P, 李双铃, 等. SSR分子标记在花生杂种鉴定中的应用[J]. 福建农林大学学报(自然科学版), 2015,44 (4): 350-354.
[26] 尹亮, 任艳, 石延茂, 等. 利用AhMITE1转座子分子标记鉴定栽培花生杂交F1代种子真伪[J]. 山东农业科学, 2015, 47(12): 1-5.
[27] 曹广英, 吴琪, 王云云, 等. 利用SSR标记鉴定花生杂交F1代真假杂种[J]. 山东农业科学, 2016, 48(1): 7-10,15.
[28] 韩燕, 马登超, 刘译阳, 等. 利用特异性SNP位点鉴定花生杂交F1代真假杂种[J]. 山东农业科学, 2016, 48(4): 14-17.
[29] Janila P, Pandey M, Shasidhar Y, et al. Molecular breeding for introgression of fatty acid desaturase mutant alleles (ahFAD2AandahFAD2B) enhances oil quality in high and low oil containing peanut genotypes [J]. Plant Science, 2016, 242: 203-213.
[30] 徐平丽, 唐桂英, 付春, 等. 高通量检测花生油酸含量相关基因AhFAD2等位变异的方法 [J]. 农业生物技术学报, 2016,24(9): 1364-1373.
[31] 付留洋, 李丽娜, 李文静, 等. 花生栽培种(ArachishypogaeaL.)与野生种Arachismacedoi杂种F1细胞遗传分析[J]. 中国油料作物学报, 2016, 38 (3): 300-306.
[32] 李丽娜, 杜培, 付留洋, 等. 花生栽培种与野生种(Arachisoteroi)人工杂交双二倍体的创制和鉴定[J]. 作物学报, 2017, 43 (1): 133-140.
[33] 李丽娜, 付留洋, 秦利, 等. 花生栽培种与野生种(Arachisstenosperma)种间杂种的创制、鉴定与遗传分析[J]. 中国油料作物学报, 2017, 39(2): 137-144.
[34] 赵术珍, 侯蕾, 李长生, 等. 分子标记辅助回交选育高油酸花生新种质[J]. 中国油料作物学报, 2017, 39(1):30-36.
[35] 于明洋, 孙明明, 郭悦, 等. 利用回交法快速选育高油酸花生新品系[J]. 作物学报, 2017, 43(6): 855-861.
[36] Zhao S Z,Li A Q,Li C S, et al. Development and application of KASP marker for high throughput detection ofAhFAD2 mutation in peanut [J]. Electronic Journal of Biotechnology, 2017, 25:9-12.
[37] Norden A J,Gorbet D W,Knauft D A,et al. Variability in oil quality among peanut genotypes in the Florida breeding program[J]. Peanut Science, 1987, 14(1): 7-11.
[38] Moore K, Knauft D. The inheritance of high oleic acid in peanut [J]. J. Hered., 1989, 80: 252-253.
[39] Chu Y, Ramos L, Holbrook C C, et al. Frequency of a loss-of-function mutation in oleoyl-PC desaturase (ahFAD2A) in the mini-core of the U.S. peanut germplasm collection [J]. Crop Science, 2007, 47(6): 2372-2378.
[40] Chu Y, Holbrook C C, Ozias-Akins Peggy. Two Alleles ofahFAD2Bcontrol the high oleic acid trait in cultivated peanut [J]. Crop Science, 2009, 49(6): 2029-2036.
[41] 陈静. 高油酸花生遗传育种研究进展[J]. 植物遗传资源学报, 2011, 12(2): 190-196.
[42] 王传堂, 王秀贞, 唐月异, 等. 中国高油酸花生种质创制、品种选育进展与建议[J]. 花生学报, 2015, 44 (2): 49-53.
[43] 王传堂, 王秀贞, 唐月异, 等. 花生属区组间杂种高油酸自然突变体的结实特性和脂肪酸成分分析[J]. 花生学报, 2016, 45(1): 48-52.
[44] 王传堂, 王秀贞, 唐月异, 等. 高油酸大花生新品种花育961的选育[J]. 花生学报, 2014, 43(4): 60-62.
[45] 迟晓元,陈明娜,潘丽娟,等. 花生高油酸育种研究进展[J]. 花生学报, 2014, 43(4): 32-38.
[46] 任丽, 谷建中, 邓丽, 等. 高产高油酸花生品种开农176的选育[J]. 中国种业, 2016(4): 59-60.
[47] 王传堂, 唐月异, 王秀贞, 等. 7个高油酸花生新品种的丰产性和脂肪酸成分评价[J]. 山东农业科学, 2016,48(5): 31-34.
[48] 李栓柱,宋江春,王建玉,等. 高油酸花生遗传育种研究进展[J]. 作物杂志, 2017(3): 6-12.
[49] Barkley N A, Chamberlin K, Wang M L, et al. Development of a real-time PCR genotyping assay to identify high oleic acid peanuts(ArachishypogaeaL.) [J]. Molecular Breeding, 2010, 25(3): 541-548.
[50] Barkley N A, Wang M L , Pittman R N. A real-time PCR genotyping assay to detectFAD2ASNPs in peanuts (ArachishypogaeaL.) [J]. Electronic Journal of Biotechnology, 2011, 14(1): 9-10.
[51] Chen Z B, Wang M L, Barkley N A, et al. A simple allele-specific PCR assay for detectingFAD2 alleles in both A and B genomes of the cultivated peanut for high-oleate trait selection [J]. Plant Molecular Biology Reporter, 2010, 28(3): 542-548.
[52] Yu H T, Yang W Q, Tang Y Y, et al. An AS-PCR assay for accurate genotyping ofFAD2A/FAD2Bgenes in peanuts (ArachishypogaeaL.) [J]. Grasas Y Aceites, 2013, 64(4): 395-399.
[53] Patel M, Jung S, Moore K et al. High-oleate peanut mutants result from a MITE insertion into theFAD2 gene[J]. Theoretical and Applied Genetics, 2004, 108(8): 1492-1502.
[54] 雷永,姜慧芳,文奇根,等.ahFAD2A等位基因在中国花生小核心种质中的分布及其与种子油酸含量的相关性分析[J]. 作物学报, 2010, 36(11): 1864-1869.
[55] 陈静, 白鑫, 胡晓辉,等.利用CAPS标记推测花生品种(系)FAD2位点基因型的研究[J]. 核农学报, 2013, 27(1): 28-32.
[56] 廖伯寿,雷永,姜慧芳,等. 一种花生高油酸回交育种后代基因型的快速精准鉴定方法:中国, CN 105200142 A. 201510675886.4[P].
[57] Mienie C M S, Pretorius A E. Application of marker-assisted selection forahFAD2AandahFAD2Bgenes governing the high-oleic acid trait in South African groundnut cultivars (ArachishypogaeaL.) [J]. African Journal of Biotechnology, 2013, 12(27): 4283-4289.
AdvancesonIdentificationofTrueF1HybridsandahFAD2GenotypingTechniqueinPeanut
Yuan Mei1, Wang Chuantang1, Han Suoyi3, Ren Yan1,Yin Liang1, Shi Yanmao1, Xu Ping1, Wang Xiaohua1, Li Shuangling1, Zheng Yixiong2
(1.ShandongPeanutResearchInstitute/ShandongProvincialKeyLaboratoryofPeanut/KeyLaboratoryofPeanutBiologyandGeneticImprovement,MinistryofAgriculture,Qingdao266100,China; 2.SchoolofAgronomy,ZhongkaiUniversityofAgricultureandEngineering,Guangzhou510225,China; 3.InstituteofEconomicalCrops,HenanAcademyofAgriculturalSciences,Zhengzhou450002,China)
The identification of true F1hybrids is critical for peanut genetics and crossing breeding. The molecular marker methods for identification of true F1hybrids were summarized. The identification methods onahFAD2 gene conferring high oleic acid content in peanut and their application in identification of F1hybrids and high oleic acid breeding were detailed.
Peanut; Hybrid identification; High oleic acid content;ahFAD2;SNP;AhMITE;SSR
S565.203.53
A
1001-4942(2017)10-0143-08
10.14083/j.issn.1001-4942.2017.10.031
2016-11-18;
2017-09-25
科技部“863”计划子课题(2013AA102602);国家自然科学基金项目(31471533);山东省农业科学院科技创新重点项目(CXGC2016B02);国家花生产业技术体系项目(CARS-14);山东省农业科学院青年基金项目(2015YQN21)
袁美(1972—),女,博士,研究员,研究方向:花生生物技术育种。E-mail: yuanbeauty@126.com
李双铃(1971—),男,副研究员,研究方向:花生育种与科研管理。E-mail:lisl7105@163.com 郑奕雄(1962—),男,博士,教授,研究方向:花生遗传育种。E-mail: gdsscqs@163.com
