基于谱特征嵌入的脑网络状态观测矩阵降维方法
2017-10-21代照坤王文哲王亚楠
代照坤,刘 辉,王文哲,王亚楠
(昆明理工大学 信息工程与自动化学院,昆明 650504)
(*通信作者电子邮箱liuhui621@126.com)
基于谱特征嵌入的脑网络状态观测矩阵降维方法
代照坤,刘 辉*,王文哲,王亚楠
(昆明理工大学 信息工程与自动化学院,昆明 650504)
(*通信作者电子邮箱liuhui621@126.com)
针对基于功能核磁共振(fMRI)重构的脑网络状态观测矩阵维数过高且无特征表现的问题,提出一种基于谱特征嵌入(Spectral Embedding)的降维方法。该方法首先计算样本间相似性度量并构造拉普拉斯矩阵;然后对拉普拉斯矩阵进行特征分解,选取前两个主要的特征向量构建2维特征向量空间以达到数据集由高维向低维映射(降维)的目的。应用该方法对脑网络状态观测矩阵进行降维并可视化在二维空间平面,通过量化类别有效性指标对可视化结果进行评价。实验结果表明,与主成分分析(PCA)、局部线性嵌入(LLE)、等距映射(Isomap)等降维算法相比,使用该方法得到的脑网络状态观测矩阵低维空间的映射点有明显的类别意义表现,且在类别有效性指标上与多维尺度分析(MDS)和t分布随机邻域嵌入(t-SNE)降维算法相比,同一类样本间平均距离Di指数分别降低了87.1%和65.2%,不同类样本间平均距离Do指数分别提高了351.3%和25.5%;在多个样本上的降维可视化结果均有一定的规律性体现,该方法的有效性和普适性得以验证。
高维数据降维;功能脑网络;脑网络;状态观测矩阵;谱特征嵌入算法;动态特性
0 引言
人类大脑由上千亿个神经细胞组成,相当于银河系的星体总数,它长期以来保持着神秘色彩,人们对其知之甚少。近些年世界上各个国家纷纷启动人类大脑计划或与人类大脑相关的研究计划,可见研究人类大脑的重要性。如今功能核磁共振成像(functional Magnetic Resonance Imaging, fMRI)技术的普遍应用更为研究人员进一步探索大脑功能性活动变化提供了必要的无侵入手段,fMRI可通过探测大脑血氧水平依赖(Blood-Oxygen-level-dependent,BOLD)信号变化达到观察大脑活动变化的目的[1]。
现有的基于fMRI脑成像研究有很多是在静息态下进行,即实验时要求被试者放松,不要刻意去想某件事情,此类研究发现,即使在休息时,多个大脑区域之间也表现出了高度相关的自发活动[2-5],针对这一问题,脑网络动态特性的研究逐渐成为多数学者探索的热点。
马士林等[6]采用成组独立成分分析(Independent Component Analysis, ICA)的方法来构建动态脑功能连接网络,并对精神分裂症患者和正常被试的数据进行分类识别;高晴等[7]利用动态因果模型,通过研究运动想象和运动执行的主要激活脑区(M1和SMA的脑功能动态网络),深入了解运动想象的神经机制;王昱青[8]利用分级聚类分析(Hierarchical Clustering Analysis, HCA)与邻域相关(Neighborhood Correlation, NC)以及独立成分分析相结合的方法来探测相关脑区功能性动态网络的关联特性;Yu等[9]通过对比分析健康人和精神分裂病人脑功能连接的动态属性,揭示了精神分裂病人异常的大脑活动表现;Suk等[10]使用隐马尔可夫模型(Hidden Markov Model, HMM)来估计基于静息态fMRI构建的脑功能网络的动态特性。已有研究更侧重于对动态脑网络的构建或者对部分脑区甚至疾病状态脑功能动态特性的分析,但在分析全脑网络动态特征在时间上的演变特性方面还有待进一步研究。
本文对脑网络的动态特性研究是基于滑动时间窗口技术构建的全脑网络状态观测矩阵[11]展开的,且由于脑网络状态观测矩阵的维数过高,不便于对人脑网络动态特征进行辨识和分析,所以本文对其降维方法展开研究,旨在分析人脑网络动态特征在时间上的演变是否状态可分。在此利用谱特征嵌入(Spectral Embedding)算法[12]对脑网络观测矩阵进行降维分析,该方法采用流形学习,从图论的原理出发,对脑网络状态观测矩阵构造无向加权图G=(V,E),由样本间相似性度量得到度矩阵D并构造出拉普拉斯矩阵L(L=D-W),对拉普拉斯矩阵L进行特征分解,选取主要的特征向量构建一个k(k≥2)维特征向量空间达到数据集由高维向低维映射(降维)的目的。与目前主流的主成分分析(Principal Component Analysis, PCA)[13]、t分布随机邻域嵌入(t-distributed Stochastic Neighbor Embedding, t-SNE)[14]、局部线性嵌入(Locally Linear Embedding, LLE)[15]等高维数据降维方法应用于脑网络状态观测矩阵的降维效果对比,实验结果显示,应用本文算法降维可视化后显示出明显合理的类别效果,并在类别有效性指标上优于基于t-SNE等算法的降维可视化结果,这为进一步描述人类大脑在时间上的动态演变特性提供了必要的实验基础。
1 脑网络状态观测矩阵的构建和表达
根据AAL(Anatomical Automatic Labeling)模板将全脑共划分成90个脑区,通过fMRI在采样周期内分别采集90个脑区的BOLD时间序列信号,如图1所示,从左至右为脑网络状态观测矩阵的构建过程。在信号采集的基础上,用步长为L的滑动窗口对BOLD信号在N个时间点上进行分割,得到N个脑网络状态观测窗口(对应于图1中的窗口1至窗口N);然后在每个窗口内利用皮尔逊相关系数,计算每两个脑区之间的信号相关度,得到一个90×90的相关度矩阵;由于每两个脑区信号相关度在计算过程中会重复一次,所以得到的相关度矩阵是沿对角线对称的结构,在此取主对角线以上的元素并且首尾相连组成一个1×4 005维的向量,即脑网络状态观测向量;最后,随着滑动窗口的进行会依次得到N个1×4 005的脑网络状态观测向量,由这些向量得到本文的研究对象:N×4 005的脑网络状态观测矩阵。

图1 脑网络状态观测矩阵构建过程Fig. 1 Construction process of brain network state observation matrix
2 算法的原理和类别有效性指标的建立
2.1 基于谱特征嵌入的脑网络状态观测矩阵降维算法
谱特征嵌入(Spectral Embedding)是一种流形学习算法,不同于目前主流的降维算法,该算法以图论原理为基础,对以图形为基础构建的相关度矩阵映射效果较为显著。本文的研究对象正是基于fMRI图像分析两两脑区间的BOLD信号强度相关性重构的脑网络状态观测矩阵,因此从原理上Spectral Embedding算法是适用于研究脑网络状态观测矩阵的。在此基础上,本文基于Spectral Embedding的脑网络状态观测矩阵降维算法具体实施步骤如下:
1)假设本文第1章中构建的N个脑网络状态观测向量是分布在高维空间中的N个样本点,对需要降维的N个脑网络状态观测向量构造无向加权图G=(X,E),其中X表示N个样本点,且X中每个样本xi的取值服从高斯分布(i∈[1,N]),eij∈E(i,j∈[1,N])表示每两个样本点xi和xj的连接边。
2)设置参数n_neighbors=h,h∈[1,N],n_neighbors表示构造度矩阵时设置的近邻系数;每两个样本点的相似性用其之间边的权值wij∈[0,1]表示,则无向加权图G的相似性矩阵W∈RN×N定义为:
(1)
(2)
其中:dist(xi,xj)表示两个样本点xi和xj之间的测地线距离,σ为高斯分布的尺度参数。
3)由式(1)、(2)可得样本点度的定义:
(3)
由度的定义di组成度矩阵D:
D=diag(d1,d2,…,dN)
(4)
4)由式(4)构造样本集的拉普拉斯矩阵L并进行特征分解,即:
Lθ=λDθ
(5)
其中:特征分解求得的特征值1≥λ1≥λ2≥…≥λN≥0,对应特征向量θ1,θ2,…,θN。选取主要的特征向量θ1,θ2,…,θk构建一个k维特征向量空间X∈RN×k用以表示原数据集,以达到降维的目的。对于脑网络状态观测矩阵需要降到2维并可视化后观察其是否有类别意义表现,所以取k=2,从而实现将4 005维脑网络状态观测矩阵数据映射到2维。
5)若降维后可视化没有明显类别意义表现,则反馈调整参数n_neighbors值h,重复步骤2)~4)以达到合理的降维可视化效果。
2.2 类别有效性指标的建立
针对如何量化指标评价基于Spectral Embedding算法将脑网络状态观测矩阵降维可视化结果的问题,本文提出一种基于计算同一类样本间平均距离Di和不同类样本间平均距离Do的方法。具体构建步骤如下:
1)假设降维后可视化结果能够将数据样本划分为若干个通常是不相交的子集,每个子集称为一个“类”。针对本文第1章中构建的脑网络状态观测矩阵,则样本集X={x1,x2,…,xN}包含N个无标记样本,每个样本xi=(xi1;xi2;…;xi4 005)是一个4 005维向量,则降维算法将样本集X降到2维得到低维空间下原数据集表示yj=(yj1,yj2);对其可视化后表现出m种类别信息{Lr|r=1,2,…,m},每一类别含有n个样本数据。其中i,j,m,n∈[1,N],且为整数,定义:
(6)
(7)
(8)
其中:dist(yi,yj)用于计算降维后同一类内每两个样本之间的欧氏距离,则dsum对应于同类样本间欧氏距离的和;Cen代表同类间样本的中心点,dist(Ceni,Cenj)用于计算每两个中心点间的欧氏距离,dcen则对应于不同类间所有中心点欧氏距离的和。
2)基于式(6)~(8)可导出类别有效性指标:
①同一类样本间平均距离,Di指数:
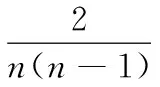
(9)
②不同类样本间平均距离,Do指数:
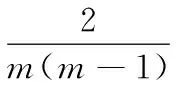
(10)
3)由式(9)、(10)得出:对于降维可视化后得到的类别m,如果Do指数值越大并且Di指数值越小(即[IDo=max(IDo)]&[IDi=min(IDi)]),则类别有效性指标越好。
3 实验与分析
3.1 实验数据与实验平台
3.1.1 实验数据
本文利用美国通用电气(GE)公司制造的3.0 Tesla 磁共振成像仪对三个健康人(性别、年龄不详)的fMRI数据进行采集。静息态功能成像要求被试者保持不动的平躺姿势,闭眼但不进入睡眠状态,头脑清醒,不进行特别的思维活动,数据采集过程使用头部正交线圈、梯度回波序列-回波平面成像,重复时间TR=1 500 ms,回波时间TE=30 ms,采集矩阵64×64,视野FOV=256 mm×256 mm,扫描层厚4 mm,层间距0 mm,翻转角Flipangle为60°,共采集时间点个数N=200。随后对成像的数据用Resting State Pipeline平台进行预处理,预处理参数设置如下:
剔除前4个不稳定时间点后N=196,滑动窗口大小M=20,窗口移动步长L=1,根据图1脑网络状态观测矩阵的构建过程,则会生成(N-M+1)即177个状态观测向量。由此三组健康人的fMRI数据便会生成三组177×4 005的脑网络状态观测矩阵,将这三组数据分别标记为A、B、C。
3.1.2 实验平台
本文实验中所用的计算机是Intel Pentium CPU G2020 @2.90 GHz,8 GB内存,500 GB硬盘。由于近些年来研究学者多把Python作为开发工具来实现脑科学领域相关算法[16],所以本文算法也利用Python编程实现。
PyQt是Qt与Python的成功融合,本文基于Qt框架并结合Python第三方库PyQt4设计实现了基于Spectral Embedding算法的脑网络降维可视化平台(Human Brain Network Dimensionality Reduction Visualization Platform based on Spectral Embedding),如图2所示,该平台集成了降维维数设置、参数设置、数据转换、Spectral Embedding降维及可视化等功能。

图2 基于Spectral Embedding算法的脑网络降维可视化平台界面Fig. 2 Interface of human brain network dimensionality reduction visualization platform based on Spectral Embedding
3.2 算法参数调节实验
本文首先对3.1.1节中构建的脑网络状态观测矩阵A应用3.1.2节中所开发平台进行降维实验,可视化采用对降维后映射到2维坐标平面的样本点按时间序列进行编号处理(编号顺序为0~176),并且将不同类别用不同符号表示予以区分。实验过程中Spectral Embedding算法的参数选择会直接影响最后降维可视化的效果,因此本文采用假设检验的方法就参数n_neighbors取值大小对降维可视化结果影响进行大量实验,以得到同组数据最为明显的类别结果,充分发挥本文算法对脑网络状态观测矩阵的降维效果。
参数n_neighbors表示使用最近邻法构造亲和度矩阵时设置的近邻系数,其取值范围为n_neighbors∈(0, 177],且为整数,本文就n_neighbors的取值从1~177分别进行177次实验观察降维后可视化结果,发现当n_neighbors取值为21时,降维可视化结果显示出明显的类别效果(按可视化样本集编号分为:0~52、53~121和122~176三类),且优于本组数据样本参数n_neighbors取其他值的结果。在此选取其中n_neighbors值为1、10、20、21、22、23、30、40、70和100等10组实验结果进行展示,如图3所示。
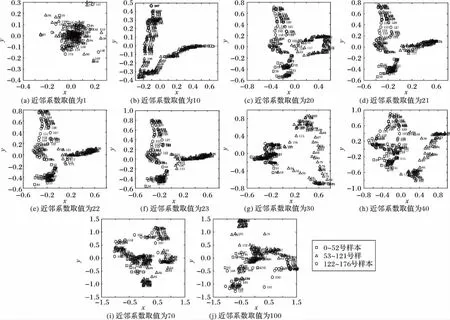
图3 参数n_neighbors调节实验结果Fig. 3 Experimental results of adjusting the parameter n_neighbors
3.3 对比实验
3.3.1 降维效果对比实验
本文对数据A首先应用四种主流降维算法分别进行降维实验,分别为主成分分析(PCA)、等距映射(Isometric Mapping, Isomap)算法、随机森林(Random Trees)和局部线性嵌入(LLE)。多次实验的最佳降维可视化输出效果如图4所示,其中177个二维空间样本点用符号标注与图3相同。
3.3.2 类别指标对比实验
由于实验过程中采用多维尺度分析(Multi-dimensional scaling, MDS)和t分布随机邻域嵌入(t-SNE)[17]算法分别对脑网络状态观测矩阵降维后可视化的结果均显示有类别信息,为了更充分地说明本文算法对脑网络状态观测矩阵降维的优越性,参照2.2节给出的类别有效性指标,将本文算法与MDS和t-SNE算法分别采用类别有效性指标Di指数和Do指数计算,并进行对比实验,实验过程如下:
1)本文对数据A分别采用MDS算法、t-SNE算法和本文算法进行降维,并通过多次实验选择最佳可视化输出效果,其中MDS算法的降维维度n_components=2,初始化运行smacof算法的时间常数n_init=10,单次运行smacof算法的最大迭代次数max_iter=300;t-SNE算法的降维维度n_components=2,学习率learning_rate=100,优化的最大迭代次数n_iter=1 000。实验结果如图5所示。
2)对上述算法将脑网络状态观测矩阵A降维后二维空间内的177个数据点形成的三种类别效果,分别采用类别有效性指标进行计算后结果如表1。
3.4 不同被试的脑数据降维实验
本文就脑网络状态观测矩阵降维及可视化进一步采用B和C两组数据进行实验,旨在进一步验证Spectral Embedding算法对脑网络状态观测矩阵数据降维的有效性和普遍适用性。实验中Spectral Embedding算法应用于数据B和C的n_neighbors参数分别设置为32和27,结合数据A降维可视化结果,三组数据A、B、C可视化后结果以及类别有效性指标计算结果如图6和表2所示。
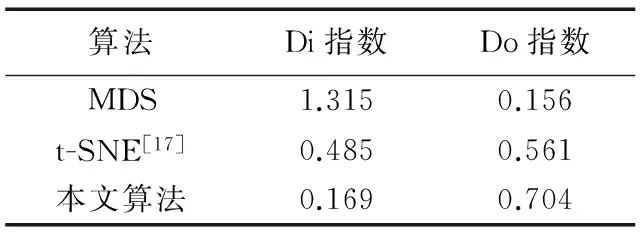
表1 三种算法降维可视化类别有效性指标对比Tab. 1 Category validity indicator comparison of dimensionality reduction and visualization based on three algorithms
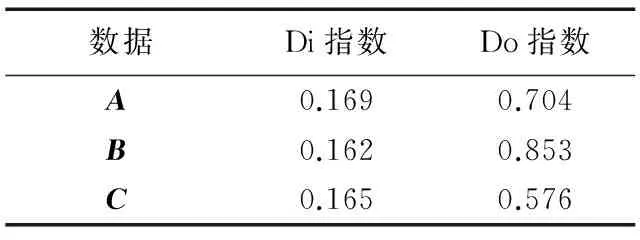
表2 数据A、B、C类别有效性指标计算结果Tab. 2 Category validity indicator calculation results of data A, B and C

图4 本文算法与4种对比算法的降维可视化结果对比Fig. 4 Comparison of dimensionality reduction and visualization results between the proposed algorithm and other four contrast algorithms
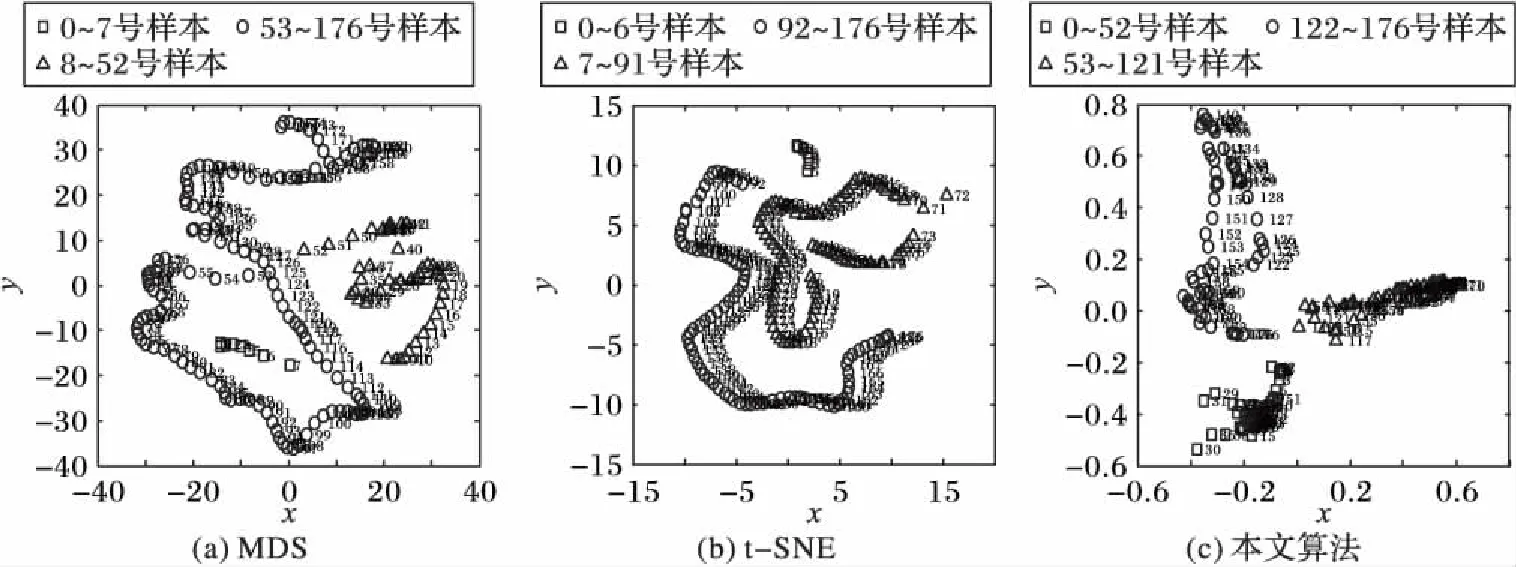
图5 本文算法与另2种对比算法的降维可视化结果对比Fig. 5 Dimensionality reduction and visualization results between the proposed algorithm and other two contrast algorithms

图6 数据A、B、C应用本文算法降维可视化结果Fig. 6 Dimensionality reduction and visualization results of data A, B and C based on Spectral Embedding
3.5 实验结果分析
以上实验结果分析如下:
1)从3.2节实验结果图3可以看出,当n_neighbors取值为21时,使用本文算法将数据A从4 005维映射到二维空间平面后,不同时间点的样本具有明显的可区分性,重叠区域较少,三种类别和时间顺序强相关;而当参数n_neighbors取其他值时标记的类别样本点分布混乱,没有明显类别意义表现,说明适当的算法参数选取对验证本文算法有效性的重要程度。
2)从3.3节对比实验结果得出以下分析结论:
①由图4可以看出,对数据A分别采用PCA、Isomap、LLE、Random Trees算法降维可视化后,二维空间映射的样本点无明显类别特征表现,相近时间内标记符号的数据点无任何分布规律,且出现相互交叉分布现象;而从本文算法对数据A进行降维后的可视化结果看,相近时间点的数据分布集中,用符号标记的样本点类别信息明显,不同类之间断点清晰可见,无交叉分布现象。所以与其他四种降维算法相比,本文算法更适用于对脑网络状态观测矩阵进行降维分析。
②由图5可以看出,虽然MDS算法和t-SNE算法分别对数据A进行降维可视化后表现出一定的类别特征,但采用类别有效性指标对降维可视化结果进行评价后可以由表1看出,采用本文算法降维得到的Di指数比另外两种算法分别降低了87.1%和65.2%,Do指数比另外两种算法分别提高了351.3%和25.5%。也就是说在有明显类别意义的前提下,本文算法降维得到的类别有效性指标明显比另外两种算法好,即从量化的指标上说明本文算法对脑网络状态观测矩阵降维效果优于MDS和t-SNE算法降维效果。
综合以上对比实验结果,由于Spectral Embedding算法以图论原理为基础,对以图形为基础构建的相关度矩阵映射效果较为显著,而本文的研究对象正是基于fMRI图像分析两两脑区间的BOLD信号强度相关性重构的脑网络状态观测矩阵,因此从算法原理上Spectral Embedding是适用于研究脑网络状态观测矩阵降维的;其他降维算法则不具备这种特点,所以针对脑网络状态观测矩阵的降维效果没有本文所采用的Spectral Embedding算法好。
3)由图6可以看出,应用Spectral Embedding算法对三组不同的脑网络观测矩阵进行降维并可视化后均有明显的类别信息表示,同类间样本点分布较为集中,且这种分布规律与时间强相关;不同类之间具有清晰的断点,这从实验的角度证明了本文算法普遍适用于对脑网络状态观测矩阵进行降维分析。
综合以上实验结果,可以证明基于fMRI重构的静息态人脑网络动态特征在时间上的演变具有类别意义,能为进一步研究构建人类大脑动态演变模型和分析病态大脑演变特性提供必要的实验基础和依据。
4 结语
本文将Spectral Embedding降维算法应用于人脑网络状态观测矩阵降维实验中,实验结果和类别有效性指标显示,本文方法对脑网络状态观测矩阵降维可视化后在二维空间的分布有较明显的类别意义,且在基于MDS和t-SNE算法对脑网络状态观测矩阵降维可视化后同样具有类别意义的前提下,采用本文方法的类别有效性指标明显优于MDS和t-SNE两种方法。
以本文为基础,后续研究将不断优化基于PyQt的脑网络状态观测矩阵降维及可视化平台,使之逐渐发展成为脑网络动态特性研究的综合型平台;围绕脑网络动态特性建模展开研究,加入疾病对照组进行实验分析,进一步验证人类大脑网络的动态演变特性,为医学上关于脑疾病研究的发展提供必要的理论和技术支持。
References)
[1] BISWAL B, YETKIN F Z, HAUGHTON V M, et al. Functional connectivity in the motor cortex of resting human brain using echo-planar MRI [J]. Magnetic Resonance in Medicine, 1995, 34(4): 537-541.
[2] LOWE M J, DZEMIDZIC M, LURITO J T, et al. Correlations in low-frequency BOLD fluctuations reflect cortico-cortical connections [J]. Neuroimage, 2000, 12(5): 582-587.
[3] BUCKNER R L, VINCENT J L. Unrest at rest: default activity and spontaneous network correlations [J]. NeuroImage, 2007, 37(4): 1091-1096.
[4] VAN DEN HEUVEL M, MANDL R, POL H H. Normalized cut group clustering of resting-state fMRI data [J]. PLOS ONE, 2008, 3(4): e2001.
[5] WANG X, WANG Q. A novel image encryption algorithm based on dynamic S-boxes constructed by chaos [J]. Nonlinear Dynamics, 2014, 75(3): 567-576.
[6] 马士林,梅雪,李微微,等.fMRI动态功能网络构建及其在脑部疾病识别中的应用[J].计算机科学,2016,43(10):317-321. (MA S L, MEI X, LI W W, et al. Building of fMRI dynamic functional connectivity network and its applications in brain diseases identification[J]. Computer Science, 2016, 43(10): 317-321.)
[7] 高晴,陈华富.基于动态因果模型的运动执行和运动想象脑网络研究[J].电子科技大学学报,2010,39(3):457-460. (GAO Q, CHEN H F. Study of brain networks during motor execution and imagery using dynamic causal model [J]. Journal of University of Electronic Science and Technology of China, 2010, 39(3): 457-460.)
[8] 王昱青.基于运动任务的脑功能动态网络的整合研究[D].西安:西安电子科技大学,2011:5-6. (WANG Y Q. Integration research of brain function dynamic network based on motion task[D]. Xi’an: Xidian University, 2011: 5-6.)
[9] YU Q, ERHARDT E B, SUI J, et al. Assessing dynamic brain graphs of time-varying connectivity in fMRIdata: application to healthy controls and patients with schizophrenia [J]. NeuroImage, 2015, 107: 345-355.
[10] SUK H-I, WEE C-Y, LEE S-W, et al. State-space model with deep learning for functional dynamics estimation in resting-state fMRI [J]. NeuroImage, 2016, 129: 292-307.
[11] 马洒洒,王彬,薛洁,等.基于同步多维数据流的脑网络动态特征辨识方法研究[J/OL].计算机应用研究, 2016 [2016- 11- 28]. http://www.cnki.net/kcms/detail/51.1196.TP.20161128.1600.062.html. (MA S S, WANG B, XUE J, et al. Research of dynamic characteristic identification method for human brain network based on multidimensional synchronization data flow [J/OL]. Application Research of Computers, 2016 [2016- 11- 28]. http://www.cnki.net/kcms/detail/51.1196.TP.20161128.1600.062.html.)[12] 韩丽,颜震,徐建国,等.基于显著特征谱嵌入的三维模型相似性分析[J].模式识别与人工智能,2015,28(12):1119-1126. (HAN L, YAN Z, XU J G, et al. Three-dimensional model similarity analysis based on salient features spectral embedding [J]. Pattern Recognition and Artificial Intelligence, 2015, 28(12): 1119-1126.)
[13] REICH D, PRICE A L, PATTERSON N. Principal component analysis of genetic data [J]. Nature Genetics, 2008, 40(5): 491-492.
[14] MAATEN L, HINTON G. Visualizing data using t-SNE [J]. Journal of Machine Learning Research, 2008, 620(1): 267-284.
[15] DONOHO D L, GRIMES C. Hessian eigenmaps: locally linear embedding techniques for high-dimensional data [J]. Proceedings of the National Academy of Sciences, 2003, 100(10): 5591-5596.
[16] TORBEN-NIELSEN B. An efficient and extendable Python library to analyze neuronal morphologies [J]. Neuroinformatics, 2014, 12(4): 619-622.
[17] 董迎朝,王彬,马洒洒,等.基于t-SNE的脑网络状态观测矩阵降维方法研究[J/OL].计算机工程与应用, 2017 [2017- 02- 16]. http://d.wanfangdata.com.cn/Periodical/pre_83f9a8c2-08d4-4731-8f7c-778f3d088a54. (DONG Y Z, WANG B, MA S S, et al. Dimension reduction method research of brain network status observation matrix based on t-SNE [J/OL]. Computer Engineering and Applications, 2017 [2017- 02- 16]. http://d.wanfangdata.com.cn/Periodical/pre_83f9a8c2-08d4-4731-8f7c-778f3d088a54.)
This work is partially supported by the National Natural Science Foundation of China (61263017), the Teaching Reform Project of Kunming University of Science and Technology (10968397).
DAIZhaokun, born in 1990, M. S. candidate. His research interests include real-time dynamic characteristic analysis of human brain network, dimensionality reduction of high dimensional data.
LIUHui, born in 1984, Ph. D., associate professor. His research interests include real-time computer control, image processing, pattern recognition.
WANGWenzhe, born in 1991, M. S. candidate. His research interests include image processing.
WANGYanan, born in 1990, M. S.candidate.Her research interests include high dimensional data reduction, data mining.
DimensionreductionmethodofbrainnetworkstateobservationmatrixbasedonSpectralEmbedding
DAI Zhaokun, LIU Hui*, WANG Wenzhe, WANG Yanan
(FacultyofInformationEngineeringandAutomation,KunmingUniversityofScienceandTechnology,KunmingYunnan650504,China)
As the brain network state observation matrix based on functional Magnetic Resonance Imaging (fMRI) reconstruction is high-dimensional and characterless, a method of dimensionality reduction based on Spectral Embedding was presented. Firstly, the Laplacian matrix was constructed from the similarity measurement between the samples. Secondly, in order to achieve the purpose of mapping (reducing dimension) datasets from high dimension to low dimension, the first two main eigenvectors were selected to construct a two-dimensional eigenvector space through Laplacian matrix factorization. The method was applied to reduce the dimension of the matrix and visualize it in two-dimensional space, and the results were evaluated by category validity indicators. Compared with the dimensionality reduction algorithms such as Principal Component Analysis (PCA), Locally Linear Embedding (LLE), Isometric Mapping (Isomap), the mapping points in the low dimensional space got by the proposed method have obvious category significance. According to the category validity indicators, compared with Multi-Dimensional Scaling (MDS) and t-distributed Stochastic Neighbor Embedding (t-SNE) algorithms, the Di index (the average distance among within-class samples) of the proposed method was decreased by 87.1% and 65.2% respectively, and the Do index (the average distance among between-class samples) of it was increased by 351.3% and 25.5% respectively. Finally, the visualization results of dimensionality reduction show a certain regularity through a number of samples, and the effectiveness and universality of the proposed method are validated.
dimensionality reduction of high dimensional data; functional brain network; brain network; state observation matrix; Spectral Embedding algorithm; dynamic characteristics
TP391.4
A
2017- 01- 13;
2017- 03- 08。
国家自然科学基金资助项目(61263017);昆明理工大学教学改革项目(10968397)。
代照坤 (1990—),男,陕西渭南人,硕士研究生,主要研究方向:人脑网络实时动态特性分析、高维数据降维; 刘辉(1984—),男,陕西渭南人,副教授,博士,CCF会员,主要研究方向:实时计算机控制、图像处理、模式识别; 王文哲(1991—),男,河南平顶山人,硕士研究生,主要研究方向:图像处理; 王亚楠(1990—),女,河南信阳人,硕士研究生,主要研究方向:高维数据降维、数据挖掘。
1001- 9081(2017)08- 2410- 06
10.11772/j.issn.1001- 9081.2017.08.2410
