基于SSR标记的黑龙江省绿豆品种遗传多样性分析及指纹图谱构建
2017-10-19,,2,,*
, ,2,,*
(1.黑龙江八一农垦大学食品学院,黑龙江大庆 163319;2.国家杂粮工程技术研究中心,黑龙江大庆 163319)
基于SSR标记的黑龙江省绿豆品种遗传多样性分析及指纹图谱构建
赵雅楠1,王颖1,2,张东杰1,*
(1.黑龙江八一农垦大学食品学院,黑龙江大庆 163319;2.国家杂粮工程技术研究中心,黑龙江大庆 163319)
目的:为明确黑龙江地区绿豆品种遗传多样性水平并构建SSR指纹图谱,为绿豆育种研究及品种鉴定奠定基础。方法:基于SSR荧光标记技术,利用筛选得到的14对SSR引物对黑龙江地区35份绿豆品种进行遗传多样性分析及SSR指纹图谱构建。结果:14对SSR引物在35个绿豆品种中共检测到49个等位基因,每对引物检测到2~7个,平均3.5个;多态性信息含量PIC值介于0.106~0.761之间,平均为0.398。参试品种遗传相似系数变幅介于0.6966~1.0000之间,平均为0.894,其中在0.82~0.86之间的遗传相似系数占全部数据的51.2%。在遗传相似系数为0.83的阈值处可将参试材料分为三大类群。构建了35份具有品种特异性的绿豆SSR指纹图谱及分子身份证,具有唯一性,可用于绿豆品种鉴定。结论:黑龙江地区绿豆品种遗传多样性水平不够丰富,SSR指纹图谱具可应用于绿豆品种真伪鉴定。
SSR分子标记技术,绿豆,遗传多样性分析,指纹图谱构建
绿豆(Vignaradiate(L.)Wilczek)是我国重要的粮食作物,富含蛋白质、维生素等营养成分,同时还含有黄酮类、生物碱类等活性成分,具有清热解毒、名目降压等功效,是一种深受欢迎的药食同源作物[1-2]。我国绿豆栽培已有2000多年历史,产量及种植面积均居世界前列,主产区主要集中在黄淮流域及东北地区,其中内蒙古、吉林、安徽、黑龙江等地区种植较多。然而绿豆一直被视为小杂粮作物,研究基础较薄弱,且在长期生产种植过程中优良亲本的频繁交流导致遗传基础狭窄,品种间相似度较高。随着人们对绿豆需求量的不断增加,假冒伪劣产品层出不穷,使得绿豆品种鉴定、原产地保护及溯源研究面临严峻挑战[3]。
随着分子生物学的快速发展,SSR分子标记技术在农作物遗传多样性分析及指纹图谱构建方面得到了长足的发展。Sonkar[4]等利用4对SSR引物对36个水稻品种进行遗传多样性分析,将参试品种分为两大主要类群,明确了各品种间的遗传关系;Molla[5]等利用5对SSR引物对42份孟加拉绿豆品种进行分析,证明SSR技术可为绿豆品种鉴定、遗传多样性分析提供理论基础;段艳凤[6]等利用10对SSR引物构建了88个通过审定的马铃薯品种的指纹图谱,具有唯一性,可将参试品种完全区分。Kassahun[7]等利用39对SSR引物构建了12份高粱品种的指纹图谱;胡晓辉[8]等利用50对SSR引物构建了山东省46份审定花生品种的指纹图谱,为花生种质资源管理及育种实践奠定理论基础;薛建峰[9]等利用171对SSR引物构建了国内外31份具有品种特异性的蓖麻指纹图谱,可用于品种区分鉴定。
黑龙江省是我国绿豆主产区之一,但目前关于该地区绿豆SSR遗传多样性分析及指纹图谱构建的研究鲜有报道。基于此,本研究利用14对多态性丰富的SSR引物对黑龙江地区35份绿豆品种样品进行遗传多样性分析并构建了具有品种特异性的指纹图谱,旨在初步明确黑龙江绿豆种质的遗传分化程度,建立一种高效、快速的绿豆品种鉴定方法,为绿豆新基因挖掘、育种创新及品种鉴定、溯源管理奠定基础。
1 材料与方法
1.1材料与仪器
本研究选取黑龙江地区35个绿豆品种为材料,均取自中国农业科学院作物科学研究所 详情见表1;选取NCBI数据库(http://www.ncbi.nlm.nih. gov/)和中国农业科学院作物科学院研究所提供的100对SSR引物进行初筛。普通引物由上海生工生物工程有限公司合成;荧光引物 由美国ABI公司合成。植物基因组DNA提取试剂盒、Taq酶、dNTPs、Buffer、Mg2+等 均购于北京天根生物技术有限公司。
ABI Prism 3730XL型基因分析仪 上海杰李生物技术有限公司;Applied Biosystems® PCR基因扩增仪 北京昊诺斯科技有限公司;德国Retsch(莱驰)MM400冷冻混合研磨仪 弗尔德(上海)仪器设备有限公司;KL05R高速冷冻离心机 湖南凯达科学仪器有限公司;α1900S紫外可见分光光度计 上海谱元仪器有限公司;Alpha 凝胶成像系统 美国ProteinSimple公司;SW-CJ-1D超净工作台 上海皓庄仪器有限公司;LDZX-40B立式自动电热压力蒸汽灭菌锅 河北蓝梦生物医药科技有限公司。
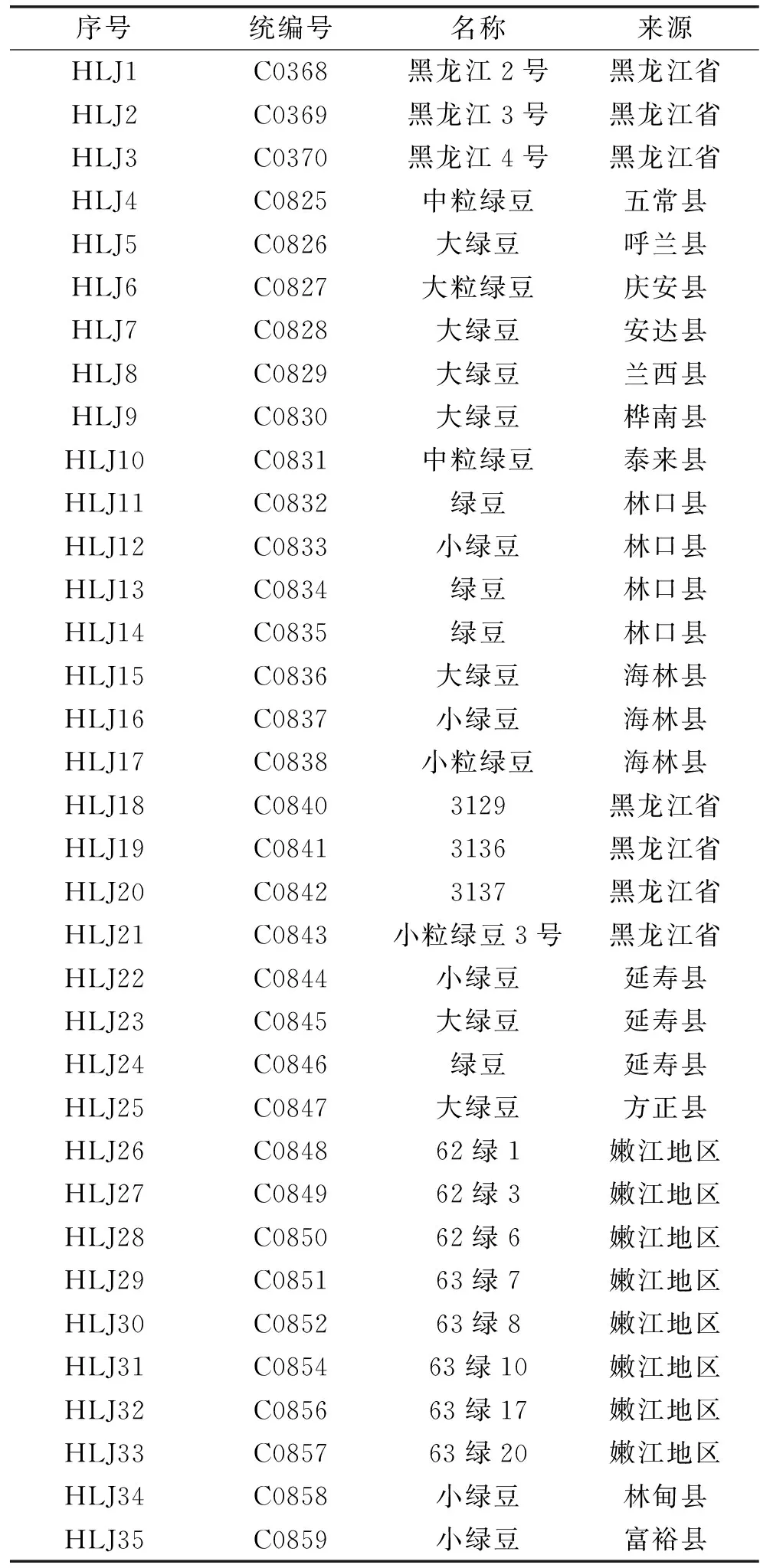
表1 参试绿豆材料Table 1 Origin of mungbean resources
1.2实验方法
1.2.1 绿豆基因组DNA提取及检测 每个样品取室温下(22~25 ℃)种植15 d左右的新鲜嫩叶,液氮环境下在球磨仪中打碎,采用新型植物基因组提取试剂盒(DP320)提取DNA,利用1%琼脂糖凝胶电泳进行检测,用紫外分光光度计测定浓度,将提取得到的DNA母液稀释成25 ng/μL于4 ℃环境下保存备用。
1.2.2 SSR-PCR扩增及引物初筛 SSR-PCR总反应体系为20 μL,包括ddH2O 7.2 μL,MIX 10 μL,引物0.6 μL,DNA模板2 μL,Taq酶0.2 μL。SSR-PCR扩增程序:94 ℃预变性5 min;94 ℃变性30 s,54 ℃复性35 s,72 ℃延伸40 s,共35个循环;最终72 ℃延伸3 min。后于6%变性聚丙烯酰胺凝胶进行垂直电泳分析,银染后观察。随机挑选10份绿豆为模板,对100对引物进行初筛,选取条带稳定、多态性丰富的引物用于大批量分析研究。
1.2.3 SSR荧光标记毛细管电泳检测及数据处理 用超纯水稀释荧光标记PCR产物以确定进样浓度,纯化稀释后的PCR产物去除其中的蛋白质、盐分等杂质,将甲酰胺与分子量内标按100∶1体积比混匀,取15 μL加入上样板中,再加入1 μL稀释10倍的PCR产物。最后使用3730XL测序仪进行毛细管电泳,利用Genemarker软件分析原始数据,根据目标峰位置与内标LIZ500进行比较,读出每个样品在各个等位基因位点的片段大小。
1.2.4 数据处理 根据分析结果,利用POPGENE[10]软件计算有效等位基因数(Ne)、期望杂合度(He)、观察杂合度(Ho)、多态性信息含量(PIC)等遗传参数;基于遗传相似系数,采用UPGMA法[11]构建参试个体的聚类图进行亲缘关系分析。
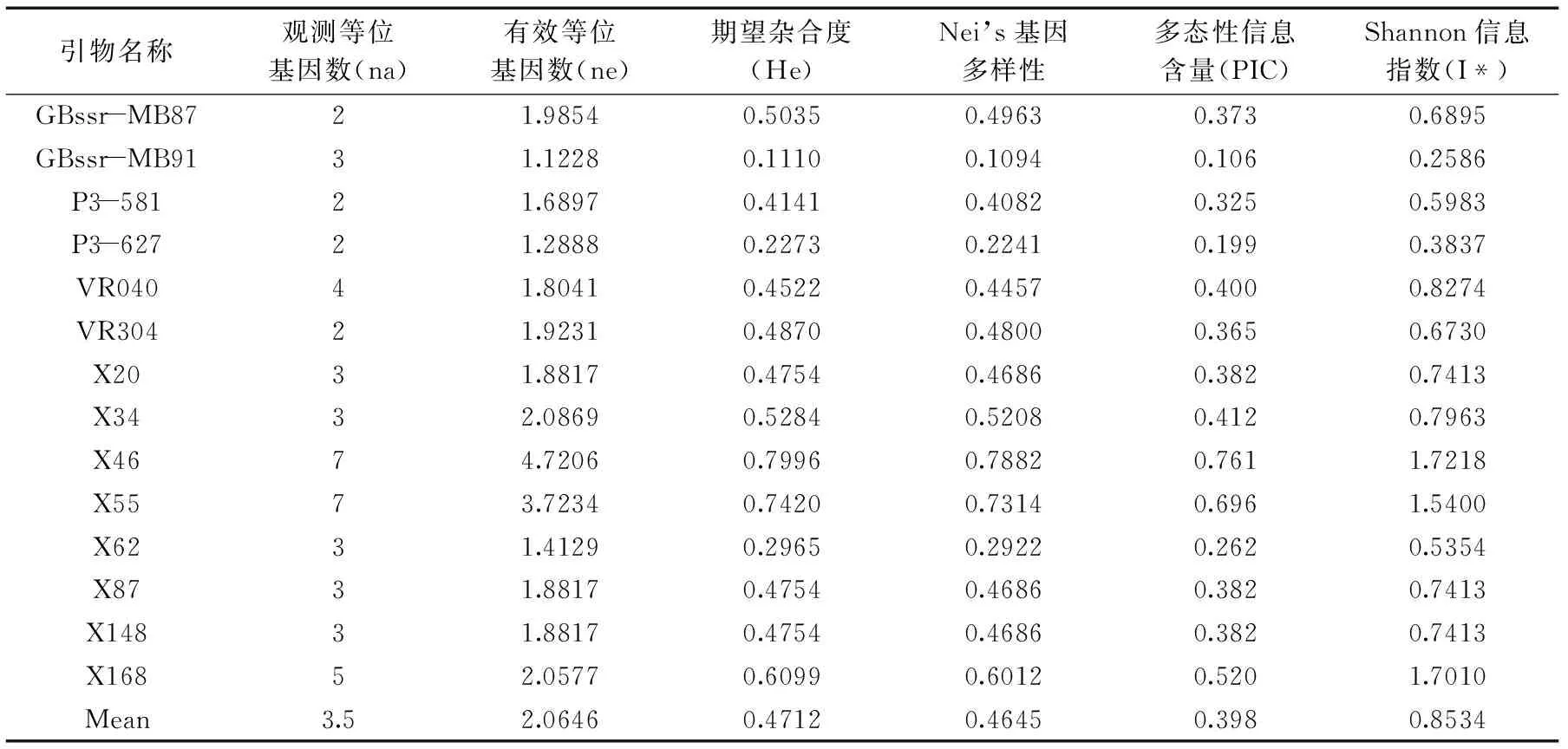
表2 基于14个SSR分子标记的黑龙江地区绿豆品种多态性信息Table 2 Genetic diversity of mungbean from Heilongjiang province based on 14 SSRs
2 结果与分析
2.1SSR引物的多态性分析
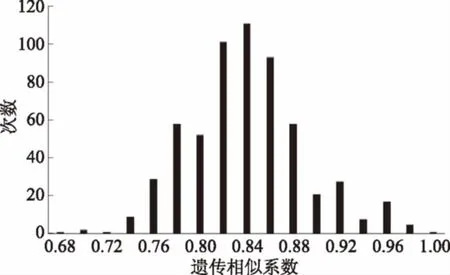
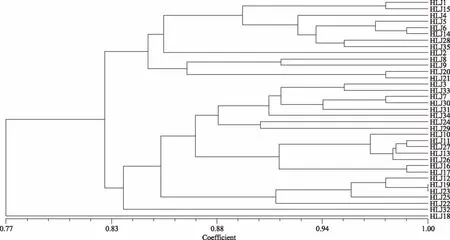
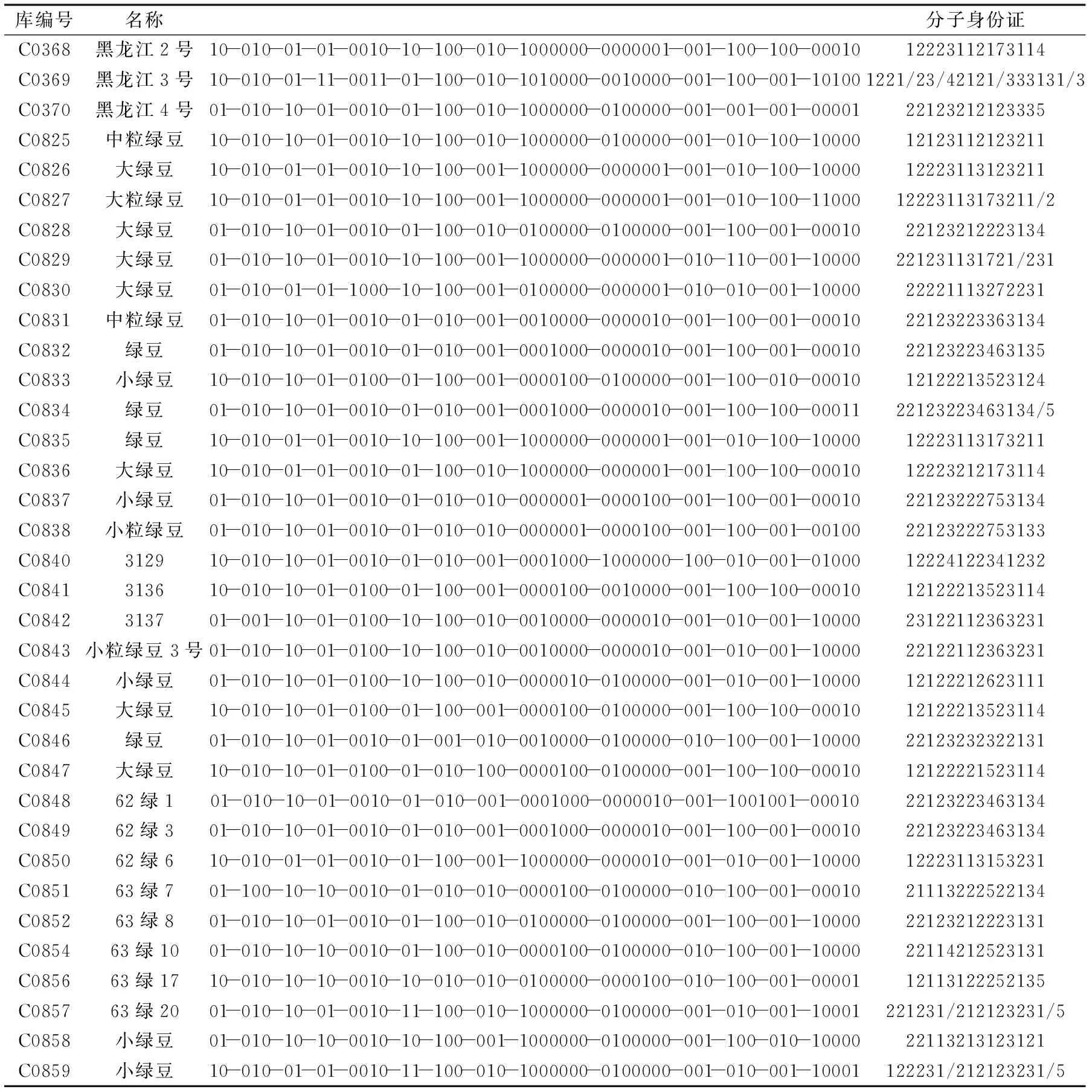
对100对绿豆SSR引物进行初筛,最终选取14对多态性丰富、条带稳定单一的核心引物,用于全部参试样品的遗传多样性分析及指纹图谱构建。部分引物筛选结果如图1所示。14对SSR引物共扩增出49个等位基因,每对引物扩增出2~7个不等,平均为3.5个;有效等位基因数变幅介于1.1228~4.7206之间,平均为2.0646;期望杂合度变幅介于0.1110~0.7996之间,平均为0.4712;Nei’s基因多样性指数变幅介于0.1094~0.7882之间,平均为0.4645。多态性信息含量PIC值变幅介于0.106~0.761之间,平均为0.398。其中引物X46、X55、X168为高度多态性位点(PIC>0.5),多态性丰富,GBssr-MB91、P3-627属低度多态性位点(PIC<0.25),检测能力较差,其余9对引物均为中度多态性位点(0.25 图1 部分引物筛选结果Fig.1 The amplified results of some primer selection 2.2遗传多样性分析 依据14对SSR引物检测到的49个等位基因,利用NTSYSpc2.0统计分析软件按Nei’s的方法计算,共获得遗传相似系数595个,变幅介于0.6966~1.0000之间,平均为0.894。其中小绿豆(C0858)和3129(C0840)之间的遗传相似系数最小(0.6966),说明二者遗传相似度较低,亲缘关系较远,而3136(C0841)和大绿豆(C0845)之间的遗传相似系数最大,为1.0000,14对核心引物并没有将其区分开,表明其亲缘关系较近,也可能是“同种异名”的情况。如图2所示,以0.02为组距对595个遗传相似系数进行次数分布分析,35份参试绿豆品种的遗传相似系数近似呈正态分布,主要集中在0.82~0.86之间,共分布了305个,占全部数据的51.26%。表明黑龙江地区绿豆资源遗传相似度较高,没有相互间遗传差异较大的品种。 图2 遗传相似系数的次数分布Fig.2 The distribution of genetic similarity 图3 黑龙江地区绿豆资源个体UPGMA聚类图Fig.3 UPGMA dendrogram of mungbean individuals from Heilongjiang province 2.3聚类分析 通过UPGMA法利用遗传相似系数对参试品种进行聚类分析,在遗传相似系数为0.83的阈值处可将参试个体分为三个组群。第一大类群包括黑龙江2号(C0368)、大绿豆(C0836)等10个品种;第二大类群包括黑龙江4号(C0370)、63绿20(C0857)等24个品种,3129(C0840)单独聚为一类,为第三大类群,说明该品种与其他品种间的遗传距离较大,亲缘关系较远。进一步分析,在遗传相似系数为0.88的阈值处,可将第二大类群分为三个亚类。第一亚群的遗传多样性水平相对较高,品种间亲缘关系相对较远,而第二、三亚群品种间遗传相似程度较高,特别是第三亚群中3136(C0841)和大绿豆(C0845),二者在遗传相似系数为1.0的水平上聚为一类,说明它们的遗传背景相同,但不排除“同种异名”的情况。虽然黑龙江地区绿豆品种可明显的被分为三大类群,但参试材料间的遗传相似系数大部分都在0.80以上,说明该地区绿豆品种相似度较高,在育种过程中亲本材料间的遗传背景比较窄。 2.4SSR指纹图谱及分子身份证构建 整理14对引物对黑龙江地区35份绿豆材料扩增得到的电泳峰图,在同一峰值处有带记为1,无带记为0,构建参试绿豆品种在14个微卫星标记上的DNA指纹图谱数据库[12]。同时,按表2引物顺序,对每对引物在35份参试绿豆品种中扩增出的等位基因按片段大小升序排列,依次编号。将每个样品在14对引物上的扩增条带数据串联起来,即得到由至少14位数字(有杂合带时则多于14位)组成的分子身份证。如黑龙江2号(C0368)的分子身份证为12223112173114,第一个数字是“1”,即黑龙江2号(C0368)在1号引物GBssr-MB87上扩增出在片段排列中排位第1的片段,其余13位依此类推。黑龙江地区绿豆品种SSR指纹数据及分子身份证详情见表3。 表3 黑龙江地区绿豆品种SSR指纹数据及分子身份证Table 3 Fingerprint date and molecular identification of 35 mungbean varieties at 14 SSR loci 分子生物学的不断发展为作物遗传多样性分析及指纹图谱构建提供了新的途径,尤其是SSR标记技术因其多态性丰富、稳定性强、操作简便等特点更加适用于遗传分析研究[13-15]。以往传统的SSR标记技术都是利用聚丙烯酰胺凝胶电泳进行检测,耗时费力,效率较低,检测结果易受环境和操作影响,且银染显影后无法确定片段大小,因此在不同批次分析中易产生误差,降低检测的精确性[16-17]。而SSR荧光标记技术以DNA测序仪为平台,利用荧光染料标记SSR扩增产物,可精确检测扩增产物的片段长度,具有灵敏度高、检测结果准确、可实现自动化等特点[19]。许鲲[19]、王瑞[20]、程本义[21]等分别利用SSR荧光标记毛细管检测技术对油菜、高粱及稻米进行遗传多样性分析及SSR指纹图谱构建,均得出上述结论。本研究基于SSR荧光标记技术对黑龙江地区绿豆品种进行检测,直接读取片段大小,研究结果更加客观可靠。 本研究利用SSR荧光标记技术,以14对SSR引物对黑龙江地区35份绿豆品种进行分析,共检测到49个等位基因,每对引物检测到2~7个,平均为3.5个;多态性信息含量变幅介于0.106~0.761之间,平均为0.398,这与刘岩[22]等利用40对引物分析全国272份绿豆资源得到平均等位基因数为3.1、平均PIC值为0.35的结果基本一致,但与其他豇豆属作物研究相比结果略低,宫慧慧[23]等利用18对SSR引物对国内外80份小豆品种进行分析,每对引物检测到的等位基因数为3~13个,平均为5.2个,PIC值介于0.23~0.83之间,平均为0.64。这一方面可能是由于绿豆基因组结构决定其多态性水平较低,另一方面与研究者所用参试材料的遗传背景、数量及对选用引物检测到的位点数目及共显性位点有效性等因素有密切联系。同时,本研究选用的引物中X45和X55的PIC值(0.761,0.696)和等位基因数(7,7)均高于前人研究,多态性较丰富,可作为绿豆资源研究的骨干引物。 遗传多样性分析发现,黑龙江地区绿豆品种的遗传相似系数介于0.6966~1.0000之间,平均为0.894,大部分集中在0.82~0.86之间,表明参试品种间遗传相似度较高,亲缘关系较近。这可能是因为本研究材料均取自黑龙江地区,在长期种植过程中优良亲本频繁利用,导致其在DNA水平上相似度较高,整体表现为该地区遗传背景比较狭窄。同时,品种3136(C0841)和大绿豆(C0845)在遗传相似系数为1.0的水平上聚为一类,考虑二者之间的亲缘关系较近,甚至可能是“同种异名”情况。聚类分析结果显示,在遗传相似系数为0.83处,可将参试品种分为3个类群,其中3129(C0840)单独聚为一类,与其他品种的遗传距离较大,考虑其可能来自黑龙江以外的其他地区。同时黑龙江2号(C0368)来源未知,根据聚类图分析其可能与大绿豆(C0836)亲缘关系较近,推测其可能与绿豆(C0836)一样均来自海林县或其附近地区。 传统的品种鉴定主要是依靠田间种植鉴定,耗时长,易受环境影响。因此,建立品种DNA指纹图谱,快速、准确鉴定品种的真实性及种子纯度具有重要意义。本研究基于SSR荧光标记技术构建了黑龙江地区35份绿豆品种的SSR指纹数据库及分子身份证,可将33个品种完全区分,数据具有特异性和唯一性,可为绿豆品种真伪鉴定提供依据。特别是分子身份证的构建,将指纹数据数字化,更加直观,方便。同时,分子身份证也可为品种亲缘关系判定提供参考。如小绿豆(C0837)和小粒绿豆(C0838)的分子身份证分别为“22123222753134”和“22123222753133”,相似度较高,而二者之间的遗传相似系数也较大,为0.988(文中未体现),表明其亲缘关系较近,遗传背景相似。 本研究基于SSR技术对黑龙江地区35个绿豆品种进行遗传多样性分析,从分子水平上揭示了黑龙江地区绿豆品种的遗传多样性水平。研究发现,该地区绿豆品种遗传多样性不够丰富,遗传背景狭窄,今后应在保护优质资源的同时着力引进国内外绿豆资源丰富地区的种质以拓展该地区遗传背景。同时,指纹图谱及分子身份证的构建为品种鉴定提供了新的思路和途径,为名优品种保护、品种真伪鉴定及原产地溯源研究提供重要依据。 [1]张会娟,胡志超,吕小莲,等. 我国绿豆加工利用概况与发展分析[J]. 江苏农业科学,2014,42(1):234-236. [2]田茜,张文兰,李群,等. 绿豆的品质特性及综合利用研究进展[J]. 中国农学通报,2016,32(9):77-82. [3]赵雪英,王宏民,李赫,等. 绿豆种质资源的ISSR遗传多样性分析[J]. 植物遗传资源学报,2015,16(6):1277-1282. [4]Sonkar S,Singh S K,Vennela P R,et al. Molecular marker based genetic diversity analysis in rice(Oryza sativa L.)using SSR markers[J]. 2016,9(1):45. [5]Md. Rezwan Molla. Genetic Diversity Analysis and DNA Fingerprinting of Mungbean(Vigna radiata L.)Genotypes Using SSR Markers[J]. Journal of Plant Sciences,2016,6(4):153-164. [6]段艳凤,刘杰,卞春松,等. 中国88个马铃薯审定品种SSR指纹图谱构建与遗传多样性分析[J]. 作物学报,2009,35(8):1451-1457. [7]Bantte K,Mogus Y. DNA Fingerprinting and Genetic Relationship among Ethiopian Sorghum[Sorghum bicolor(L.)Moench]Lines[J]. International Journal of Plant Breeding & Genetics,2016,10(1):38-44. [8]胡晓辉,毛瑞喜,苗华荣,等. 山东省46个花生品种SSR指纹图谱构建与遗传多样性分析[J]. 核农学报,2016(10):1925-1933. [9]薛建峰,谭美莲,严明芳,等 我国部分蓖麻品种遗传资源SSR分析及DNA指纹图谱[J]. 中国油料作物学报,2015(1):48-54. [10]F C. Yeh,R C. Yang,T Boyle. POPGENE,microsoft windows-based freeware for population genetic analysis release 1.31[M]. Edmonton:University of Alberta,1999. [11]Sun M,Sun M. Genetic diversity in three colonizing orchids with contrasting mating systems[J]. American Journal of Botany,1997,84(2):224-232. DOI:10.2307/2446084 [12]李小华,叶胜海,赵小燕,等. 浙江省46个粳稻新品种SSR指纹图谱构建及遗传多样性分析[J]. 浙江农业学报,2014(5):1158-1163. [13]S Martins,F Simões,D Mendonça,et al. Chloroplast SSR genetic diversity indicates a refuge for Corylus avellana in northern Portugal[J]. Genetic Resources and Crop Evolution,2013,60(4):1289-1295. [14]Smit R,Toit E S D,Vorster B J. RAPD and SSR genetic diversity analysis of Moringa oleifera[J]. South African Journal of Botany,2013,86(3):182. [15]Vincent Njung’ea,Santosh Deshpandeb,Moses Siambia,et al. SSR genetic diversity assessment of popular pigeonpea varieties in Malawi reveals unique fingerprints[J]. Electronic Journal of Biotechnology,2016,21:65-71. [16]Chandra A,Grisham M P,Pan Y B. Allelic divergence and cultivar-specific SSR alleles revealed by capillary electrophoresis using fluorescence-labeled SSR markers in sugarcane[J]. Genome,2014,57(6):363. [17]Gao Y,Tian L M,Liu F Z,et al. Using the SSR Fluorescent Labeling to Establish SSR Fingerprints for 92 Cultivars in Pyrus[J]. Acta Horticulturae Sinica,2012. [18]李锐,白建荣,张丛卓,等.利用SSR荧光标记分析山西糯玉米农家种的遗传多样性[J].山西农业科学,2016,44(10):1433-1436. [19]许鲲,李锋,吴金锋,等.SSR荧光标记毛细管电泳法与国家冬油菜区试指纹鉴定平台的构建[J].中国油料作物学报,2014,36(2):150-159. [20]王瑞,张福耀,程庆军,等. 利用SSR荧光标记构建20个高粱品种指纹图谱[J]. 作物学报,2015,41(4):658-665. [21]程本义,夏俊辉,龚俊义,等. SSR荧光标记毛细管电泳检测法在水稻DNA指纹鉴定中的应用[J]. 中国水稻科学,2011,25(6):672-676. [22]刘岩,程须珍,王丽侠,等. 基于SSR标记的中国绿豆种质资源遗传多样性研究[J]. 中国农业科学,2013,7(20):459-463. [23]宫慧慧,谢华,马荣才,等. 利用SSR分析小豆种质遗传多样性[J]. 农业生物技术学报,2008,16(5):872-880. GeneticdiversityanalysisandDNAfingerprintsconstructionbasedonfluorescentlabeledSSRMarkersformungbeanvarieties(VignaradiateL.)fromheilongjiangprovince ZHAOYa-nan1,WANGYing1,2,ZHANGDong-jie1,* (1.College of Food Science,Heilongjiang Bayi Agricultural University,Daqing 163319,China;2.National Coarse Cereals Engineering Research Center,Daqing 163319,China) Objective:The project of this study was to analysis the genetic diversity and establish SSR fingerprinting database of mungbean varieties from Heilongjiang province to lay the foundation for breeding and variety identification of mung bean.Methods:The 35 mungbean varieties were used as materials to analyze the genetic diversity and construct DNA fingerprint maps by 14 SSRs.Results:By using 14 pairs of SSR primers,49 alleles were detected out from 35 mungbean varieties,and each pair of primers detected out 2~7 alleles with an average of 3.5.The polymorphic information content values(PIC)ranged from 0.106 to 0.761,with a mean of 0.298. The genetic diversity coefficient among 35 mungbean varieties varied from 0.6966~1.0000 with an average of 0.83,and the genetic similarity coefficient of 51.2% tested materials ranged from 0.82 to 0.86. UPGMA clustering results showed that 35 mungbean varieties were clustered into 3 groups at 0.83 of genetic similarity coefficient. Establishing the SSR fingerprinting database and molecular identification of 35 mungbean cultivars from Heilongjiang province,providing reference for variety identification.Conclusion:The genetic background of mung bean varieties in Heilongjiang was narrow,and the level of genetic diversity was low.SSR fingerprints and molecular identification were constructed using SSR marker technique,which was of great significance for genetic authenticity,identity validation,traceability management and protection of origin of mungbean varieties. SSR Markers;mungbean varieties;genetic diversity analysis;fingerprints construction TS201.3 A 1002-0306(2017)19-0148-06 10.13386/j.issn1002-0306.2017.19.028 2017-03-29 赵雅楠(1993-),女,硕士研究生,研究方向:农产品加工及贮藏工程,E-mail:zyn658@163.com。 *通讯作者:张东杰(1966-),男,博士,教授,研究方向:农产品加工及贮藏工程,E-mail:byndzdj@126.com。 黑龙江省应用技术与开发重大项目(GA14B104);大庆科技局创新项目《杂粮及其深加工分析测试技术平台建设》(sjh-2013-65);黑龙江省研究生创新科研项目资金(YJSCX2017-Y50)。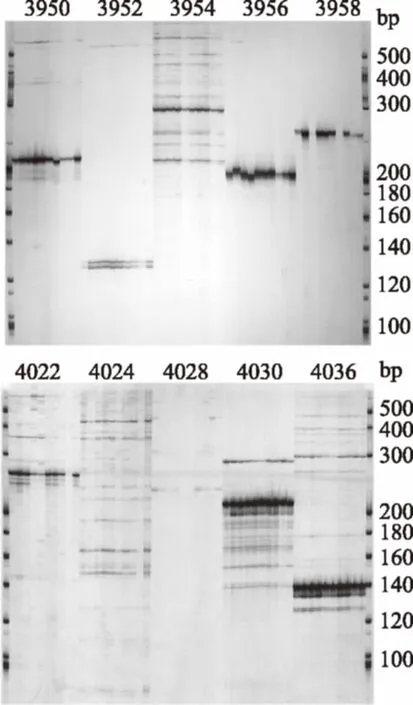
3 讨论
4 结论
