海洋琼胶降解菌FG15基因组从头测序分析
2017-10-16曾鸿俏张力雄刘明明方再光
曾鸿俏,张力雄,刘明明,方再光
(海南大学 热带生物资源教育部重点实验室/海南省热带水生生物技术重点实验室,海南 海口 570228)
海洋琼胶降解菌FG15基因组从头测序分析
曾鸿俏,张力雄,刘明明,方再光
(海南大学 热带生物资源教育部重点实验室/海南省热带水生生物技术重点实验室,海南 海口 570228)
为充分了解琼胶降解菌FG15的基因功能和代谢途径,本研究使用SOAP de novo 2.04对Illumina Hiseq2000平台测序产生的基因片段进行了拼接和组装,同时利用Glimmer 3.02来预测基因的开放性阅读框,并采用蛋白质直系同源基因簇(COG)、基因本体数据库(GO)以及京都基因和基因组百科全书(KEGG)的数据来预测其基因功能,获得了代谢途径.结果表明:FG15的基因组大小为5.10 Mb,G+C含量为44.58%,共有38条Scaffolds,4 922个开放性阅读框,82个tRNA,2个rRNA; 通过COG分析可将菌株FG15注释到22种COG功能类型,主要包括细胞代谢、细胞信号转导等;利用GO注释可将FG15注释到3大类39个GO功能亚类上;KEGG分析能将其定位到154个代谢通路中,包括物质代谢、次生代谢产物的生物合成等.次生代谢产物的合成代谢途径精确显示,FG15能合成青霉素和头孢菌素,且其与抗生素抗性实验的结果一致.研究结果为FG15功能基因组学的研究和相关次级代谢产物的生物合成途径以及异源表达的研究提供了理论基础.
琼胶降解菌;琼胶寡糖;基因组从头测序;基因功能;代谢途径
能够降解琼脂的微生物主要来自于海洋,河流、土壤和污水中也有少量分布.海洋中的红藻,如石花菜(Gelidiumamansii)和江蓠(Gracilaria)等植物的表面通常能分离到琼胶降解菌(Agarivoranssp.).琼胶酶是琼胶降解菌分泌的水解酶,它主要可分为α和β两类,α-琼胶酶产生以3,6-内醚-α-L半乳糖为还原性末端的琼寡糖;β-琼胶酶则产生以β-D-半乳糖为还原性末端的新琼寡糖[1].琼胶寡糖除了拥有普通寡糖的性质以外,它还具有抗癌、抗氧化、抗炎和抗淀粉老化等特性,因而具有极大的开发潜力[2].
制备琼胶寡糖的方法主要有化学法和生物酶法,但由于化学降解法存在条件不易控制、产物不均一、环境污染严重、产物分析和回收难等缺点,因而限制了其在工业上的应用.而使用酶水解法来生产琼胶寡糖,其消耗的能量较少,反应条件亦易于控制,同时该法还具有高效性和专一性,能够克服化学降解法中存在的问题[3].因此,将生物催化技术应用于工业生产成为一种公认的绿色环保技术,且随着对琼胶酶的研究越来越深入,用酶水解法来制备琼胶寡糖已经发展为一种新的方向.
DNA测序经历了三代技术的发展:第一代测序技术是Sanger测序法,第二代测序技术是高通量测序,第三代测序技术则是单分子测序.全基因组de novo测序,也称为基因组从头测序,属于第二代测序技术,它是先在没有任何参照序列的情况下进行基因组测序,然后再通过生物信息学拼接和组装已经获得的序列,进而获得其基因图谱.从头测序有助于充分了解物种的分子进化、基因组成以及基因调控.
尹群健等[4]从海南岛的热带海洋环境中分离和纯化出1株革兰氏阴性琼胶酶高产菌,并对其进行了分子鉴定及酶学性质分析,结果表明,FG15对氨苄青霉素、羧苄青霉素和头孢菌素均不敏感;通过16S rDNA序列分析和BLAST同源性比对发现,FG15菌株的16S rDNA序列与船蛆杆菌(Teredinibacterturnerae)和噬琼胶菌(Agarivoranssp.)对应序列的同源性最高;FG15与噬琼胶菌属的亲缘关系最近;初步鉴定FG15为噬琼胶菌属(Agarivoranssp.); FG15所产的琼胶酶的最适温度为36 ℃;最适pH=7.5;产酶主要为胞外酶.本研究通过全基因组从头测序技术,分析了海洋琼胶降解菌FG15的基因组分,并通过与蛋白质直系同源基因簇(Cluster of Orthologous Groups of proteins, COG)、基因本体数据库(Gene Ontology, GO)以及京都基因和基因组百科全书(Kyoto Encyclopedia of Genes and Genomes, KEGG)的数据进行比对,预测了其基因功能,本研究为通过基因改造来获得琼胶寡糖高产菌株和抗生素工程菌提供了理论依据.
1 材料与方法
1.1菌株以海南陵水热带海洋环境分离和鉴定的琼胶降解菌(Agarivoranssp.)FG15[4]为实验对象.
1.2培养基X改良培养基:0.5%的蛋白胨,0.1%的酵母粉,0.1% 的KH2PO4,0.05%的KCl,0.05%的 MgSO4·7H2O,0.7%~2.0%的琼胶,pH=7.2.
1.3全基因组测序在海南陵水沿海采样,于无菌条件下将所采集到的样品研磨,用无菌海水稀释成梯度菌悬液,分别涂布到X固体培养基上,然后在28 ℃恒温培养3~5 d,待平板长出单菌落,挑FG15菌株的单菌落接入X液体培养基中,并于28 ℃和150 r·min-1下摇床过夜培养,在其质量浓度大于20 ng·uL-1且为纯培养后,提取纯基因组DNA,待达到深圳华大基因研究院的送样标准后,将样品寄送到华大基因研究院,并由该院进行全基因组的测序.
1.3.1 序列组装在通过Illumina Hiseq2000平台测序后,使用SOAP denovo 2.04短序列组装软件进行组装.于reads(高通量测序平台产生的序列)之间的Overlap区,通过reads拼接来获得Contigs,构建454 Paired-end库以确定来自同一转录本的不同Contig的先后顺序,然后利用先后顺序已知的Contigs来共同组成Scaffold.序列组装的目的是为了去除接头、引物及低质量的数据,并通过优化参数Kmer值来获得最好的组装结果.
1.3.2 基因预测基因的ORFs是利用Glimmer 3.02软件来预测;rRNA是利用RNAmmer 1.2(http://www.cbs.dtu.dk/services/RNAmmer/)来预测;tRNA是利用tRNAscan-SE 1.23(http://gtrnadb.ucsc.edu/)来预测;sRNA(small RibonucleicAcid)小核糖核酸是通过与Rfam10.1数据库进行比对来寻找;串联重复序列则是利用TRF(Tandem Repeat Finder)4.04(http://rfam.sanger.ac.uk/)来预测;微卫星以及小卫星序列却是根据重复单元的长度及数目来进行筛选.
1.3.3 基因组功能注释通过blastx,将基因序列与NR数据库(Non-redundant GenBank)、京都基因和基因组百科全书(Kyoto Encyclopedia of Genes and Genomes, KEGG)、SwissProt、蛋白质直系同源基因簇(Cluster of Orthologous Groups of Proteins, COG)等的数据进行比对,可得到与给定序列具有高度相似性的蛋白,从而得到该基因的蛋白功能注释信息.根据NR数据库(Non-redundant GenBank)的注释信息,可通过预测得到基因本体数据库(Gene Ontology, GO) 的注释信息,再通过网络基因本体论注解绘图工具(Web Gene Ontology Annotation Plot, WEGO)可对其进行详细的功能分类,然后根据蛋白质直系同源基因簇(Cluster of Orthologous Groups of Proteins, COG)数据库的注释信息可预测其功能分类,最后根据KEGG注释信息可进一步得到基因的代谢途径.
2 结果与分析
2.1基因组装结果分析于软件SOAPdenovo 2.04测序组装后,其结果如表1所示,FG15基因组大小为5 103 kb;共有38个scaffolds,最大长度为1 841 kb,最小长度为506 kb.将所有拼接得到的Contigs按照从大到小的顺序排序,其累加片段长度达到所有Contigs总长度的50%时,其所对应的Contig长度为N50值;同理,将所有拼接得到的Contigs按照从大到小的顺序排序,其累加片段长度达到所有Contigs总长度的90%时,其所对应的Contig长度为N90值.N50值和N90值是评价序列组装好坏的一个指标,一般来说,N50值越大,说明组装得越好,大片段的比例就越高[5].本研究的结果显示:N50大小为742 kb,N90则为116 kb.
在基因组注释中要判断组装是否已达到了要求,除了可用一些统计指标来表述组装的完整性和连续性之外,最重要的就是N50值.但对于基因预测而言,N50达到基因的平均长度是一个合理的目标,它预示基因中约有50%的基因有望包括在单个scaffold或者Contig中;而对于N90而言,它预示基因中约有90%的基因有望包括在单个scaffold或者Contig中,这样会得到一个完整的基因序列[6].
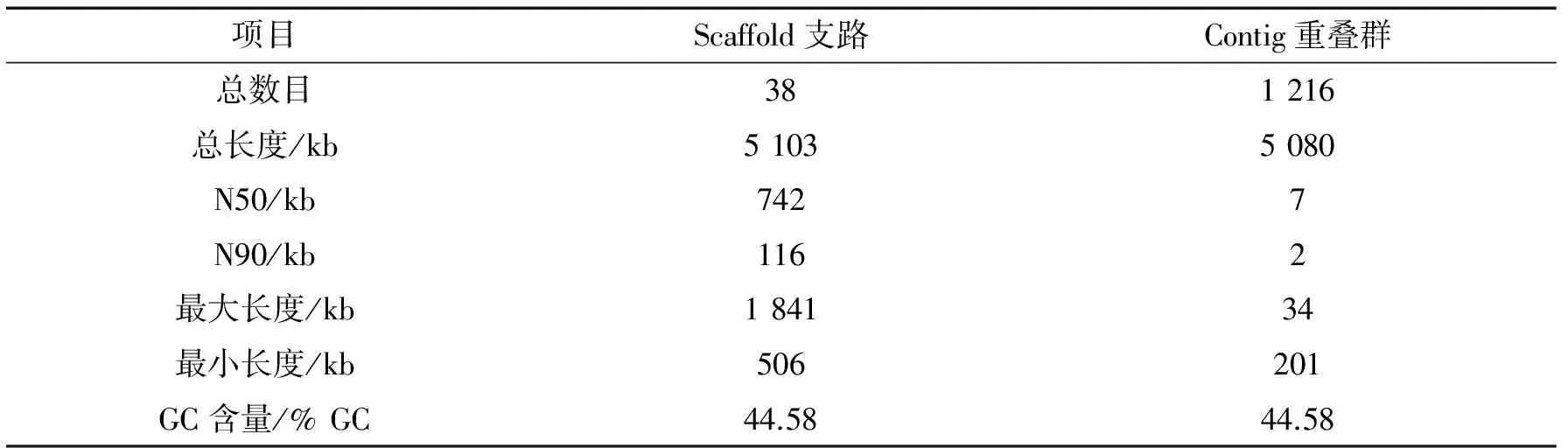
表1 FG15的组装结果
2.2基因预测结果的分析通过基因预测、重复序列预测、非编码RNA预测等方法可获取测序菌株的基因组组成情况.通过基因组组分的分析发现,FG15的基因组大小为5.10 Mb,G+C含量为44.58%,含有4 922个基因,其总长度为4.36 Mb,平均长度为885 bp,占基因组全长的85.35%.对于96个串联重复序列,其总长为22.55 kb,占基因组全长的0.44%,有37个小卫星序列,16个微卫星序列,82个tRNA,2个rRNA以及13个sRNA.基因预测结果的分析表明:FG15具有功能基因组学研究的理论价值.

基因匹配数量:A:RNA的加工与修改(1条);B:染色质结构与动力学(1条);C:能量产生与转化(230条);D:细胞周期调控、细胞分裂、染色体分配(38条);E:氨基酸转运与代谢(353条);F:核苷酸转运与代谢(81条);G:糖类运输与代谢(186条);H:辅酶转运与代谢(153条);I:脂质转运与代谢(114条);J:翻译、核糖体结构与生物转化(186条);K:转录作用(257条);L:复制、重组与修复(143条);M:细胞壁/细胞膜/细胞被膜源(184条);N:细胞运动(127条);O:翻译后修饰、蛋白质转换、分子伴侣(147条);P:无机离子转运与代谢(204条);Q:次生代谢产物生物合成、运输与分解代谢(61条);R:通用功能预测(388条);S:未知功能(274条);T:信号转导机制(190条);U:细胞内运输、分泌物与膜泡运输(110条);V:防卫机制(59条)
图1 FG15的COG功能分类
2.3功能注释结果的分析
2.3.1 COG功能分类FG15的COG蛋白的功能注释信息结果如图1,由图1可知,FG15的功能注释结果可分为22类,共有3 487条基因,其中,FG15最多的功能为通用功能(388条),占11.12%;氨基酸转移与代谢功能(353条),占10.12%;糖类的运输与代谢功能(186条),占5.33%;能量产生与转化功能(230条),占6.59%;信号转导机制功能(190条),占5.45%.在FG15的功能归类中所占比例较少的有:RNA加工与修改功能(1条),占0.03%;染色质结构与动力学功能(1条),占0.03%;细胞周期调控、细胞分裂和染色体分配功能(38条),占1.09%;核苷酸转运与代谢功能(81条),占2.32%;次生代谢产物的生物合成、运输与分解代谢功能(61条),占1.75%;防卫机制功能(59条),占1.69%.
2.3.2 GO功能注释GO的功能分类结果如图2所示,其主要包括生物过程、细胞组分和分子功能3大类.GO的功能分类结果显示,细胞过程和代谢过程在生物过程中十分活跃;细胞和细胞部位在细胞过程中占主导地位;整合和催化活性在分子功能中起重要作用;在FG15菌株的基因组中,与生物过程有关的基因为173 060条,与细胞组分有关的基因为3 008条,对应到分子功能的基因有3 776条.

图2 FG15 的GO 功能注释
2.3.3 KEGG代谢通路KEGG的注释信息结果如图3所示,由图3可知,参与FG15的KEGG代谢通路的基因有33类,共有4 101条基因,其中,参与膜运输代谢途径的基因最多,共有661条基因,占16.11%;其次为糖代谢,共有428条基因,占10.43%;再次为氨基酸代谢,共有408条基因,占9.94%.
代谢途径的分析结果如图4和图5所示,结果表明:基因组涵盖了糖类代谢、能量代谢、脂类代谢、核苷酸和氨基酸代谢、次生代谢产物的合成代谢等途径.糖代谢途径中都包含了糖酵解途径(见图4)和磷酸戊糖途径,此外,本研究还发现FG15可以利用果糖、甘露糖、半乳糖、淀粉、蔗糖,N-多聚糖,脂多糖等.在次生代谢产物的合成代谢途径中, FG15能合成青霉素、头孢菌素(见图5)、新生霉素、链霉素、安沙霉素类和四环素等.根据代谢途径的酶和基因的异同,在此用不同颜色来表示选中的不同代谢途径及酶基因的差异.
3 讨 论
本研究结果显示,FG15的基因组大小为5.10 Mb,G+C的含量为44.58%,共有38个scaffolds,1 216个contigs,37个小卫星序列,16个微卫星序列,82个tRNA,2个rRNA.嗜琼胶卵链菌(Catenovulumagarivorans)YM01T的 G+C含量和白色噬琼胶菌(Agarivoransalbus)MKT 106的G+C含量分别为44.8%[7],48%~50%[8],FG15的G+C含量与之相比比较接近.
在COG的注释信息中,FG15含有22种COG功能类型,主要包括细胞代谢、细胞信号转导等,其中,共有3406条预测基因有相应的COG功能注释.在对各功能类别的基因数目进行统计时发现,

基因匹配数量,从上至下依次为:神经系统(2);免疫系统(2);分泌系统(1);环境适应性(11);内分泌系统(17);消化系统(10);循环系统(3);外源性物质的降解和代谢(151);核苷酸代谢(138);多酮类化合物的代谢(74);其它氨基酸的代谢(67);辅酶因子的代谢(164);脂质代谢(173);多糖生物合成和代谢(92);酶家族代谢(91);能量代谢(217);糖代谢(428);其它次生物质的生物合成和代谢(22);氨基酸代谢(408);神经变性疾病(18);新陈代谢疾病(5);传染性疾病(53);免疫系统疾病(3);癌症(7);遗传信息-翻译(221);遗传信息-转录(138);复制和修复(288);折叠,排序和降解(110);信号分子相互作用(10);信号转导(223);膜运输代谢(661);细胞运动(276);细胞增殖和消亡(17);
图3 FG15的KEGG代谢通路分类图
其中基因数目较多的类别主要有通用功能预测、各种结构的生物转化、复制、重组和修复、翻译、转录、各种物质的转运与代谢、各种机制的发生等.这与刘君彦等[9]关于啤酒易感乳杆菌功能基因的研究有很大的相似之处.
本研究中,通过GO注释可将FG15基因组注释到3大类39个COG的功能亚类上,与生物过程有关的基因有173 060条,与细胞组分有关的基因有3 008条,对应到分子功能的基因有3 776条.在代谢产物合成途径和比较基因组学的研究方面,何晓峰等[10]关于小单孢菌DSM 803全基因组测序及分析的研究在基因数量、大小等代谢合成途径上有一定的差异,但是,在主要的基因结构和功能作用方面却又有相似之处,例如,在功能基因组学的研究中,研究均发现其具有很高的GC含量,且COG和GO的基因结构和功能分类都有相似之处,这为后续的功能基因组学研究以及生产利用提供了较为坚实的理论基础.在转录物生物学过程的功能类型中,细胞过程和代谢过程占绝大部分,其次是刺激反应和生物调节过程;而在转录物细胞组分的功能类型中,细胞和细胞分离所占的比例最高,其次是细胞器和生物膜,然而蛋白结合和催化活性却是转录物分子功能类型中的主体部分.
KEGG分析能将其定位到154个代谢通路中,包括物质代谢、次生代谢产物生物合成等,其中,参与糖代谢的基因有428条,而参与氨基酸代谢的基因有408条.
次级代谢产物是指微生物生长到一定阶段后才产生的十分复杂的化学结构,该生物无明显的生理功能或并非是微生物生长和繁殖所必需的物质[11].FG15次生代谢产物的合成代谢途径精确显示,其能合成青霉素和头孢菌素,并且在菌株的抗生素抗药性实验中已证实,FG15对氨苄青霉素、羧苄青霉素和头孢菌素都不敏感,所得结果与理论相符.

图4 FG15 的糖酵解途径

图5 FG15 青霉素和头孢菌素的生物合成通路
目前,在NCBI上公布的白色噬琼胶菌属,其一般只被鉴定到属,并未被鉴定到种,这极大地限制了白色噬琼胶菌属的功能基因组学的研究,究其原因,这可能是其基因组DNA有较高GC含量,从而致使GC高含量区域的DNA片段未能得到很好地扩增的缘故[12].通过构建PCR-free文库可以在一定程度上消除PCR扩增的偏爱性,但仅依靠一种测序技术仍然很难获得基因组的完成图.可喜的是近年来出现了三代测序技术,如单分子实时测序仪的出现,它将成为复杂基因组测序的理想平台[13],因为三代测序技术不再需要进行PCR扩增,并且其读长可达到20 kb以上,因此,二代和三代测序技术联合应用来共同解析物种全基因组序列将成为主流.
由于测序成本的原因,因此本研究只采用了二代测序技术来对FG15进行全基因组的从头测序,但随着未来测序成本的降低,今后还需通过补充三代测序数据,以便将FG15菌株的基因组序列拼接成完成图.
[1] Arnott S, Fulmer A, Seott W E, et al. The agarose double helix and its function in agarose gel structure [J]. Mol Biol, 1974,90(2): 269-284.
[2] 吴国汉.琼胶酶生产菌的筛选、鉴定及其产酶条件的优化[D].福建:集美大学,2011.
[3] 缪伏荣,李忠荣.琼胶的降解及其产物的开发应用[J]. 现代农业科技,2007,12(5):156-158.
[4] 尹群健,陈潇骁,杨宏胜,等.产琼胶酶菌株Agarivoranssp. FG15的筛选鉴定与酶学性质[J].中国酿造,2014,33(2):17-22.
[5] Morimoto N, Arita M, Suyama A, et al. Solid phase DNA solution to the Hamiltonian path problem[J]. Discrete Mathematics and Theoretical Computer Science, 1999,48(3): 193-206.
[6] 黄勇.基于高通量测序的微生物基因组学研究[D].北京:军事医学科学院,2013.
[7] 严书林.3株新菌的分类鉴定及新型耐热琼胶酶基因的克隆表达[D].青岛:中国海洋大学,2011.
[8] Midori K, Akira Y.Agarivoransalbusgen. nov. sp. nov.aγ-proteo bacterium isolated from marine animals[J]. International Journal of Systematic and Evolutionary Microbiology, 2004,54: 693-697.
[9] 刘君彦,李琳,李冰,等.De novo测序技术在啤酒易感乳杆菌全基因组研究中的应用[J].现代食品科技,2015,31(11):155-162.
[10] 何晓峰,杨慧林,张志斌,等.小单孢菌(Micromonosporarosaria)DSM 803 全基因组测序分析[J].基因组学与应用生物学,2016,35(8):2 055-2 063.
[11] 杨慧林,张志斌,颜日明,等.东乡野生稻内生放线菌Streptomycessp. PRh5的全基因组测序及序列分析[J].微生物学通报,2015,42 (4):801-809.
[12] 肖前程, 彭思露, 李尔汉, 等. 茂原链霉菌DSM 40847 全基因组测序及序列分析[J]. 基因组学与应用生物学, 2014,33(4): 729-736.
[13] 张得芳,马秋月,尹佟明,等.第三代测序技术及其应用[J].中国生物工程杂志,2013,33(5):125-131.
Abstract:In order to fully understand gene function and metabolic pathways of FG15, its genome wasdenovosequenced by Illumina Hiseq2000 sequencing system and assembled by SOAPdenovo2.04. Its open reading frames (ORFs), rRNA, and tRNA were predicted by Glimmer 3.02, RNAmmer 1.2, and tRNAscan-SE 1.23, respectively. The function of the genes and the metabolic pathways were annotated by Cluster of Orthologous Groups of proteins (COG), gene ontology (GO), and Kyoto Encyclopedia of Genes and Genomes (KEGG). The analysis results from Glimmer 3.02, RNAmmer 1.2, and tRNAscan-SE 1.23 showed that FG15 genome contain 5.10 Mb with a G+C content of 44.58%, a total of 38 scaffolds, 4 922 ORFs, 82 tRNA and 2 rRNA. COG analysis results showed that the genes can be assigned into 22 kinds of COG function, including cell metabolism, cell signal transduction; GO analysis results showed that the genes can be assigned into the 3 major categories, which include 39 GO function class; KEGG analysis results showed that the genes can be assigned into 154 metabolic pathways, including material metabolism, secondary metabolite biosynthesis. The synthesis and metabolism of secondary metabolites analysis results showed that FG15 can synthesize penicillin and cephalosporin, consistent with the results of antibiotic resistance test. Our findings can provide genome sequence information for future functional genomics, biosynthetic pathways and heterologous expression of secondary metabolites of FG15.
Keywords:Agar-degrading bacteria; agar oligosaccharide; genomedenovosequencing; gene function; metabolic pathway
GenomedenovoSequencingofAgar-degradingMarineStrainFG15
Zeng Hongqiao, Zhang Lixiong, Liu Mingming, Fang Zaiguang
(Key Laboratory of Tropical Biological Resources of Ministry of Education, Key Laboratory of Biotechnology of Tropical Aquatic Organisms of Hainan Province, Hainan University, Haikou 570228, China)
Q5
A DOl:10.15886/j.cnki.hdxbzkb.2017.0041
2017-03-20
国家自然科学基金项目(40666001);海南大学青年基金(qnjj1205);校县合作项目(02005001)
曾鸿俏(1990-),男,海南临高人,海南大学海洋学院2014级在读硕士研究生,E-mail:zhqcyy@qq.com
方再光(1975-),男,博士后,副教授,研究方向:海洋微生物功能基因及热带大型藻类培养技术的研究工作,E-mail:guangyan0508@163.com
1004-1729(2017)03-0260-08
