黄藤2个NAC基因的分子特征及其SSR分子标记开发*
2017-10-14孙化雨李利超赵韩生杨意宏王思宁高志民
孙化雨 李利超 赵韩生 杨意宏,2 王思宁 高志民
(1. 国际竹藤中心 国家林业局竹藤科学与技术重点开放实验室 北京 100102; 2. 河北农业大学园艺学院 保定 071001)
黄藤2个NAC基因的分子特征及其SSR分子标记开发*
孙化雨1李利超1赵韩生1杨意宏1,2王思宁1高志民1
(1. 国际竹藤中心 国家林业局竹藤科学与技术重点开放实验室 北京 100102; 2. 河北农业大学园艺学院 保定 071001)
【目的】 棕榈藤是重要的森林植物,纤鞭是其重要的攀援器官,也是重要的分类依据。研究黄藤NAC(NAM,ATAF和CUC)转录因子基因的分子特征并开发SSR分子标记,以期为棕榈藤的分子育种和辅助分类提供参考依据。【方法】 以黄藤为材料,借助转录组数据,采用同源克隆的方法从黄藤中分离NAC基因,采用生物信息学方法进行基因结构、蛋白性质与结构分析和SSR位点预测,采用实时定量PCR技术分析基因的组织表达特性,利用PAGE电泳和测序技术分析SSR分子标记在不同棕榈藤样品中的通用性和多态性。【结果】 从黄藤叶片中获得了2个NAC同源基因DjNAC3 (GenBank登录号: KU556738)和DjNAC4(GenBank登录号: KX579750),二者的开放阅读框长度分别为729 bp和1 326 bp,对应的基因组序列为850 bp和1 441 bp,均包含2个外显子和1个内含子。DjNAC3和DjNAC4编码的蛋白分别为242 aa和441 aa。蛋白结构分析表明,DjNAC3和DjNAC4具有典型的NAC转录因子结构特征,属于NAC家族的CUC亚家族,但二者之间的相似系数仅为23.6%,表明它们在黄藤生长发育过程中可能具有不同的功能。DjNAC3和DjNAC4在不同组织中的表达模式存在明显差异,DjNAC3在发育成熟纤鞭中的表达丰度最高,叶片中的表达丰度最低,而DjNAC4则在发育成熟的钩刺中表达丰度最高,而发育初期的钩刺中最低。在DjNAC3和DjNAC4的基因组序列中分别包含1个SSR位点,其中前者的SSR位点位于内含子区域,为(TA)6,后者的SSR位点位于第1个外显子区域,为(GCA)5。根据DjNAC3和DjNAC4中SSR位点旁侧序列设计引物,以黄藤和另外20个不同棕榈藤样品的基因组DNA为模板进行扩增,PAGE电泳结果分析表明,引物具有较高的通用性,且扩增产物具有多态性。6个样品的测序结果证实,用DjNAC3设计引物的扩增产物测序获得的SSR位点序列存在着一定的差异,既包括SSR类型变异、重复次数变化等多态性,又有SSR位点缺失的现象; 而根据DjNAC4设计引物扩增获得的SSR位点序列差异主要为重复次数的变化。【结论】 黄藤DjNAC3和DjNAC4基因的基因结构、表达模式、SSR位点等均存在明显的差异,这表明它们在黄藤生长发育中可能具有不同的功能,二者所包含的SSR分子标记具有通用性和多态性,可以作为分子标记应用于棕榈藤的辅助分类和分子辅助育种。
黄藤;NAC; 基因表达; SSR开发
Abstract: 【Objective】 Rattan is one of the important forest plants, cirri and flagella are important organs for its climbing habit, which are also the important basis for rattan classification. In order to provide a basis for molecular breeding and SSR-assisted classification of rattan, study on the molecular characteristics of NAC (NAM, ATAF and CUC) transcription factor genes inDaemonoropsjenkinsianaand the development of SSR markers was performed in this paper.【Method】 With the aid of transcriptome data, the sequences ofNAChomologue genes inD.jenkinsianawere isolated by PCR method. The analyses of gene structure, protein properties and structure as well as the prediction of SSR locus inNACgenes were conducted using bioinformatics method. The tissue specific expression ofNACgenes in different tissues were analyzed using real-time quantitative PCR (qPCR). To evaluate the universality and polymorphism of the developed SSR markers, PAGE electrophoresis and sequencing analyses were used on the basis of products amplified with different rattan samples.【Result】 Two homologous genes,DjNAC3 (GenBank No. KU556738) andDjNAC4 (GenBank No. KX579750), were obtained from leaves ofD.jenkinsiana, of which open reading frame (ORF) were 729 bp and 1 326 bp, respectively. The genomic sequence corresponding to the ORFs ofDjNAC3 andDjNAC4 were 850 bp and 1 441 bp, which all contained two exons and one intron. The proteins encoded byDjNAC3 andDjNAC4 were 242 aa and 441 aa respectively. Protein structure analysis showed that DjNAC3 and DjNAC4 had the typical structural features of NAC transcription factors, which were belonged to the CUC subfamily of NAC family. However, the similarity coefficient between DjNAC3 and DjNAC4 was only 23.6%, indicating that they might have different functions in the growth process ofD.jenkinsiana. The expression patterns ofDjNAC3 andDjNAC4 in different tissues were obviously different.DjNAC3 was expressed in developed cirri with the highest level, and the lowest in leaves, while that ofDjNAC4 was the highest abundance in developed barbs and the lowest in early developed barbs. The SSR loci were detected in the genomic sequences ofDjNAC3 andDjNAC4, the SSR locus of (TA)6was located in the intron region ofDjNAC3 and that of (GCA)5was in the first exon ofDjNAC4. SSR primers were designed according to the flanking sequences of SSR loci inDjNAC3 andDjNAC4. Genomic DNAs ofD.jenkinsianaand other 20 different rattan samples were selected as templates for amplification. PAGE electrophoresis analysis showed that the primers had universality and polymorphism in the samples. The sequencing result of amplification products from six different templates further confirmed the polymorphism such as variation of SSR type, the number change of repetitions, and SSR locus missing, which were found in the sequences generated by the primer pair designed based onDjNAC3. The main difference in the sequences generated by the primer pair designed according toDjNAC4 was the number change of repetitions.【Conclusion】 There are significant differences betweenDjNAC3 andDjNAC4, such as gene structures, gene expression patterns, and SSR locus, which means thatDjNAC3 andDjNAC4 might play different roles in the growth and development ofD.jenkinsiana. The universality and polymorphism of SSR markers developed fromDjNAC3 andDjNAC4 indicate that they can serve as molecular markers for rattan classification and molecular-assisted breeding.
Keywords:Daemonoropsjenkinsiana;NAC; gene expression; SSR development
NAC (NAM,ATAF和CUC)是植物所特有的转录因子家族,其成员数量众多。NAC家族成员的N端相对保守,都由约150个氨基酸组成结构域,且被分为5个亚结构域(A、B、C、D和E),但其C端的序列呈现出高度多样化,富含丝氨酸、谷氨酸等(Olsenetal., 2005)。迄今为止,对许多物种全基因组范围内的NAC转录因子进行了鉴定,包括拟南芥(Arabidopsisthaliana)(117个)、水稻(Oryzasativa)(151个)(Nuruzzamanetal., 2010)、大豆(Glycinemax)(152个)(Leetal., 2011)、白菜(Brassicarapa)(204个)(Liuetal., 2014)、玉米(Zeamays)(152个)(Shirigaetal., 2014)、鹰嘴豆(Cicerarietinum)(71个)(Haetal., 2014)、番茄(Solanumlycopersicum)(104个)(Suetal., 2015)和毛竹(Phyllostachysedulis)(125个)(黎帮勇等, 2015)等。NAC转录因子的功能多样,参与许多生物学过程,包括花发育、次生壁形成、细胞分裂、顶端分生组织的形成、叶片衰老,以及生物和非生物胁迫应答(Olsenetal., 2005; Tranetal., 2010; Nakashimaetal., 2012; Nuruzzamanetal., 2013; Banerjeeetal., 2015)等,在植物的生长发育过程中起着重要的调控作用。
随着研究的不断深入,越来越多NAC在植物中的功能被揭示。NAM基因在矮牵牛(Petuniahybrida)的分生组织和原基边界表达,是其胚胎和花形成所需的(Soueretal., 1996); 拟南芥CUC2的表达决定其叶缘锯齿的程度(Nikovicsetal., 2006); 玉米ZmCUC3在顶端分生组织外围表达,边界明显,部分细胞将形成一个新的叶原基(Zimmermannetal., 2005)。拟南芥NAC转录因子NTL8参与赤霉酸介导的盐信号调控(Kimetal., 2008),而且通过FLOWERING LOCUST调节拟南芥盐响应开花(Kimetal., 2007),冷激活NAC转录因子调控诱导拟南芥病原体的抗性反应(Seoetal., 2010),AtNAC2作为乙烯和生长素信号转导通路下游的转录因子,参与盐应激反应和侧根发育调控(Heetal., 2005)。拟南芥NAC转录因子AtNAP在叶片衰老中发挥着重要作用,AtNAP的诱导表达导致拟南芥早熟衰老(Guoetal., 2006),ABA-AtNAP通过控制气孔的开闭来调节乙烯刺激呼吸,实现对果实的衰老调控(Kouetal., 2012)。在祖野麦(Triticumturgidumssp.dicoccoides)中NAC转录因子(NAM-B1)可以加速衰老,提高叶片的营养转运形成籽粒(Uauyetal., 2006)。水稻OsNAC19是一种转录激活因子,参与水稻响应稻瘟病菌感染,可能在茉莉酸甲酯介导的信号传导途径中起重要作用(Linetal., 2007)。欧洲油菜(Brassicanapus)BnaNAC55通过激活一些活性氧(Reactive Oxygen Species, ROS)以及与防御有关的基因的表达,来调控ROS的积累和细胞死亡(Niuetal., 2016)。
棕榈藤属于棕榈科(Palmae)植物,是重要的热带森林资源,其藤茎是优良的非木材森林产品之一,是编制各种高档家具及工艺品的理想材料,具有很高的经济价值。由于棕榈藤资源极其匮乏,其原料是国内外市场上的紧缺物资。绝大多数棕榈藤具有极强的攀缘性,总是和其他的树木纠缠在一起,使其成为森林的伴生物种。黄藤(Daemonoropsjenkinsiana)为棕榈科鳞果亚科(Lepidocaryoideae)省藤族(CALAMEAE)黄藤属(Daemonorops)植物,分布于广东东南部、香港、海南及广西西南部,为我国棕榈科单属单种植物,叶羽状全裂,茎初时直立,后攀援,其攀援主要借助于叶片顶端延伸而成的具爪状刺的纤鞭。研究表明NAC参与叶片形态建成的调控(Zimmermannetal., 2005; Nikovicsetal., 2006),目前虽然对黄藤的生长特性以及形态特征具有比较详细的描述(江泽慧等, 2013),但关于黄藤的生长发育的分子基础尚属空白。因此,本研究以黄藤为对象,从叶片中分离克隆了2个NAC基因,并对其分子特征进行深入分析,研究其表达模式,并开发SSR分子标记,以期为深入研究其功能和分子标记辅助育种提供参考。
1 材料与方法
1.1 试验材料 黄藤样品取自中国林业科学研究院热带林业研究所(广州,龙洞),分别采集叶片、发育初期的纤鞭、发育成熟的纤鞭、发育初期的钩刺、发育成熟的钩刺,放入RNAlater® RNA Stabilization Reagent(QIAGEN,德国)中,带回实验室备用。另外从中国科学院西双版纳植物园分别采集的20个藤种(表1)叶片,用硅胶干燥处理后带回实验室备用。
1.2 DNA提取、RNA分离与cDNA的合成 采用改良CTAB法提取不同藤种的基因组DNA(高志民等, 2006),存-20 ℃备用。采用改良Trizol法提取黄藤叶片、发育初期钩刺、发育成熟钩刺、发育初期纤鞭和发育成熟纤鞭的总RNA(Gaoetal., 2006),按照反转录试剂盒(Promega, 美国)说明书分别合成cDNA。

表1 本研究所用藤种Tab.1 The list of rattan accessions used in this study
1.3 基因克隆与分析 根据黄藤转录组数据(NCBI SRA 注册号: SRR3089417、SRR3089429、SRR3089432、SRR3089433、SRR3089434、SRR3089435、SRR3089436和SRR3089437)中预测的NAC转录因子基因序列设计引物,Teng-NAC3-F和Teng-NAC3-R,以及Teng-NAC4-F和Teng-NAC4-R(表2),由生工生物工程股份有限公司合成。分别以黄藤cDNA和基因组DNA为模板进行扩增。PCR反应体系为: 5×Prime STARTMBuffer 4 μL,dNTP Mixture (2.5 mmol·L-1)2 μL,正向引物(10 μmol·L-1)0.5 μL,反向引物(10 μmol·L-1)0.5 μL,cDNA/基因组DNA 2 μL,Prime STAR HS DNA polymerase 0.2 μL,DMSO 1 μL,加水补至总体积为20 μL。PCR反应程序为: 98 ℃ 4 min; 98 ℃ 30 s,60 ℃ 30 s,72 ℃ 1 min 50 s(基因组DNA扩增延伸为2 min 30 s),反应循环35个; 72 ℃ 10 min,4 ℃保存。用胶回收试剂盒(Biomega, 美国)回收PCR产物,进行加A反应,反应体系为: 10×LA PCR Buffer 1.0 μL,dATP(2.5 mmol·L-1)1.0 μL,回收产物7.9 μL,LATaq酶0.1 μL; 反应程序: 70 ℃ 30 min。用加A产物连接 pGEM-T easy载体(Promega, 美国),转化大肠杆菌(Escherichiacoli)DH5α,选取阳性克隆,酶切检测分析后,送生工生物工程股份有限公司测序。
用DNAstar对测序获得的序列进行初步分析。用NCBI在线软件BLAST (http:∥blast.ncbi.nlm.nih.gov/Blast.cgi)程序进行序列同源性比较。蛋白结构域预测用TMHMM 2.0 (http:∥myhits.isb-sib.ch/cgi-bin/motif_scan)来完成。对获得基因编码的蛋白采用Phyre2.0 (http:∥www.sbg.bio.ic.ac.uk/~phyre2/html/page.cgi?id=index)进行三级结构建模。

表2 PCR扩增所用引物Tab.2 List of PCR primer sequences
1.4 基因表达分析 分别利用定量引物qNAC3-F和qNAC3-R、qNAC4-F和qNAC4-R(表2)对2个NAC基因在叶片、发育初期的纤鞭、发育成熟的纤鞭、发育初期的钩刺、发育成熟的钩刺中的表达变化情况进行定量分析。反应在耶拿qTOWER2.2仪器上进行,总体系(10.0 μL): 5.0 μL LightCycler®480 SYBR Green I Master Mix (Roche,美国),0.8 μL cDNA,正、反向引物各0.2 μL,3.8 μL ddH2O。反应程序: 95 ℃ 5 min; 95 ℃ 30 s,60 ℃ 10 s,40个循环。定量分析所用内参基因为γ-tubulin(GenBank No. KX793703)。反应重复3次,结果用2-ΔΔCt法(Livaketal., 2001)分析。
1.5 SSR位点分析 利用SSRIT(Simple Sequence Repeat Identification Tool)在线软件 (Temnykhetal., 2001)对获得的黄藤NAC转录因子基因序列进行SSR搜索,SSR类型最多核苷酸数量设置为4个,重复次数等于或大于5。
根据DjNAC3和DjNAC4基因组序列分别设计SSR引物(ssrNAC3-F和ssrNAC3-R、ssrNAC4-F和ssrNAC4-R),分别以21个不同藤种(表1)的基因组DNA为模板,进行扩增,PCR产物用8%的PAGE电泳进行分析。回收以伸长钩叶藤、老挝钩叶藤、狭叶黄藤、黄藤属种1、勐腊鞭藤、斑岭省藤6个藤种基因组DNA为模板扩增获得的目的片段,并连接到pGEM-T easy载体,转化大肠杆菌,并送公司测序。
2 结果与分析
2.1DjNAC的克隆与基因结构分析 对应黄藤cDNA和基因组DNA模板,引物Teng-NAC3-F和Teng-NAC3-R的PCR扩增产物电泳结果显示,分别约在0.7 kb和0.8 kb有1条目的条带,同时引物Teng-NAC4-F和Teng-NAC4-R分别约在1.3 kb和1.4 kb有1条目的条带。分别回收目的条带测序结果表明,引物Teng-NAC3-F和Teng-NAC3-R扩增的cDNA为731 bp,包含1个729 bp的开放阅读(ORF),ORF对应的基因组序列为850 bp,包含2个外显子和1个121 bp内含子(454-574 bp),将该基因cDNA命名为DjNAC3 (GenBank登录号: KU556738)。引物Teng-NAC4-F和Teng-NAC4-R扩增的cDNA为1 334 bp,包含1个1 326 bp的ORF,ORF对应的基因组序列为1 441 bp,包含2个外显子和1个107 bp的内含子(875-981 bp),将该基因cDNA命名为DjNAC4 (GenBank登录号: KX579750)。DjNAC3和DjNAC4的开放阅读(ORF)对应的基因组所包含的内含子均符合GT-AG剪切原则(Mooreetal., 1993),基因结构如图1所示。
2.2 DjNAC蛋白性质与结构分析DjNAC3编码1个242 aa的蛋白,预测的分子量为27.95 kDa,等电点为8.323。DjNAC4编码1个441 aa的蛋白,预测的分子量为49.66 kDa,等电点为7.082。Blast分析表明,DjNAC3和DjNAC4具有典型的NAC转录因子结构特征,N端保守性较强,由大约150个高度保守的氨基酸残基组成,包含A、B、C、D、E 5个亚结构域,C端的氨基酸序列保守性较弱。DjNAC3和DjNAC4均属于CUC (development-related NAC)亚家族(Zhuetal., 2015),但二者之间的相似系数仅为23.6%,以c3ulxA_为模板构建DjNAC3和DjNAC4的三级结构(图2),结构上的差异意味着它们在黄藤生长发育过程中可能具有不同的功能。
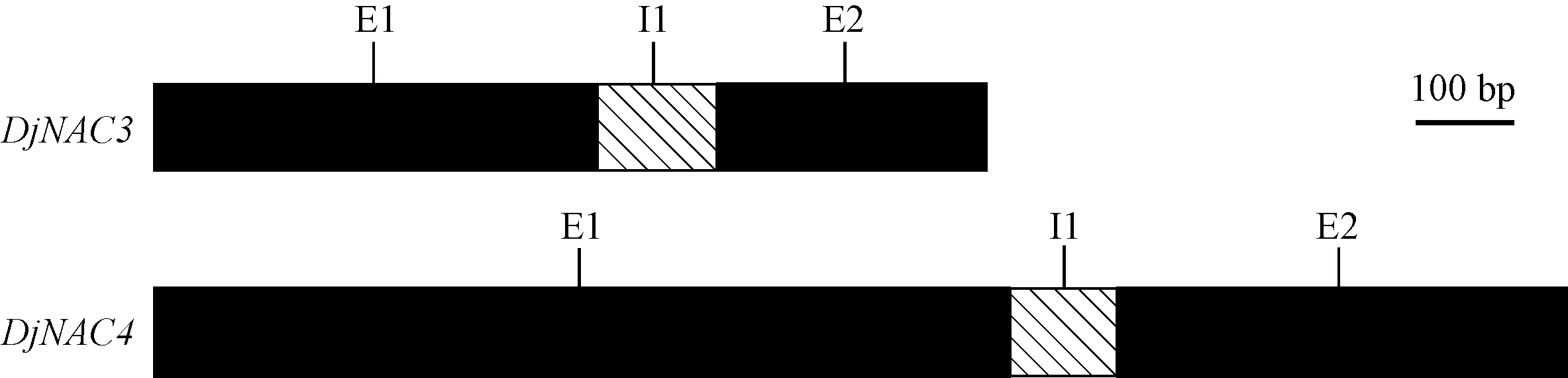
图1 DjNAC3和DjNAC4的基因结构Fig.1 Gene structures of DjNAC3 and DjNAC4E: 外显子; I: 内含子。E: Exon; I: Intron.
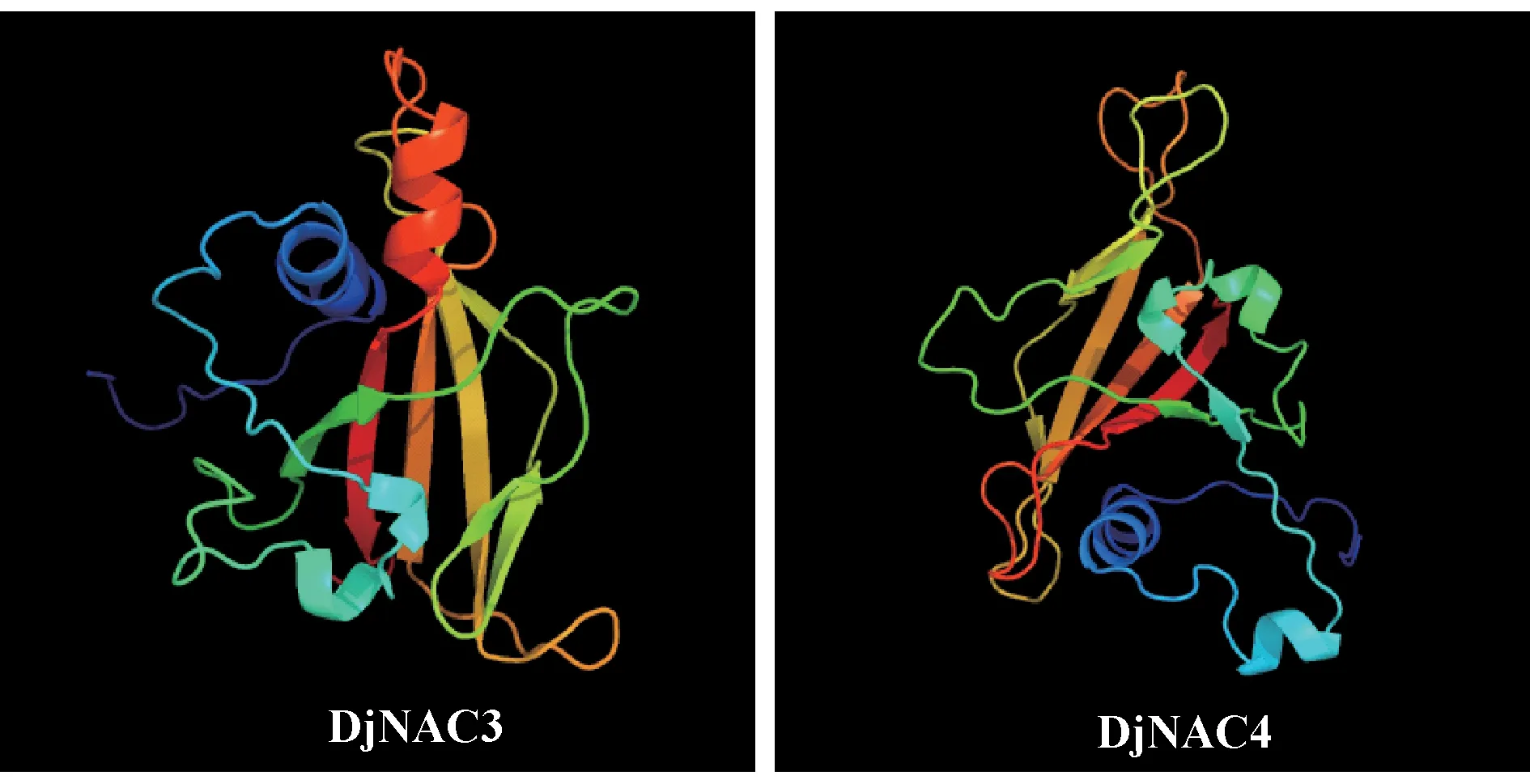
图2 DjNAC3和DjNAC4蛋白的三级结构Fig.2 Tertiary structures of DjNAC3 and DjNAC4
2.3DjNAC的表达分析 以黄藤叶片、发育初期钩刺、发育成熟钩刺、发育初期纤鞭和发育成熟纤鞭的cDNA为模板,进行实时定量PCR分析,检测DjNAC在不同组织中的表达模式。结果显示,DjNAC3和DjNAC4的表达模式存在着明显的差异。DjNAC3在叶片中的表达丰度最低,远低于钩刺和纤鞭,而纤鞭中的表达又高于钩刺,以发育成熟的纤鞭中的表达丰度最高;DjNAC4则在发育成熟的钩刺中表达丰度最高,发育成熟的纤鞭次之,叶片和发育初期的纤鞭中均较多,而发育初期的钩刺中最低(图3)。由此表明,DjNAC3和DjNAC4在黄藤叶片发育过程中可能具有不同的功能。
2.4 SSR位点分析与分子标记开发 SSR搜索结果表明,在DjNAC3和DjNAC4的基因组序列中分别找到1个SSR位点,其中前者的SSR位点位于内含子区域,SSR类型为TA,重复次数为6; 后者的SSR位点位于第1个外显子区域,SSR类型为GCA,重复次数为5(表3)。
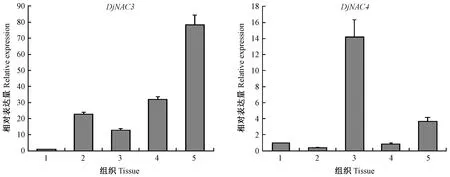
图3 DjNAC3和DjNAC4在黄藤不同组织中的表达分析Fig.3 Expression analysis of DjNAC3 and DjNAC4 in different tissues of Daemonorops jenkinsiana1: 叶片; 2: 发育初期钩刺; 3: 发育成熟钩刺; 4: 发育初期纤鞭; 5: 发育成熟纤鞭。1: Leaves; 2: Early developed barbs; 3: Developed barbs; 4: Early developed cirri; 5: Developed cirri.
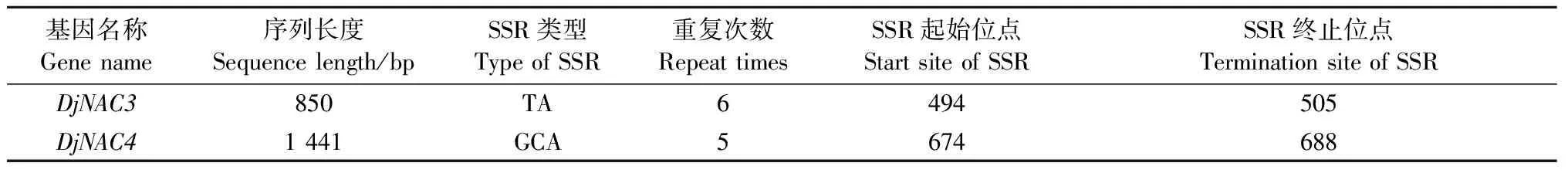
表3 SSR位点分析Tab.3 Analysis of SSR loci

图4 不同藤种样品SSR扩增产物的测序分析Fig.4 Analysis of SSR sequences of PCR products amplified from six rattan samplesA: ssrNAC3-F和ssrNAC3-R扩增产物序列; B: ssrNAC4-F和ssrNAC4-R扩增产物序列。编号与表1中藤种相对应。A: Sequences of products amplified by ssrNAC3-F and ssrNAC3-R; B: Sequences of products amplified by ssrNAC4-F and ssrNAC4-R. The No. represents the species in Tab.1 correspondingly.
为进一步验证SSR位点的可靠性,分别回收了伸长钩叶藤、老挝钩叶藤、狭叶黄藤、黄藤属种1、勐腊鞭藤、斑岭省藤6个藤种的PCR产物进行测序。结果表明,引物对ssrNAC3-F和ssrNAC3-R的扩增产物在205~220 bp之间,不同藤种样品中的SSR序列存在着一定的差异,既包括SSR类型变异、重复次数变化等多态性,又有SSR位点缺失的现象。如狭叶黄藤、黄藤属种1与黄藤的SSR位点均为(TA)6,而伸长钩叶藤、老挝钩叶藤和勐腊鞭藤的SSR位点均为(TA)4,少了2个TA重复,但在斑岭省藤中SSR位点却缺失了,同时在SSR位点之后的30个碱基各种之间存在着不同程度的多态性(图4A)。引物对ssrNAC4-F和ssrNAC4-R的扩增产物在331~346 bp之间,其中狭叶黄藤、黄藤属种1与黄藤的SSR位点均为(GCA)5,而伸长钩叶藤、老挝钩叶藤、勐腊鞭藤和斑岭省藤的SSR位点分别为(GCA)8、(GCA)9、(GCA)7和(GCA)4,不同属间存在明显的多态性(图4B)。
2.5 SSR分子标记的不同藤种通用性分析 为验证SSR位点的通用性和多态性,利用SSR引物(ssrNAC3-F和ssrNAC3-R、ssrNAC4-F和ssrNAC4-R)(表2),以黄藤和另外20个不同藤种的基因组DNA为模板进行扩增。PAGE电泳结果表明,所有PCR产物均有明显的目的条带,表明2个分子标记在不同藤种中具有通用性(图5)。
引物对ssrNAC3-F和ssrNAC3-R的21个样品扩增产物约在220 bp均有一明显特异条带(图5A),其中钩叶藤属的伸长钩叶藤和老挝钩叶藤的目的条带大小一致,而细钩叶藤的明显偏大; 黄藤属的长柄黄藤、狭叶黄藤和黄藤种1的带型一致,而黄藤的目的条带明显偏小; 省藤属中除了褐鳞省藤和勐捧省藤带型一致、种2(C. sp. 2)和种3(C. sp. 3)的带型较一致外,其他藤种的带型差异明显。引物对ssrNAC4-F和ssrNAC4-R的扩增产物则约在350 bp均有一明显特异条带(图5 B),其中钩叶藤属3个藤种的目的条带大小一致; 黄藤属的长柄黄藤、狭叶黄藤和种1(D. sp. 1)的带型一致,而黄藤的目的条带明显偏小; 省藤属柳条省藤和褐鳞省藤带型目的条带差异明显,而褐鳞省藤和勐捧省藤的相一致,其余藤种的带型差异明显,但元江省藤(C. sp.)与种1(C. sp. 1)、种2(C. sp. 2)和种3(C. sp. 3)的基本一致。由此表明,2个SSR分子标记在不同藤种中具有多态性,尤其是不同属的不同藤种之间更为明显。
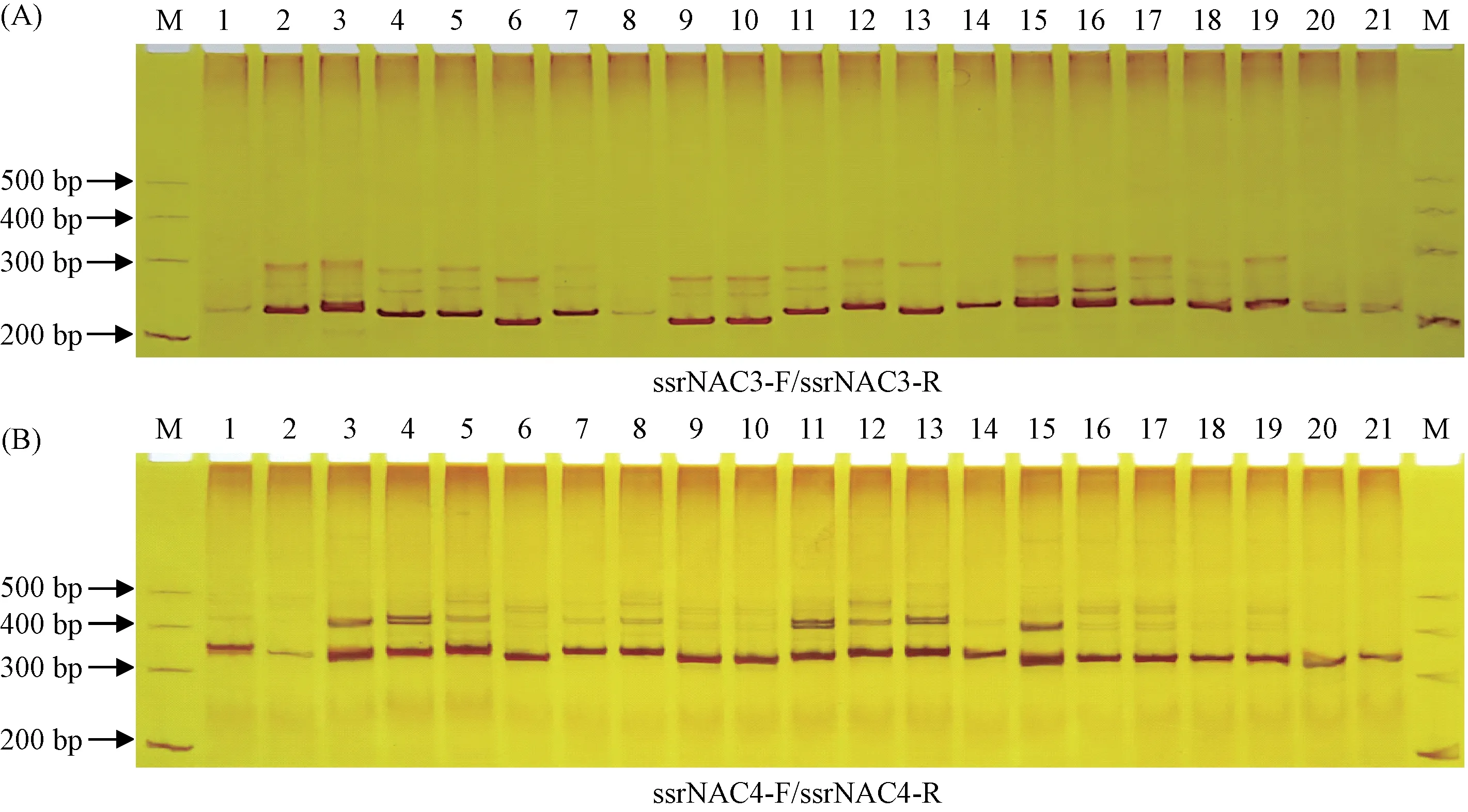
图5 不同藤种样品SSR引物扩增产物的PAGE电泳分析Fig.5 PAGE analysis of PCR products amplified from different rattan samples using SSR primersA: ssrNAC3-F和ssrNAC3-R的扩增产物; B: ssrNAC4-F和ssrNAC4-R的扩增产物。M: DNA分子量标记; 1-21: 不同藤种样品。A. PCR products of ssrNAC3-F and ssrNAC3-R; B. PCR products of ssrNAC4-F and ssrNAC4-R. M: DNA ladder; 1-21: Different rattan samples.
3 讨论
NAC转录因子对植物不同组织器官的系统发育具有重要作用,是植物形态建成中不可或缺的调控因子。根据蛋白结构特征将被子植物的NAC划分为6个大的家族,每个家族包含不同的亚家族,即家族Ⅰ(VND和NST/BRN/SMB)、Ⅱ(NAC1、CUC和ORE)、Ⅲ(NTL、NAC2和TMM)、Ⅳ(FEZ/JUB和LOV1)、Ⅴ(XND1、胁迫相关家族和NARS)和家族Ⅵ(其他) (Pereira-Santanaetal., 2015)。本研究从黄藤叶片中分离得到的DjNAC3和DjNAC4基因均属于NAC家族的CUC亚家族。基因结构的差异,意味着它们具有不同的功能,DjNAC3和DjNAC4在叶片、发育初期钩刺、发育成熟钩刺、发育初期纤鞭和发育成熟纤鞭中的基因表达模式差异进一步证实了这一点。纤鞭是棕榈藤植物为适应森林攀援生活形成的最为重要的器官,是由叶轴顶端延伸出去形成的,或是叶鞘发育形成。黄藤的叶片属于大型羽状复叶(全裂),其纤鞭由叶轴顶端延伸发育而成,纤鞭上具有轮生的钩刺,利于绞缠攀爬。DjNAC3和DjNAC4在黄藤叶片的不同部位均有表达,且存在明显差异,但其功能如何,在黄藤叶片生长发育中是怎么实现其调控作用的,有待于进一步深入研究。
SSR标记作为一种有效的分子标记已被广泛应用于遗传多样性分析、系统发育、辅助分类和育种研究等。DjNAC3和DjNAC4的基因组中均具有1个SSR位点,并证实在21个不同棕榈藤样品中具有通用性,且在不同属种间具有多态性,这为进一步研究棕榈藤的遗传多样性和辅助分类提供了分子标记。另外研究表明,SSR位点在基因序列中所处位置不同,其功能也存在着差异,如位于5′非翻译区的SSR 可以调控基因的转录和翻译,位于基因内部的SSR具有较强的选择压力,可以调控基因的表达,位于内含子区域的SSR可以影响基因的转录,位于3′非翻译区的SSR可以引起mRNA的延伸 (Lietal., 2004)。DjNAC3和DjNAC4的SSR位点,分别位于内含子区域和外显子区域,这对二者的基因表达可能具有不同的影响,具体情况需要进一步试验验证。
随着现代生物技术的快速发展,利用基因工程手段开发利用植物中具有调控作用的关键NAC 转录因子,实现植物性状的定向改良已成为现实。水稻SNAC1转入小麦(Triticumaestivum)导致转基因植物生长迟缓(Saadetal., 2013),毛竹PeNAC在拟南芥中过量表达促进转基因植株的侧根发育(Wangetal., 2016),在水稻中过量表达穇子(Eleusinecoracana)的EcNAC67能够提高水稻的耐盐和抗旱能力(Rahmanetal., 2016),大麦(Hordeumvulgare)中过量表达HvNAC005导致提早成熟(Christiansenetal., 2016)。棕榈藤植物作为重要的森林资源之一,其潜在的价值远远没有得到开发利用,随着对其生长发育分子基础研究的不断深入,诸如NAC家族的转录因子等具有重大经济价值的功能基因将不断被挖掘出来,对其性状的调控将更具有操作性。因此,未来棕榈藤的分子育种有着广阔的前景。
4 结论
本研究从黄藤中克隆了2个NAC转录因子家族成员基因DjNAC3和DjNAC4,二者在基因结构、编码蛋白特征等方面具有明显不同的分子特征,在黄藤叶片形态建成中可能具有不同的调控功能。同时,DjNAC3和DjNAC4的序列中均包含1个SSR位点,分别为(TA)6和(GCA)5,据此开发的分子标记在不同的棕榈藤中具有较高的通用性和多态性,可应用于棕榈藤辅助分类和分子辅助育种。
高志民, 范少辉, 高 健, 等. 2006. 基于CTAB法提取毛竹基因组DNA的探讨. 林业科学研究, 19(6): 725-728.
(Gao Z M, Fan S H, Gao J,etal. 2006. Extract genomic DNA fromPhyllostachysedulisby CTAB-based method. Forest Research, 19(6):725-728. [in Chinese])
江泽慧,王康林. 2013. 中国棕榈藤. 北京: 科学出版社, 90-92.
(Jiang Z H, Wang K L. 2013. Rattan in China. Beijing: Science Press, 90-92.[in Chinese])
黎帮勇, 胡尚连, 曹 颖, 等. 2015. 毛竹NAC 转录因子家族生物信息学分析. 基因组学与应用生物学, 34(8): 1769-1777.
(Li B Y, Hu S L, Cao Y,etal. 2015. Bioinformatics analysis of NAC gene family in moso bamboo. Genomics Applied Biol, 34(8): 1769-1777. [in Chinese])
Banerjee A, Roychoudhury A. 2015. WRKY proteins: signaling and regulation of expression during abiotic stress responses. Sci World J, 2015: 807560.
Christiansen M W, Matthewman C,Podzimska-Sroka D,etal. 2016. Barley plants over-expressing the NAC transcription factor geneHvNAC005 show stunting and delay in development combined with early senescence. J Exp Bot, 67 (17): 5259-5273.
Gao Z M, Li X P, Li L B,etal. 2006. An effective method for total RNA isolation from bamboo. Chinese For Sci Tech, 5(3): 52-54.
Guo Y, Gan S. 2006. AtNAP, a NAC family transcription factor, has an important role in leaf senescence. Plant J, 46(4): 601-612.
Ha C V, Esfahani M N, Watanabe Y,etal. 2014. Genome-wide identification and expression analysis of the CaNAC family members in chickpea during development, dehydration and ABA treatments. PLoS One, 9(12): e114107.
He X J, Mu R L, Cao W H,etal. 2005. AtNAC2, a transcription factor downstream of ethylene and auxin signaling pathways, is involved in salt stress response and lateral root development. Plant J, 44(6): 903-916.
Kim S G, Kim S Y, Park C M. 2007. A membrane-associated NAC transcription factor regulates salt-responsive flowering via FLOWERING LOCUST inArabidopsis. Planta, 6(3): 647-654.
Kim S G, Lee A K, Yoon H K,etal. 2008. A membrane-bound NAC transcription factor NTL8 regulates gibberellic acid-mediated salt signaling inArabidopsisseed germination. Plant J, 55(1):77-88.
Kou X, Watkins C B, Gan S S. 2012.ArabidopsisAtNAP regulates fruit senescence. J Exp Bot, 63(17): 6139-6147.
Le D T, Nishiyama R, Watanabe Y,etal. 2011. Genome-wide survey and expression analysis of the plant-specific NAC transcription factor family in soybean during development and dehydration stress. DNA Res, 18(4): 263-276.
Li Y C, Korol A B, Fahima T,etal. 2004. Microsatellites within genes: structure, function, and evolution. Mol Biol Evol, 21(6): 991-1007.
Lin R M, Zhao W S, Meng X B,etal. 2007. Rice geneOsNAC19 encodes a novel NAC-domain transcription factor and responds to infection byMagnaporthegrisea. Plant Sci, 172(1): 120-130.
Liu T K, Song X M, Duan W K,etal. 2014. Genome-wide analysis and expression patterns of NAC transcription factor family under different developmental stages and abiotic stresses in Chinese cabbage. Plant Mol Biol Report, 32(5): 1041-1056.
Livak K J, Schmittgen T D. 2001. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCtmethod. Methods, 25: 402-408.
Moore M J, Query C C, Sharp P A. 1993. Splicing of precursors to mRNA by the spliceosome∥ Gesteland R F, Atkins J F. The RNA world. Cold Spring Harbor: Cold Spring Harbor Laboratory Press, 303-357.
Nakashima K, Takasaki H, Mizoi J,etal. 2012. NAC transcription factors in plant abiotic stress responses. Biochim Biophys Acta, 1819(2): 97-103.
Nikovics K, Blein T, Peaucelle A,etal. 2006. The balance between theMIR164AandCUC2 genes controls leaf margin serration inArabidopsis. Plant Cell, 18(11): 2929-2945.
Niu F, Wang C, Yan J,etal. 2016. Functional characterization of NAC55 transcription factor from oilseed rape (BrassicanapusL.) as a novel transcriptional activator modulating reactive oxygen species accumulation and cell death. Plant Mol Biol, 92(1/2): 89-104.
Nuruzzaman M, Manimekalai R, Sharoni A M,etal. 2010. Genome-wide analysis of NAC transcription factor family in rice. Gene, 465(1/2): 30-44.
Nuruzzaman M, Sharoni A M, Kikuchi S. 2013. Roles of NAC transcription factors in the regulation of biotic and abiotic stress responses in plants. Front Microbiol, 4: 248.
Olsen A N, Ernst H A, Leggio L L,etal. 2005. NAC transcription factors: structurally distinct, functionally diverse. Trends Plant Sci, 10(2): 79-87.
Pereira-Santana A, Alcaraz L D, Castao E,etal. 2015. Comparative genomics of NAC transcriptional factors in angiosperms: Implications for the adaptation and diversification of flowering plants. PLoS One, 10(11): e0141866.
Rahman H, Ramanathan V, Nallathambi J,etal. 2016. Over-expression of a NAC 67 transcription factor from finger millet (EleusinecoracanaL.) confers tolerance against salinity and drought stress in rice. BMC Biotechnol, Suppl 1: 35.
Saad A S, Li X, Li H P,etal. 2013. A rice stress-responsive NAC gene enhances tolerance of transgenic wheat to drought and salt stresses. Plant Sci, 203/204: 33-40.
Seo P J, Kim M J, Park J Y,etal. 2010. Cold activation of a plasma membrane-tethered NAC transcription factor induces a pathogen resistance response inArabidopsis. Plant J, 61(4): 661-671.
Shiriga K, Sharma R, Kumar K,etal. 2014. Genome-wide identification and expression pattern of drought-responsive members of the NAC family in maize. Meta Gene, 2: 407-417.
Souer E, van Houwelingen A, Kloos D,etal. 1996. Thenoapicalmeristemgene of petunia is required for pattern formation in embryos and flowers and is expressed at meristem and primordia boundaries. Cell, 85(2): 159-170.
Su H, Zhang S, Yin Y,etal. 2015. Genome-wide analysis of NAM-ATAF1,2-CUC2 transcription factor family inSolanumlycopersicum. J Plant Biochem Biotechnol, 24(2): 176-183.
Temnykh S, DeClerck G, Lukashova A,etal. 2001. Computational and experimental analysis of microsatellites in rice (OryzasativaL.): frequency, length variation, transposon associations, and genetic marker potential. Genome Res, 11(8): 1441-1452.
Tran L S, Nishiyama R,Yamaguchi-Shinozaki K,etal. 2010. Potential utilization of NAC transcription factors to enhance abiotic stress tolerance in plants by biotechnological approach. GM Crops, 1(1): 32-39.
Uauy C, Distelfeld A, Fahima T,etal. 2006. A NAC gene regulating senescence improves grain protein, zinc, and iron content in wheat. Science, 314(5803): 1298-1301.
Wang L, Zhao H, Chen D,etal. 2016. Characterization and primary functional analysis of a bambooNACgene targeted by miR164b. Plant Cell Rep, (6):1371-1383.
Zhu G, Chen G, Zhu J,etal. 2015. Molecular characterization and expression profiling of NAC transcription factors inBrachypodiumdistachyonL. PLoS One, 10(10): e0139794.
Zimmermann R, Werr W. 2005. Pattern formation in the monocot embryo as revealed by NAM and CUC3 orthologues fromZeamaysL. Plant Mol Biol, 58(5): 669-685.
(责任编辑 徐 红)
MolecularCharacteristicsandSSRMarkerDevelopmentofTwoNACGenesfromDaemonoropsjenkinsiana
Sun Huayu1Li Lichao1Zhao Hansheng1Yang Yihong1,2Wang Sining1Gao Zhimin1
(1.KeyOpenLaboratoryontheScienceandTechnologyofBambooandRattanofStateForestryAdministrationInternationalCenterforBambooandRattanBeijing100102; 2.CollegeofHorticulture,HebeiAgriculturalUniversityBaoding071001)
S718.46
A
1001-7488(2017)08-0132-09
10.11707/j.1001-7488.20170815
2016-09-12;
2016-10-24。
“十二五”农村领域国家科技计划项目“竹藤种质资源创新利用研究”第一课题“竹藤优异种质创制创新与种苗培育标准化示范”(2015BAD04B01)。
*高志民为通讯作者。
