基于稳定同位素标记和平行反应监测的蛋白质组学定量技术用于肝癌生物标志物的筛选和验证
2017-09-14王素兰高华萍
王素兰, 高华萍, 张 菁, 叶 翔
(浙江大学附属邵逸夫医院下沙院区, 浙江 杭州 310018)
研究论文
基于稳定同位素标记和平行反应监测的蛋白质组学定量技术用于肝癌生物标志物的筛选和验证
王素兰, 高华萍, 张 菁, 叶 翔*
(浙江大学附属邵逸夫医院下沙院区, 浙江 杭州 310018)
肝癌是全球第五大恶性肿瘤,其五年生存率极低,及早地发现与诊断对肝癌的临床治疗具有重要意义。通过结合体外稳定同位素标记的蛋白质组学相对定量技术和基于平行反应监测的靶向蛋白质组学定量技术,建立了一种癌症生物标志物的筛选和验证方法。该方法被用于肝癌组织的差异蛋白质筛选和后期验证,共筛选到70个在癌组织中显著变化的蛋白质,并对其中7个蛋白质进行了验证。所验证的蛋白质包括已在临床使用的肝癌标志物甲胎蛋白(AFP)和文献报道的潜在肝癌生物标志物热休克蛋白(HSP90)、脂肪酸结合蛋白5(FABP5)和乙醇脱氢酶4(ADH4),说明了该方法的可靠性。该文所筛选的差异蛋白质可以为肝癌生物标志物研究和临床验证提供参考;该方法还可用于其他癌症样品的差异蛋白质筛选和验证。
蛋白质组学;稳定同位素标记;平行反应监测;肝癌
肝癌是肝细胞发生癌变从而引发的一种癌症,是全球最为常见的恶性肿瘤之一[1]。我国是肝癌发病率高的国家之一,据统计2012年全球因肝癌死亡的患者中我国占51.4%左右,且死亡率仍未得到有效控制[2]。在原发性肝癌中,70%~85%为肝细胞癌(hepatocellular carcinoma, HCC)。肝癌的5年生存率仅为3%~5%,目前肝癌最有效的治疗方法是手术切除或肝移植,然而大部分患者就诊时已属中晚期,失去了最佳治疗时机,临床上有效治疗的患者仅为30%左右。因此,及早地发现与诊断肝癌对于肝癌病情的控制和治疗有着重要的意义。甲胎蛋白(alpha-fetoprotein, AFP)是肝癌早期诊断最常用的生物标志物[3],其诊断敏感度为55%~60%,是目前效果最好的生物标志物之一。然而AFP的使用存在诸多限制,如很多慢性肝炎和肝硬化患者的AFP也会升高。因此,发现新的肝癌生物标志物以弥补AFP的限制,或与AFP联合使用以提高准确率,对肝癌的早期诊断至关重要。
蛋白质组学定量技术是肿瘤生物标志物筛选的常用技术之一,能够对临床样品中的差异变化蛋白质进行高通量筛选和后期验证。稳定同位素标记法是蛋白质组学最为常用的相对定量方法,广泛应用于生物、医学、农学等各个领域。该方法包括iTRAQ(isobaric tags for relative and absolute quantitation)、TMT(tandem mass tags)和同位素二甲基标记等,主要用于前期的差异蛋白质高通量筛选。平行反应监测(parallel reaction monitoring, PRM)是一种靶向蛋白质组学方法,可有目的地检测目标蛋白质,具有选择性和灵敏度高等优点,主要用于后期差异蛋白质的规模化验证[4]。目前已有较多文献使用PRM进行差异蛋白质验证[5-8]。
本文采用基于TMT标记的高通量蛋白质组相对定量方法对肝癌组织和癌旁组织中的差异蛋白质进行了高通量的筛选,随后对筛选出的差异蛋白质进行了平行反应监测的靶向蛋白质组学定量验证。本文以肝癌组织样品为例,旨在建立一套癌症生物标志物筛选和验证的蛋白质组学方法。
1 实验部分
1.1主要仪器、试剂与材料
1260高效液相色谱仪(安捷伦公司,美国),Q-Exactive Plus高分辨质谱仪、EASY-nLC 1000超高效液相色谱系统、SpeedVac SPD131DDA真空浓缩仪(赛默飞公司,美国)。
TMT标记试剂盒购自赛默飞公司(美国),胰蛋白酶购自Promega公司(美国), BCA试剂盒购自碧云天生物技术有限公司。蛋白酶抑制剂、四乙基溴化铵(TEAB)、甲酸、乙腈、尿素、碘代乙酰胺和二硫苏糖醇等试剂均购自Sigma-Aldrich公司(美国)。
本实验所使用的样本是肝细胞癌组织和癌组织外周大于2 cm处的正常组织。样品的取得和使用完全合法,并符合本院伦理委员会的相关规定。样品采集后保存于-80 ℃冰箱备用。本实验一共使用了3例肝癌样品和3例癌旁样品,3位患者的临床TNM(tumor-node-metastasis,原发灶-淋巴结-转移)分期分别为4A、4B和4B, AFP均呈阳性。
1.2蛋白质提取和酶解
称取适量组织样品,使用预冷的磷酸盐缓冲液清洗3次,将样品转移至液氮预冷的研钵中,加液氮充分研磨至粉末后,加入4倍体积裂解液(8 mol/L尿素,100 mmol/L TEAB, 2%(质量分数)蛋白酶抑制剂),超声裂解。于4 ℃、20 000 g离心5 min,去除细胞碎片,上清液采用BCA试剂盒测定蛋白质浓度。
取适量蛋白质溶液,加入二硫苏糖醇至终浓度为10 mmol/L,于56 ℃反应30 min。随后加入碘代乙酰胺至终浓度为20 mmol/L,室温避光反应30 min。最后加入TEAB缓冲液将样品中的尿素浓度稀释至2 mol/L以下。将蛋白酶和蛋白质以1∶50的质量比加入胰蛋白酶,于37 ℃酶解过夜(约16 h)。蛋白酶解液经C18固相萃取柱除盐后,在真空离心浓缩仪中冻干溶剂。将肽段样品于-20 ℃保存,备用。
1.3肽段标记和预分离
以0.1 mol/L TEAB缓冲液(pH 8.5)溶解肽段,根据TMT试剂盒操作说明标记肽段:标记试剂解冻后用40 μL乙腈溶解,与肽段混合后室温孵育2 h,标记后将肽段混合,然后进行除盐处理,冷冻干燥后备用。
肽段在安捷伦1260高效液相色谱仪上完成预分离,采用高pH反相色谱法,色谱柱为Thermo C18-AQ(250 mm×2.1 mm, 3 μm)。流动相A为2%(v/v)乙腈水溶液,并添加氨水至pH 9.5;流动相B为98%(v/v)乙腈水溶液。操作如下:肽段分离梯度时长为48 min,流动相由5%B逐渐上升为40%B,每4 min收集一个组分,共分为12个组分,经真空冷冻干燥后备用。
1.4液相色谱-质谱联用分析
肽段用含0.1%(v/v)甲酸和3%(v/v)乙腈的水溶液溶解后上样至EASY-nLC 1000超高效液相色谱系统进行分离。流动相A为含0.1%(v/v)甲酸和3%(v/v)乙腈的水溶液;流动相B为含0.1%(v/v)甲酸和97%(v/v)乙腈的水溶液。流动相梯度设置:0~46 min, 6%B~23%B; 46~54 min, 23%B~35%B; 54~57 min, 35%B~80%B; 57~60 min, 80%B,流速维持在300 nL/min。肽段经由超高效液相系统分离后进入Q-Exactive质谱进行分析。离子源电压设置为2.0 kV,肽段母离子及其二级碎片都使用静态轨道肼(Orbitrap)进行检测。一级质谱扫描范围设置为m/z350~1 600,扫描分辨率设置为70 000;二级质谱扫描范围固定起点为m/z100, Orbitrap扫描分辨率设置为17 500。数据采集模式为数据依赖式扫描(DDA)模式,在一级质谱中选择信号强度最高的前20个母离子进入高能碰撞诱导解离(HCD)碰撞池碎裂,碎裂能量设置为30。自动增益控制(AGC)设置为5×104,信号阈值设置为1×104离子/s,最大离子注入时间(Maximum IT)设置为200 ms,动态排除时间设置为30 s。
1.5平行反应监测
肽段用含0.1%(v/v)甲酸和3%(v/v)乙腈的水溶液溶解后上样至EASY-nLC 1000超高效液相色谱系统进行分离,色谱条件同1.4节。肽段经由超高效液相系统分离后进入Q-Exactive质谱进行分析,离子源电压设置为2.0 kV,肽段母离子及其二级碎片都使用Orbitrap进行检测。一级质谱扫描范围设置为m/z350~1 000,扫描分辨率设置为35 000;二级质谱Orbitrap扫描分辨率设置为17 500。数据采集模式使用PRM模式,HCD碰撞池的碎裂能量设置为27。一级质谱AGC设置为3×106,最大离子注入时间设置为20 ms;二级质谱AGC设置为1×105,最大离子注入时间设置为auto,隔离窗口(isolation window)设置为m/z2.0。
1.6TMT标记定量数据检索
二级质谱数据使用Maxquant(1.4.1.2版本)进行检索。检索参数如下:数据库为SwissProt人库,在数据库中加入了常见的污染库用于消除鉴定结果中污染蛋白质的影响;酶切方式设置为Trypsin/P;最大容忍漏切位点为2;肽段最小长度设置为7个氨基酸;肽段最大修饰数设为5;第一次检索(First search)母离子质量误差设为20 ppm (10-6),主体检索(Main search)母离子质量误差设为5 ppm,二级碎片离子的质量误差设置为20 ppm。固定修饰设置为半胱氨酸烷基化,可变修饰为甲硫氨酸的氧化和蛋白质N端的乙酰化。定量方法设置为TMT 6标,蛋白质鉴定的假阳性率(FDR)设置为小于1%。
1.7PRM数据处理
肽段参数:蛋白酶设置为Trypsin[KR/P],最大漏切位点数设置为2,肽段长度设置为8~25个氨基酸残基,设置半胱氨酸烷基化和甲硫氨酸氧化为可变修饰,最大修饰数设置为3。母子离子对(Transition)参数:母离子电荷设置为2、3,子离子电荷设置为1、2,离子类型设置为b、y、p。碎片离子选择从第3个开始到最后一个,离子匹配的质量误差设置为0.02 Da。
1.8生物信息分析
对筛选得到的差异变化蛋白质进行了GO(基因本论,gene ontology)注释、KEGG(京都基因与基因组百科全书,Kyoto encyclopedia of genes and genomes)注释、亚细胞定位等分析,对差异蛋白质进行了基于GO和KEGG的功能富集,并进行了相应的聚类分析。对差异蛋白质进行了蛋白质-蛋白质相互作用网络分析。
2 结果与讨论
2.1基于TMT标记的蛋白质组学定量方法用于肝癌生物标志物的筛选
使用TMT标记的蛋白质组学相对定量方法进行了肝癌生物标志物的高通量筛选,实验流程见图1。分别提取3例肝癌组织和3例癌旁组织的蛋白质后,加入胰蛋白酶酶解成肽段,然后使用TMT 6标试剂分别对6个肽段样品进行标记;标记后的样品进行1∶1∶1∶1∶1∶1(质量比)混合,随后采用高pH值流动相的液相色谱预分离成12个组分以降低样品复杂程度;最后将每个组分分别上样至液相色谱-质谱联用仪上进行分析。经过数据库检索,鉴定到4 113个蛋白质,其中可定量的蛋白质是3 069个。共筛选到了247个在肝癌组织中差异变化的蛋白质(3组样品的相对定量比值均在1.2倍以上),其中有70个蛋白质的差异变化倍数在3组样品中均大于1.5倍(38个显著下调,32个显著上调),结果见附表1(http://www.chrom-China.com/UserFiles/File/sp1705022-supporting%20info.pdf)。这些在肝癌组织中发生显著变化的蛋白质有可能成为肝癌的生物标志物。但是,由于TMT、iTRAQ等方法存在定量比值压缩效应[9,10],这些差异变化蛋白质的相对定量比值是否准确需要用更为精确的蛋白质定量方法或者生化方法进行后期验证。

图 1 基于TMT和PRM技术的肿瘤生物标志物筛选与验证流程示意图Fig. 1 Workflow for TMT labeling and PRM-based proteomic quantification for cancer biomarker screening and validationTMT: tandem mass tags; PRM: parallel reaction monitoring.
2.2功能注释、功能富集和蛋白质-蛋白质相互作用网络分析
为了研究本文筛选到的差异变化蛋白质以探知肝癌组织在蛋白质组水平的病理状态,我们对这些差异蛋白质分别进行了深入的生物信息分析。首先,我们对上调和下调的蛋白质分别进行了亚细胞定位的功能注释分析,结果见图2。从图2可以看出,显著下调的蛋白质主要分布于细胞质中,而显著上调的蛋白质则集中分布于细胞核和胞外基质中。
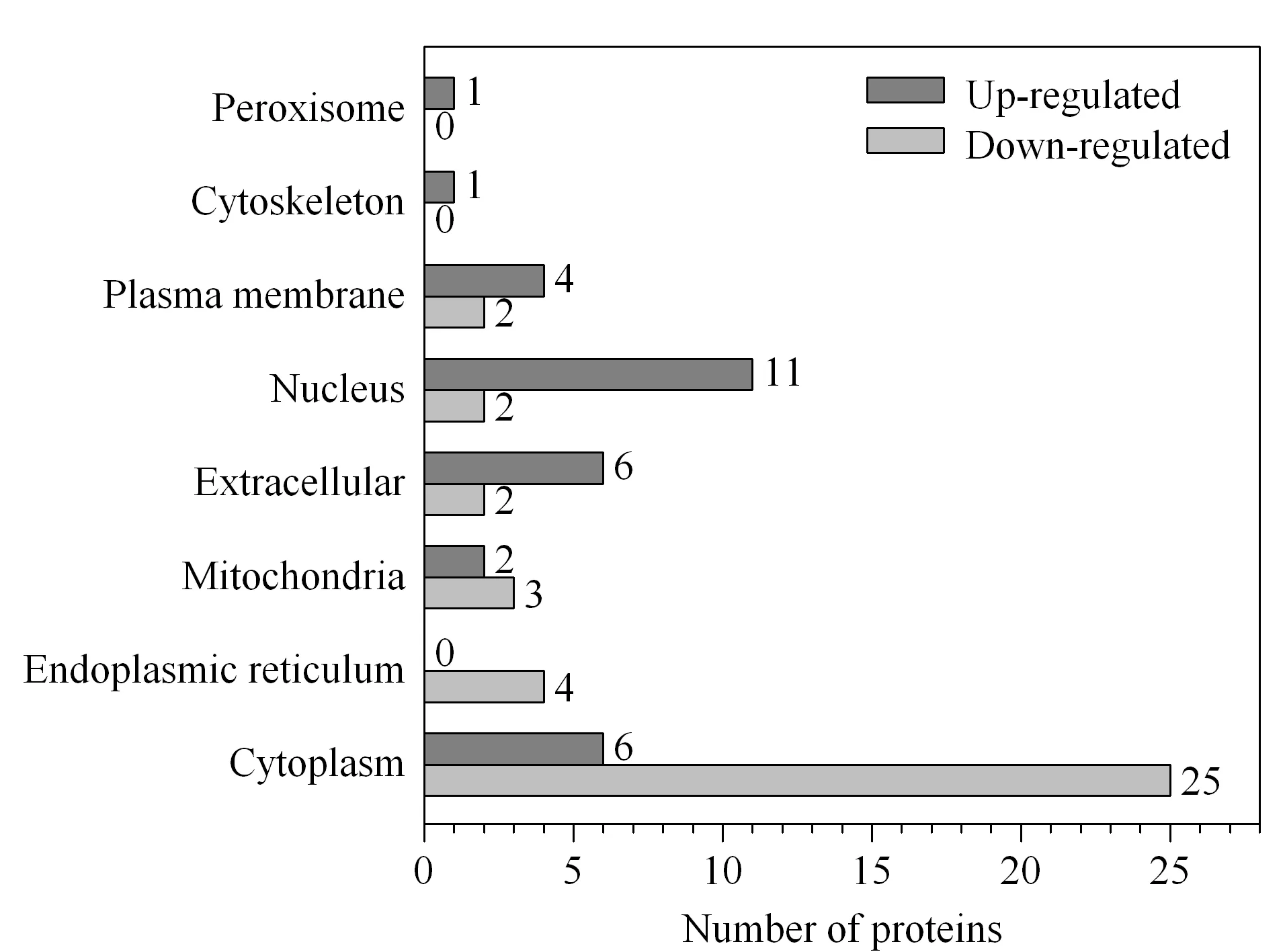
图 2 差异表达蛋白质的亚细胞定位分析Fig. 2 Subcellular localization analysis of up- and down-regulated proteins
随后,我们又对差异表达的蛋白质进行了基于GO和KEGG的功能富集分析,结果见图3。从GO富集分析结果中我们发现,显著上调的蛋白质主要参与细胞核内的DNA重组、染色质结合、基因表达及胞外基质的构成等功能,而显著下调的蛋白质则主要分布于细胞质中,并与催化、代谢、糖链结合等功能相关。KEGG富集分析发现,上调的蛋白质主要与碱基切除修复、ECM受体相互作用、PI3K-Akt信号通路、酒精中毒等信号通路相关,而下调的蛋白质则主要与淀粉和蔗糖代谢、糖酵解、化学致癌、脂肪酸降解、视黄醇代谢、果糖和甘露糖代谢等通路相关。
最后,我们对差异表达蛋白质进行了蛋白质-蛋白质相互作用网络分析,结果见图4。我们发现,表达上调的蛋白质显著富集于剪接体上,而表达下调的蛋白质则主要富集于淀粉和蔗糖代谢、视黄醇代谢和脂肪酸降解等过程。以上分析结果揭示了肝癌组织中显著变化蛋白质的分布和功能,可以为后期肝癌生物标志物的筛选以及肝癌的蛋白质组学研究提供参考。
2.3差异蛋白质的PRM验证
虽然TMT和iTRAQ等体外标记定量方法可以很好地用于组织样品的差异蛋白质筛选,但有文献[9,10]报道该方法由于肽段共洗脱的原因,会存在定量比值压缩效应,造成定量差异低于实际差异,且压缩效应与样品复杂程度相关。虽然在本文中我们采用了高pH值液相色谱预分离的方法以降低每个组分的复杂程度来减少肽段共洗脱的情况,但是仍然无法避免有些肽段会存在共洗脱而干扰定量结果。此外,基于TMT标记的发现蛋白质组学方法只适合于前期的差异蛋白质高通量筛选,最终所筛选到的蛋白质是否在临床中有实际意义,仍然需要进行后期验证。在传统的实验体系中一般采取Western blotting的方法进行蛋白质验证,该方法操作简单、实用。但是,由于该方法依赖于抗体,需要首先购买到高选择性的抗体,对于某些没有合适抗体的蛋白质无法使用,且单次验证的蛋白质数量有限,无法进行高通量的验证实验。本文中我们采用了靶向蛋白质组学的方法进行后期差异蛋白质验证。靶向蛋白质组学技术可有目的地检测目标蛋白质,具有选择性和灵敏度高等优点,被认为可补充和替代Western blotting用于差异蛋白质的后期验证[11]。该方法能够进行高通量、高准确度的蛋白质定量,可同时对多达100个蛋白质进行验证实验。PRM是近期发展起来的一种靶向蛋白质组学方法,目前已有较多文献使用该方法进行差异蛋白质验证[5-8]。
我们在差异变化大于1.5倍的70个蛋白质中选取了7个蛋白质进行PRM靶向蛋白质组学验证。AFP是目前最为常用的肝癌生物标志物,在肝癌病人体内会显著升高[3]。此外,文献[12-15]报道热休克蛋白(HSP90)和脂肪酸结合蛋白5(FABP5)在癌症患者体内会显著升高,而乙醇脱氢酶4(ADH4)在肝癌患者体内显著降低,均有潜力成为肝癌生物标志物。这4个蛋白质在本文的TMT差异蛋白质筛选实验中均被定量检测到且变化趋势与文献报道一致。另外,我们还在差异变化蛋白质列表中选择了3个文献没有报道的蛋白质进行后续验证,分别是纤粘蛋白(FN1)、脂肪酸结合蛋白1(FABP1)和乙醇脱氢酶1B (ADH1B)。
PRM的定量结果及与TMT定量结果的比较见表1和附表2(http://www.chrom-China.com/UserFiles/File/sp1705022-supporting%20info.pdf)。从结果中可以发现,TMT和PRM的定量结果总体趋势非常一致,其中AFP、HSP90AB1、FABP5和FN1 4个蛋白质在肝癌组织中显著上调,而FABP1、ADH1B和ADH4 3个蛋白质在肝癌组织中显著下调。该结果说明了实验体系的稳定性和定量的准确性。但是,从结果中我们也可以看出TMT实验仍然有一部分存在定量比例压缩效应,包括FN1、FABP5、FABP1和ADH1B等。由此可见,使用TMT和iTRAQ定量方法筛选的差异蛋白质必须经过后期的准确验证。本文筛选到的差异变化蛋白质有可能成为肝癌组织的标志蛋白质,但是否具有临床价值仍然需要使用大规模的临床样品进行验证,本文的结果将为肝癌标志物的临床筛选提供有效依据。

图 3 基于GO和KEGG注释的HCC差异表达蛋白质功能富集分析Fig. 3 Enrichment analysis of the differentially expressed proteins in HCC based on GO and KEGGGO: gene ontology; KEGG: Kyoto encyclopedia of genes and genomes. HCC: hepatocellular carcinoma.

图 4 肝癌差异表达蛋白质的相互作用网络图Fig. 4 Protein-protein network of the differentially expressed proteins in HCC a. the proteins enriched in pathways of spliceosome, starch and sucrose metabolism and retinol metabolism; b. the protein-protein network. C1, P1, C2, P2, C3, P3: sample numbers; C: cancer; P: paracancerous.
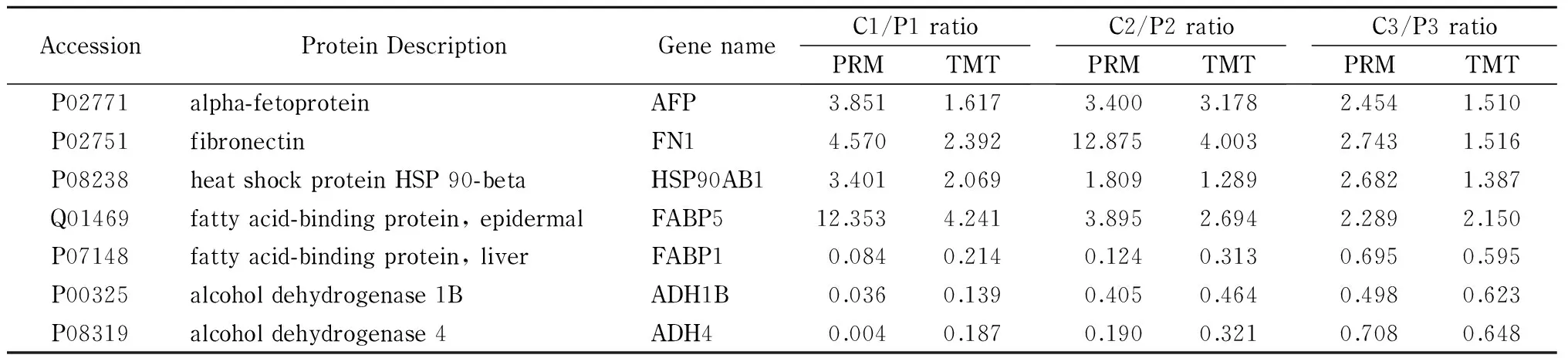
AccessionProteinDescriptionGenenameC1/P1ratioPRMTMTC2/P2ratioPRMTMTC3/P3ratioPRMTMTP02771alpha-fetoproteinAFP3.8511.6173.4003.1782.4541.510P02751fibronectinFN14.5702.39212.8754.0032.7431.516P08238heatshockproteinHSP90-betaHSP90AB13.4012.0691.8091.2892.6821.387Q01469fattyacid-bindingprotein,epidermalFABP512.3534.2413.8952.6942.2892.150P07148fattyacid-bindingprotein,liverFABP10.0840.2140.1240.3130.6950.595P00325alcoholdehydrogenase1BADH1B0.0360.1390.4050.4640.4980.623P08319alcoholdehydrogenase4ADH40.0040.1870.1900.3210.7080.648
3 结论
本文中我们通过结合基于TMT标记的高通量蛋白质组相对定量方法和基于PRM的靶向蛋白质组定量方法,对肝癌组织和癌旁组织中的差异蛋白质进行了高通量的筛选和验证,建立了一种基于蛋白质组学定量技术的癌症生物标志物筛选和验证方法。实验证明该方法能够很好地用于差异蛋白质的筛选,且定量准确,所筛选到的蛋白质包含临床已经使用的和文献报道的肝癌生物标志物。本文所筛选到的差异变化蛋白质将为肝癌标志物的临床筛选提供有效依据。此外,该方法可用于各种其他癌症样品的差异蛋白质筛选和验证,具有较大的应用价值。
[1] Farazi P A, DePinho R A. Nat Rev Cancer, 2006, 6(9): 674
[2] Wei K R, Peng X B, Liang Z H, et al. China Cancer, 2015, 24(8): 621
魏矿荣, 彭侠彪, 梁智恒, 等. 中国肿瘤, 2015, 24(8): 621
[3] Gao W F, Zheng J S, Sun B. Contemporary Medicine, 2010, 16(32): 19
高文峰, 郑加生, 孙斌. 当代医学, 2010, 16(32): 19
[4] Peterson A C, Russell J D, Bailey D J, et al. Mol Cell Proteomics, 2012, 11: 1475
[5] Kim H J, Lin D, Lee H J, et al. Mol Cell Proteomics, 2016, 15(2): 682
[6] Urisman A, Levin R S, Gordan J D, et al. Mol Cell Proteomics, 2017, 16(2): 265
[7] Uzozie A C, Selevsek N, Wahlander A, et al. Mol Cell Proteomics, 2017, 16(3): 407
[8] Jung S, Choi J, Rousseaux M, et al. Mol Cell Proteomics, 2017, 16(4): 581
[9] Savitski M M, Mathieson T, Zinn N, et al. J Proteome Res, 2013, 12(8): 3586
[10] Zhang B, Wang J, Wang X J, et al. Nature, 2014, 513(7518): 382
[11] Aebersold R, Burlingame A L, Bradshaw R A. Mol Cell Proteomics, 2013, 12(9): 2381
[12] Yim S H, Chung Y J. Cancers, 2010, 2(2): 809
[13] Ohata T, Yokoo H, Kamiyama T, et al. J Clin Oncol, 2014, 32(15): e15110
[14] Ohata T, Yokoo H, Kamiyama T, et al. J Clin Oncol, 2015, 33(3): S310
[15] Wei R R, Zhang M Y, Rao H L, et al. Med Oncol, 2012, 29(4): 2737
Stable isotope labeling and parallel reaction monitoring-based proteomic quantification for biomarker screening and validation of hepatocellular carcinoma
WANG Sulan, GAO Huaping, ZHANG Jing, YE Xiang*
(Sir Run Run Shaw Hospital Xiasha Branch, Zhejiang University, Hangzhou 310018, China)
Liver cancer is the fifth most common cancer with extremely low five year survival rate. Early diagnosis is of great importance for cancer therapy. In this work, stable isotope labeling-based relative quantitative proteomics and parallel reaction monitoring-based target proteomics were combined for cancer biomarker screening and validation. By using this strategy, 70 significantly changed proteins in hepatocellular carcinoma tissues were obtained, among which seven proteins were further validated. The validated proteins contain the clinically used hepatocellular carcinoma (HCC) biomarker alpha-fetoprotein (AFP) and the reported biomarker candidates Heat shock protein HSP 90-beta (HSP90), fatty acid-binding protein, epidermal (FABP5) and alcohol dehydrogenase 4 (ADH4), which demonstrated the robustness of the strategy. The proteins identified in this work could be benefit for further HCC biomarker screening and clinical validation. Moreover, this strategy could be further applied to other cancer types.
proteomics; stable isotope labeling; parallel reaction monitoring; hepatocellular carcinoma (HCC)
10.3724/SP.J.1123.2017.05022
2017-05-30
.Tel:(0571)87887161,E-mail:Yesxiang@126.com.
O658
A
1000-8713(2017)09-0934-07
*