新疆核桃野生种及4 个栽培品种的遗传多样性分析
2017-08-31张宏祥张明理桂东伟
张宏祥, 张明理,2,①, 桂东伟, 姜 黎
(1. 中国科学院新疆生态与地理研究所 中国科学院干旱区生物地理与生物资源重点实验室, 新疆 乌鲁木齐 830011;2. 中国科学院植物研究所, 北京 100093; 3. 新疆策勒荒漠草地生态系统国家野外科学观测研究站, 新疆 策勒 848300)
新疆核桃野生种及4 个栽培品种的遗传多样性分析
张宏祥1, 张明理1,2,①, 桂东伟3, 姜 黎1
(1. 中国科学院新疆生态与地理研究所 中国科学院干旱区生物地理与生物资源重点实验室, 新疆 乌鲁木齐 830011;2. 中国科学院植物研究所, 北京 100093; 3. 新疆策勒荒漠草地生态系统国家野外科学观测研究站, 新疆 策勒 848300)
以新疆地区种植的4个核桃(JuglansregiaLinn.)栽培品种(包括新栽培品种‘温185’和‘新新2’以及老栽培品种‘新丰’和‘扎343’)及巩留野核桃自然保护区生长的野生核桃为研究对象,对其cpDNA的psbK-psbI区间和mtDNA的COX2 intron Ⅰ区间以及nrDNA的ITS和ETS区间的DNA片段序列进行了比较分析,并对其MP、ML和UPGMA系统发育树进行了分析;此外,还基于SSR分子标记结果对其进行了遗传多样性指数、UPGMA系统发育树和遗传分组分析。结果表明:野生种与4个栽培品种的cpDNA和mtDNA片段序列无碱基变异,而其nrDNA的片段序列却存在3个碱基变异,但4个栽培品种间无碱基变异。以麻核桃(J.hopeiensisHu)为外类群,基于上述4个DNA片段序列构建的MP、ML和UPGMA系统发育树的聚类结果一致,均表现为4个栽培品种聚为一组,而野生种和麻核桃则分别单独聚为一组。野生种的观测杂合度、预期杂合度和固定指数分别为0.383、0.448和0.153,4个栽培品种的上述3个遗传多样性指数分别为0.428~0.576、0.423~0.619和-0.043~0.234。基于SSR分子标记结果的UPGMA系统发育树和分组数为5的遗传分组结果均表明:野生种和品种‘温185’分别单独为一组;品种‘新新2’和‘新丰’为一组;而品种‘扎343’也单独为一组,但与品种‘新新2’和‘新丰’遗传关系较近。遗传分组结果还表明:分组数为3更利于明确品种‘扎343’的分组地位,此时,其与品种‘新新2’和‘新丰’为一组。综合分析结果表明:核桃4个栽培品种间的遗传差异较小,且老栽培品种的遗传多样性总体上高于新栽培品种;野生种与栽培品种间具有明显的遗传差异,说明在育种或栽培过程中核桃种质资源的遗传多样性可能会逐渐降低,并且,该野生种可为核桃的分子育种提供天然的基因库资源。
核桃; 野生种; 栽培品种; 遗传多样性; DNA片段序列; SSR分子标记
核桃(JuglansregiaLinn.)又名胡桃,隶属胡桃属(JuglansLinn.),为“世界四大坚果”树种之一,拥有悠久的栽培历史。在选育过程中,核桃新品种通常较老品种具有更高的经济价值[1]。但是,随着核桃新品种的不断选育,为了达到形态和遗传的一致性,各品种间的遗传多样性却逐渐丧失。这种现象已经在一些动物的育种过程中得到实验验证[2-4]。然而,从种质资源角度来看,被替代的一些核桃老品种的某些特殊遗传特性仍可用于培育核桃新品种或改良已有的核桃品种,因此,对于选育核桃新品种而言,保护和利用好新、老品种的种质资源十分重要。相关研究人员已经对核桃与其同属近缘种间的亲缘关系[5-6]、野生种群间的遗传多样性[7-8]以及栽培品种间的亲缘关系[9-11]等进行了研究,认为核桃与泡核桃(J.sigillataDode)间存在遗传渗入[5],且其栽培品种存在多个属于不同地理和生态类型的遗传组[10-11]。但是,这些研究并没有对核桃新品种与老品种间的亲缘关系及其与野生种间的亲缘关系进行分析,而明确上述关系对于培育核桃新品种及其品种改良均具有重要作用。
新疆是中国重要的核桃种植区和野生核桃分布地,具有丰富的核桃种质资源。目前,新疆地区种植了多个优良的核桃品种,尤以‘温185’(‘Wen185’)和‘新新2’(‘Xinxin2’)等薄皮核桃品种为代表。另外,在新疆巩留野核桃自然保护区还分布有原始野生核桃种群。鉴于此,作者对新疆地区种植的4个核桃栽培品种(包括2个新栽培品种和2个老栽培品种)和巩留野核桃自然保护区生长的野生核桃cpDNA的psbK-psbI区间和mtDNA的COX2 intron Ⅰ区间以及nrDNA的ITS和ETS区间的DNA片段序列进行比较分析和系统发育树分析,并对上述5个供试材料进行SSR分子标记分析,在SSR分子标记结果基础上进行遗传多样性指数、系统发育树和遗传分组分析,以期明确供试核桃野生种与4个栽培品种间的亲缘关系,并评估其遗传多样性,以揭示人工选育对核桃种质资源遗传多样性的影响,为核桃遗传育种研究提供参考资料。
1 材料和方法
1.1 材料
供试的2个核桃新栽培品种‘温185’和‘新新2’以及2个核桃老栽培品种‘新丰’(‘Xinfeng’)和‘扎343’(‘Za343’)植株均种植在新疆温宿核桃林场,并且均从温宿木本粮油林场核桃园的早实核桃实生群体中选育而来[12],供试的核桃野生种植株则生长在新疆巩留野核桃自然保护区内,5个材料依次编号为1、2、3、4、5,采样植株数分别为25、25、18、17、19。每个植株选取1枚嫩叶,共采集104枚叶片,将采集的叶片立即放入装有硅胶的自封袋中干燥并带回实验室,于室温条件下保存、备用。
扩增反应使用的所有试剂均购自生工生物工程(上海)股份有限公司,所有引物合成和序列分析也均由该公司完成;使用的PCR仪为ABI Verit96梯度PCR仪(美国ABI公司)。
1.2 方法
1.2.1 基因组总DNA的提取 每个单株取经硅胶干燥的叶片约50 mg,采用CTAB法[13]提取叶片中的基因组总DNA。
1.2.2 DNA片段序列的扩增和分析 每个材料随机选取5个单株的基因组总DNA样品,对mtDNA的COX2 intron Ⅰ区间、cpDNA的psbK-psbI区间以及nrDNA的ITS和ETS区间的DNA片段序列进行PCR扩增,扩增体系总体积为30 μL,扩增体系的具体组分和扩增程序均按照Zhang等[14]的条件完成,4个DNA片段扩增使用的引物序列见表1。对扩增出的所有DNA片段进行测序,将所有原始序列进行人工校正和Clustal X比对[15],并将获得的DNA片段序列上传至GenBank数据库(GenBank登录号为KY809038至KY809057)。
表1 用于新疆核桃4个栽培品种和1个野生种不同DNA片段扩增的引物序列
Table 1 Sequences of primers used for amplifying different DNA fragments of four cultivars and one wild species ofJuglansregiaLinn. in Xinjiang
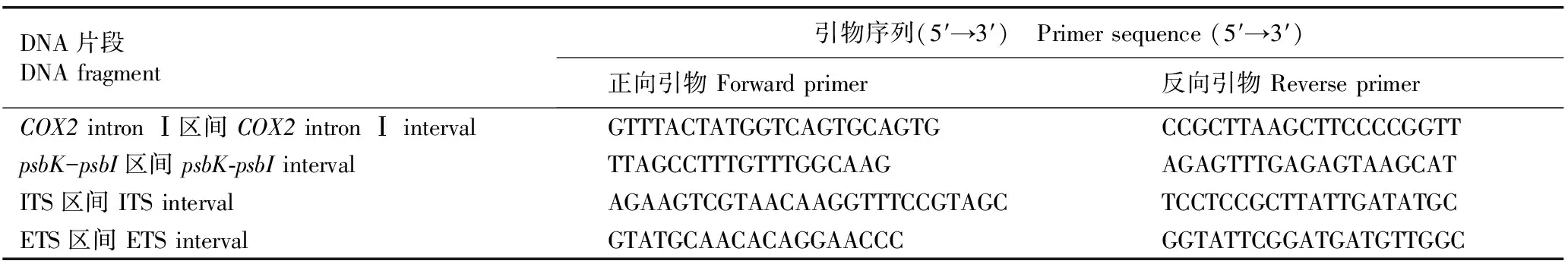
DNA片段DNAfragment引物序列(5′→3′) Primersequence(5′→3′)正向引物Forwardprimer反向引物ReverseprimerCOX2intronⅠ区间COX2intronⅠintervalGTTTACTATGGTCAGTGCAGTGCCGCTTAAGCTTCCCCGGTTpsbK-psbI区间psbK⁃psbIintervalTTAGCCTTTGTTTGGCAAGAGAGTTTGAGAGTAAGCATITS区间ITSintervalAGAAGTCGTAACAAGGTTTCCGTAGCTCCTCCGCTTATTGATATGCETS区间ETSintervalGTATGCAACACAGGAACCCGGTATTCGGATGATGTTGGC
1.2.3 SSR分子标记分析 根据相关文献[16-17]设计SSR引物,经过预实验,最终筛选出具有多态性且容易扩增的5对SSR引物,并在引物序列的5′端添加荧光信号,各引物序列见表2。对供试所有单株的基因组总DNA样品分别进行PCR扩增,扩增体系总体积为30 μL,扩增体系的具体组分和扩增程序也按照Zhang等[14]的条件完成。对所有扩增出的DNA片段进行测序,并通过GeneMapper软件对测定的序列进行校正。
表2 用于新疆核桃4个栽培品种和1个野生种SSR分子标记分析的引物序列
Table 2 Sequences of primers used for SSR molecular marker analysis of four cultivars and one wild species ofJuglansregiaLinn. in Xinjiang

引物编号Primercode引物序列(5′→3′) Primersequence(5′→3′)正向引物Forwardprimer反向引物ReverseprimerWGA001ATTGGAAGGGAAGGGAAATGCGCGCACATACGTAAATCACWGA004TGTTGCATTGACCCACTTGTTAAGCCAACATGGTATGCCAWGA032CTCGGTAAGCCACACCAATTACGGGCAGTGTATGCATGTAWGA321TCCAATCGAAACTCCAAAGGGTCCAAAGACGATGATGGAWGA376GCCCTCAAAGTGATGAACGTTCATCCATATTTACCCCTTTCG
1.3 数据处理及分析
首先,基于测定的DNA片段序列确定核桃4个栽培品种和1个野生种间的碱基变异信息;然后,以麻核桃(J.hopeiensisHu)为外类群,基于测定的4个DNA片段序列数据,使用MEGA 6.0 软件[18]、采用MP、ML和UPGMA 3种方法构建系统发育树,并采用靴带检验法计算各分支的靴带支持率,重复计算1 000次。
基于SSR分子标记结果,运用GenAlEx 6.5软件[19]计算每个核桃栽培品种和野生种的遗传多样性指数,包括差异等位基因数、观测杂合度、期望杂合度和固定指数;根据SSR分子标记数据计算不同样品间的遗传距离,运用MEGA 6.0软件构建UPGMA系统发育树。
基于SSR分子标记结果,运用STRUCTURE 2.3软件[20]进行Bayesian遗传结构分析。分组数(K)设为1~8,分别进行10次独立运算;Burn-in指数设为100 000,并执行500 000步的Markov Chain Monte Carlo模拟;依据估算模型的后验概率〔lnP(D)〕,按照Evanno等[21]的方法计算ΔK值,并根据ΔK值的最大值来确定供试核桃4个栽培品种和1个野生种的最优分组数。运用CLUMPP 1.1.2软件[22]将STRUCTURE 2.3软件的10次运算结果进行合并,并采用DISTRUCT 1.1软件[23]制图。
2 结果和分析
2.1 DNA片段序列的扩增结果及系统发育树分析
在基于新疆核桃4个栽培品种和1个野生种的4个DNA片段序列比对后构建的联合数据矩阵中,序列总长度为2 678 bp。在母系遗传的cpDNA和mtDNA片段序列中,4个栽培品种与野生种间无碱基变异;而在双亲遗传的nrDNA片段序列中,野生种与4个栽培品种间存在3个碱基变异,但4个栽培品种间无碱基变异。
比较结果显示:基于上述4个DNA片段序列的联合数据矩阵,运用MP、ML和UPGMA方法构建的新疆核桃4个栽培品种和1个野生种的系统发育树(图1)的聚类结果完全一致。由图1可见:供试核桃的4个栽培品种聚为一组,而野生种和外类群麻核桃则分别单独聚为一组,并且,系统发育树中各分支的靴带支持率均很高。
2.2 基于SSR分子标记的相关分析
2.2.1 遗传多样性指数分析 新疆核桃4个栽培品种和1个野生种4个遗传多样性指数(包括等位基因数、观测杂合度、预期杂合度和固定指数)的比较结果见表3。

1: ‘温185’ ‘Wen185’; 2: ‘新新2’ ‘Xinxin2’; 3: ‘新丰’ ‘Xinfeng’; 4: ‘扎343’ ‘Za343’; 5: 野生种 Wild species; 6: 麻核桃Juglans hopeiensis Hu. 每个分支上的3个数据依次代表MP、ML和UPGMA系统发育树的靴带支持率 Three datums above each branch successively indicate bootstrap values of MP, ML and UPGMA phylogenetic trees.图1 基于4个DNA片段序列的新疆核桃4个栽培品种和1个野生种的系统发育树Fig. 1 Phylogenetic tree of four cultivars and one wild species of Juglans regia Linn. in Xinjiang based on four DNA fragment sequences



编号1)Code1)等位基因数Allelenumber观测杂合度Observedheterozygosity预期杂合度Expectedheterozygosity固定指数Fixationindex1240 428±0 1840 423±0 1210 0742240 576±0 1590 539±0 074-0 0433170 555±0 1480 569±0 0940 1034160 516±0 1250 619±0 0860 2345180 383±0 0780 448±0 0760 153
1)1: ‘温185’ ‘Wen185’; 2: ‘新新2’ ‘Xinxin2’; 3: ‘新丰’ ‘Xinfeng’; 4: ‘扎343’ ‘Za343’; 5: 野生种 Wild species.
由表3可见:新栽培品种‘温185’和‘新新2’的等位基因数最多,均为24;老栽培品种‘新丰’和‘扎343’的等位基因数较少,分别为17和16;野生种的等位基因数居中,为18。‘新新2’的观测杂合度最高(0.576),野生种最低(0.383),‘新丰’和‘扎343’较高,而‘温185’较低;‘扎343’的预期杂合度最高(0.619),‘温185’最低(0.423),‘新丰’和‘新新2’较高,而野生种较低;‘新新2’的固定指数为-0.043,其余3个栽培品种和野生种的固定指数均为正值,其中,‘扎343’的固定指数最高(0.234),野生种和‘新丰’次之(分别为0.153和0.103),说明‘扎343’、‘新丰’和野生种具有较高水平的近交繁殖特征。
2.2.2 UPGMA系统发育树分析 基于SSR分子标记构建的新疆核桃4个栽培品种和1个野生种的UPGMA系统发育树见图2。

1: ‘温185’ ‘Wen185’; 2: ‘新新2’ ‘Xinxin2’; 3: ‘新丰’ ‘Xinfeng’; 4: ‘扎343’ ‘Za343’; 5: 野生种 Wild species.图2 基于SSR分子标记的新疆核桃4个栽培品种和1个野生种的UPGMA系统发育树Fig. 2 UPGMA phylogenetic tree of four cultivars and one wild species of Juglans regia Linn. in Xinjiang based on SSR molecular marker
由图2可见:在构建的UPGMA系统发育树中,野生种和新栽培品种‘温185’分别单独为一组;新栽培品种‘新新2’与老栽培品种‘新丰’为一组;老栽培品种‘扎343’也单独为一组,但是其与‘新新2’和‘新丰’的遗传关系较近。
2.2.3 遗传分组分析 STRUCTURE 2.3软件的分析结果表明:后验概率〔lnP(D)〕随分组数(K)的增大而逐渐升高(图3-A),根据ΔK值确定的最优分组数为5(图3-B)。
遗传分组结果表明:K值为5时,新疆核桃4个栽培品种和1个野生种的分组结果(图4-A)与其基于SSR分子标记结果的UPGMA系统发育树的聚类结果相同,即野生种单独为一组;新栽培品种‘温185’单独为一组;新栽培品种‘新新2’与老栽培品种‘新丰’为一组;老栽培品种‘扎343’的分组比较混乱(内部个体形成2组,其中一组与品种‘新新2’和‘新丰’一组近缘,另一组则单独为一组),或单独为一组。可见,K值为5的分组结果不利于明确品种‘扎343’的分组地位。
为了更清晰地明确品种‘扎343’的分组地位,对K值为3时的分组情况进行了分析(图4-B),结果表明:品种‘扎343’、‘新新2’和‘新丰’为一组,而野生种和品种‘温185’分别单独为一组。
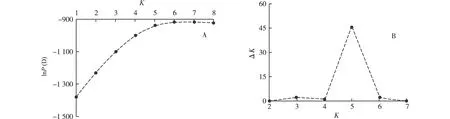
lnP(D): 后验概率 Posterior probability; K: 分组数 Cluster number.图3 新疆核桃4个栽培品种和1个野生种的分组分析Fig. 3 Analysis on cluster of four cultivars and one wild species of Juglans regia Linn. in Xinjiang
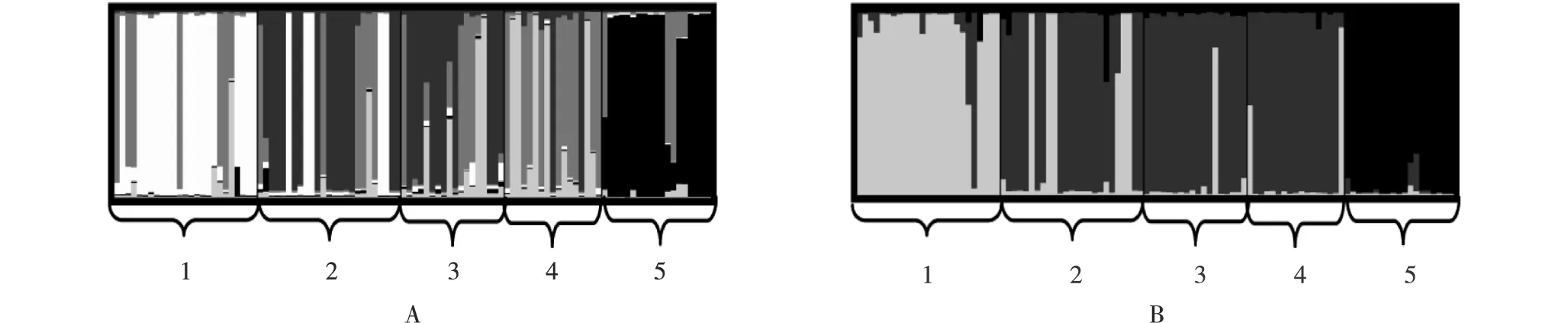
1: ‘温185’ ‘Wen185’; 2: ‘新新2’ ‘Xinxin2’; 3: ‘新丰’ ‘Xinfeng’; 4: ‘扎343’ ‘Za343’; 5: 野生种 Wild species.图4 分组数为5(A)和3(B)时新疆核桃4个栽培品种和1个野生种的遗传分组结果Fig. 4 Genetic cluster results of four cultivars and one wild species of Juglans regia Linn. in Xinjiang with cluster number of 5 (A) and 3 (B)
3 讨论和结论
不同品种间的亲缘关系和遗传多样性差异是评估园艺植物种质资源状况的重要依据。本研究采用cpDNA的psbK-psbI区间、mtDNA的COX2 intron Ⅰ区间以及nrDNA的ITS和ETS区间4个DNA片段序列和SSR分子标记对新疆核桃4个栽培品种(包括2个新栽培品种和2个老栽培品种)和1个野生种(生长在新疆巩留野核桃自然保护区)间的亲缘关系和遗传多样性差异进行了研究。结果表明:基于4个DNA片段序列构建的MP、ML和UPGMA系统发育树的聚类结果完全一致,4个栽培品种直接聚为一组,说明他们具有相同的基因型,并且各栽培品种间的遗传差异较小。
基于SSR分子标记构建的UPGMA系统发育树和分组数(K)为3时的分组结果均显示:新栽培品种‘新新2’与老栽培品种‘新丰’和‘扎343’为一组,而新栽培品种‘温185’却单独为一组,说明品种‘新新2’继承了2个老栽培品种的种质资源,而‘温185’与2个老栽培品种间的种质资源却存在明显差异。‘扎343’和‘新丰’是20世纪70年代至80年代选育出的核桃优良栽培品种,而‘温185’和‘新新2’则是20世纪80年代末至90年代基于更高标准选育出的更加优良的核桃新栽培品种[24-25]。这就导致有些新栽培品种可能继承了老栽培品种的一些种质资源,还有些新栽培品种与老栽培品种间的种质资源可能存在一定的遗传差异。
遗传分组结果表明:K值为5时,老栽培品种‘扎343’表现出比较特殊的遗传结构,说明‘扎343’存在特殊的种质资源,这些特殊的种质资源应该受到保护和保存,以便在日后的分子育种过程中得以利用。王国安等[12]曾利用老栽培品种‘扎343’二次果核桃的早实性从其实生群体中选育出优良的矮化核桃单株,从而培育出核桃矮化新品种。实际上,核桃的野生种与栽培品种同属于1个物种,因此,他们在母系遗传的mtDNA的COX2 intron Ⅰ区间和cpDNA的psbK-psbI区间的片段序列间保留了遗传同一性。而基于双亲遗传的nrDNA的ITS和ETS区间的片段序列以及基于SSR分子标记结果的系统发育树和遗传分组结果却显示新疆核桃野生种与栽培品种间具有显著的遗传差异,说明新疆核桃野生种可为其分子育种提供天然的基因资源,这些基因资源对于核桃的品种改良和新品种培育具有重大作用。然而,本研究中,新疆核桃野生种的观测杂合度(0.383)较低,固定指数却较高(0.153),说明该野生种群的遗传多样性较低,但种群内的近交繁殖水平却较高,表明新疆核桃野生种群已经出现遗传同一性现象,急需开展保育工作。
Gunn等[5]认为,人类的居住环境和家族谱系关系等人类活动对核桃在地理空间上的遗传变异有较大影响。此外,为了获取较高的经济价值和遗传稳定性,人们不断开展的定向选择育种也会对核桃的种质资源造成一定的影响。本研究中,2个核桃老栽培品种的遗传多样性水平高于2个核桃新栽培品种,说明在核桃育种过程中可能会逐渐降低其种质资源的遗传多样性水平。而且,供试的3个核桃栽培品种‘温185’、‘新丰’和‘扎343’的固定指数均为正值,说明这3个核桃栽培品种内部存在较高水平的近交繁殖现象,表明选择性育种或者栽培过程中的无性繁殖可能会增加核桃种质资源的纯合度。
本研究中,K值为5时,老栽培品种‘扎343’的内部衍生出2个遗传类型,其中,一个类型与新栽培品种‘新新2’和老栽培品种‘新丰’近缘,另一个类型则自成一组。这可能是因为供试的多数核桃栽培品种的观测杂合度较高,在遗传分组时若设置的K值较大,则会造成品种‘扎343’内部的部分个体形成新的分组,因此,在对核桃栽培品种进行遗传分组时,应设置合适的K值,否则不利于探查不同核桃栽培品种间的遗传分组情况。
[1] 张 强, 虎海防, 李西萍. 七个新疆良种核桃品质评价分析[J]. 北方园艺, 2010, 34(19): 16-17.
[2] 赵广泰, 刘贤德, 王志勇, 等. 大黄鱼连续4代选育群体遗传多样性与遗传结构的微卫星分析[J]. 水产学报, 2010, 34(4): 500-507.
[3] 陈雪峰, 杨国梁, 孔 杰, 等. 人工养殖与选育对罗氏沼虾遗传多样性的影响[J]. 水生生物学报, 2012, 36(5): 866-873.
[4] LI Z X, LI J, WANG Q Y, et al. The effects of selective breeding on the genetic structure of shrimpFenneropenaeuschinensispopulations[J]. Aquaculture, 2006, 258: 278-282.
[5] GUNN B F, ARADHYA M, SALICK J M, et al. Genetic variation in walnuts (JuglansregiaandJ.sigillata; Juglandaceae): species distinctions, human impacts, and the conservation of agrobiodiversity in Yunnan, China[J]. American Journal of Botany, 2010, 97: 660-671.
[6] WANG H, PAN G, MA Q G, et al. The genetic diversity and introgression ofJuglansregiaandJuglanssigillatain Tibet as revealed by SSR markers[J]. Tree Genetics and Genomes, 2014, 11: 1-11.
[7] 王东升, 辛 红, 邢世岩, 等. 基于AFLP标记的山东省5个野核桃群体的遗传多样性分析[J]. 植物资源与环境学报, 2013, 22(3): 63-69.
[8] 张 捷, 李勤霞, 张 萍, 等. 基于SRAP分子标记新疆野核桃的遗传多样性分析[J]. 植物遗传资源学报, 2016: 17(2): 239-245.
[9] CHEN L N, MA Q G, CHEN Y K, et al. Identification of major walnut cultivars grown in China based on nut phenotypes and SSR markers[J]. Scientia Horticulturae, 2014, 168: 240-248.
[10] 李 超, 罗淑萍, 秦 伟, 等. 新疆核桃亲缘关系的ISSR分析[J]. 新疆农业科学, 2010, 47(9): 1722-1727.
[11] 刘朝斌, 张文辉. 核桃种质资源遗传多样性的SSR分析[J]. 西北植物学报, 2012, 32(6): 1118-1125.
[12] 王国安, 张 强, 阿卜杜许库尔·牙合甫. 新温609、新温724等6个核桃矮化品种的选育[J]. 新疆农业科学, 2011, 48(7): 1183-1188.
[13] DOYLE J J. A rapid DNA isolation procedure for small quantities of fresh leaf tissue[J]. Phytochemical Bulletin, 1987, 19: 11-15.
[14] ZHANG H X, ZHANG M L, WANG L N. Genetic structure and historical demography ofMalussieversiiin the Yili Valley and the western mountains of the Junggar Basin, Xinjiang, China[J]. Journal of Arid Land, 2015, 7: 264-271.
[15] THOMPSON J D, GIBSON T J, PLEWNIAK F, et al. The Clustal X windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Research, 1997, 25: 4876-4882.
[16] DANGL G S, WOESTE K, ARADHYA M K, et al. Characterization of 14 microsatellite markers for genetic analysis and cultivar identification of walnut[J]. Journal of the American Society for Horticultural Science, 2005, 130: 348-354.
[17] WOESTE K, BURNS R, RHODES O, et al. Thirty polymorphic nuclear microsatellite loci from black walnut[J]. Journal of Heredity, 2002, 93: 58-60.
[18] TAMURA K, STECHER G, PETERSON D, et al. MEGA6: molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology and Evolution, 2013, 30: 2725-2729.
[19] PEAKALL R, SMOUSE P E. GenAlEx 6.5: genetic analysis in Excel. Population genetic software for teaching and research—an update[J]. Bioinformatics, 2012, 28: 2537-2539.
[20] PRITCHARD J K, STEPHENS M, DONNELLY P. Inference of population structure using multilocus genotype data[J]. Genetics, 2000, 155: 945-959.
[21] EVANNO G, REGNAUT S, GOUDET J. Detecting the number of clusters of individuals using the software STRUCTURE: a simulation study[J]. Molecular Ecology, 2005, 14: 2611-2620.
[22] JAKOBSSON M, ROSENBERG N A. CLUMPP: a cluster matching and permutation program for dealing with label switching and multimodality in analysis of population structure[J]. Bioinformatics, 2007, 23: 1801-1806.
[23] ROSENBERG N A. DISTRUCT: a program for the graphical display of population structure[J]. Molecular Ecology Resources, 2004, 4: 137-138.
[24] 张树信. 核桃新品种新早丰、新萃丰及新巨丰的选育[J]. 中国果树, 1990(3): 76-78.
[25] 张树信. 新疆核桃良种资源[J]. 新疆农业科学, 1989(3): 30-31.
(责任编辑: 佟金凤)
Analysis on genetic diversity of four cultivars and wild species ofJuglansregiain Xinjiang
ZHANG Hongxiang1, ZHANG Mingli1,2,①, GUI Dongwei3, JIANG Li1
(1. Key Laboratory of Biogeography and Bioresource in Arid Land, Xinjiang Institute of Ecology and Geography, Chinese Academy of Sciences, Urumqi 830011, China; 2. Institute of Botany, Chinese Academy of Sciences, Beijing 100093, China; 3. Cele National Station of Observation and Research for Desert-Grassland Ecosystem, Cele 848300, China),J.PlantResour. &Environ., 2017, 26(2): 10-16
Taking four cultivars (including newly cultivar of ‘Wen185’ and ‘Xinxin2’ and old cultivar of ‘Xinfeng’ and ‘Za343’) ofJuglansregiaLinn. planted in Xinjiang region and wildJ.regiagrown in Gongliu Wild Walnut Nature Reserve as research objects, the DNA fragment sequences ofpsbK-psbIinterval of cpDNA andCOX2 intron Ⅰ interval of mtDNA, and ITS and ETS intervals of nrDNA were comparatively analyzed, and their MP, ML and UPGMA phylogenetic trees were analyzed. Otherwise, based on SSR molecular marker results, their genetic diversity indexes, UPGMA phylogenetic tree and genetic cluster were also analyzed. The results show that there is no base variation of fragment sequences of cpDNA and mtDNA among wild species and four cultivars, while there are three base variations of fragment sequences of their nrDNA, but there is no base variation among four cultivars. TakingJ.hopeiensisHu as an outer group, the cluster results of MP, ML and UPGMA phylogenetic trees constructed based on above four DNA fragment sequences are identical, all appear that four cultivars are clustered into one group, while wild species andJ.hopeiensisare clustered into independent group, respectively. The observed heterozygosity, expected heterozygosity and fixation index of wild species are 0.383, 0.448 and 0.153, respectively, and the above three genetic diversity indexes of four cultivars are 0.428-0.576, 0.423-0.619 and -0.043-0.234, respectively. The results of UPGMA phylogenetic tree based on SSR molecular marker results and genetic cluster with cluster number of 5 both show that wild species and cultivar ‘Wen185’ are independent group, respectively, and cultivar ‘Xinxin2’and ‘Xinfeng’ are one group, while cultivar ‘Za343’ is also an independent group, but it has a closer genetic relationship with cultivar ‘Xinxin2’and ‘Xinfeng’. The genetic cluster results also show that cluster number of 3 is more suitable for defining the cluster status of cultivar ‘Za343’, right now, it is one group with cultivar ‘Xinxin2’and ‘Xinfeng’. The result of comprehensive analysis indicates that the genetic difference among four cultivars ofJ.regiais small, and in general, the genetic diversity of old cultivars is higher than that of newly cultivars. In addition, there are obvious genetic differences between wild species and cultivars, meaning that genetic diversity of germplasm resource ofJ.regiamight decrease gradually during breeding or cultivation processes, and the wild species can provide natural gene pool resource for molecular breeding ofJ.regia.
JuglansregiaLinn.; wild species; cultivar; genetic diversity; DNA fragment sequence; SSR molecular marker
2016-08-30
中国科学院西部之光项目(XBBS-2014-18); 中国科学院科技服务网络计划(STS)项目(KFJ-SW-STS-176); 中国科学院西部之光项目(XBBS-2014-12); 国家自然科学基金资助项目(31500271)
张宏祥(1986—),男,湖南沅陵人,博士,助理研究员,主要从事干旱区植物资源保护与植物地理研究。
①通信作者E-mail: zhangml@ibcas.ac.cn
Q946-33; S664.1.024
A
1674-7895(2017)02-0010-07
10.3969/j.issn.1674-7895.2017.02.02
