利用12S rRNA基因标记鉴定鱼翅真伪
2017-08-23黄娅琳
黄娅琳
(南京森林警察学院, 江苏 南京 210023; 国家林业局森林公安司法鉴定中心, 江苏 南京 210023)
利用12S rRNA基因标记鉴定鱼翅真伪
黄娅琳
(南京森林警察学院, 江苏 南京 210023; 国家林业局森林公安司法鉴定中心, 江苏 南京 210023)
为检测鱼翅样本真伪及鲨鱼种属,通过提取送检鱼翅、鱼翅羹样本DNA,扩增其线粒体DNA上的用于动物种属鉴定的12S rRNA基因片段,并进行DNA测序和序列分析,测序结果在GenBank上进行BLAST搜索,与数据库中相关物种序列进行同源性分析。结果表明,在送检的23份样本中,18份鱼翅羹样本、2份粉丝状鱼翅、1份翅状鱼翅中均未成功提取到DNA,未扩增出目的基因片段,而从2份鱼翅样本中成功地提取到了基因组总DNA,并成功扩增出了12S rRNA基因片段,这2份样本的12S rRNA基因片段的碱基序列分别与黑边鳍真鲨(Carcharhinuslimbatus)、斜锯牙鲨(Rhizoprionodonterraenovae)的同源性达到99%。对鉴定出含有鲨鱼成分的2份样本进行高温泡发处理,高温泡发实验表明,1份样本(22号)有明胶析出。研究证实,12SrRNA基因序列测定技术能准确、快速鉴定待检样本中是否含有鲨鱼成分,结合高温泡发时是否有明胶析出可综合判定鱼翅真伪。
鱼翅; 真伪; 12S rRNA; 基因标记; 种属鉴定
鱼翅是由某些种类的鲨鱼或鳐鱼的胸鳍、背鳍和尾鳍部分经过一系列加工而制成细丝状软骨的淡干品[1]。从鲨身体上割下的新鲜鱼鳍经去砂、去骨、干燥成片状后称为明翅,明翅煮后将细丝状软骨(翅筋)抽出,干燥成形后称为翅饼[2]。近年来,鱼翅越来越多地走进了寻常百姓的餐桌,成为商业晚宴、大型聚会的必点菜肴。同时,在鱼翅高昂价格的引诱下,各种造假和掺假鱼翅应运而生,极大地扰乱了市场秩序,侵害了消费者的合法权益。目前我国对鱼翅制品的鉴定还没有国家标准和行业标准,关于鱼翅的真假鉴别报道较少[3-5],主要靠行业内专家和正规酒楼的厨师根据多年的经验,通过眼看、手摸、口尝的方法对鱼翅的真假和优劣进行鉴别。利用DNA条形码技术或基因分子标记技术检测鱼翅类食品中鲨鱼成分国内外也仅有较少文献报道[2,4-11],目前还没有利用12S rRNA基因标记鉴别鱼翅真伪的报道,本研究通过提取疑似鱼翅样本DNA,扩增其线粒体DNA(mitochondrial DNA;mtDNA)上的12S rRNA基因片段,并对其进行DNA序列分析,以确定鱼翅样本真伪及鲨鱼种属,从而破获一起恶劣的食品造假案。
1 材料与方法
1.1 实验材料与仪器设备
本研究对浙江省消费者权益保护委员会送检的18份疑似鱼翅羹样本和5份疑似鱼翅样本进行实验。18份疑似鱼翅羹样本分别编号1~18号,5份鱼翅样本分别编号19~23号,如图1。
NanoDrop 2000型超微量蛋白核酸检测仪,美国Thermo公司;Veriti型PCR仪,美国ABI公司;3130xl型测序仪,美国ABI公司;Micro17R型台式高速冷冻离心机,美国Thermo公司;GeneGenius型全自动凝胶成像系统,英国Syngene公司;电泳仪、电泳槽,美国Bio -Rad公司。DNA提取试剂盒,日本Takara公司;PCR 产物纯化试剂盒,日本Takara公司;Big Dye Terminator Cycle Sequencing Kit Ver. 3.1型DNA测序试剂盒,美国ABI公司。

图1 送检的疑似鱼翅羹及鱼翅样本Fig.1 Suspected shark fin samples
1.2 实验方法
1.2.1 样本DNA的提取
从疑似鱼翅羹中取粉丝状样本,用纯净水将其表面的油脂及佐料冲洗干净,用干净的吸水纸吸干表面水分后,剪取约30 mg置于1.5 mL EP管中;固状疑似鱼翅样本剪取约20 mg 置于1.5 mL EP管中,将样本剪碎。样本基因组总DNA 均用DNA提取试剂盒(Takara公司)提取,提取方法参照试剂盒说明书。组织消化时间延长为10 h。将样品总DNA置于-20 ℃保存,待后续实验用。
1.2.2 DNA浓度检测及电泳检测
吸取2 μL所提取的DNA溶液加到NanoDrop 2000型超微量蛋白核酸检测仪的侦测台上,根据所测得的OD260值计算DNA的浓度。对鉴定出含DNA的样本进行琼脂糖凝胶电泳检测,电泳凝胶浓度为1.2%。
1.2.3 PCR扩增
扩增线粒体DNA上12S rRNA基因片段采用通用引物L1091和H1478[12],扩增反应在Veriti PCR 仪上进行。12S rRNA基因片段采用以下反应体系:总反应体积为25 μL,其中含2.5 pmol/μL引物各2 μL,2.5 mMdNTPs 2 μL,1.25 U Ex Taq酶0.25 μL,10×buffer 2.5 μL,25 mM MgCl21.5 μL,10~20 ng/μL模板1~2 μL,其他为灭菌蒸馏水。上述反应体系在94 ℃预变性3 min然后进入如下循环:94 ℃变性30 s,56 ℃退火30 s,72 ℃延伸1 min,以上一共38个循环,循环结束后72 ℃延伸7 min。扩增产物经1% 琼脂糖凝胶电泳,在凝胶成像系统(GENEGENUS)上检测、成像。
1.2.4 PCR扩增片段测序
PCR产物用PCR 产物(小量)纯化试剂盒纯化。以纯化产物为模板DNA,用ABI公司测序试剂盒进行测序PCR扩增(操作按试剂盒说明),扩增产物经甲酰胺变性后,上ABI公司3130xl测序仪进行测序。
1.2.5 BLAST搜索及序列对比
将测序结果在GenBank中进行BLAST搜索,并进行同源性分析比对,以确定鲨鱼种属。
1.2.6 高温泡发实验
一届届的学生与自己所在学校的校园建筑朝夕相处,难免每个人都会在学校的某个角落里,有着特别的记忆。一间教室、一间宿舍里都有可能有着一段难忘的回忆,但是这些故事情感与共鸣感的传播只局限在当事人的人际交往圈中,无法达到多个人际交往圈的重叠并产生更高程度上的情感共鸣。我们团队所做的校园漫游社交APP正是为此而生。团队旨在将这个APP建立成为一个情感寄托与联系的平台,为还在学校的以及已经毕业的莘莘学子,创造一个记录服务校园生活,分享交流对学校的情感,稳固学生与学校的关系,建立牢固关系纽带的一个记录分享服务型平台。
将含鲨鱼成分的疑似鱼翅样本剪取约50 g放置到70 ℃清水中浸泡30 min后,观察有无黏稠状明胶溢出。
1.2.7 淀粉检测实验
将含鲨鱼成分的疑似鱼翅样本剪取约50 g放置到70 ℃清水中浸泡30 min后,向浸泡液中滴入10%的碘溶液,观察是否有蓝色出现。
2 结果与分析
2.1 电泳检测结果及分析
1~21号样本通过超微量蛋白核酸检测仪检测,未检测到DNA,表明其提取的基因组总DNA溶液中不含DNA或DNA含量低。22号、23号样本的基因组总DNA的浓度分别为1 ng/μL和8 ng/μL。
由图2可知,23号样本DNA裂解液中检测到基因组总DNA,22号样本DNA裂解液通过琼脂糖凝胶电泳未检测到基因组总DNA,推测是由于22号样本DNA含量过低的缘故,未达到琼脂糖凝胶电泳的检测极限的缘故。
以22号和23号样本基因组总DNA为模板,扩增线粒体DNA上12S rRNA基因片段,结果如图3。
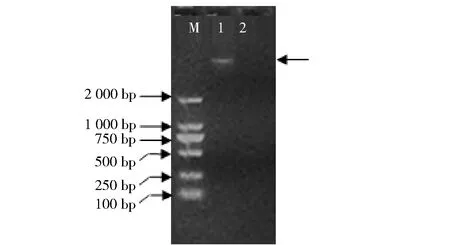
M为DL2000 分子量标记; 1列为23号样本总DNA;2列为22号样本总DNA。图2 样本基因组总DNA琼脂糖电泳Fig.2 Electropherogram of genome total DNA
由图3可见,检材通过PCR,得到了清晰的PCR
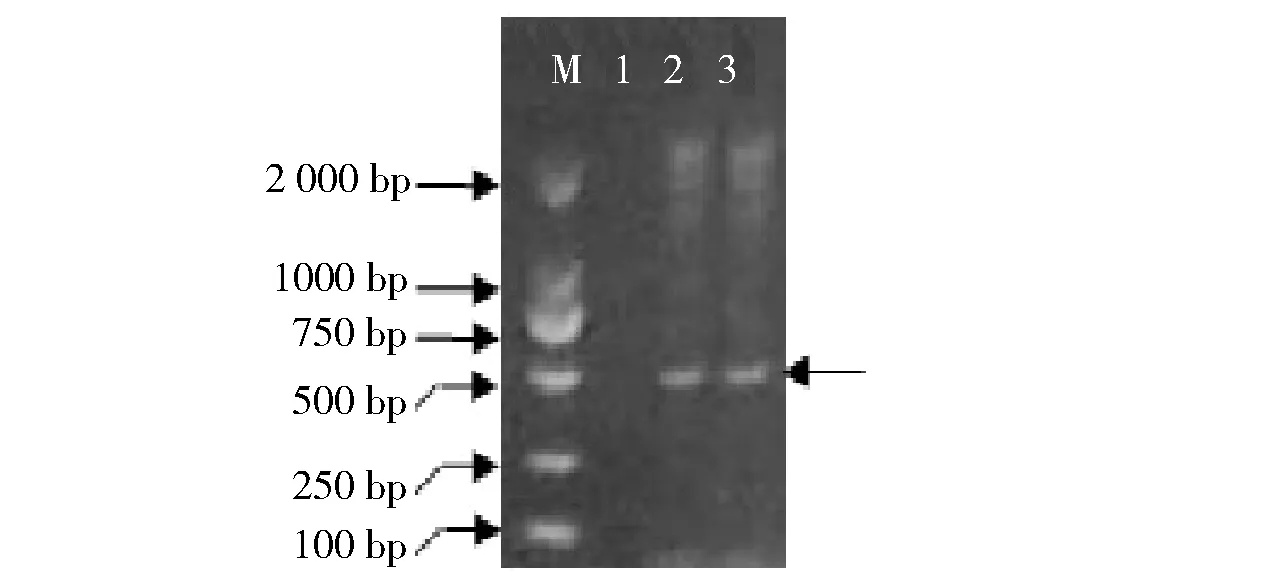
M列为DL2000分子量标记;1列为空白对照;2列为23号样本PCR产物;3列为22号样本PCR产物。图3 12S rRNA基因片段PCR扩增产物电泳Fig.3 Electropherogram of PCR product of 12S rRNA gene
产物(箭头所示),空白对照无扩增产物。
将PCR产物纯化后经3130xl测序仪进行测序,所得测序结果如图4。

图4 样品的mtDNA 12S rRNA基因序列Fig.4 Sequence of mtDNA 12S rRNA
由图4可知,22号和23号样本DNA扩增产物分别为379 bp和400 bp的基因序列。扩增产物序列经BLAST搜索、同源性分析,发现22号和23号样本DNA保守片段序列分别与黑边鳍真鲨(Carcharhinuslimbatus)、斜锯牙鲨(Rhizoprionodonterraenovae)线粒体DNA 12S rRNA基因序列的部分片段的同源性均高达99%,比对结果如图5和图6。

Sbjct为黑边鳍真鲨(Carcharhinus limbatus)12S rRNA基因部分序列;Query为22号样本12S rRNA基因部分序列。图5 黑边鳍真鲨(Carcharhinus limbatus)和22号样本12S rRNA基因序列比对Fig.5 Comparison of sample 22 sequence with 12S rRNA gene sequence of Carcharhinus limbatus

Sbjct为斜锯牙鲨(Rhizoprionodon terraenovae)12S rRNA基因部分序列;Query为23号样本12S rRNA基因部分序列。图6 斜锯牙鲨(Rhizoprionodon terraenovae)和23号样本12S rRNA基因序列比对(截自NCBI/blast)Fig.6 Comparison of sample 23 sequence with 12S rRNA gene sequence of Rhizoprionodon terraenovae
由图5和图6可知,第22、23号送检样本的种属来源分别被鉴定为黑边鳍真鲨(Carcharhinuslimbatus)、斜锯牙鲨(Rhizoprionodonterraenovae)。高温泡发实验结果表明22号样本经70 ℃清水浸泡30分钟后,有大量黏稠透明状明胶溢出,表明该样本含有明胶成分,推测22号样本是用鲨鱼肉打成粉和明胶混合后仿制而成的仿翅针。23号样本无明胶析出,表明不含明胶成分,向其热水浸泡液中滴入10%的碘溶液后溶液未变成蓝色,表明其中不含淀粉成分,因此23号样本被鉴定为真鱼翅。
3 讨 论
仿制鱼翅通常是以明胶、化学添加剂(淀粉配以一些色素)等作为主要原料制成,不含有鲨鱼成分,其在口感和外观等方面与天然的鱼翅很相似。此外,也有将鲨鱼肉料打成粉,并与明胶等混合可做成仿翅针,与真翅针形状极为相似,价格却比真翅针便宜很多[3]。现在市场上许多地方出售的假鱼翅产品制作工艺已经相当成熟,制作出的假鱼翅已经与真鱼翅在外观上很相近,无法从外形上辨别真伪,故本研究利用DNA检验技术,针对mtDNA上12S rRNA基因片段,利用通用引物进行PCR扩增,获得了高纯度的目的基因片段。并利用GenBank上丰富的序列资源,通过BLAST搜索、同源性比对,确定本案送检的22号、23号样品中分别含有黑边鳍真鲨(Carcharhinuslimbatus)、斜锯牙鲨(Rhizoprionodonterraenovae)成分。
本研究在基因位点的选择上,选用了线粒体DNA上的12S rRNA基因位点,由于疑似鱼翅检材常经过人工干燥或高温烹饪,通过细胞核DNA是无法提取到符合鉴定需求长度的DNA片段,通常高于200 bp以上的片段就无法扩增出来,而线粒体DNA具有环状双链的稳定结构、在细胞中拷贝数多等优点,非常适合降解、陈旧检材的鉴定。此外,线粒体12S rRNA基因的进化速率较快,不同物种间序列差异大,有利于设计针对目标物种的高特异性引物,且扩增该基因的稳定性和可重复性好。12S rRNA基因是在种内个体间的序列差异很小,而种间的序列差异却较大的DNA 片段,正是物种鉴别的理想标记。美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)的GenBank中有大量的12S rRNA基因片段比对序列资源,为未知物种的鉴定提供了极大的方便[13-14]。
本实验所检的从各大餐馆、酒楼购买的鱼翅羹样本(1~18号)、粉丝状鱼翅样本(19~21号)均不含鲨鱼成分,为假鱼翅样本;22号为用鲨鱼肉料打成粉,并与明胶等混合可做成的仿翅针,23号样本不含明胶和淀粉成分,含有斜锯牙鲨(Rhizoprionodonterraenovae)成分,为真鱼翅。本研究表明利用12S rRNA基因标记技术结合高温泡发实验能准确鉴定鱼翅真伪,为打击不法商贩和保证食品安全提供帮助。
[1] 杨维湘,林长治,赵丕扬.海味干货大全[M].上海:世界图书出版公司,2005:152-153.
[2] 黄文胜,韩建勋,董洁,等.FINS方法鉴定鱼翅和鲨鱼软骨的鲨鱼种类[J].食品科技,2011,36(11):265-271. HUANG W S,HAN J X,DONG J,et al.Species identification of shark fins and cartilages with FINS method[J].Food Science and Technology,2011,36(11):265-271.
[3] 郭云霞,冒海琳,张舒亚, 等.鱼翅的分类及鉴别方法[J].食品工业,2011(6): 96-99. GUO Y X, MAO H L, ZHANG S Y, et al. Classification and identification of shark fin[J]. Food Industry, 2011(6): 96-99.
[4] 郭云霞,张舒亚,谌鸿超,等.SYBR Green实时荧光PCR检测食品中鲨鱼源性成分真实性方法的建立[J].食品与生物技术学报,2012,31(12):1300-1306. GUO Y X,ZHANG S Y,SHEN H C,et al.Authentication of shark derived material in food using SYBR green fluorescence real -time PCR[J].Journal of Food Science and Biotechnology,2012,31(12):1300-1306.
[5] 覃芳芳,王德莲,冼燕萍,等.鱼翅类食品中鲨鱼成分 PCR 鉴定方法研究[J].现代食品科技,2014,30(4):274-278. QIN F F,WANG D L,XIAN Y P,et al.Identification of shark fins in food with PCR method[J].Modern Food Science and Technology,2014,30(4):274-278.
[6] ARMANI A,TINACCI L,XIONG X,et al. Fish species identification in canned pet food by BLAST and forensically informative nucleotide sequencing (FINS) analysis of short fragments of the mitochondrial 16s ribosomal RNA gene (16S rRNA)[J]. Food Control, 2015, 50: 821-830.
[7] BLANCO M, PÉREZ -MARTN R,SOTELO C. Identification of shark species in seafood products by forensically informative nucleotide sequencing (FINS)[J]. Journal of Agricultural and Food Chemistry, 2008, 56(21): 9868-9874.
[8] ABERCROMBIE D L,CLARKE S C,SHIVJI M S. Global -scale genetic identification of hammerhead sharks: appli -cation to assessment of the international fin trade and law enforcement[J]. Conservation Genetics, 2005, 6(5): 775-788.
[9] CHAPMAN D D,ABERCROMBIE D L,DOUADY C J, et al. A streamlined, bi -organelle, multiplex PCR approach to species identification: application to global conservation and trade monitoring of the great white shark,Carcharodoncarcharias[J]. Conservation Genetics, 2003, 4: 415-425.
[10] WONG H K,SHIVJIM S,HANNERR H. Identifying sharks with DNA barcodes:assessing the utility of a nucleotide diagnostic approach[J]. Molecular Ecology Resources, 2009, 9(1): 243-256.
[11] PINHAL D,GADIG O B,WASKO A P,et al. Discrimination of shark species by simple PCR of 5S rDNA repeats[J]. Genetics and Molecular Biology, 2008, 31(1): 361-365.
[12] KOCHER T,THOMAS W,MEYER A,et al. Dynamics of mitochondrial DNA evolution in animals: amplification and sequencing with conserved primers[J]. Proceedings of the National Academy of Sciences of the United States of America, 1989, 86(16): 6196-6200.
[13] 黄璐琦,唐仕欢,李军德,等.动物药材分子鉴定研究策略[J].中国中药杂志,2011,36(3):234-236. HUANG L Q,TANG S H,LI J D,et al.Research strategy on molecular identification of animal medical material[J].China Journal of Chinese Materia Medica,2011,36(3):234-236.
[14] 黄娅琳.mtDNA 12S rRNA基因序列测定在腐烂动物肌肉样本鉴定中的应用[J].刑事技术,2005(4):10-11. HUANG Y L.Identification of animal putrid intramuscular swatch by sequencing of patial 12S rRNA gene[J].Forensic Science and Technology,2005(4):10-11.
(责任编辑:李 宁)
Identification of Shark Fin Using 12S rRNA Gene Marker
HUANG Yalin
(NanjingForestPoliceCollege,Nanjing210023,China;WildlifeMaterialEvidenceAppraisalCenteroftheStateForestryAdministration,Nanjing210023,China)
To detect authenticity and species of 23 suspected shark fin samples. DNAs from either shark fin or shark fin derived soup were extracted, and then 12S rRNA gene of mitochondrial DNA that was special for animal species identification was amplified. PCR product was sequenced and homology comparison was deployed by BLAST search in GenBank database with related species sequences. The results showed that, among a total of 23 suspected shark fin samples, 18 soup -sourced, two vermicelli -like, and a fin -like one were failed to extract DNA or amplify target fragments, while the remaining two samples were successful to extract genome DNA and amplify the corresponding 12S rRNA gene fragments. Sequence analysis indicated nucleotide sequences of the 12S rRNA gene fragment from the two samples matched 99% identity toCarcharhinuslimbatusandRhizoprionodonterraenovae, respectively. For those samples containing shark components, high temperature soaking treatment was carried out. High temperature soaking treatment showed gelatin was precipitated in a sample (22#).These results indicated 12S rRNA gene sequence determination could accurately and quickly identify whether the samples contained shark ingredients. If combined with the high temperature soaking treatment to observe whether there was a gelatin precipitation, we can comprehensively judge the authenticity of shark’s fin.
shark fin; authenticity; 12S rRNA; gene marker; species identification
10.3969/j.issn.2095 -6002.2017.03.010
2095 -6002(2017)03 -0066 -05

黄娅琳. 利用12S rRNA基因标记鉴定鱼翅真伪[J]. 食品科学技术学报,2017,35(3):66-70.
HUANG Yalin. Identification of shark fin using 12S rRNA gene marker[J]. Journal of Food Science and Technology, 2017,35(3):66-70.
2016 -11 -17
中央高校基本科研业务费专项资金资助项目(LGYB201518)。
黄娅琳,女,副教授,博士,主要从事野生动物DNA鉴定及相关遗传学方面的研究。
TS254.7
A
