普通菜豆籽粒大小与形状的QTL定位
2017-08-22耿庆河王兰芬王述民
耿庆河 王兰芬 武 晶 王述民
中国农业科学院作物科学研究所,北京 100081
普通菜豆籽粒大小与形状的QTL定位
耿庆河 王兰芬 武 晶 王述民*
中国农业科学院作物科学研究所,北京 100081
普通菜豆是世界上最重要的食用豆类作物之一,其籽粒大小和形状与产量及外观品质密切相关。本研究以来自安第斯基因库的大粒品种龙270709和来自中美基因库的小粒品种F5910配置杂交组合,获得的F2分离群体分别在哈尔滨大田与北京昌平温室种植,对百粒重、粒长、粒宽、粒厚、长宽比和长厚比6个籽粒性状进行了相关性分析和QTL定位。相关性分析表明,百粒重与粒长、粒宽、粒厚、长宽比、长厚比5个衡量籽粒大小和形状的性状均显著正相关。利用基于完备区间作图方法的IciMapping 4.1进行QTL定位,哈尔滨环境下定位到38个与百粒重、粒长、粒宽、粒厚、长宽比、长厚比相关的QTL,表型贡献率介于2.39%~17.37%之间,分布在除第1染色体外的其余10条染色体上;北京昌平环境下定位到21个上述性状的QTL,表型贡献率介于5.92%~22.53%之间,分布在第1、第3、第6、第7、第8、第9和第11染色体上。其中,百粒重QTLSW7与SW7’,SW6.1与SW6’,粒长QTLSL6.1与SL6.1’,粒厚QTLSH11与SH11’在2个环境下的标记区间重叠或者重合,SW7、SW6.1、SL6.1、SW6’和SL6.1’的表型贡献率在10%以上。
普通菜豆;籽粒大小;粒形;数量性状位点(QTL)
普通菜豆(Phaseolus vulgaris L.)是世界上种植面积最大的食用豆类,在非洲和美洲的部分地区作为一种重要的食物来源被广泛种植[1-3]。在我国,普通菜豆是重要的出口创汇作物,主要分布在黑龙江、内蒙古、山西、陕西、四川、贵州、云南等省区。籽粒是普通菜豆主要的消费对象,在长期饮食习惯和文化的影响下,不同地区的人们对籽粒的大小和形状形成了不同的偏好,籽粒大小和形状作为重要的品质性状深刻影响着普通菜豆的商业价值[2]。同时,籽粒大小也是普通菜豆产量要素之一,在长期育种实践中扮演着不可或缺的角色。因此,解析普通菜豆籽粒大小的遗传基础将有效地促进普通菜豆的育种进程。
籽粒性状在水稻中的研究较为深入,已鉴定出大量相关基因,其中包括控制粒长和粒重的GS3[4]基因,控制粒宽和粒重的GW2[5]基因,控制粒宽和粒重的qSW5[6]基因等,与粒重相关的基因GS3、GS5、qSW5、GW5、GW8、GIF1在驯化和育种进程中与产量密切相关[4-10],这些基因的分离和鉴定极大地促进了水稻的育种进程。普通菜豆籽粒大小QTL定位的研究基础相对薄弱,而且大部分集中在国外。普通菜豆的籽粒大小是多基因控制的数量性状[11],遗传基础相对复杂,籽粒大小又可进一步划分为粒长、粒宽、粒厚等遗传组分[12]。1923年,Sax[11]报道了与百粒重连锁的P基因。随着分子标记的不断发展,遗传连锁作图已成为解析数量性状遗传基础的重要方法。2006年,Blair等[13]利用哥伦比亚大粒栽培种与野生种杂交获得的 BC2F3:5群体以及SSR、SCAR、朊蛋白标记构建的连锁图谱定位到10个与百粒重相关的QTL,位于第2、第3、第6、第7、第8、第9、第10和第11染色体上;2010年,Pérez-Vega等[2]利用来自2个基因库间的材料构建的RIL群体在第2、第3、第6、第8和第10染色体上定位到5个与粒长相关的QTL,在第3、第6和第7染色体上定位到3个与粒宽相关的QTL,在第6、第8染色体上定位到2个与粒厚相关的QTL,以及在第6、第8染色体上定位到3个与百粒重相关的QTL; 2014年,Fernando等[14]利用2个来自安第斯基因库的亲本杂交获得的185个RILs定位到6个与粒长相关的QTL,位于第1、第2、第6、第7和第10染色体上,3个与粒宽相关的QTL,位于第2、第7和第9染色体上,2个与粒厚相关的QTL,位于第2、第9染色体上,以及4个与百粒重相关的QTL,位于第1、第6和第9染色体上。之前的报道中所用的定位群体的亲本多来自同一基因库,亲本间的多态性较低,在一定程度上影响了QTL定位的准确度与精确度。中美起源中心(主要包括墨西哥、危地马拉、哥伦比亚等国家)和安第斯起源中心(主要包括秘鲁南部、玻利维亚、阿根廷等国家和地区)由于长期地理隔离导致生殖隔离而形成了2个独立的基因库,在形态特征方面,安第斯起源中心的栽培种籽粒大小(百粒重大于40 g)普遍大于中美起源中心栽培种的籽粒大小(百粒重25~40 g,或小于25 g),因此发掘不同起源中心种质的优异籽粒性状基因有利于推进普通菜豆的育种进程。普通菜豆2个基因库典型材料全基因组测序的完成为解析重要农艺性状的遗传基础提供了新的契机[3,15]。
本研究采用分别来自安第斯基因库和中美基因库的龙270709和F5910为亲本配制杂交组合,获得F2分离群体作为定位群体,有利于发掘来自不同基因库的新基因。根据普通菜豆全基因组序列设计SSR标记,有利于QTL信息的比对和综合分析。多环境F2定位数据利于获得稳定的QTL。本研究为进一步精细定位籽粒性状相关基因及分子标记辅助育种奠定了良好基础。
1 材料与方法
1.1 材料及田间试验
以龙270709为母本,F5910为父本构建F2群体。龙270709为2005年黑龙江省农业科学院育成品系,来自安第斯基因库,生长习性为矮生有限型,生育期100 d左右,籽粒较大,百粒重约70 g;F5910为哥伦比亚品种,来自中美基因库,生长习性为矮生无限型,生育期90 d左右,籽粒较小,百粒重约20 g。亲本在粒长、粒宽、粒厚等籽粒大小相关性状间均存在较大差异。2015年5月中旬,在黑龙江省农业科学院国家现代农业示范区种植400株F2单株,每行10株,株距44 cm,行距50 cm,田间管理与当地大田管理相同,346株单株最终用于本试验;同年11月底,在北京中国农业科学院作物科学研究所昌平试验基地温室种植的包括188个F2单株的群体用于本试验,种植密度及田间管理同上,温室温度设置为白天28℃±2℃,夜晚20℃±2℃。成熟后,单株收取干籽粒,用于表型鉴定。
1.2 性状调查
收获后调查百粒重(seed weight,SW)、粒长(seed length,SL)、粒宽(seed width,SWI)、粒厚(seed height, SH)、长宽比(seed length-to-width ratio,SLW)和长厚比(seed length-to-height ratio)。粒长为平行于种脐,上下2个顶点之间的距离,粒宽为垂直于种脐,上下2个面之间的距离,粒厚为种脐到它对立面的距离[14],长宽比=粒长/粒宽,长厚比=粒长/粒厚。从每个单株取健康籽粒20粒,采用SC-G自动考种分析仪测量粒长、粒厚,采用游标卡尺测量粒宽,利用电子分析天平测量百粒重。
1.3 统计分析
采用SAS9.2软件和Microsoft Excel 2007进行各性状间相关性分析,以及基本统计量的计算。
1.4 QTL定位
在开花前,取亲本和 F2单株幼嫩叶片,以CTAB法提取叶片DNA[16]。本实验室根据2014年公布的普通菜豆全基因组序列(https://phytozome. jgi.doe.gov/)[3],开发了2000余对SSR标记。筛选获得的亲本间多态性标记用于本试验基因型的鉴定。利用IciMapping 4.1计算标记间的遗传距离,设置LOD≥3,利用 Kosambi作图函数作图,利用JoinMap 4根据遗传距离绘制连锁图谱[17-19]。采用高用明等[20]推荐的 QTL命名方法命名。利用IciMapping 4.1检测F2群体籽粒大小和形状QTL,采用完备区间作图方法,设置检测步长1.0 cM和1000次排列检验确定QTL的临界值(P<0.05)[17-18]。
2 结果与分析
2.1 表型变异及相关分析
从表1可以看出,母本龙270709的百粒重、粒长、粒宽、粒厚、长宽比、长厚比均高于父本F5910,其中百粒重的差异最为明显。母本270709百粒重在黑龙江哈尔滨和北京昌平温室2个环境下的均值差别较大,可能是品种特性与环境因素共同作用的结果。F2分离群体在2个环境下不同性状间均存在较大程度的分离,变异系数介于5.17%~20.35%之间,其中百粒重的变异系数最大,2个地点分别为16.62%和20.35%,长厚比的变异系数最小,分别为5.17%和6.62%。各性状的表型值为连续变异,接近正态分布(图1)。
相关性分析结果(表2)显示,百粒重分别与粒长、粒宽、粒厚、长宽比及长厚比显著正相关。粒长分别与粒宽、粒厚、长宽比和长厚比显著正相关。粒宽与粒厚显著正相关,与长宽比显著负相关,与长厚比在哈尔滨环境(环境1,下同)下显著正相关,在北京昌平环境下(环境2,下同)相关性不显著。粒厚与长宽比显著正相关,与长厚比相关性不显著。长宽比与长厚比显著正相关。
2.2 遗传图谱构建
利用哈尔滨种植的346株F2和北京昌平种植的188株F2分别构建了连锁图谱(图2和图3)。346株F2构建的遗传图谱包括100个SSR标记,分布于11条染色体上,每条染色体上的标记个数5~17个,连锁图谱总长439.88 cM,标记间平均遗传距离4.40 cM,有2对标记间的遗传距离大于20.00 cM,分别位于第10染色体P10S167~P10S186(31.91 cM)和第4染色体P4S2~P4S22(22.28 cM),其余标记遗传距离均小于20 cM。188株F2构建的遗传图谱包括88个SSR标记,与前者相比,第5、第6、第7、第9和第11染色体分别缺少了4、4、2、1、1个标记,为F2基因型缺失较严重的标记,其余标记均与前者相同,每条染色体上的标记个数3~13个,连锁图谱总长421.94 cM,标记间平均遗传距离4.80 cM,标记间遗传距离大于20 cM的2对标记同样位于第10染色体P10S167~P10S186(31.18 cM)和第4染色体P4S2~ P4S22(26.57 cM),其余标记的遗传距离均小于20 cM。
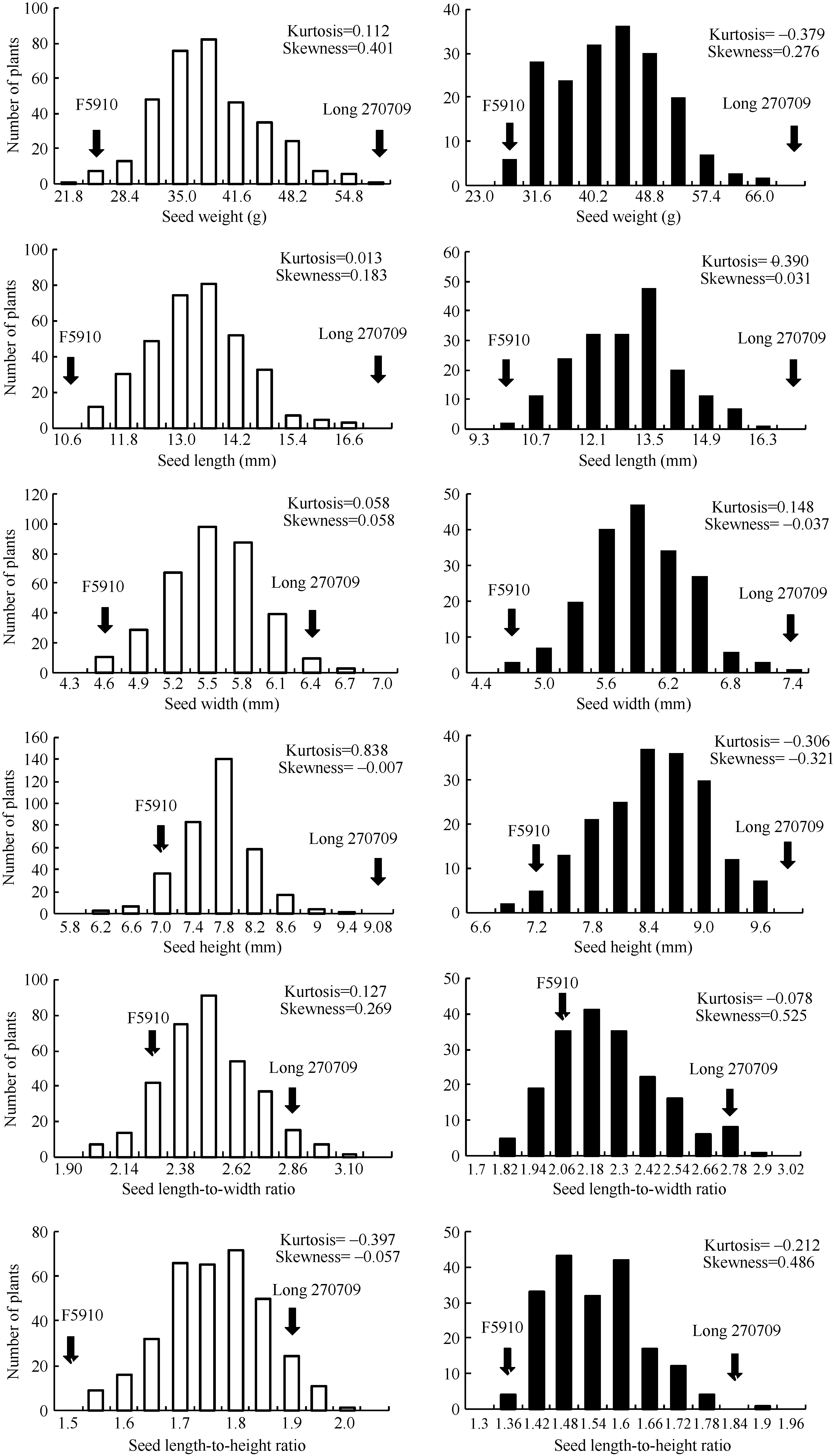
图1 群体籽粒大小与形状相关性状频率分布图Fig.1 Frequency distribution of seed size and shape traits of F2population in Harbin and Beijing
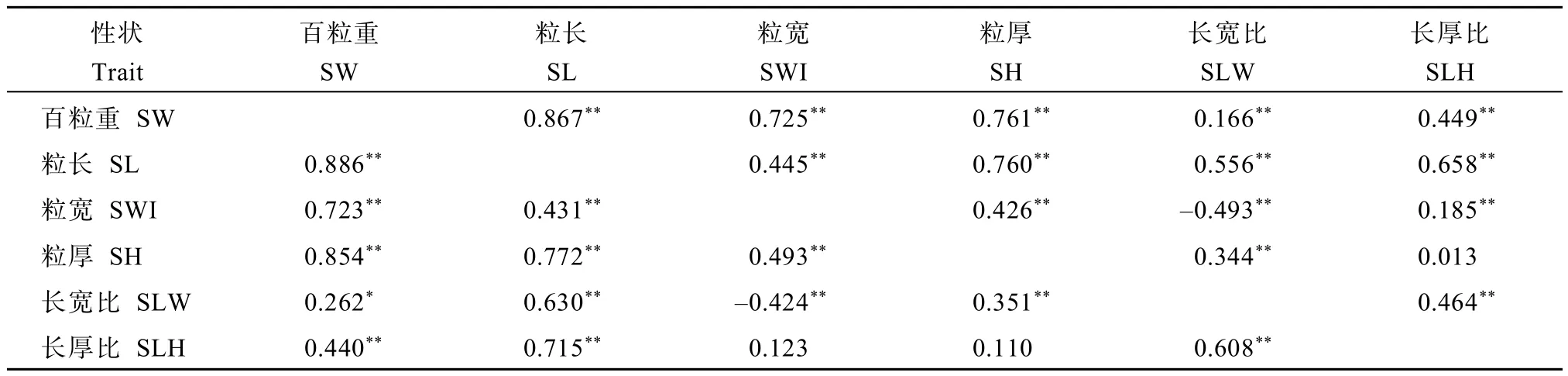
表2 籽粒性状间的相关性分析Table 2 Correlation coefficients for seed traits in common bean
2.3 QTL定位
利用IciMapping 4.1进行1000次排列获得QTL显著的LOD阈值均为3.0。哈尔滨环境下定位到的QTL在11条染色体上的分布如图2所示,北京昌平环境下定位到的QTL在11条染色体上的分布如图3所示。2个环境下QTL的定位结果见表3,其中哈尔滨环境下共定位到38个与百粒重、粒长、粒宽、粒厚、长宽比和长厚比相关的QTL,北京昌平环境下共定位到21个与上述6个性状相关的QTL。
2.3.1 百粒重 环境1中共检测到7个与百粒重相关的QTL(SW7、SW6.1、SW6.2、SW9、SW4、SW2和SW10),分布在第2、第4、第6、第7、第9和第10染色体上,表型贡献率介于3.88%~10.97%之间,加性效应均为正,表明增效等位基因均来自大粒亲本龙270709。第7染色体上的SW7,表型贡献率最大,为10.97%。第6染色体上的SW6.1和SW6.2,表型贡献率分别为10.79%和7.76%,遗传效应较大。SW9、SW4、SW2、SW10分别位于第9、第4、第2和第10染色体上,表型贡献率分别为 5.56%、5.27%、4.42%和3.88%,遗传效应较小。
环境2中共检测到4个与百粒重相关的QTL (SW6’、SW7’、SW11’、SW9’),分布在第6、第7、第11和第9染色体上,表型贡献率分别为15.88%、8.22%、8.16%和7.85%,加性效应均为正。
比较2个环境下的定位结果比较,第6、第7和第9染色体上均检测到百粒重QTL,SW7(P7S85~ P7S129)与SW7’(P7S85~P7S140)标记区间存在重合,与标记P7S85的遗传距离均为0 cM。SW6.1与SW6’均位于标记P6S108~P6S266之间,与标记P6S108的遗传距离分别为 4.50 cM和 0.32 cM。SW9 (P9S147~P9S155)与 SW9’(P9S136~P9S147)标记区间相邻,与标记P9S147的遗传距离分别为2.17 cM和0.47 cM。SW4、SW2、SW10环境贡献率均小于SW9,未在环境2中检测到,SW6.2贡献率比SW7和SW6.1小,比SW9大,在环境2中未被检测到。
2.3.2 粒长 环境1中共检测到8个与粒长相关的QTL(SL6.1、SL7、SL6.2、SL9、SL5、SL10、SL2和SL4),分布在第2、第4、第5、第6、第7、第9和第10染色体上,除SL5外,其余位点加性效应均为正。SL6.1和SL7遗传效应较大,表型贡献率分别为17.37%和12.53%。其余6个QTL(SL6.2、SL9、SL5、SL10、SL2、SL4)表型贡献率分别为8.57%、4.46%、4.25%、3.96%、2.94%和2.64%。
在环境2定位到5个粒长QTL(SL6.1’、SL7’、SL6.2’、SL1’和SL9’),分布于第1、第6、第7和第9染色体上,表型贡献率介于6.01%~22.53%之间, SL1’加性效应为负值,其余均为正值。SL6.1’与SL7’遗传效应较大,表型贡献率分别为 22.53%和10.14%。SL6.2’、SL1’、SL9’表型贡献率分别为9.10%、7.14%和6.01%。
2个环境下定位结果比较分析,均在第6、第7、第9染色体上定位到粒长QTL。SL6.1与SL6.1’均位于标记P6S108~P6S266之间,与标记P6S108之间的遗传距离分别为 4.50 cM 和 0.32 cM。SL7 (P7S85~P7S129)和 SL7’(P7S140~P7S153)与标记P7S85的遗传距离分别为 3 cM 和 4 cM。SL9 (P9S86~P9S129)和 SL9’(P9S136~P9S147)与标记P9S147间的遗传距离分别为4.83 cM和0.47 cM。SL6.2与SL6.2’均位于第6染色体,但是距离较远,与标记P6S185之间的遗传距离分别是7.00 cM和28.82 cM。另外,在环境2定位到位于第1染色体的SL1’并没有在环境1中检测到,环境1中检测到的的SL5、SL10、SL2、SL4表型贡献率相对较小,没有在环境2中检测到。
2.3.3 粒宽 在环境1定位到3个粒宽QTL (SWI2、SWI6、SWI5),分布于第2、第5和第6条染色体上,表型贡献率介于4.36%~7.68%之间,除SWI5加性效应为负值外,其余位点加性效应均为正值。

图2 哈尔滨环境下(环境1)定位到的QTL在连锁群上的分布Fig.2 Distribution of QTL for seed size and shape in linkage groups in Harbin,Heilongjiang(environment 1)
在环境2定位到3个粒宽QTL(SWI11’、SWI8’和SWI9’),分布于第8、第9、第11染色体上,表型贡献率介于8.22%~10.72%之间,加性效应均为正值。2个群体中定位到的粒宽QTL表型贡献率均没有超过10%,同时也没有位于相同染色体上的位点,表明粒宽受环境影响较大。

图3 北京昌平环境下(环境2)定位到的QTL在连锁群上的分布Fig.3 Distribution of QTL for seed size and shape in linkage groups in Beijing(environment 2)

表3 哈尔滨环境(环境1)和北京昌平(环境2)的QTL定位结果Table 3 QTL detected in Harbin,Heilongjiang(environment 1)and Changping,Beijing(environment 2)
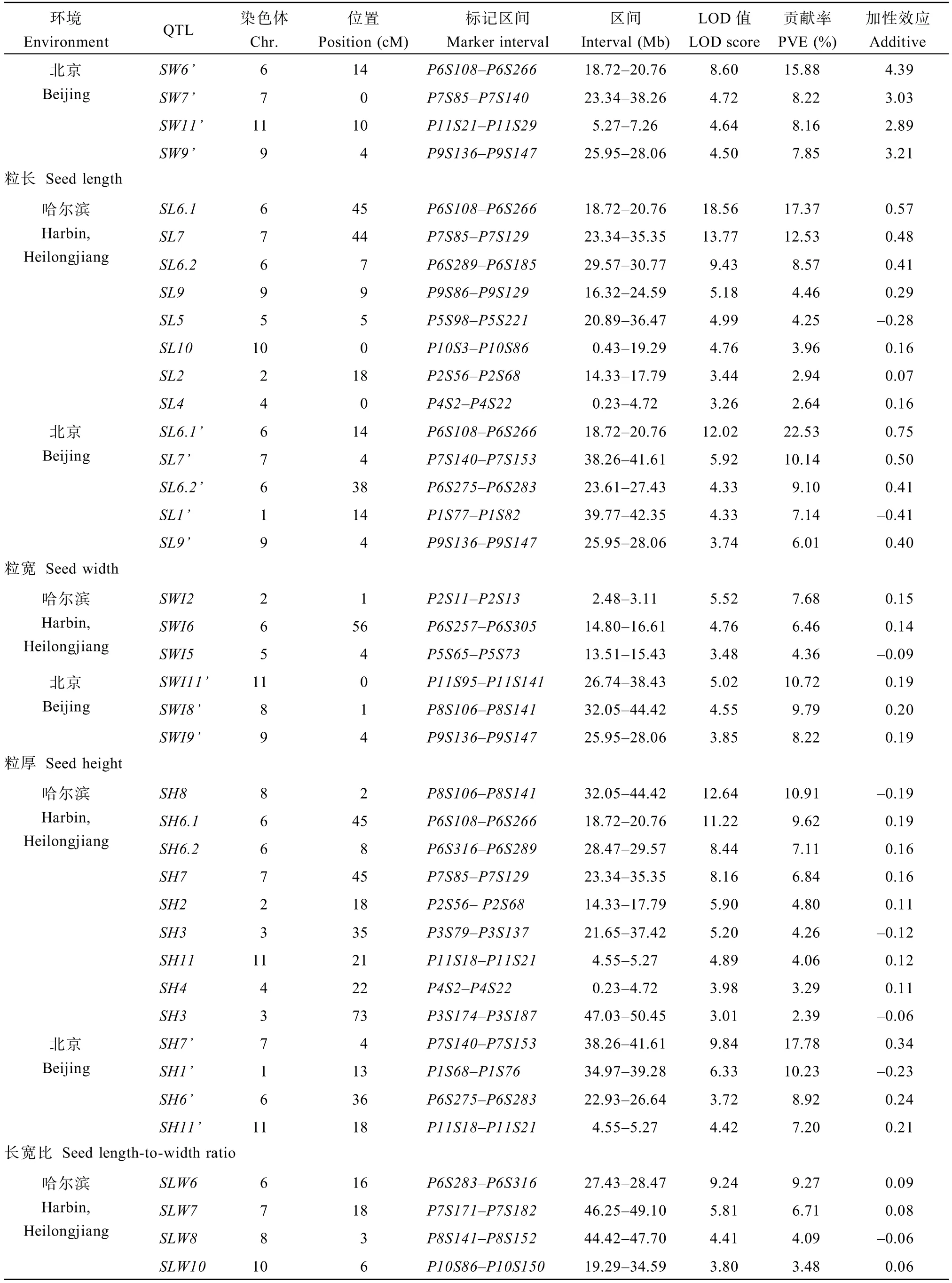
(续表3)
2.3.4 粒厚 在环境1定位到9个粒厚QTL (SH8、SH6.1、SH6.2、SH7、SH2、SH3.1、SH11、SH4和SH3.2),分布于第2、第3、第4、第6、第7、第8和第11染色体上,表型贡献率介于3.29%~ 10.91%之间,在第3和第6染色体上分别检测到2个QTL,在其余染色体上均检测到1个QTL。SH8和SH3加性效应为负值,其余位点遗传效应均为正值。SH8表型贡献率最大,为10.91%,SH6.1表型贡献率次之,为9.62%。其余位点(SH6.2、SH7、SH2、SH3.1、SH11、SH4、SH3.2)表型贡献率分别为7.11%、6.84%、4.80%、4.26%、4.06%、3.29%和2.39%。在环境2定位到4个粒厚QTL(SH7’、SH1’、SH6’、SH11’),分布于第1、第6、第7、第11染色体上,SH1’加性效应为负值,其余位点加性效应均为正值。表型贡献率在10%以上的位点有2个,分别是SH7’(17.78%)和SH1’(10.23%)。SH6’和SH11’表型贡献率分别为8.92%和7.20%。

(续表3)
环境1中检测到的SH8表型贡献率最大,但在环境2中并未被检测到。SH6.1(P6S108~P6S266)和SH6’(P6S275~P6S283)与标记P6S108间的遗传距离分别为4.50 cM和23.68 cM。SH6.2(P6S316~P6S289)和SH6’(P6S275~P6S283)与标记P6S289间的遗传距离分别为0.08 cM和21.22 cM。可见SH6’与SH6.1和SH6.2距离都比较远。SH7(P7S85~P7S129)和SH7’(P7S140~P7S153)与标记P7S85间的遗传距离分别为 0 cM和 4 cM。SH11与 SH11’均位于标记P11S21-P11S18之间,与标记P11S18间的遗传距离分别为0.32 cM和0.90 cM。
2.3.5 长宽比 环境1中定位到4个长宽比QTL (SLW6、SLW7、SLW8、SLW10),分布于第6、第7、第8、第9染色体上,SLW8加性效应值为负,其余均为正,表型贡献率最大的QTL是SLW6,为9.27%,最小的是SLW10,为3.48%。环境2中定位到3个长宽比QTL(SLW6’、SLW8’、SLW7’),分布于第6、第7、第8染色体,SLW8’加性效应值为负,其余为正,3个长宽比QTL(SLW6’、SLW8’、SLW7’)表型贡献率分别为20.29%、9.88%和7.31%。
2个环境下均在第6、第7、第8染色体上定位到长宽比QTL。在第6染色体上定位到的SLW6 (P6S283~P6S316)和SLW6’(P6S266~P6S309)标记区间不同且不相邻,与标记P6S316间的遗传距离为8.08 cM和36.22 cM,相距较远。在第7染色体上定位到的SLW7(P7S171~P7S182)与SLW7’(P7S153~ P7S171)标记区间相邻,与标记P7S182间的遗传距离分别为5.41 cM和11.80 cM。在第8染色体上定位到的SLW8(P8S141~P8S152)和SLW8’(P8S106~ P8S141)标记区间相邻,与标记P8S152间的连锁距离分别是4.11 cM和4.21 cM。
2.3.6 长厚比 环境1中定位到7个与长厚比相关的QTL(SLH3、SLH7、SLH6、SLH5、SLH8、SLH9和SLH10),分布在第3、第5、第6、第7、第8、第9和第10染色体上,加性效应值均为正,表型贡献率介于2.83%~10.99%之间。环境2中定位到2个与长厚比相关的QTL(SLH6’、SLH3’),位于第6和第3染色体上,表型贡献率分别为31.59%和5.92%,加性效应值均为正。
2个环境下均在第3和第6染色体上定位到长厚比 QTL,位于第 3染色体上的 SLH3(P3S79~ P3S137)与 SLH3’(P3S169~P3S171)标记区间不同,与标记P3S171间的遗传距离分别为20.98 cM和0.76 cM,相距较远。位于第 6染色体上的SLH6(P6S309~P6S275)与SLH6’(P6S108~P6S266)标记区间不同,与P6S108间的遗传距离分别是7.90 cM和0.32 cM。
3 讨论
3.1 QTL定位结果分析
普通菜豆的籽粒特性是重要的农艺性状,直接影响着普通菜豆的产量和商品性。一直被研究者关注,截至目前在菜豆中已经定位到大量的与籽粒特性相关的QTL[2,13-14,21]。本研究在哈尔滨环境下共定位到38个与百粒重、粒长、粒宽、粒厚、长宽比和长厚比相关的QTL,在昌平环境下共定位到21个与上述性状相关的QTL。其中,位于第2染色体的百粒重QTL SW2,标记区间P2S11~P2S13,在染色体上的物理位置为2.48~3.11 Mb,与Blair等[13]定位到的百粒重QTL sw2.2(2.819 Mb)位置重叠;位于第6染色体标记 P6S289~P6S185之间的百粒重 QTL SW6.2,物理位置在29.57~30.77 Mb之间,与Blair等[13]检测到的百粒重QTL sw6.1(27.44 Mb)位置重叠;位于第1染色体上的粒长QTLSL1’,标记区间P1S77~P1S82,物理位置在39.77~42.35 Mb之间,与Fernando等[14]检测到的SL1.2PP(32.41 Mb)位置相近。定位结果的一致性一方面说明定位结果的可靠性,另一方面也说明这些位点的重要性。我们还注意到在第4染色体上未见前人报道与籽粒大小相关的位点,而本研究在这一染色体上检测到与百粒重、粒长、粒厚相关的QTL,位于标记P4S2~P4S22之间。
同一群体多环境表型鉴定及QTL分析,可以检测到相对稳定可靠的位点。特别是贡献率(加性效应)较大的QTL更容易在不同环境条件下被连续检测到[22]。本研究中2个环境下共检测到4对标记区间重叠的QTL:百粒重QTL SW7和SW7’分别位于标记区间P7S85~P7S129和 P7S85~P7S140,百 粒 重 QTL SW6.1(SW6’)位于标记区间P6S108~P6S266,粒长QTL SL6.1(SL6.1’)位于标记区间 P6S108~P6S266,粒厚 QTL SH11(SH11’)位于标记区间 P11S18~ P11S21。其中3对QTL至少在一个环境下表型贡献率在 10%以上,SW7(SW7’)表型贡献率 10.91% (8.22%),SW6.1(SW6’)表型贡献率10.79%(15.88%), SL6.1(SL6.1’)表型贡献率17.37%(22.53%)。这种具有较高贡献率且在不同的环境下可连续检测到的QTL可以更好地用于分子标记辅助选择育种[23]。4对QTL加性效应均为正,表明增效等位基因均来自母本龙270709,这将有利于发掘和利用来自安第斯基因库的普通菜豆优异种质的籽粒大小和形状基因。除此之外,有3对QTL标记区间虽然不重叠但是相邻,分别位于第7、第8、第9染色体上,位于第7染色体上的长宽比QTL SLW7和SLW7’分别位于标记区间P7S171~P7S182和P7S153~P7S171,位于第8染色体上的长宽比QTL SLW8和SLW8’分别位于标记区间P8S141~P8S152和P8S106~P8S141,位于第9染色体上的百粒重QTL SW9和SW9’分别位于标记区间P9S147~P9S155和P9S136~P9S147。SLW8(SLW8’)、SW9(SW9’)与SLW7(SLW7’)表型贡献率均介于4%~10%之间,分别为4.09%(9.88%)、5.56%(7.85%)和6.71%(7.31%)。基因型与环境互作可能是导致这类情况出现的主要原因,尤其是对于表型贡献率相对较小的位点,环境的影响更为明显。精细定位或者标记选择时可考虑将其标记区间扩大来利用这些位点。
另外,由于环境2所用群体比环境1所用群体相对较小,环境1中检测到的表型贡献率较小的位点在环境2中并未被检测到,如SW2、SW4、SW10、SL2、SL4、SL5、SL10、SH2、SH3和SH4,包括与百粒重、粒长和粒厚相关的QTL,集中分布在第2、第3、第4、第5染色体上。群体大小影响检测的效率,群体扩大,效应值较小的位点也能被检测到[24]。
3.2 染色体区段多效性分析
本研究中出现不同性状定位在相同或相近染色体区段的现象。这种现象较为普遍,在小麦、玉米、大豆等作物中均有类似报道[25-29]。Tuberosa等[30]认为“一因多效”可能是这种现象出现的主要原因,例如水稻中的GS3基因既控制粒长,又与百粒重有关, GW2同时与粒宽和百粒重相关[4-5]。另外,QTL紧密连锁也可能是导致这种结果的一个原因。本研究中,第 6染色体 P6S108~P6S266区段、第 7染色体P7S85~P7S129区段内均同时检测到与百粒重、粒长、粒厚和长厚比均相关的QTL;第4染色体P4S2~P4S22区段检测到与百粒重、粒长和粒厚相关的QTL;第2染色体P2S11~P2S13区段内检测到与百粒重、粒宽相关的QTL,P2S56~P2S68区段内检测到与粒长、粒厚相关的QTL;第10染色体P10S3~ P10S86区段内检测到与百粒重、粒长、长厚比相关的QTL;第6染色体P6S275~P6S283与第7染色体P7S140~P7S153区段内均同时检测到与粒长、粒厚相关的QTL;第9染色体P9S136~P9S147区段内同时检测到与百粒重、粒长、粒宽相关的QTL。百粒重与粒长、粒厚、粒宽、长厚比相关的QTL在同一染色体区段被同时检测到,这为解释百粒重与粒长、粒厚、粒宽及长厚比之间显著的相关性提供了一定的遗传基础。
4 结论
黑龙江哈尔滨环境中定位到38个与百粒重、粒长、粒宽、粒厚、长宽比和长厚比相关的QTL,北京昌平环境下定位到上述性状的QTL共21个,其中百粒重QTLSW7(SW7’)和SW6.1(SW6’),粒长QTL SL6.1(SL6.1’),粒厚QTLSH11(SH11’)在2个环境下被重复检测到,4个位点增效等位基因均来自安第斯基因库的大粒亲本龙270709,可作为精细定位的重要候选区段以及标记辅助选育的位点。
[1]张赤红,曹永生,宗绪晓,王志刚,王述民.普通菜豆种质资源形态多样性鉴定与分类研究.中国农业科学,2005,38: 27~32
Zhang C H,Cao Y S,Zong X X,Wang Z G,Wang S M. Morphological diversity and classification of common bean (Phaseolus vulgaris L.)germplasm resource in China.Sci Agric Sin,2005,38:27–32(in Chinese with English abstract)
[2]Pérez-Vega E,Pañeda A,Rodríguez-SuárezC,Campa A, Giraldez R, Ferreira J J. Mapping of QTLs for morpho-agronomic and seed quality traits in a RIL population of common bean(Phaseolus vulgaris L.).Theor Appl Genet,2010, 120:1367–1380
[3]Schmutz J,McClean P E,Mamidi S,Wu G A,Cannon S B, Grimwood J,Jenkins J,Shu S Q,Song Q J,Chavarro C, Torres-Torres M,Geffroy V,oghaddam S M,Gao D Y,Abernathy B,Barry K,Blair M,Brick M A,Chovatia M,Gepts P,Goodstein D M,Gonzales M,Hellsten U,Hyte D L,Jia G F,Kelly J D, Kudrna D,Lee R,Richard M M S,Miklas P N,Osorno J M, Rodrigues J,Thareau V,Urrea C A,Wang M,Yu Y,Zhang M, Wing R A,Cregan P B,Rokhsar D S,Jackson S A.A reference genome for common bean and genome-wide analysis of dual domestications.Nat Genet,2014,46:707–713
[4]Fan C C,Xing Y Z,Mao H L,Lu T T,Han B,Xu C G,Li X H, Zhang Q F.GS3,a major QTL for grain length and weight and minor QTL for grain width and thickness in rice,encodes a putative transmembrane protein.Theor Appl Genet,2006,112: 1164–1171
[5]Song X J,Huang W,Shi M,Zhu M Z,Lin H X.A QTL for rice grain width and weight encodes a previously unknown RING-type E3 ubiquitin ligase.Nat Genet,2007,39:623–630
[6]Shomura A,Izawa T,Ebana K,Ebitani T,Kanegae H,Konishi S, Yano M.Deletion in a gene associated with grain size increased yields during rice domestication.Nat Genet,2008,40:1023–1028
[7]Weng J F,Gu S H,Wan X Y,Gao H,Guo T,Su N,Lei C L, Zhang X,Cheng Z J,Guo X P,Wang J L,Jiang L,Zhai H Q,Wan J M.Isolation and initial characterization of GW5,a major QTL associated with rice grain width and weight.Cell Res,2008,18: 1199–1209
[8]Wang E T,Wang J J,Zhu X D,Hao W,Wang L Y,Li Q,Zhang L X,He W,Lu B R,Lin H X,Ma H,Zhang G Q,He Z H.Control of rice grain-filling and yield by a gene with a potential signature of domestication.Nat Genet,2008,40:1370–1374
[9]Li Y B,Fan C C,Xing Y Z,JiangY H,Luo L J,Sun L,Shao D,Xu C J.Natural variation in GS5 plays an important role in regulating grainsize and yield in rice.Nat Genet,2011,43:1266–1269
[10]Wang S K,Wu K,Yuan Q B,Liu X Y,Liu Z B,Lin X Y,Zeng R Z,Zhu H T.Control of grain size,shape and quality by OsSPL16 in rice.2012,Nat Genet,44:950–954
[11]Sax K.The association of size differences with seed-coat pattern and pigmentation in PHASEOLUS VULGARIS.Genetics,1923,8: 552–560
[12]Vallejos C E,Chase C D.Linkage between isozyme markers and a locus affecting seed size in Phaseolus vulgaris L.Theor Appl Genet,1991,81:413–419
[13]Blair M W,Iriarte G,Beebe S.QTL analysis of yield traits in an advanced backcross population derived from a cultivated Andean ×wild common bean(Phaseolus vulgaris L.)cross.Theor Appl Genet,2006,112:1149–1163
[14]Yuste-Lisbona F J,González A N,Capel C,Garcia-Alcazar M, Capel J,Ron A M D,Lozano R,Santalla M.Genetic analysis of single-locus and epistatic QTLs for seed traits in an adapted× nuña RIL population of common bean(Phaseolus vulgaris L.). Theor Appl Genet,2014,127:897–912
[15]Vlasova A,Capella-Gutiérrez S,Rendón-Anaya M,Hernández-Oñate M E.Minoche A,Erb I,Câmara F,Prieto-Barja P,Corvelo A,Sanseverino W,Westergaard G,Dohm J C,Pappas Jr G J, Saburido-Alvarez S,Kedra D,Gonzalez I,Cozzuto L, Gómez-Garrido J,Aguilar-Morón M A,Andreu N,Aguilar O, Garcia-Mas J,Zehnsdorf M,Vázquez M P,Delgado-Salinas A, Delaye L,Lowy E,Mentaberry A,Vianello-Brondani R P,García J,Alioto T,Sánchez F,Himmelbauer H,Santalla M,Notredame C, Gabaldón T,Herrera-Estrella A,Guigó R.Genome and transcriptome analysis of the Mesoamerican common bean and the role of gene duplications in establishing tissue and temporal specialization of genes.Genome Biol,2016,17:1–18
[16]Murray M G,Thompson W F.Rapid isolation of high molecular weight plant DNA.Nucl Acids Res,1980,8:4321–4325
[17]Li H H,Ye G Y,Wang J K.A modified algorithm for the improvement of composite interval mapping.Genetics,2007,175: 361–374
[18]Meng L,Li H H,Zhang L Y,Wang J K.QTL IciMapping: integrated software for genetic linkage map construction and quantitative trait locus mapping in biparental population.Crop J, 2015,3:269–283
[19]Wan X Y,Wan J M,Jiang L,Wang J K,Zhai H Q,Weng J F, Wang H L,Lei C L,Wang J K,Zhang X.QTL analysis for rice grain length and fine mapping of an identified QTL with stable and major effects.Theor Appl Genet,2006,112:1258–1270
[20]高用明,朱军,宋佑胜,何慈信,石春海,邢永忠.水稻永久F2群体抽穗期QTL的上位性及其与环境互作效应的分析.作物学报,2004,30:849–854
Gao Y M,Zhu J,Song Y S,He C X,Shi C H,Xing Y Z.Use of permanent F2population to analyze epistasis and their interaction effects with environments for QTLs controlling heading date in rice.Acta Agron Sin,2004,30:849–854(in Chinese with English abstract)
[21]Hoyos-Villegas V,Song Q,Wright E M,Beebe S E,Kelly D J. Joint linkage QTL mapping for yield and agronomic traits in a composite map of three common bean RIL populations.Crop Sci, 2016,56:1–18
[22]Tanksley S D.Mapping ploygenes.Annu Rev Genet,1993,27: 205–233
[23]陈秀华,于丽娟,罗黎明,陈洪梅,刘丽.玉米分子标记辅助育种及标记开发研究进展.中国农业科技导报,2016,18(1): 26–31
Chen X H,Yu L J,Luo L M,Chen H M,Liu L.Research progress on maize molecular marker-assisted breeding and marker development.J Agric Sci Technol,2016,18(1):26–31(in Chinese with English abstract)
[24]赵洪波,李明丽,鲁绍雄,连林生,李国治.群体规模和性状遗传力对F2设计下QTL定位效果的影响.云南农业大学学报, 2007,22(2):159–163
Zhao H B,Li M L,Lu S X,Lian L S,Li G Z.Study on the effects of population size and trait heritability on the accuracy of QTL mapping under F2design.J Yunnan Agric Univ,2007,22(2): 159–163(in Chinese with English abstract)
[25]Groos C,Robert N,Bervas E,Charmet G.Genetic analysis of grain protein-content,grain yield and thousand-kernel weight in bread wheat.Theor Appl Genet,2003,106:1032–1040
[26]张坤普,徐宪斌,田纪春.小麦籽粒产量及穗部相关性状的QTL定位.作物学报,2009,35:270–278
Zhang K P,Xu X B,Tian J C.QTL mapping for grain yield and spike related traits in common wheat.Acta Agron Sin,2009,35: 270–278(in Chinese with English abstract)
[27]李文福,刘宾,彭涛,袁倩倩,韩淑晓,田纪春.利用DH和IF2两个群体进行小麦粒重、粒型和硬度的QTL分析.中国农业科学,2012,45:3453–3462
Li W F,Liu B,Peng T,Yuan Q Q,Han S X,Tian J C.Detection of QTL for kernel weight,grain size,and grain hardness in wheat using DH and immortalized F2population.Sci Agric Sin,2012, 45:3453–3462(in Chinese with English abstract)
[28]覃鸿妮,晏萌,王召辉,郭莹,王辉,孙海燕,刘志斋,蔡一林.玉米籽粒中花色苷和黑色素含量的QTL分析.作物学报, 2012,38:275–284
Qin H N,Yan M,Wang Z H,Guo Y,Wang H,Sun H Y,Liu Z Z, Cai Y L.QTL mapping for anthocyanin and melanin contents in maize kernel.Acta Agron Sin,2012,38:275–284(in Chinese with English abstract)
[29]郑德波,杨小红,李建生,严建兵,张士龙,贺正华,黄益勤.基于SNP标记的玉米株高及穗位高QTL定位.作物学报, 2013:549–556
Zheng D B,Yang X H,Li J S,Yan J B,Zhang S L,He Z H, Huang Y Q.QTL identification for plant height and ear height based on SNP mapping in maize(Zea mays L.).Acta Agron Sin, 2013,39:549–556(in Chinese with English abstract)
[30]Tuberosa R,Salvi S,Sanguineti M C,Landi P,Maccaferri M, Conti S.Mapping QTLs regulating morpho-physiological traits and yield:casestudies,shortcomings and perspectivesin drought-stressed maize.Ann Bot,2002,89:941–963
QTL Mapping for Seed Size and Shape in Common Bean
GENG Qing-He,WANG Lan-Fen,WU Jing,and WANG Shu-Min*
Institute of Crop Science,Chinese Academy of Agricultural Sciences,Beijing 100081,China
Common bean is one of the most important food legumes worldwide.Seed size and shape have a great effect on yield and seed quality.This research used an F2population derived from a cross of Andean cultivar Long 270709´Mesoamerican cultivar F5910 planted in Harbin,Heilongjiang and Beijing respectively to analyze the correlation among seed weight,seed length, seed width,seed height,seed length-to-width ratio and seed length-to-height ratio,and to detect QTL related to these traits.The correlation analysis showed that seed weight had a significant correlation with other traits related to seed size and shape.Software IciMapping 4.1 based on inclusive composite interval mapping was used to identify the additive QTL for seed size and shape.A total of 38 QTL were detected on 10 chromosomes except the first chromosome in Harbin environment with the PVE between 2.39%and 17.37%.A total of 21 QTL were detected on seven chromosomes(Chr.1,Chr.3,Chr.6,Chr.7,Chr.8,Chr.9,Chr.11)in Beijing environment with the PVE between 5.92%and 22.53%.Among them,four pairs of QTL were detected on the same marker interval,including seed weight QTL SW7 and SW7’,seed weight QTL SW6.1 and SW6’,seed length QTL SL6.1 and SL6.1’,and seed height QTL SH11 and SH11’.The PVE of SW7,SW6.1,SL6.1,SW6’,and SL6.1’was larger than 10%.
Common bean;Seed size;Seed shape;Quantitative trait locus(QTL)
(
):2016-12-21;Accepted(接受日期):2017-04-20;Published online(网络出版日期):2017-04-27.
10.3724/SP.J.1006.2017.01149
本研究由国家自然科学基金项目(31471559),国家现代农业产业技术体系建设专项(CARS-09)和国家科技支撑计划项目(2013BAD01B05-2-4)资助。
This study was supported by the National Natural Science Foundation of China(31471559),the China Agriculture Research System (CARS-09),and the National Key Technology R&D Program of China(2013BAD01B05-2-4).
*通讯作者(Corresponding author):王述民,E-mail:wangshumin@caas.cn
联系方式:E-mail:17710428642@163.com
URL:http://kns.cnki.net/kcms/detail/11.1809.S.20170427.0948.016.html
