近红外光谱法快速测量平菇菌丝总蛋白含量
2017-07-29李瑾韩建东秦宏伟任海霞任鹏飞万
李瑾+韩建东+秦宏伟+任海霞+任鹏飞+万鲁长+姚强+宫志远
摘要:本文以平菇平板培养菌丝总蛋白含量为指标,在1 000~1 799 nm近红外光谱区域采集光谱信息,采用化学计量学法建立菌株各参数的偏最小二乘法(PLS)定量预测模型。结果表明:最佳光谱预处理方法为Savitzky-Golay平滑+Savitzky-Golay导数+多元散射校正(MSC)+均值中心化,所建定量模型的校正集相关系数、校正标准差(SEC)、验证集相关系数、预测标准差(SEP)、主因子数、SEP/SEC均在合理范围,模型真实值与预测值的相关系数为0.67263,模型可靠性、稳健性和预测效果较好,可用于菌丝蛋白质含量检测。
关键词:近红外光谱;平菇;菌丝蛋白含量;定量模型
中图分类号:S646.1+40.1 文献标识号:A 文章编号:1001-4942(2017)07-0145-05
Abstract Using the total protein content in mycelia of oyster mushroom cultured in plate medium as the index, the spectral information in 1 000~1 799 nm region was collected to establish a quantitative prediction model for the parameters of strains through partial least squares regression combined with chemometrics. The results showed that the optimal spectral pretreatment method was the combination of Savitzky-Golay smoothness + Savitzky-Golay derivative + MSC + Mean-Centering. Parameters of the quantitative model including RC, SEC, RP, SEP, MF, SEP/SEC were all in the reasonable regions. The correlation coefficient of the real value and predictive value of the model was 0.67263. The prediction model had better reliability, robustness and predictive effects, so it could be used for protein content detection in mycelia.
Keywords Near infrared spectroscopy; Oyster mushroom; Protein content in mycelia; Quantitative model
平菇是山東省食用菌栽培的主要菇种之一,年产量超过160万吨,占全省食用菌总产量1/3以上,在全国仅次于河南省。平菇产业发展最基本的条件是生产菌株,而选育高品质、高抗病、高转化率的平菇菌种是食用菌科研的根本目标。对大多数菌株来说,同培养条件下,特定时间内菌体积累的蛋白质含量往往与出菇时间、出菇品质紧密相关,因而测定平菇菌体蛋白质含量就成为平菇种质资源鉴定以及品种选育的重要方式之一。目前测定平菇菌体中的蛋白含量主要使用紫外分光光度计法、凯氏定氮法和旋光法等[1],这些方法可靠性高,但所需样品量大、采样过程对样品产生污染损耗,无法多次连续测量,测定步骤繁琐、费用高。而在平菇育种过程中,一般采用平板培养的方式对大量种质资源、突变体、杂交后代材料进行鉴定和筛选,如果采用传统分析方法意味着巨大的工作量和试验结果同步性不足,因而需要快速、无损无污染的检测方法即时监控菌体蛋白质含量变化过程。
近红外光谱(Near Infrared,NIR) 分析技术近年来在农业育种、质量检测领域发展迅速,其原理是利用光谱仪获取有机物在近红外光谱区的特征振动吸收信息,与其在化学分析法基础上取得的测定值对应,通过数学方法建立近红外光谱分析模型,待测样品通过该模型快速与光谱信息库内模型比对计算,给待测参数定量,从而一次性获取样品中多种化学成分含量数值,是一种简便无损无需生化试验操作的即时检测手段[2]。
因为平板培养是食用菌育种筛选优良新品种、测定菌株形态和生理生化指标最常用的方法之一,研究组以70个平板培养模式下的平菇菌株为对象,用分光光度计法测定其蛋白质含量,利用SupNIR-2720近红外光谱分析仪,优化光谱预处理方法,建立平菇菌体蛋白质含量的相关模型,并对建模效果进行评价,以期为平菇蛋白质含量指标的准确快速检测提供依据,为平菇种质资源评价筛选提供一种快速、简便、无损的分析方法。
1 材料与方法
1.1 材料和仪器
供试材料:平菇为山东省农业科学院农业资源与环境研究所菌种库保藏的70个菌株。平板培养基:葡萄糖20 g、蛋白胨10 g、琼脂20 g、磷酸二氢钾3 g、硫酸镁1.5 g,加水至1 000 mL[3]。每个菌株接种3个平板,25℃避光培养9 d。
仪器设备:近红外光谱分析仪(杭州聚光科技SupNIR-2720 )、紫外分光光度计(北京普析T6新世纪)、恒温培养箱(上海博讯)、生物样品研磨器(法国Bertin Precellys Evolution )、烘箱(淄博新华医疗)、高速冷冻离心机(Eppendorf 5810 )
1.2 检测方法
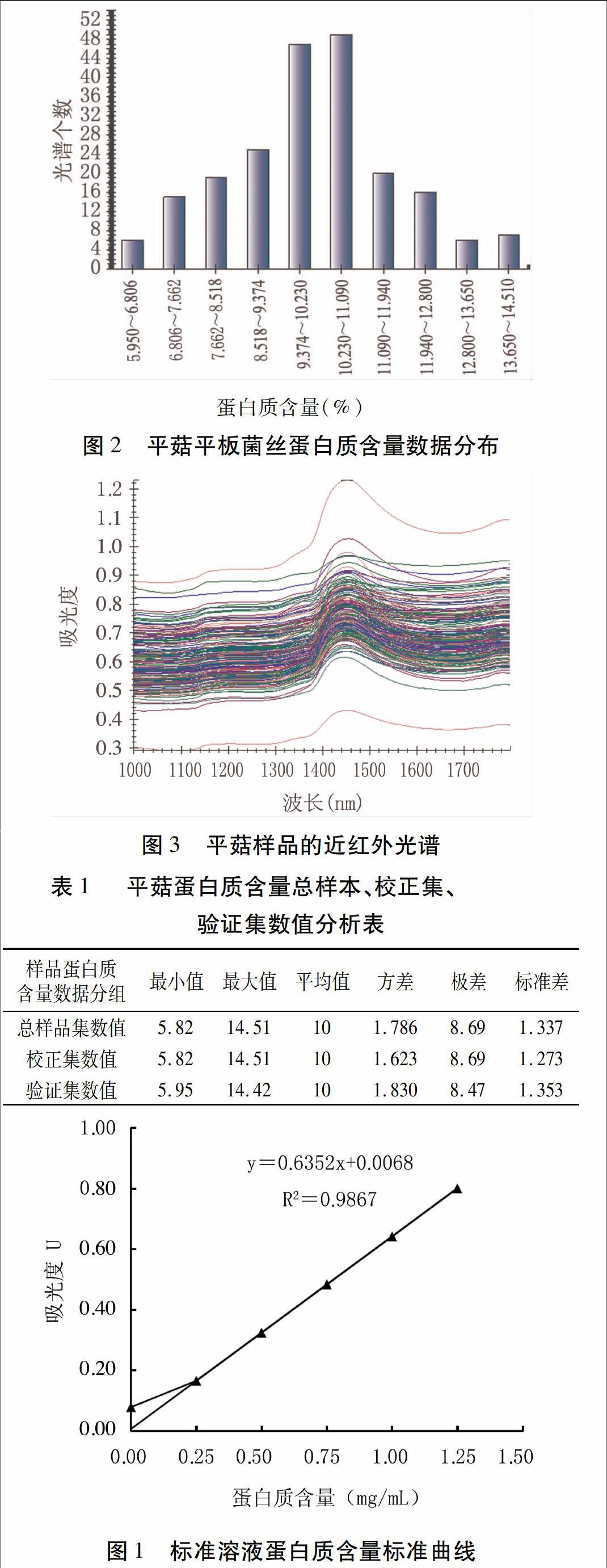
1.2.1 平板菌体蛋白质含量测定 采用标准曲线法。首先从5.00 mg/mL标准蛋白质溶液中用移液器分别吸取0.5、1.0、1.5、2.0、2.5 mL,用0.9% NaCl溶液分别稀释至10 mL,以0.9% NaCl溶液为参比,在280 nm处分别测定各标准溶液的吸光度,以标准溶液吸光度U为纵坐标、蛋白质含量(mg/mL)为横坐标绘制标准曲线[4](图1),可得标准溶液蛋白质含量标准曲线公式:y=0.6352x+0.0068,R2=0.9867 。
制备检测样品:每个菌株平板刮取500 mg菌丝放入加研磨珠的研磨管,液氮冷冻,放入生物样品研磨器5 000 r/min、5 min研磨2轮,再加入0.9%NaCl溶液1.0 mL,4℃、10 000 r/min离心10 min,取上清液0.1 mL,用0.9% NaCl溶液稀释至10 mL,得到待测样品。将紫外分光光度计波长设为280 nm,检测样品吸光度,每样品做3份平行取平均值,代入标准曲线计算对应样品蛋白质浓度[5]。根据样品蛋白质浓度计算平板菌体蛋白质含量。
1.3 光谱数据采集
提前12 h将样品平板从恒温箱中取出,放入25℃烘箱除去样品的冷凝水。在光谱采集过程中保持25℃环境温度、35%环境湿度,使待测样品温度和湿度基本一致[6]。设置光谱扫描范围为1 000~1 799 nm,分辨率为10 nm,波长准确性为0.2 nm,波长重复性为±0.05 nm,吸光度噪声小于50 μA。将待测平板放在检测槽正中,面朝上并保证旋转扫描区域都在平板之内,样品旋转一周为5 s,重复扫描采集3次形成一条反射光谱储存。每个菌株采集3 个平板光谱信息,作为原始光谱,并将光谱信息转换成吸光度值储存[7]。
1.4 光谱预处理
将各样品含量数据与光谱数据一一对应并进行关联,使用RIMP化学计量学分析軟件进行光谱处理,采用k-s分类方法将样品集划分为校正集和验证集,采用80∶20的马氏距离分配[8],将样品随机划分为168个校正集和42个验证集。
1.5 模型建立与评价方法
采用标准化、求导、平滑、信号校正等11 种预处理方法处理光谱。先用168个校正样品集在全光谱范围下应用偏最小二乘法(partia least squares method,PLS)建立校正模型,再将验正样品集进行外部验证,以校正标准差(SEC)、交互验证标准差(SECV)、预测标准差(SEP)、校正集相关系数(RC)和验证集相关系数(RP)、主因子数(MF)为参考标准,选出最优样品预处理方法[9]。根据一般模型评价的规则,在主因子数较少,SEP/SEC<1.2条件下,校正标准差、交互验证标准差、预测标准差越小且相互间越接近,说明近红外分析结果与化学分析结果越吻合,可信度越高;校正集相关系数和验证集相关系数越接近1,则模型预测值与化学分析值越接近,模型的准确度越高。以上述参数作为评价指标对模型进行优化时,应剔除个别异常值[10]。
2 结果与分析
2.1 化学分析结果
210个样品蛋白含量试验数据范围为 5.950%~14.510%,平均值为 10.000%,将其输入RIMP软件做参比值分析,参比值分布区见图2,经对比发现数据参比值变化幅度较大、分布范围比较宽,说明样品具有较好的代表性,建立的模型适用性较高[11]。
2.2 光谱数据预处理
经扫描获得210份平菇样品的近红外光谱图(图3)。从中可看出,光谱在1466 nm附近有最大吸收,该处位于蛋白质特征吸收谱段[122]。各样品的光谱波形很相似,但并不完全重合,这表明不同样本之间重现性良好,又存在差异。为了消除样品检测过程误差对建模的影响,需要对光谱进行预处理[13]。
2.2.1 校正集、验证集样品分析 由表1可见,校正集和验证集蛋白质含量数据的最大值、最小值及平均值与总样品数据接近,化学值分布的离散程度较高,表明校正集和验证集的分组合理,其数据相对于总样品具有代表性;验证集数据标准差、平均值与校正集偏差小,且上下限均包含在校正集数据内,因而验证集可用于对校正集所建模型的验证[14]。
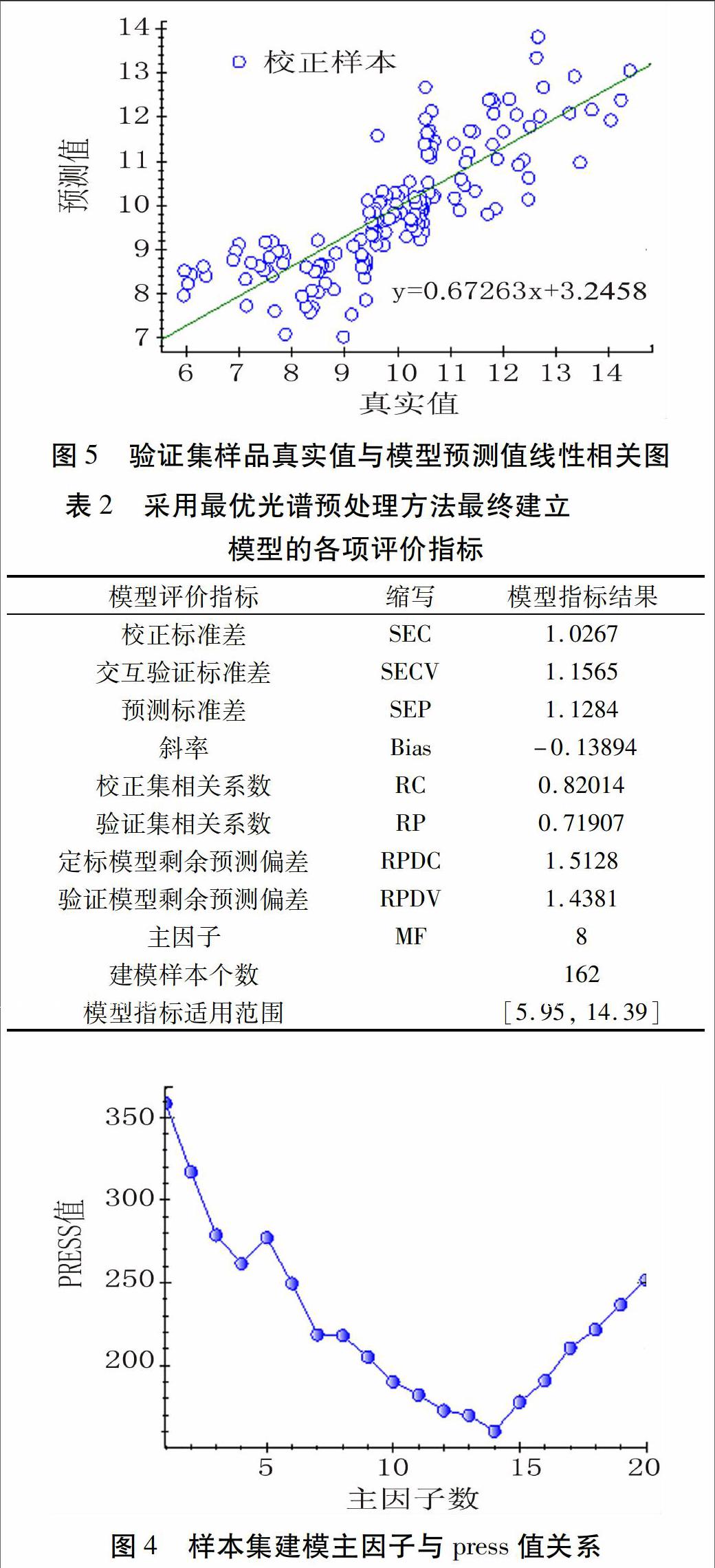
2.2.2 光谱预处理方法的选择与最优模型构建 通过RIMP软件中的自动优化功能,对最佳光谱预处理方法进行筛选,最终确定在1 000~1 799 nm光谱范围内,Savitzky-Golay平滑+Savitzky-Golay导数+多元散射校正(MSC)+均值中心化为最优光谱预处理方法,采用PLS方法建立样品集分析预测模型,用验证集样品对模型进行验证,并依据相应参评指标的优劣筛选出最优光谱预处理方法,最优参评指标见表2。图4显示当主因子数为14的时候,得到的press值最小,但综合试验数据分析,主因子数为8时,模型能够包含样品集绝大多数光谱信息和蛋白含量信息,模型的预测偏差最小,因此模型最佳主因子数为8。
2.2.3 模型验证结果 用样品集建立的模型对验证集进行预测,其回归效果如图5所示。从中可以看出真实值与预测值分布在直线两侧,其相关系数为0.67263,说明预测效果比较好,化学法和近红外仪器法测定结果无显著差异,近红外测定结果准确可靠。
3 讨论与结论
近红外光谱作为一种间接性鉴定技术,能够无损、快速、准确给出成分复杂物质的特异图谱,作为定性和定量分析的依据[16,17],该方法如果用于生产过程实时定量监测,将成为食用菌育种、生产自动化条件下质量控制的理想工具。
本研究以70个平菇菌株的210组数据为样本,采集样本在1 000~1 799 nm区域近红外光谱,以平板菌丝蛋白质含量为参考指标,通过化学分析方法得到与近红外光谱信息对应建模数据,分别输入软件,采用Savitzky-Golay平滑+Savitzky-Golay导数+多元散射校正(MSC)+均值中心化作为光谱预处理方法,应用偏最小二乘法(PLS)建立平菇蛋白质含量预测模型,经验证该模型真实值与预测值的相关系数为0.67263,预测结果符合有效范围,可用于平菇菌体蛋白质含量的快速无损检测。
后续试验中发现,在控制环境并严格操作流程的重复性试验条件下,大部分预测结果与实际试验数据偏差均在试验许可范围内,但建模样本数量、参考指标样本的分布广度、样品状态、水含量等都是影响模型准确度的重要因素,需要不断加入新的样品进行模型补充矫正,进一步提高模型的稳定性和准确性[15]。
参 考 文 献:
[1]石礼娟.基于可见光/近红外光谱的稻米质量快速无损检测研究[D].武汉:华中农业大学,2011.
[2]宦克为.小麦内在品质近红外光谱无损检测技术研究[D].长春:长春理工大学,2014.
[3]张玉森,姚霞,田永超,等.应用近红外光谱预测水稻叶片氮含量[J]. 植物生态学报,2010(6):704-712.
[4]吕慧. 基于近红外光谱技术的大米品质分析与种类鉴别[D].合肥:安徽农业大学,2011.
[5]于燕波. 近红外光谱分析技术在转基因水稻识别和高油棉籽筛选中的应用研究[D].北京:中国农业大学,2014.
[6]毕京翠,张文伟,肖应辉,等. 应用近红外光谱技术分析稻米蛋白质含量[J]. 作物学报,2006,32(5):709-715.
[7]汪成龙. 基于多源信息融合的马铃薯分级无损检测方法研究[D].武汉:华中农业大学,2014.
[8]刘培. 油菜品质多参数近红外检测技术的建立与应用[D].北京:中国农业科学院,2008.
[9]高居荣,樊广华,李圣福,等. 近红外光谱技术分析小麦品质的应用研究[J]. 实验技术与管理,2009(3):42-44.
[10]席志勇. 基于近紅外光谱技术荞麦无损检测方法研究[D].昆明:昆明理工大学,2013.
[11]俞永华.近红外光谱技术定量测定基质参数研究[J]. 光谱学与光谱分析,2011(11):2928-2931.
[12]宋丽华. 花生籽仁蛋白质含量近红外光谱模型的建立及育种应用[D].保定:河北农业大学,2011.
[13]秦利,刘华,杜培,等. 基于近红外光谱法的花生籽仁中蔗糖含量的测定[J]. 中国油料作物学报,2016,38(5):666-671.
[14]姚霞,汤守鹏,曹卫星,等. 应用近红外光谱估测小麦叶片氮含量[J].植物生态学报,2011,35(8):844-852.
[15]唐月明. 稻谷品种和品质的光谱快速无损检测研究[D].杭州:浙江大学,2008.
[16]王刚. 花椒挥发油含量近红外光谱无损检测研究[D].重庆:西南大学,2008.
[17]林新. 绿茶主要成分近红外光谱分析方法研究[D].武汉:华中农业大学,2008.
