饲粮添加苜蓿黄酮对奶牛瘤胃菌群的影响
2017-07-21占今舜邬彩霞刘明美苏效双詹康赵国琦
占今舜,邬彩霞,刘明美,3,苏效双,詹康,赵国琦*
(1.扬州大学动物科学技术学院,江苏 扬州 225009;2.江西省农业科学院畜牧兽医研究所,江西 南昌 330200;3.江苏联合职业技术学院淮安生物工程分院,江苏 淮安 223200)
饲粮添加苜蓿黄酮对奶牛瘤胃菌群的影响
占今舜1,2,邬彩霞1,刘明美1,3,苏效双1,詹康1,赵国琦1*
(1.扬州大学动物科学技术学院,江苏 扬州 225009;2.江西省农业科学院畜牧兽医研究所,江西 南昌 330200;3.江苏联合职业技术学院淮安生物工程分院,江苏 淮安 223200)
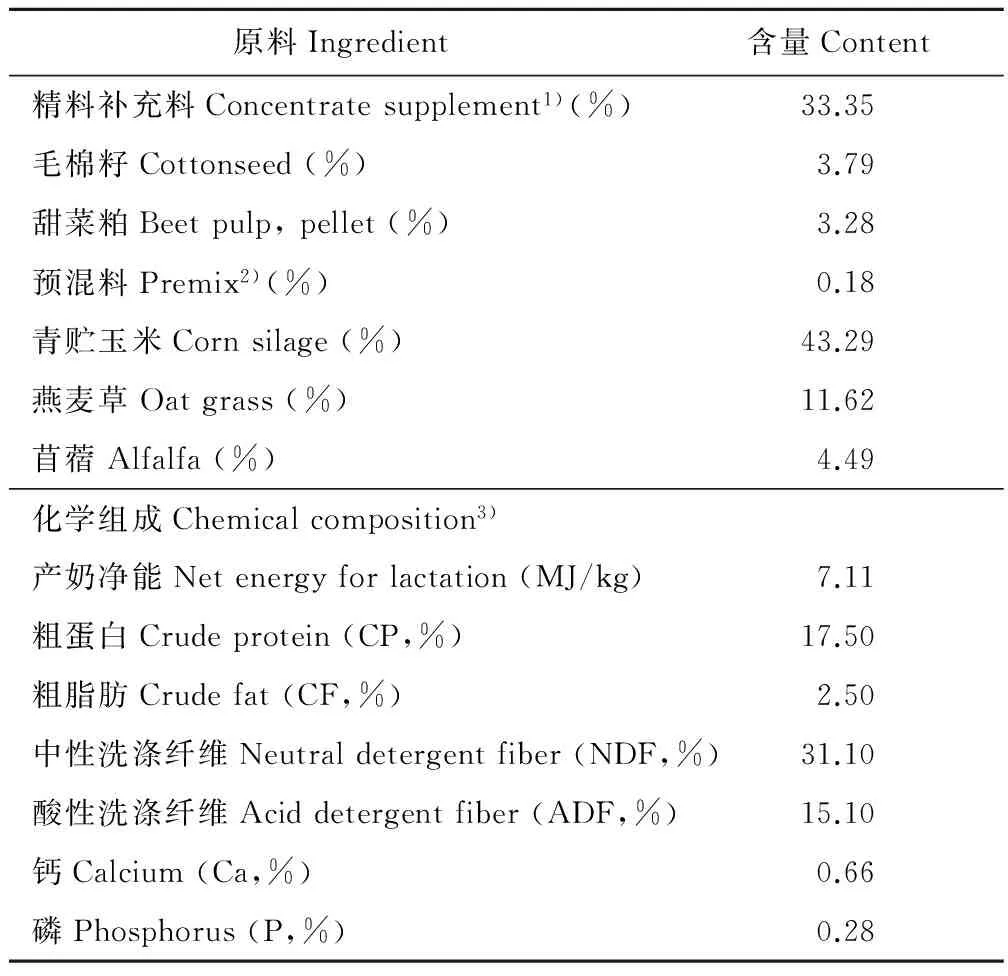
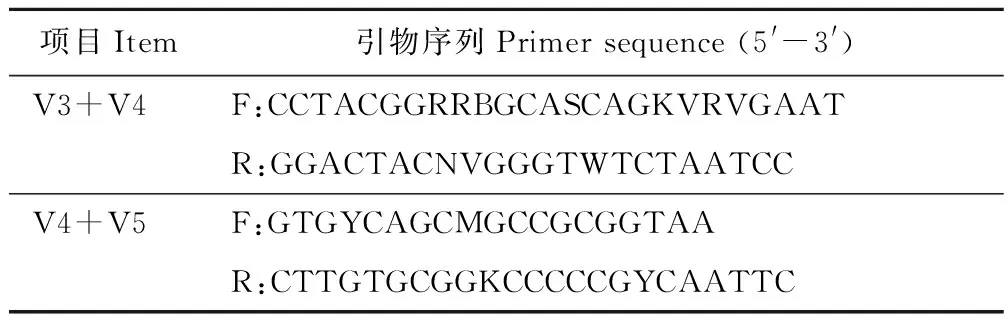
本试验旨在研究苜蓿黄酮对奶牛瘤胃菌群结构和组成的影响。选取4头装有瘘管的头胎荷斯坦奶牛,试验采用4×4拉丁方设计,每头奶牛每天饲喂全混合饲粮(TMR),其中分别添加0 g(A组)、20 g(B组)、60 g(C组) 和 100 g(D组)苜蓿黄酮。试验分为4期,每期24 d。每期采集瘤胃液,提取总DNA后在Illumina MiSeq 平台上进行测序。结果为:1)4个处理组共产生52747个OTU,共享16142个OTU,占总OTU数量的30.60%,其中试验A组的OTU数量最高。2)Shannon指数随着苜蓿黄酮添加剂量的升高呈先上升后下降的趋势,试验C组的Simpson指数显著高于试验D组和A组(P<0.05),但各处理组间的ACE和Chao 1指数无显著性差异。3)各试验组间的细菌门的数量没有显著性差异,但试验C组的细菌属的数量显著高于试验B组和试验A组(P<0.05)。在门水平上,SR1门相对丰度随着苜蓿黄酮添加水平的升高呈线性升高的趋势(0.05 苜蓿;黄酮类化合物;微生物;瘤胃;奶牛 反刍动物瘤胃是一个厌氧的生态系统,里面存在大量的细菌、原虫、真菌和古生菌等微生物,它是一个自然纤维素降解系统[1]。因此,研究瘤胃中的微生物种类和数量的改变有助于了解反刍动物消化代谢的作用机理。黄酮类化合物是植物次生代谢产物,广泛存在于植物中[2]。苜蓿(Medicagosativa)中含有丰富的蛋白质、维生素等营养物质,被广泛应用在奶牛生产中。黄酮类物质是苜蓿草中含量比较丰富的次生代谢产物,其在提高奶牛生产中可能发挥着重要的作用。Orhana 等[3]发现,黄酮类化合物能够抑制绿农假单胞菌、鲍曼不动杆菌、金黄色葡萄球菌、粪肠球菌等细菌和克鲁斯假丝酵母等真菌以及病毒。研究发现,富含黄酮化合物的印度苦楝树(Azadirachtaindica)种子提取物和意大利蓟(Carduuspycnocephalus)叶子提取物能够降低瘤胃原虫的数量,而含黄酮化合物的问荆草(Equisetumarvense)提取物、贯叶金丝桃(Hypericumperforatum)花提取物和鼠尾草(Salviajaponica)叶提取物对原虫的数量没有影响[4-6]。另外,据Oskoueian等[7]研究报道,黄酮、杨梅酮、儿茶素、芦丁和山奈酚能够降低瘤胃微生物数量,柚皮苷和槲皮素能够抑制产甲烷菌和原虫。以上结果表明,黄酮类化合物能够调节瘤胃微生物菌群,而且不同来源和结构的黄酮类化合物对瘤胃微生物的影响结果不同。目前,苜蓿黄酮对奶牛瘤胃微生物的影响研究尚未见报道。研究微生物生态学的方法有很多,高通量测序技术由于其能全面地反映样品微生物结构的优点,而被广泛应用于研究反刍动物瘤胃微生物[8]。因此,本试验通过16s DNA高通量测序方法研究苜蓿黄酮对奶牛瘤胃微生物区系的影响,探讨苜蓿黄酮是如何改变微生物区系来影响瘤胃的消化代谢,为苜蓿黄酮的应用提供一些参考价值。 1.1 试验设计和饲养管理 试验于2015年4月在上海光明荷斯坦牧业有限公司星火奶牛二场进行。选取4头装有瘤胃、小肠和回肠瘘管的头胎荷斯坦奶牛[平均体重为(500±25) kg,泌乳天数为(79±6) d],试验采用4×4拉丁方设计,每头奶牛每天饲喂全混合饲粮(total mixed ration,TMR)中分别添加0 g(A组)、20 g(B组)、60 g(C组) 和 100 g(D组)的苜蓿黄酮(含量为50%,由陕西绿清生物工程有限公司提供)作为试验处理。进行4期动物饲养试验,每期为24 d,其中预饲期为10 d。奶牛采用拴式饲养方式,自由饮水,每天饲喂3次,分别为06:30、13:30 和19:30,每日挤奶3次,时间分别为10:00、16:00和22:00。饲粮配方及营养水平见表1。粗蛋白、纤维和钙、磷等参照张丽英[9]的方法进行检测,产奶净能参照杨凤[10]的方法进行计算。 1.2 样品采集 每个试验期的第18天,在晨饲前半个小时打开牛瘤胃瘘管采集瘤胃液。在瘤胃的前后、左右采集瘤胃内容物,然后将内容物经过4层纱布过滤获得瘤胃液。将瘤胃液装入冻存管中,放在液氮中冻存,后转移到-80 ℃进行保存。 1.3 瘤胃微生物DNA提取 将瘤胃液放在超纯水中进行解冻,吸取300 μL的瘤胃液进行微生物DNA的提取。提取方法参照试剂盒(TIANamo Stool DNA Kit,天根)上的说明书进行,将提取的DNA 样品在Qubit 2.0 荧光光度计(Invitrogen,美国)上进行浓度的测定,并用0.8%琼脂糖凝胶进行纯度的检测。取5~50 ng瘤胃微生物总DNA,用MetaVxTM文库的制备试剂盒进行文库的制备(GENEWIZ,美国)。根据金唯智公司自主合成的V3、V4和V5区引物进行测序。设计通用引物如表2 所示。 第一次PCR扩增采用25 μL反应体系,体系组成为:TransStart Buffer (10×),2.5 μL;TransStart Taq (2.5 U/μL),0.5 μL;dNTPs (2.5 mmol/L),2.0 μL;(3+8) primer mix (1×),2.5 μL;DNA(30~50 ng),3.0 μL;Molecular water,14.5 μL。扩增反应程序为:94 ℃预变性3 min;94 ℃变性5 s,57 ℃退火90 s,72 ℃延伸10 s,进行14个循环,最后72 ℃ 延伸 5 min,10 ℃ 保存。第二次PCR扩增采用50 μL反应体系,体系组成为:TransStart Buffer (10×),2.5 μL;TransStart Taq (2.5 U/μL),0.5 μL;dNTPs (2.5 mmol/L),2.0 μL;primer cocktail (1×),4.0 μL;D50×/D70×,6.0 μL;Molecular water,10.0 μL;(3+8) amplification production,25.0 μL。扩增反应程序为:94 ℃预变性3 min;94 ℃变性5 s,60 ℃退火30 s,72 ℃延伸10 s,进行10个循环,最后72 ℃ 延伸 5 min,10 ℃ 保存。PCR产物经过质量检测后,根据说明书上所记载的步骤,在Illumina MiSeq仪器上进行DNA文库的构建,然后使用2×250 或 2×300双端进行测序。 表1 饲粮组成和营养水平(干物质基础)Table 1 Nutrient level and composition of feed (dry matter basis) 1) 精料补充料包含Concentrate supplement contained:玉米Corn 158.30 g/kg,豆粕Soybean meal 35.30 g/kg,大豆皮Soybean hull 52.90 g/kg,酒糟Distillers dried grains with soluble 54.90 g/kg,啤酒糟Brewer’s grain 16.70 g/kg,食盐Sodium chloride 2.20 g/kg,碳酸钙Calcium carbonate 3.50 g/kg,碳酸氢钙Calcium hydrophosphate 4.40 g/kg,碳酸氢钠Sodium bicarbonate 3.30 g/kg. 2) 预混料每kg含The premix contained the following per kg of diets:维生素A Vitamin A 3000 IU,维生素D Vitamin D 31400 IU,维生素E Vitamin E 30 IU,铁Iron 100 mg,铜Copper 10 mg,锌Zinc 35 mg,锰Manganese 20 mg,碘Iodine 0.30 mg,硒Selenium 0.10 mg,钴Cobalt 0.08 mg. 3) 产奶净能为计算值,其他为实测值。 Net energy for lactation was calculated and the others were measured. 表2 V3、V4和V5区引物序列Table 2 Sequence of primer for V3, V4 and V5 1.4 数据分析 通过软件CASAVA(V1.8.2),对测序结果原始图像数据进行图像碱基识别,经过质量分析,得到测序数据。通过软件Qiime(v1.7),使用 UCLUST方法进行operational taxonomic unit(OTU)聚类,对在97%的相似水平下所有序列进行OTU划分并进行生物信息统计分析。然后根据ACE(http://www.mothur.org/wiki/Ace)和Chao(http://www.mothur.org/wiki/Chao)指数来分析环境群落的物种丰度,根据Simpson(http://www.mothur.org/wiki/Simpson)和Shannon(http://www.mothur.org/wiki/Shannon)指数分析群落的多样性。通过weighted uniFrac距离矩阵进行 PCoA 作图分析,使用 UPGMA(unweighted pair group method with arithmetic mean)聚类方法,基于 weighted unifrac距离矩阵,将样品进行聚类。 α多样性等数据采用SAS 9.2软件的 PROC GLM 程序进行方差分析。分析模型为Yijkl=μ+Pi+Cj(l)+Tk+Eijk,其中Yijkl为因变量值,μ为总体均值,Pi为试验期效应(i=1~4),Cj(l)为第l拉丁方内第j试验奶牛的随机效应(j=1~4),Tk为饲粮处理效应(k=1~4),Eijk为随机误差。数据采用线性和二次线性回归分析以及Duncan法进行多重比较,P≤0.05 表示差异显著,而0.05 2.1 苜蓿黄酮对细菌丰富度和多样性的影响 试验4期16个样品共产生2771189条有效序列,经过优化后得到2487653条优质序列,平均序列长度为437 nt。试验A组的有效序列和优质序列均最高,随着苜蓿黄酮添加剂量的升高,有效序列和优质序列呈先降低后升高趋势(表3)。从图1可知,各试验组的样品稀释曲线趋于平缓,随着测序深度的增加,OTU数量将不再增加,说明测序数量比较合理。基于97%相似性的原则进行OTU分析(图2),4个处理组共产生52747个OTU,各组分别产生OTU数量为36421(A)、34185(B)、34756(C)和36310(D),其中共享16142个OTU,占总OTU数量的30.60%。从表3中可知,各组的ACE和Chao1指数无显著性差异,说明苜蓿黄酮对细菌丰度无显著影响。随着苜蓿黄酮添加剂量的升高,Shannon指数呈先上升后下降的趋势;试验C组的Simpson指数显著高于试验D组和A组(P<0.05),且随着苜蓿黄酮添加剂量的升高呈先上升后降低趋势;试验C组细菌属的数量显著高于试验B组和A组(P<0.05)。说明苜蓿黄酮能够影响细菌的多样性。 表3 苜蓿黄酮对细菌丰富度和多样性的影响Table 3 Effect of flavonoids from alfalfa on the richness and diversity of microbe 注:ACE和Chao1指数表示细菌丰富度,香浓和辛普森指数表示细菌的多样性。A、B、C和D表示试验组分别为0,20,60和100 g苜蓿黄酮组。不同小写字母表示差异显著(P<0.05),相同字母或无字母表示差异不显著(P>0.05)。下同。 Note: ACE and Chao1 indexes show the richness of microbe; Shannon and Simpson indexes show the diversity of microbe. A, B, C and D show the experimental groups with 0, 20, 60 and 100 g alfalfa flavonoids, respectively. In the same row, values with different lowercase letters mean significant differences (P<0.05), the same letter or no letter mean no significant differences (P>0.05).The same below. 图1 各个样品的稀释曲线Fig.1 Rarefaction curves of all samples 图2 OTU 维恩图Fig.2 OTU venn 表4 苜蓿黄酮对细菌组成和结构的影响(门水平)Table 4 Effect of flavonoids from alfalfa on composition and structure of microbe (at the phyla level) % 表5 苜蓿黄酮对细菌组成和结构的影响(属水平)Table 5 Effect of flavonoids from alfalfa on composition and structure of microbe (at the genus level) % 2.2 苜蓿黄酮对细菌组成和结构的影响 在门水平上(表4),随着苜蓿黄酮的添加水平的升高,SR1门相对丰度呈线性升高趋势(0.05 2.3 聚类分析 通过主坐标分析可知(图3),各试验组的样品均能很好地分开,对各试验组进行聚类分析(图4),发现试验A和B组细菌群落结构比较相似,试验C和D组细菌群落结构比较相似。结果说明,苜蓿黄酮对瘤胃菌群结构具有影响作用。 图3 PcoA分析Fig.3 Analysis by principal co-ordinate analysis 图4 UPGMA 聚类树分析Fig.4 Analysis by UPGMA tree 高通量测序技术可以快速、高效地了解瘤胃微生物群落结构和种类,已广泛应用于研究反刍动物瘤胃微生物菌群[11-12]。黄酮类化合物具有抑制细菌生长的作用,而瘤胃微生物生态群落比较复杂,因此,通过高通量测序技术可以更全面地了解苜蓿黄酮对瘤胃微生物区系的影响。 Shannon和Simpson指数是用来估算微生物多样性的指标。Simpson指数值越大,说明群落多样性越高;Shannon指数越大,说明群落多样性越高。在本试验中,Shannon和Simpson指数随苜蓿黄酮添加剂量的升高呈先上升后降低的趋势。另外,细菌属的数量随苜蓿黄酮添加水平的升高而呈先升高后降低的趋势。结果说明,瘤胃细菌的多样性随苜蓿黄酮添加水平(20~60 g)的升高而升高,但添加100 g时,瘤胃细菌的多样性下降。研究发现,黄酮类化合物能够通过抑制细菌核酸的合成和破坏细菌细胞膜,进而产生抗菌作用[13]。而且,黄酮的抑菌作用随着浓度增加而增强[14]。因此,苜蓿黄酮添加剂量超过60 g,降低瘤胃细菌多样性的原因可能是黄酮浓度高,发挥强抑菌和杀菌作用,进而降低瘤胃中细菌的种类和多样性。0 g组和20 g组细菌门的数量和属的数量比较接近,60 g组和100 g组细菌门的数量和属的数量比较接近。因此,0 g组和20 g组细菌群落的结构相似度比较高,60 g组和100 g组的细菌群落的结构相似度比较高。 Kim等[15]研究发现,反刍动物瘤胃微生物的厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)和变形菌门(Proteobacteria)数量最多,占细菌总门数量的91%。另外,对安格斯西口塔尔公牛和水牛瘤胃微生物宏基因组研究结果与其相似[16-17]。本试验得出相似的结果。互养菌门是一类革兰氏阴性厌氧细菌,广泛存在于动物肠道、土壤等厌氧环境中[18]。在本试验中,低浓度的苜蓿黄酮互养菌门相对丰度低,且随着苜蓿黄酮添加剂量的升高其相对丰度呈升高趋势。出现这种情况可能是高浓度苜蓿黄酮能够抑制与其发生竞争、拮抗细菌的生长,进而促进互养菌门细菌的生长,而低剂量发挥抑制的效果较弱所致。在本试验中,SR1门相对丰度随苜蓿黄酮添加剂量的升高呈线性升高趋势。可能是黄酮类化合物抑制了与其竞争、拮抗的细菌,进而促进其生长。 在属水平上,普雷沃菌属为反刍动物瘤胃微生物的优势菌属,其几乎可以单独完成或参与瘤胃内所有饲粮成分的降解[9]。从试验结果来看,苜蓿黄酮在一定程度上可能会有提高瘤胃普雷沃菌属数量的作用。从本试验中可知,饲粮中添加苜蓿黄酮能够提高厚壁菌门,毛螺菌科的毛螺菌属、假丁酸弧菌属和Shuttleworthia属的相对丰度,而这些属的细菌能够发酵碳水化合物。但是苜蓿黄酮可能会对该科下的Blautia属和Marvinbryantia属产生抑制作用。结果说明苜蓿黄酮可能会影响饲料中的碳水化合物的消化和代谢。厌氧弧菌属能够水解甘油三酯产生甘油和脂肪酸,并将甘油转化成丙酸和琥珀酸。从本试验的结果来看,苜蓿黄酮可能会对饲料中脂肪的消化和代谢具有影响作用,但添加剂量升高,作用会降低。互养菌属(Synergistes)是一种新发现的细菌属,从反刍动物中分离出的能够以精氨酸和组氨酸作为底物,降解动物饲料中的毒素,帮助动物生存[19]。从本试验中可知,瘤胃互养菌属相对丰度随饲粮中添加苜蓿黄酮剂量的升高呈升高趋势,说明苜蓿黄酮可能有助于降低饲料中的毒素进而可能影响奶牛的生产性能。锥形杆菌属(Pyramidobacter)是最近几年刚发现的新菌属,通过培养其主要终末代谢产物是乙酸、少量的异戊酸和痕迹量到少量的丙酸、异丁酸、琥珀酸、苯乙酸[20]。从本试验中可知,苜蓿黄酮可能具有抑制该菌属的作用,从而会影响瘤胃乙酸的生成。 1)饲粮中添加苜蓿黄酮能够影响瘤胃细菌属的数量和细菌的多样性,但对细菌丰富度没有影响。 2)在门水平上,随着苜蓿黄酮添加量的升高能够提高SR1门和互养菌门细菌相对丰度的趋势。在属水平上,苜蓿黄酮能提高SP3-e08属、U29-B03属、互养菌属、厌氧弧菌属和Shuttleworthia属细菌的相对丰度,而能够降低锥形杆菌属、Marvinbryantia属和Blautia属细菌的相对丰度。 References: [1] Yue Z B, Li W W, Yu H Q. Application of rumen microorganisms for anaerobic bioconversion of lignocellulosic biomass. Bioresource Technology, 2013, 128: 738-744. [2] Hossain M A, Kalbani M S, Farsi S A,etal. Comparative study of total phenolics, flavonoids contents and evaluation of antioxidant and antimicrobial activities of different polarities fruits crude extracts ofDaturametelL. Asian Pacific Journal of Tropical Disease, 2014, 4(5): 378-383. [3] Orhana D D, Özçelik B, Özgen S,etal. Antibacterial, antifungal, and antiviral activities of some flavonoids. Microbiological Research, 2010, 165: 496-504. [4] Patra A K, Kamra D N, Agarwal N. Effect of plant extracts oninvitromethanogenesis, enzyme activities and fermentation of feed in rumen liquor of buffalo. Animal Feed Science and Technology, 2006, 128: 276-291. [5] Broudiscou L P, Lassalas B. Effects ofLavandulaofficinalisandEquisetumarvensedry extracts and isoquercitrin on the fermentation of diets varying in forage contents by rumen microorganisms in batch culture. Reproduction Nutrition Development, 2000, 40: 431-440. [6] Soliva C R, Wimer S, Kreuaer M. Ruminal fermentation of mixed diets supplemented with St. John’s Wort (Hypericumperforatum) flowers and pine (Pinusmugo) oil or mixtures containing these preparations. Journal of Animal and Feed Sciences, 2008, 17: 352-362. [7] Oskoueian E, Abdullah N, Oskoueian A. Effects of flavonoids on rumen fermentation activity, methane production, and microbial population. BioMed Research International, 2013, (6): 1-8. [8] Myer P R, Kim M, Freetly H C,etal. Evaluation of 16S rRNA amplicon sequencing using two next-generation sequencing technologies for phylogenetic analysis of the rumen bacterial community in steers. Journal of Microbiological Methods, 2016, 127: 132-140. [9] Zhang L Y. Feed Analysis and Detection Technology of Feed Quality[M]. 3rd edition. Beijing: China Agricultural University Press, 2007. 张丽英. 饲料分析及饲料质量检测技术[M]. 第3版. 北京: 中国农业大学出版社, 2007. [10] Yang F. Animal Nutrition[M]. 2nd edition. Beijing: China Agricultural University Press, 2010. 杨凤. 动物营养学[M]. 第2版. 北京: 中国农业大学出版社, 2010. [11] Wang J W, Wang L Z, Yan T H,etal. Diversity of ruminal and fecal microbiota of goat. Chinese Journal of Animal Nutrition, 2015, 27(8): 2559-2571. 王继文, 王立志, 闫天海, 等. 山羊瘤胃与粪便微生物多样性. 动物营养学报, 2015, 27(8): 2559-2571. [12] Highlaner S K. High throughput sequencing methods for microbiome profiling: application to food animal systems. Animal Health Research Reviews, 2012, 13(1): 40-53. [13] Bahrin L G, Apostu M O, BIirsa L M,etal. The antibacterial properties of sulfur containing flavonoids. Bioorganic & Medicinal Chemistry Letters, 2014, 24: 2315-2318. [14] Tan H, Liu W. Bacteriostasis test on the flavonoid compounds fromEuphorbiahumifusaWilld.invitro. Journal of Traditional Chinese Veterinary Medicine, 2007, 4: 5-6. 谭红, 刘伟. 地锦草总黄酮的体外抑菌试验. 中兽医医药杂志, 2007, 4: 5-6. [15] Kim M, Morrison M, Yu Z. Status of the phylogenetic diversity census of ruminal microbiomes. FEMS Microbiology Ecology, 2011, 76(1): 49-63. [16] Brulej M, Antonopoulos D A, Miller M E,etal. Gene-centric metagenomics of fiber-adherent bovine rumen microbiome reveals forage specific glycoside hydrolases. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(6): 1948-1953. [17] Singh K M, Ahir V B, Tripatili A K,etal. Metagenomic analysis of surti buffalo (Bubalusbubalis) rumen: a preliminary study. Molecular Biology Reports, 2012, 39(4): 4841-4848. [18] JumasBilak E, Roudière L, Marchandin H. Description of ‘Synergistetes’ phyl. nov. and emended description of the phylum ‘Deferribacteres’ and of the family Syntrophomonadaceae, phylum ‘Firmicutes’. International Journal of Systematic & Evolutionary Microbiology, 2009, 59(5): 1028-1035. [19] Xing T, Mei L X, Li S. Research progress on relationship ofSynergistesand oral diseases. International Journal of Stomatology, 2009, 36(2): 177-179. 邢田, 梅陵宣, 李颂. 互养菌属与口腔疾病关系的研究进展. 国际口腔医学杂志, 2009, 36(2): 177-179. [20] Xiao X R, Li Y, Xiao L Y. The novel species and genus discovered and nominated from the human oral cavity in 2009-2012. West China Journal of Stomatology, 2013, 31(2): 217-220. 肖晓蓉, 李燕, 肖丽英. 2009-2012年发现的人口腔细菌新种属简介. 华西口腔医学杂志, 2013, 31(2): 217-220. Effects of alfalfa flavonoids as dietary additives on bacterial flora in the rumen of dairy cows ZHAN Jin-Shun1,2, WU Cai-Xia1, LIU Ming-Mei1,3, SU Xiao-Shuang1, ZHAN Kang1, ZHAO Guo-Qi1* 1.CollegeofAnimalScienceandTechnology,YangzhouUniversity,Yangzhou225009,China;2.InstituteofAnimalHusbandryandVeterinary,JiangxiAcademyofAgriculturalSciences,Nanchang330200,China;3.JiangsuJointInstituteofTechnologyofProfessionofHuai’anBio-engineeringBranch,Huai’an223200,China The objective of this study was to examine the effects of alfalfa flavonoids on the bacterial flora in the rumen of dairy cows. Four primiparous Holstein cows fitted with ruminal cannulas were used in a 4×4 Latin square design and were fed a total mixed ration containing 0, 20, 60, or 100 g alfalfa flavonoids per day (group A-D, respectively). The experiment had four periods and each period lasted 24 days. The ruminal fluid was collected during each period. Total bacterial DNA was extracted from the ruminal fluid, and 16S RNA sequences were obtained using the Illumina MiSeq platform. In total, 52747 operational taxonomic units (OTUs) were generated, and 16142 OTUs were shared by four groups, accounting for 30.60% of the total OTUs. The largest amount of OTUs was in group A. As the amount of alfalfa flavonoids in the diet increased, the Shannon index first increased and then decreased, but the abundance-based coverage estimator and Chao 1 indexes were unaffected. Simpson’s index was significantly higher for group C than for groups A and D (P<0.05). There were more bacterial genera in group C than in groups B and A, but the number of phyla in each group was not affected by the amount of alfalfa flavonoids in the diet. At the phylum level, the relative abundance of SR1 tended to increase linearly (0.05 alfalfa; flavonoids; microorganism; rumen; dairy cow 10.11686/cyxb2016358 2016-09-21;改回日期:2016-11-17 江苏省高校优势学科建设工程项目(PAPD),江苏省农业三新工程项目(SXGC[2016]326)和江苏省高校研究生科研创新计划项目(KYZZ_0367)资助。 占今舜(1985-),男,江西玉山人,博士。E-mail:zhanjinshun1985@163.com *通信作者Corresponding author. E-mail: gqzhao@yzu.edu.cn http://cyxb.lzu.edu.cn 占今舜, 邬彩霞, 刘明美, 苏效双, 詹康, 赵国琦. 饲粮添加苜蓿黄酮对奶牛瘤胃菌群的影响. 草业学报, 2017, 26(7): 82-89. ZHAN Jin-Shun, WU Cai-Xia, LIU Ming-Mei, SU Xiao-Shuang, ZHAN Kang, ZHAO Guo-Qi. Effects of alfalfa flavonoids as dietary additives on bacterial flora in the rumen of dairy cows. Acta Prataculturae Sinica, 2017, 26(7): 82-89.1 材料与方法
2 结果与分析







3 讨论
4 结论
