利用RAPD标记鉴定草地早熟禾种质资源的遗传多样性
2017-07-21涂明月李杰何亚丽李醒李俊袁晓君
涂明月,李杰,何亚丽,李醒,李俊,袁晓君*
(1.上海大学生命科学学院,上海市能源作物育种及应用重点实验室,上海200444;2.上海交通大学农业与生物学院,上海200240)
利用RAPD标记鉴定草地早熟禾种质资源的遗传多样性
涂明月1,李杰1,何亚丽2,李醒1,李俊2,袁晓君1*
(1.上海大学生命科学学院,上海市能源作物育种及应用重点实验室,上海200444;2.上海交通大学农业与生物学院,上海200240)
草地早熟禾是一种重要的冷季型草坪草,广泛种植于温带地区,且拥有众多的商品品种和丰富的野生资源。本研究收集了国内外72份草地早熟禾材料,对它们苗期的株高、叶宽、叶色及分蘖情况进行了考察,结果显示材料间的表型差异显著。此外,又在材料间进行了RAPD扩增产物的多态性鉴定,20条引物共扩增出115条多态性条带,多态性比率达91%。UPGMA聚类显示,材料被聚成两大类:一类为4个国内品种/系(“沪夜”、“沪青”、“沪禾2号”和“杂禾”),另一大类为剩余的所有材料。当遗传相似系数增大后,后一大类分成3组,组Ⅰ以国外引进品种为主,组Ⅱ和组Ⅲ则集中了更多的野生材料。RAPD聚类分析结果与材料背景来源之间存在一定程度的相关,但与苗期表型性状没有相关性。
草地早熟禾;RAPD;遗传多样性;聚类分析
草地早熟禾(Poapratensis)是一种广受欢迎的草坪草品种,广泛分布于温带地区,在美国北部、欧洲以及我国北部等地区都有大面积的种植。草地早熟禾具有出色的分蘖能力,其草质柔软,且拥有耐寒、耐践踏等特点,因此十分适合作为草坪种植。近年来,我国每年草坪草种的引种数量成倍增长[1],草地早熟禾品种几乎全部是从欧美国家引进。目前,国内育种者们也正陆续培育出一些品质优秀的草地早熟禾品种,如大青山[2]、沪禾[3]、沪夜、沪青[4]、青海草地早熟禾[5]等。
随着越来越多种类和数量的分子标记被开发,分子标记技术在各种作物的分子遗传图谱构建,品种鉴定与纯度分析,重要经济性状相关基因定位,以及种质资源多样性分析等领域的研究中应用已十分广泛。它在草坪草种质资源的多样性研究中也日益发挥越来越大的作用。以RAPD(random amplified polymorphic DNA)分子标记在草地早熟禾中的应用为例,国内田志宏等[6]曾利用17个RAPD引物对12份草地早熟禾品种进行了遗传多样性分析,将它们分成了5个类群;宁婷婷等[7]选用28个RAPD引物对15个草地早熟禾品种和1个加拿大早熟禾品种进行了分析,发现加拿大早熟禾的一个品种与其他草地早熟禾品种的相似性较低,单成一支;高丽娜等[8]利用69个RAPD标记对32份草地早熟禾品种也进行了遗传谱带的分析,聚类出5个类群;在国外,早在2001年Huff[9]就曾利用RAPD标记对草地早熟禾进行了研究;Johnson等[10]选取6条RAPD引物对228个草地早熟禾材料进行了聚类分析;Curley等[11]利用RAPD比较了123份草地早熟禾商业品种、种间杂交种和引种之间的遗传关系;Fard等[12]利用筛选出的17条RAPD引物对包括16份草地早熟禾在内的19份材料进行了多态性分析;Szenejko等[13]用18条RAPD引物对12个生态型,4个栽培品种和2个选育草地早熟禾材料进行了聚类分析。
此外,研究者们还利用了其他各类分子标记对草地早熟禾及其他早熟禾属植物进行了诸多研究,比如Rajasekar等[14]用15个RAPD 引物分析了27个粗茎早熟禾(Poatrivialis)品种的遗传关系。郭郁频等[15]利用ISSR(inter-simple sequence repeat)引物对34份早熟禾材料进行了遗传多样性分析。Goldman[16-17]也用ISSR标记在各早熟禾属材料中进行多态性筛选,并以此来鉴定种间杂交种。Arslan等[18]用ISSR标记研究了3个窄叶早熟禾(Poaangustifolia)和11个粗茎早熟禾材料间的遗传多样性。Honig等[19-20]开发了88个草地早熟禾的SSR(simple sequence repeats)标记,并利用其中的25个标记在247份草地早熟禾材料中进行了多态性的筛选及聚类。最近,赵闫闫等[21]也开发并利用SSR标记,对18个早熟禾材料进行了多态性筛选及指纹图谱构建。
本实验收集了72份国内外的草地早熟禾材料,除了国外引进品种和国内选育品种/系外,还包括了众多的国内外野生材料。本研究试图对它们进行一个较为全面的种质资源背景梳理,除了对苗期重要性状进行考察之外,还利用前期筛选获得的RAPD分子标记对这些材料进行了遗传多样性的鉴定。本研究结果将为草地早熟禾种质资源评价及后续的育种研究提供有用信息。
1 材料与方法
1.1 材料收集与种植
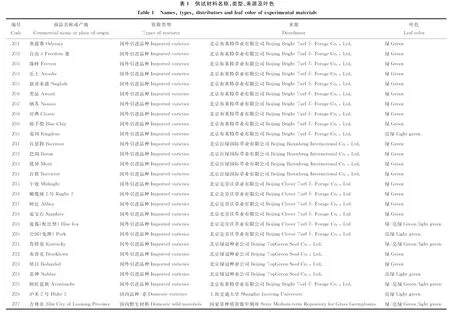
本实验共收集了国内外72份草地早熟禾材料,包括国外引进品种48份,国外野生材料10份,国内选育品种/系5份,以及吉林、甘肃、四川、贵州等地的野生材料9份(表1)。其中45份材料(Z27,Z43~Z71)由国家草种质资源中期库提供。2015年10月12日将收集的种子进行播种,每个材料约100粒,撒播于10 cm×10 cm×10 cm的塑料盆中,于上海交通大学农业与生物学院农场温室中发芽、培养。2015年11月21日,从每个材料中随机选取36株健康幼苗移栽到穴盘中,每穴一株。采用完全随机区组实验设计,共设6个区组,每个区组包括72个材料,每个材料种植一排(6株)。移栽后的植株继续置于温室中培养。隔天浇水1次,每月施Hoagland营养液1次,浇透。温室内气温在15~30 ℃,湿度40%~80%,自然光照。


1.2 苗期表型鉴定
在幼苗单株移栽当日(移栽前),测量各个材料植株的株高(发苗后未经修剪),随机选取有代表性的5株健康幼苗进行测量。2016年3月14日,对移栽单株进行叶宽和分蘖数考察,每个材料在每个区组6株中选有代表性的一株测量其叶宽并数分蘖数,6个区组即6次重复。同时,根据每个区组中同一材料6株的整体颜色表现进行叶色评价,分为绿色,亮绿色。如果该材料在6个区组中至少有5个区组颜色表现一致,即根据绝大多数表现将之记为绿色或者亮绿色,其余情况记为绿色/亮绿色。
由于一些野生材料和混合群体材料的某些表型出现了单株间的分离,所以在统计分析叶宽时,去除了Z27,Z42,Z60和Z65的数据;统计分蘖时,去除了Z27和Z65的数据。此外,因为材料Z72没有与其他材料同期播种,所以没有与其他材料一起进行株高、分蘖和叶宽的考察。
1.3 DNA的提取及RAPD多态性筛选
每个供试材料随机取10个单株,等量混合新鲜叶片,液氮冷冻,采用改良CTAB法提取基因组DNA[22]。用1.5%琼脂糖凝胶电泳对提取的基因组DNA进行检测,并稀释至40 ng/μL作为工作液,母液-20 ℃保存备用。本实验室前期曾利用6个草地早熟禾材料进行过400条RAPD引物多态性的筛选,从中选取可稳定扩增出多态性条带的24条引物供本实验使用。RAPD引物序列参照加拿大大不列颠哥伦比亚大学(University of British Columbia)公布的“NAPS Unit Standard Primers”序列。所有引物由生工生物工程(上海)有限公司合成。
PCR反应体系为10 μL:Taq DNA 聚合酶(3 U)0.08 μL,10×Buffer (Mg2+Plus) 1 μL,dNTP Mixture (25 mmol/L) 0.1 μL,引物(2 mmol/L)0.5 μL,模板DNA(40 ng)0.6 μL,ddH2O 7.72 μL;扩增程序为:95 ℃预变性3 min;95 ℃变性10 s;37 ℃引物退火35 s,72 ℃延伸50 s,循环40次;再72 ℃延伸8 min。用1.5%琼脂糖对PCR扩增产物进行检测,120 V电压,约40 min。在紫外成像系统下扫描观察并拍照。每个PCR反应至少重复两次。
1.4 数据统计与分析
用Microsoft Office Excel 2007对株高、叶宽、分蘖数的数据进行方差分析、显著性检验等统计分析,并绘制柱形图。选取在各个材料中能稳定清晰扩增的多态性位点进行统计,有带记为1,无带记为0,缺失记为999,用Excel形成0/1矩阵。利用NTSYS(Version 2.10e)软件计算品种间的遗传相似系数,再用非加权配对算术平均法(UPGMA)进行聚类分析,并生成树状图。
2 结果与分析
2.1 表型分析
根据对苗期株高的分析发现,不同材料40 d左右发芽苗的株高呈现显著差异(图1),从3.5 cm到11.0 cm不等。其中Z58,Z70,Z46,Z51,Z52和Z64的株高均超过10 cm,而Z59,Z47,Z28,Z24,Z60和Z29的株高则最矮,均不足4 cm。各材料的株高呈现正态分布,近70%的材料集中在6.5~10.0 cm。根据对苗期叶宽的分析发现,不同材料叶宽也呈现显著差异(图2),从1.78 mm到3.15 mm不等。其中Z22,Z34,Z52最宽,超过3 mm,而Z28,Z26,Z29则显著较窄,不足2 mm。
对单株移栽4个月左右的幼苗进行分蘖数分析,除了Z60之外,其他材料均有分蘖发生,80%的材料有3~5个分蘖(图3)。不同材料的分蘖数也呈现显著差异,Z49和Z56最多,超过6个,而Z45,Z67,Z47和Z14的分蘖数较少,不足3个。另根据苗期考察发现,供试材料叶片颜色大致分为两大类,绿色和亮绿色,还有一些介于两者之间(表1)。
2.2 RAPD多态性扩增与聚类分析
利用24条RAPD随机引物对72个草地早熟禾材料进行多态性扩增(图4),最终选取20条(S3,S18,S30,S32,S49,S82,S85,S92,S135,S146,S188,S229,S246,S254,S257,S285,S329,S341,S352和S400)能扩增出稳定清晰多态性条带的引物用于聚类分析。20条RAPD引物在72个品种中共扩增出126条条带,平均每条引物扩增6.3条,其中多态性条带115条。

图1 各材料苗期株高差异性比较Fig.1 Comparison of plant height at seedling stage

图3 各材料苗期分蘖数差异性比较Fig.3 Comparison of tillers number per plant at seeding stage
利用扩增效果最佳的41条多态性条带对72个材料进行UPGMA聚类分析(图5)。当遗传相似系数为0.52时,材料被分为两大类:一大类仅包含4个材料,均是上海交通大学选育的材料,分别是“沪禾2号”(Z26),“沪青”(Z28)、“沪夜”(Z29)和“杂禾”(Z42);另一大类包括了余下的68个材料。当相似系数增大到0.67时,该大类群又分成了3组:组Ⅰ包括43个材料,除3个为国内野生材料(Z72,Z27,Z61)外,其余均为国外引进品种(占比93.0%);组Ⅱ包括17个材料,由11个野生材料(3个国内,8个国外;占比64.7%)和6个国外引进品种构成;组Ⅲ包括8个材料,由1个国内选育品种,2个国外引进品种和5个野生材料(2个国内,3个国外;占比62.5%)构成。

图4 RAPD32在不同草地早熟禾材料中的扩增带型Fig.4 Amplification bands of different Kentucky bluegrass materials generated by RAPD32

图5 72个草地早熟禾材料的RAPD聚类分析Fig.5 UPGMA cluster analysis based on RAPDs of 72 Kentucky bluegrass materials *:国外引进品种Imported varieties;**:国内选育品种Domestic varieties;***:国外野生材料Wild materials abroad;****:国内野生材料Domestic wild materials.
3 讨论
3.1 RAPD在草坪草中的扩增效果
在众多种类的分子标记中,RAPD技术出现较早,由于其操作简便被大家广泛采用。但由于其引物较短,较固定标记而言稳定性欠佳,所以利用时必须要经过多轮的重复验证,以确保其准确性。本实验利用反复筛选获得的RAPD引物对材料进行了多态性条带的扩增,多态性比率达到91%,此结果与国内外同类研究报道[7-8, 10, 12-14, 18]一致,RAPD在草地早熟禾和早熟禾属植物中普遍具有很高的多态性。同时,RAPD在其他草坪草的研究中也取得了良好的扩增效果,多态性比率高。田志宏等[23]用 12个RAPD引物对从国外引进的 15 个高羊茅(Festucaarundinacea)品种进行研究,多态性带比率69.41%;李惠英等[24]利用20条RAPD引物对26份高羊茅材料进行分析,多态性条带比率达98.88%。赵念席等[25]利用16个RAPD引物对5个大针茅(Stipagrandis)种群90个个体进行扩增,多态性条比率100%。韩冰等[26]对7个不同地理分布的克氏针茅(Stipakrylovii)种群进行RAPD分析,扩增条带多态率99.22%。梁慧敏[27]利用20个RAPD对28个狗牙根(Cynodondactylon)居群进行多态性分析,多态性比率96.32%。在22个RAPD标记对16份旋花科草坪草马蹄金(Dichondrarepens)的研究[28]中,也同样获得了91.45%的高多态性比率。由此可见,RAPD分子标记在不同草中均能获得很高的多态性比率,它是理想的多态性筛选、种质资源鉴定的分子标记类型。
3.2 分子标记聚类与材料背景来源
本研究聚类分析结果显示,4个上海交通大学自育品种/系聚在一起,构成一大类。其中“沪青”和“沪夜”是两个越夏性强的材料,分别选育自“Evergreen”和“Midnight”两个国外品种[4],“沪禾2号”是它们的混合群体,“杂禾”则是多个草地早熟禾品种的混合群体,主体也是“沪青”和“沪夜”,因此它们被聚类在一起,完全符合实际的材料背景情况。与它们遗传关系较远的另一大类,在提高遗传相似系数后又被分成3组。有意思的是,组Ⅰ基本由国外引进品种构成,组Ⅱ和组Ⅲ则更多的集中了野生材料,这说明材料来源与RAPD聚类结果存在一定的相关性。赵桂琴等[29]曾利用ISSR分子标记技术对早熟禾属6种共31份种质材料进行遗传多样性分析,也同样发现来自同一地区或来源相同的材料大部分聚到一起。Farsani等[30]和Li等[31]利用ISSR标记分别对来源于伊朗和中国各地的狗牙根材料进行了种质资源的鉴定,他们都发现聚类结果与地域来源存在一定程度的相关性。Babu等[32]在利用RAPD 和 ISSR分子标记对象草(Pennisetumpurpureum)的研究中也发现,聚类结果与地域分布也存在相关。本实验结果与这些草中的研究结果相仿,说明利用分子标记对草坪草种质资源进行的聚类对了解它们的背景来源具有一定的参考价值。
3.3 苗期表型性状与分子标记聚类
综合株高、叶宽、叶色和分蘖数的考察结果,本实验搜集的材料间呈现出明显的表型差异,比如有些叶宽多分蘖(Z08、Z56、Z57),有些叶窄植株矮(Z24、Z28、Z29),有些分蘖少植株矮(Z47、Z59、Z60),有些则分蘖少植株高(Z64和Z67)……整体而言,国外引进品种多为叶片绿色、叶宽的类型,而野生材料则以叶窄、株型高的居多。可以根据这些特征来初步认识区分一些表型差异较大的材料,当然仅凭这些还很难区分那些表型相似的材料。
将本实验中涉及的苗期表型性状与分子标记聚类结果进行比对,两者无明显相关。就叶色看,引进品种/系几乎都呈绿色或者深绿色,可能是由于国外选育品种时偏爱叶色较深材料所致,而本实验中涉及的几个国内选育品种/系则无明显的颜色偏好。梁慧敏[27]曾对狗牙根进行了RAPD聚类分析,发现各类群中材料在外部形态上具有一定的相似性。而宁婷婷等[7]研究认为,早熟禾品种间的RAPD聚类关系与表型特征呈现不完全相关性。同样,Roldán-Ruiz等[33]认为多年生黑麦草(Loliumperenne)的形态特征和分子标记之间并不一致。由此可见,表型性状和分子标记聚类结果常常很难一致。在品种选育时往往会集中在少数一些重要农艺性状的改良上,而这些性状往往也正是种质资源鉴定时考察的表型特征,因此一些表型相似的材料很可能遗传背景却大相径庭。而且,作物表型性状的数量十分有限,所涉及的基因位点极少,因此它们很难反映材料的整体遗传背景;而分子标记的数量是无限的,特别是将不同类型分子标记联合使用时,可更准确地反映出材料间的遗传背景差异。
4 结论
本研究广泛搜集了目前国内外常见的72份草地早熟禾品种及野生材料,进行了一些重要苗期表型性状的考察,并利用RAPD分子标记对它们进行了聚类分析。主要结论如下:
1) 本实验搜集的草地早熟禾材料苗期表型性状差异明显,叶色主要为亮绿色和绿色两类,材料间株高、叶宽、分蘖能力差异显著。
2) RAPD引物扩增片段在草地早熟禾中表现出很高的多态性,因此它是筛选多态性的理想分子标记类型。
3) 分子标记聚类结果与材料背景来源具有一定的相关性,而与苗期表型性状无相关性。
References:
[1] Zhang R L, Zhao Q, Fan M,etal. Study progress of Kentucky bluegrass in our country. Pratacultural Science, 2005, 22(7): 67-70. 张瑞麟, 赵清, 范敏, 等. 我国草地早熟禾的研究进展. 草业科学, 2005, 22(7): 67-70.
[2] Emuhe, Hasiqiqige, Sun H L,etal. A new cool-season turfgrass variety——Kentucky bluegrass ‘Da Qing Shan’. Neimenggu Animal Science, 2000, 21(1): 43-45. 额木和, 哈斯其其格, 孙海莲, 等. 冷季型草坪新品种——大青山草地早熟禾. 内蒙古畜牧科学, 2000, 21(1): 43-45.
[3] He Y L, Chen J, Guo J F,etal. Breeding of new line “Huhe” and evaluation of germplasms of Kentucky bluegrass under artificial stresses and natural conditions in Shanghai. Journal of Shanghai Jiaotong University: Agricultural Science, 2009, 27(3): 189-195. 何亚丽, 陈杰, 郭金芳, 等. 草地早熟禾新品系“沪禾”的选育和在上海自然条件与人工逆境胁迫下的草坪特性鉴定. 上海交通大学学报: 农业科学版, 2009, 27(3): 189-195.
[4] Yuan X J, Biao Z J, He Y L. Selection of new lines ‘KBG03’ and‘ KBG04’ for summer-tolerance and turf quality evaluation of the new lines with 23 other germplasms in Kentucky bluegrass. Journal of Shanghai Jiaotong University: Agricultural Science, 2014, 32(3): 51-61. 袁晓君, 鲍真晶, 何亚丽. 草地早熟禾越夏性强新品系‘KBG03’和‘KBG04’的选育及其与23个种质资源的草坪特性比较. 上海交通大学学报: 农业科学版, 2014, 32(3): 51-61.
[5] Wang L Y, Bi Y F, Ma Y S,etal. Breeding of a new varietyPoapratensisL. cv. Qinghai for tolerance to low temperature. Chinese Journal of Grassland, 2010, 32(6): 16-20. 王柳英, 毕玉芬, 马玉寿, 等. 耐寒新品种青海草地早熟禾的选育. 中国草地学报, 2010, 32(6): 16-20.
[6] Tian Z H, Qiu Y F, Yan H,etal. Genetic diversity in Kentucky bluegrass based on random amplified polymorphic DNA (RAPD) analysis. Acta Agerstia Sinica, 2006, 14(2): 120-128. 田志宏, 邱永福, 严寒, 等. 用RAPD标记分析草地早熟禾遗传多样性. 草地学报, 2006, 14(2): 120-128.
[7] Ning T T, Zhang Z J, Jin C Z,etal. Genetic polymorphism of Bluegrass cultivars detected by RAPDs. Hereditas, 2005, 27(4): 605-610. 宁婷婷, 张再君, 金诚赞, 等. 早熟禾品种间遗传多样性分析. 遗传, 2005, 27(4): 605-610.
[8] Gao L N, Chen Y J, Gao L J,etal. Study on classification and genetic diversity of Kentucky bluegrasses by using RAPD markers. Journal of Northeast Agricultural University, 2007, 38(3): 325-329. 高丽娜, 陈雅君, 高丽娟, 等. 草地早熟禾种质资源遗传多样性的RAPD鉴定与分类研究. 东北农业大学学报, 2007, 38(3): 325-329.
[9] Huff D R. Characterization of Kentucky bluegrass cultivars using RAPD markers. International Turfgrass Society Research Journal, 2001, 9: 169-175.
[10] Johnson R C, Johnston W J, Golob C T,etal. Characterization of the USDAPoapratensiscollection using RAPD markers and agronomic descriptors. Genetic Resources and Crop Evolution, 2002, 49(4): 351-363.
[11] Curley J, Jung G. RAPD-based genetic relationships in Kentucky bluegrass: Comparison of cultivars, interspecific hybrids, and plant introductions. Crop Science, 2004, 44(4): 1299-1306.
[12] Fard J R, Zamani Z, Moghaddam M R F,etal. Evaluation of genetic diversity among some genotypes of Kentucky bluegrass by RAPD molecular markers. Horticulture Environment and Biotechnology, 2012, 53(4): 298-303.
[13] Szenejko M, Rogalski M. Characterization of morphological traits and RAPD polymorphism in selected forms of Kentucky bluegrass (PoapratensisL.). Biodiversity Research & Conservation, 2015, 37(1): 1-10.
[14] Rajasekar S, Fei S, Christians N E. Analysis of genetic diversity in Rough bluegrass determined by RAPD markers. Crop Science, 2006, 46(1): 162-167.
[15] Guo Y P, Ren Y X, Zhang Y C,etal. Genetic diversity analysis of bluegrass germplasm resources by ISSR markers. Chinese Journal of Grassland, 2014, 36(3): 28-34. 郭郁频, 任永霞, 张颖超, 等. 早熟禾种质资源ISSR遗传多样性分析. 中国草地学报, 2014, 36(3): 28-34.
[16] Goldman J. The use of ISSR markers to identify Texas bluegrass interspecific hybrids. Plant Breeding, 2008, 127(6): 644-646.
[17] Goldman J. Genetic relationships in advanced generation hybrids derived from crosses between Texas (Poaarachnifera) and Kentucky (Poapratensis) bluegrass using ISSR markers. American Journal of Plant Sciences, 2013, 4: 2440-2443.
[18] Arslan E, Tamkoç A. The application of ISSR-PCR to determine the genetic relationship and genetic diversity between narrow leaved bluegrass (Poaangustifolia) and rough bluegrass (Poatrivialis) accessions. Turkish Journal of Biology, 2011, 35: 415-423.
[19] Honig J A, Bonos S A, Meyer W A. Isolation and characterization of 88 polymorphic microsatellite markers in Kentucky bluegrass (PoapratensisL.). HortScience, 2010, 45(11): 1759-1763.
[20] Honig J A, Averello V, Bonos S A,etal. Classification of kentucky bluegrass (PoapratensisL.) cultivars and accessions based on microsatellite (Simple sequence repeat) markers. HortScience, 2012, 47(9): 1356-1366.
[21] Zhao Y Y, Yu F, Li Y,etal. Fingerprint establishment of 18Poapratensiscultivars with SSR markers. Grassland and Turf, 2016, 36(1): 31-34. 赵闫闫, 喻凤, 李媛, 等. 18个早熟禾品种SSR指纹图谱的构建. 草原与草坪, 2016, 36(1): 31-34.
[22] Clark M S. Plant Molecular Biology-A Laboratory Manual[M]. Berlin: Springer-Verlag Press, 1997.
[23] Tian Z H, Qiu Y F, Yan H,etal. Genetic diversity inFestucaarundinacesbased on random amplified polymorphic (RAPD) DNA analysis. Acta Prataculturae Sinica, 2007, 16(1): 58-63. 田志宏, 邱永福, 严寒, 等. 用RAPD标记分析高羊茅的遗传多样性. 草业学报, 2007, 16(1): 58-63.
[24] Li H Y, Lou Y H, Hu T,etal. Genetic diversity analysis of Chinese natural tall fescue (Festucaarundinacea) germplasms by RAPD. Acta Prataculturae Sinica, 2010, 19(6): 208-214. 李惠英, 娄燕宏, 胡涛, 等. 中国高羊茅种质资源遗传多样性的RAPD分析. 草业学报, 2010, 19(6): 208-214.
[25] Zhao N X, Gao Y B, Wang J L,etal. RAPD diversity ofStipagrandispopulations and its association with some ecological factors. Acta Ecologica Sinica, 2006, 26(5): 1312-1319. 赵念席, 高玉葆, 王金龙, 等. 大针茅种群RAPD多样性及其与若干生态因子的相关关系. 生态学报, 2006, 26(5): 1312-1319.
[26] Han B, Zhao M L, Yang J,etal. Genetic variation and ecological differentiation ofStipakryloviipopulation in Inner Mongolia plateau. Chinese Journal of Grassland, 2010, 32(4): 17-23. 韩冰, 赵萌莉, 杨劼, 等. 内蒙古高原克氏针茅种群的生态分化. 中国草地学报, 2010, 32(4): 17-23.
[27] Liang H M. Analysis on different populations ofCynodondactylonby RAPD. Acta Prataculturae Sinica, 2010, 19(1): 258-262. 梁慧敏. 不同居群狗牙根RAPD分析. 草业学报, 2010, 19(1): 258-262.
[28] Fu W, Tang C B, Wu J H,etal. RAPD analysis based identification ofDichondrarepensgermplasm resources. Pratacultural Science, 2010, 27(4): 106-111. 付薇, 唐成斌, 吴佳海, 等. 野生马蹄金种质资源的RAPD鉴别. 草业科学, 2010, 27(4): 106-111.
[29] Zhao G Q, Liu H, Liu M,etal. ISSR-based genetic diversity analysis ofPoaL. Acta Agerstia Sinica, 2011, 19(5): 781-786. 赵桂琴, 刘欢, 刘美. ISSR标记的早熟禾遗传多样性分析. 草地学报, 2011, 19(5): 781-786.
[30] Farsani T M, Etemadi N, Sayed-Tabatabaei B E,etal. Assessment of genetic diversity of bermudagrass (Cynodondactylon) using ISSR markers. International Journal of Molecular Sciences, 2012, 13(1): 383-392.
[31] Li H Y, Liu L, Lou Y H,etal. Genetic diversity of Chinese natural bermudagrass (Cynodondactylon) germplasm using ISSR markers. Scientia Horticulturae, 2011, 127(4): 555-561.
[32] Babu C, Sundaramoorthi J, Vijayakumar G,etal. Analysis of genetic diversity in napier grass (PennisetumpurpureumSchum) as detected by RAPD and ISSR markers. Journal of Plant Biochemistry & Biotechnology, 2009, 18(2): 181-187.
[33] Roldán-Ruiz I, van Euwijk F A, Gilliland T J,etal. A comparative study of molecular and morphological methods of describing relationships between perennial ryegrass (LoliumperenneL.) varieties. Theoretical and Applied Genetics, 2001, 103(8): 1138-1150.
Genetic diversity analysis of Kentucky Bluegrass cultivars by RAPDs
TU Ming-Yue1, LI Jie1, HE Ya-Li2, LI Xing1, LI Jun2, YUAN Xiao-Jun1*
1.SchoolofLifeScience,ShanghaiKeyLaboratoryofBio-EnergyCrops,ShanghaiUniversity,Shanghai200444,China;2.SchoolofAgricultureandBiology,ShanghaiJiaotongUniversity,Shanghai200240,China
There are many commercial cultivars and wild germplasms of Kentucky bluegrass (Poapratensis), an important cool-season turfgrass widely cultivated in temperate regions worldwide. In this study, 72 Kentucky bluegrass materials from China and elsewhere were collected and evaluated at the seedling stage by recording plant height, number of tillers, leaf width, and leaf color. All these traits differed significantly among the germplasms. Polymorphic bands were amplified from these germplasms by using random amplified polymorphic DNA (RAPD) primers. A total of 115 polymorphic bands (91%) was generated using 20 RAPD primers. The dendrogram grouped these germplasms into two main clusters. The first cluster contained four domestic varieties (‘KBG04’, ‘KBG03’, ‘Huhe 2’, and ‘Zahe’), while the second cluster contained the other varieties. The second group had a higher genetic similarity coefficient, and was divided into three subgroups: GroupⅠ consisting mainly of imported varieties, and Group Ⅱ and Group Ⅲ with mainly wild germplasms. The clustering analysis based on the RAPDs data was partly consistent with the source of the materials, but there was no correlation with seedling phenotypic traits.
Kentucky bluegrass; RAPD (random amplified polymorphic DNA); genetic diversity; clustering analysis
10.11686/cyxb2016374
2016-10-09;改回日期:2016-12-05
国家自然科学基金青年科学基金项目(31502002)和沪农科攻字(2013)第1-1号资助。
涂明月(1992-),女,安徽淮南人,在读硕士。E-mail:m18817510336@163.com
*通信作者Corresponding author. E-mail:yuanxjforever@163.com
http://cyxb.lzu.edu.cn
涂明月, 李杰, 何亚丽, 李醒, 李俊, 袁晓君. 利用RAPD标记鉴定草地早熟禾种质资源的遗传多样性. 草业学报, 2017, 26(7): 71-81.
TU Ming-Yue, LI Jie, HE Ya-Li, LI Xing, LI Jun, YUAN Xiao-Jun. Genetic diversity analysis of Kentucky Bluegrass cultivars by RAPDs. Acta Prataculturae Sinica, 2017, 26(7): 71-81.
