水稻的起源及驯化相关基因克隆研究进展
2017-07-18潘斌清王小虎钟卫国端木银熙江苏省常熟市农业科学研究所215500
俞 良 潘斌清 王小虎 钟卫国 端木银熙 (江苏省常熟市农业科学研究所 215500)
水稻的起源及驯化相关基因克隆研究进展
俞 良 潘斌清 王小虎 钟卫国 端木银熙 (江苏省常熟市农业科学研究所 215500)
现代遗传学已研究证实关键基因的变异与水稻驯化密切相关,研究水稻关键基因驯化过程,有助于弄清驯化过程中分子水平变异与自然选择间如何联系的问题。现对水稻起源及驯化相关的基因克隆研究进展进行综述,旨在为水稻驯化基因育种利用及分子设计改良育种提供参考。
水稻起源;驯化;基因克隆;研究进展
作物驯化是指人类从野生植物中选育出可栽培作物的过程。人类的主要粮食作物有水稻(Oryza sativa)、小麦(Triticum eastivum)、玉米(Zeamays)、稷(Panicummiliaceum)、薯类以及大量的蔬菜和水果等,这些都是人类驯化的产物。其中,水稻是世界上重要的粮食作物之一,是全世界50%以上人口的主食,而90%以上的水稻生产和消费都在亚洲。目前亚洲栽培的水稻大约是在1万年前从野生稻经人工驯化而来,在我国长江中下游地区,考古学家发现了这一时期人工使用水稻的证据[1-3]。在这次驯化中,种子的数量增加及大小的改变、育性的转变、株型的改变、种子形状及休眠和开花时间的调整、种皮颜色的丧失以及落粒性的丧失都是人工选择的关键,这些变化被称为“驯化综合征”(domestication syndrome)[4]。地球上物种资源丰富,其中许多对人类有价值的物种还未被人类驯化,对这些物种进行人工驯化对人类社会的发展具有重要的促进作用[1]。因此,通过研究栽培作物的驯化过程,从而认识作物的驯化机制,对现代农作物的遗传改良、新作物的驯化以及揭示作物的进化机制都具有非常重要的意义[5-7]。
目前,人类种植的水稻通常是指亚洲栽培稻(Oryza sativa L.),是目前世界上种植面积最大的粮食作物,现已被种植在除南极洲外的所有大陆[8]。迄今,虽然针对水稻的起源和驯化已开展了大量研究工作,但是对这一重要粮食作物的起源和驯化过程仍不清楚[1,8]。现通过介绍水稻起源与驯化相关基因克隆的研究进展,以期为水稻驯化基因育种利用及分子设计改良育种提供参考。
1 亚洲栽培稻的起源
水稻是研究谷类作物驯化的良好模式生物。稻属包含21个野生种及2个栽培种[9],栽培种分别为亚洲栽培稻(Oryza sativa L.)和非洲栽培稻(Oryzaglaberrima Steud.)。亚洲栽培稻遍布全球,特别集中在亚洲,是世界上最古老的农作物之一,同时也是极富多样性的作物,在全球有成千上万个品种; 非洲栽培稻则主要分布在非洲西部地区。亚洲栽培稻包含两个主要亚种:粳稻(Oryza sativa ssp. japonica)和籼稻(Oryza Sativa ssp. indica),可代表世界上大多数品种,如寿司水稻(Sushi rice)即是粳稻的一种类型,而意大利调味饭中所用的长粒水稻大多数是籼稻[10]。粳稻主要分布于东亚亚热带地区、东南亚高原和南亚高地;而籼稻则主要集中在亚洲热带地区[11]。由于水稻品种多样化,关于它的起源也一直是科学争论的话题。关于籼—粳亚种的起源有若干假说,但归纳起来主要有“籼粳同源”的单一起源论和“籼粳异源”的多起源论两种。单一起源理论认为粳稻和籼稻都是从野生稻(Oryza rufipogon)驯化而来;多起源理论模型认为粳稻和籼稻是从亚洲不同地方驯化而来。
1.1 单一起源论
目前,大多数学者认为籼、粳稻是栽培稻分化的主流,是栽培稻分化最为深刻和主要的两个方向。Oka和Chang[12]认为籼、粳稻来自共同的祖先,因为在被认为是O.sativa的祖先种O.rufipogon中,看不到籼粳分化的迹象。Oka和Morishima[13]假定籼粳起源一源论是建立在“粳型×野生稻”的杂交后代中出现似籼型个体、“籼型×野生稻”的杂交后代出现似粳型个体的结果之上,他们认为普通野生稻中不存在籼粳分化,只是潜伏着籼粳分化的可能性。Morishima等[14]和Oka[15]认为亚洲栽培稻(Oryza sativa L.)可能在不同的地区从多年生Oryza rufipogon分别被驯化成Oryza sativa的,由于驯化的选择压力而分化成indica和japonica的。Sano等[16]认为栽培稻来自一年生与多年生的中间型野生稻。但也有学者认为,多年生野生稻是栽培稻的直接祖先[17]。我国学者丁颖[18]根据籼稻在适应于湿地的习性及颖壳形状、叶枕及其他性状上都与野生稻相似的特点认为,籼稻起源于华南普通野生稻,而粳稻则是后来从籼稻分化来的,推测籼稻是基本型,粳稻是变异型。程侃声[19]与王象坤[20]等都认为“籼粳同源”,他们提出野生稻引向高山演化成粳稻,引向低洼地则演化成籼稻的假说。单一起源论假说认为,水稻的籼粳分化发生在人为驯化之后,是人为驯化的结果,但此假说没有得到同工酶和DNA分析结果的支持。Molina等[11]通过再测序630个基因片段,研究了驯化水稻的进化史,他们发现基因序列数据更吻合水稻的单一起源理论。通过大规模地对水稻基因进行再测序(gene resequencing)来追踪其几千年的进化历史,并得出水稻起源于中国的结论。该研究表明,驯化水稻(domesticated rice)可能最早出现在8 200~13 500年前的中国长江流域,从而推翻了驯化水稻可能有两个起源——印度和中国的多起源论观点。
1.2 多起源论
最早提出多起源论假说的是我国科学家周拾禄[21,22],他认为粳稻起源于我国安徽省的偏粳型野生稻,然后传播到中国各地;而籼稻是从国外传入的,很有可能是在印度驯化后传入中国。多起源论认为,籼稻和粳稻分别从各自的祖先野生稻演化而来,栽培稻的籼粳分化发生在人为驯化开始之前的野生状态。但是关于籼粳分化的起点、历史、方向等问题,仍需要进一步的研究和证据予以支持。自从现代分子生物学技术应用于栽培稻的起源地以及起源祖先种的研究以来,许多分子水平的证据支持多点多地起源的观点。这一观点往往倾向于籼稻和粳稻的差异发生在驯化之前,即现今这两个亚种的一系列差异是由于籼稻和粳稻分别起源于两个不同区域的不同祖先种群体,祖先种群体的差异即代表了驯化前的差异[23-25]。其中籼稻更有可能起源于印度东部喜马拉雅山区低海拔地区的湿热环境之中,而粳稻则更有可能起源于中国长江流域[23,25]。这一观点同时也认为异地起源的籼稻向热带和亚热带地区扩散,而粳稻则向地理上更北部的温带冷凉地区扩散,长期生长在差异极大的生态环境和气候条件更加极化了籼粳分化的程度[23]。基于以往按籼稻、粳稻、热带粳稻的分类,运用不同分子标记研究这几个类群之间的亲缘关系,认为粳稻起源于中国长江流域或中国东南部地区,籼稻起源于印度东部喜马拉雅山区低海拔地区的湿热环境之中,因为籼稻与东南亚地区热带粳稻的关系更近,之后喜马拉雅地区的籼稻由于少数民族的迁移而被引入中国南部和东南部地区,与当地的原始粳稻类型长期共存,从而形成了中国南方现在种植的籼稻品种[24-26]。
通过前人研究比较可发现,水稻的起源和驯化历史目前尚不明晰。关于亚洲栽培稻的起源是单一起源还是多起源现在还不能过早下结论。造成这一现象的主要原因是物种和群体之间的天然杂交和基因渐渗(introgression),这是非常普遍的自然现象,栽培稻之间以及栽培稻与野生稻之间持续不断的基因流和渐渗,野生稻分类模糊不清以及水稻种质资源鉴定中存在一些误判[1,3]。尽管单一起源和多起源假说都有证据支持,但大部分研究观点认为,籼稻和粳稻独立起源于不同的祖先,籼稻起源于东南亚、南亚地区的Oryza rufipogon(或Oryza nivara),粳稻起源于中国地区的Oryzarufipogon;但在驯化过程中伴随着不断的基因流和基因渐渗,一些基因片段特别是一些重要的与农艺性状相关的基因可能在水稻的一个类群中被驯化,然后又通过基因流渐渗到水稻的其他品种群中[27,28]。
2 水稻驯化性状及其驯化相关基因
在人类驯化栽培过程中,野生稻逐渐失去多年生、落粒性强、芒长、匍匐生长、部分异交、籽粒小和产量低等特征,取而代之的是一年生、落粒性适中、无芒或短芒、茎秆直立向上、自交、籽粒大小适中和产量水平高等典型的现代栽培稻的特征。人们通过亚种间以及栽培稻与野生稻间的杂交,并利用多种分子标记进行数量性状分析,已克隆出大量驯化基因,同时其功能核苷酸多态性(functional nucleotide polymorphisms, FNPs),即与农艺性状自然变异有关的DNA变异也已得到鉴定[29-47](见表1)。
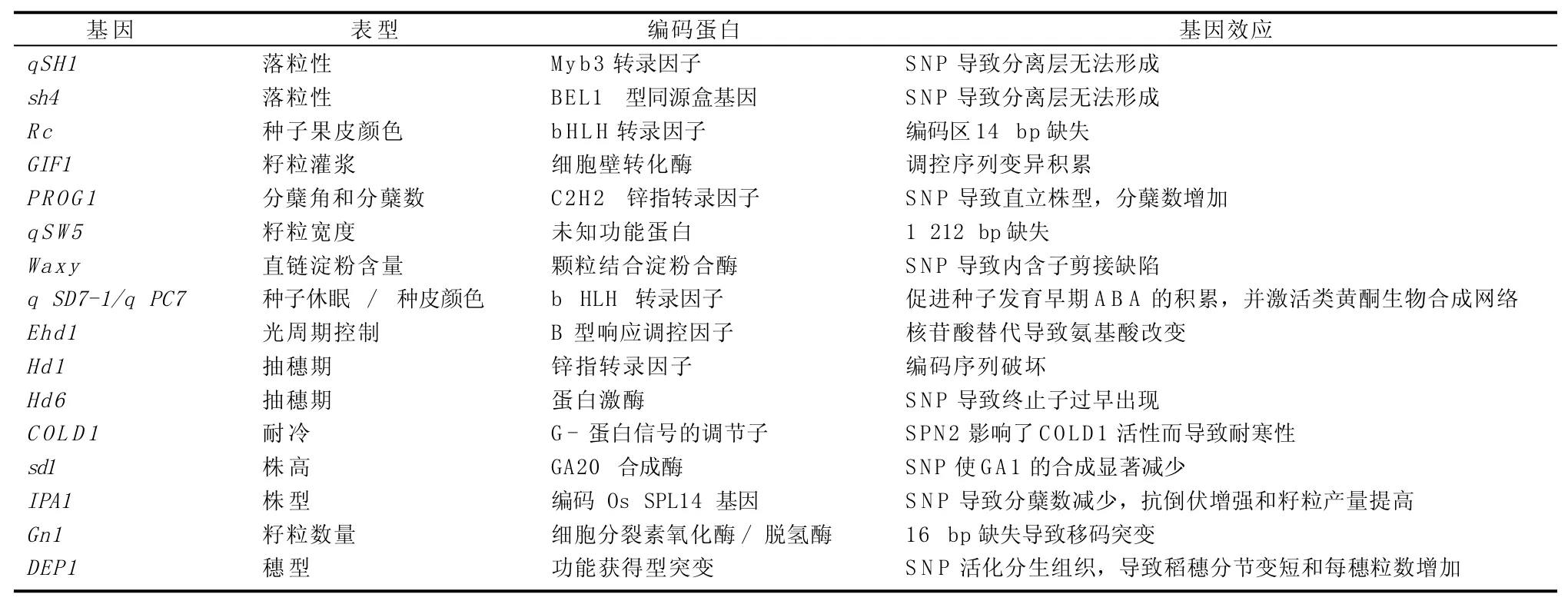
表1 已克隆的与水稻驯化相关基因
落粒性丧失是水稻驯化过程中最关键的事件,被认为是鉴定野生稻被驯化的直接形态学证据[48,49]。两个与落粒性状相关的基因qSH1[29]和sh4[30]被成功克隆,sh4是一个编码Myb3转录因子的QTL,由于第1个外显子中的单核苷酸替代产生了控制籽粒脱落的FNP,突变的等位基因导致籽粒和花梗之间的离层不能完全形成,从而减轻籽粒的脱落。sh4在所有的栽培种中都有相同的FNP,因而可能来自同一次驯化事件[30]。相比之下,粳稻的籽粒较籼稻难脱落,而qSH1正是控制这种籼粳亚种落粒性差异的QTL。qSH1编码一个BEL1型同源异型蛋白,qSH1基因5'端调节区的一个单碱基多态性(SNP)引起脱离层不能形成而导致落粒性丧失。对收集到各个水稻品种进行单体型分析和关联分析表明,该SNP与粳稻的落粒性紧密相关,这意味着水稻人工驯化过程中该SNP是人工选择的目标。对控制水稻落粒的两个位点sh4和qSH1的变异进行研究,发现不落粒sh4的等位基因在所有栽培稻中都被固定下来,多态性极低;进一步的系统发育和群体遗传分析表明,sh4等位基因为单次起源,在水稻驯化过程中通过人工选择而得以固定。相反,对qSH1的选择在栽培稻indica和japonica中都不明显。进一步的分析表明,人类对sh4基因的选择很强,以至于该基因在100年内就被固定在栽培稻的居群中,因此推测考古学所报道的不落粒表型的缓慢固定可能是由于在水稻培育的早期对变异相对较弱的选择,而不是对于sh4的选择[30]。
除落粒性状之外,还有多个与水稻驯化相关的基因被克隆,包括控制种皮颜色的Rc基因[33]、控制籽粒灌浆的GIF1基因[34]、控制分蘖角的PROG1基因[33,34]及控制籽粒形状的qSW5基因[37]、控制直链淀粉含量的Waxy基因[38]等。
Rc基因控制水稻种皮色泽,参与水稻种皮原花色素的合成,Rc基因在其第6个外显子中有大约14 bp的缺失,使其失去了生物合成花青素的功能,从而使稻米显示为白色,14bp片段缺失这一突变位点起源于粳稻亚种,跨越生殖隔离后导入籼稻亚种,同时跨越地理隔离从亚洲栽培区扩散到世界各地[37,38]。除Rc之外,Rd也参与原花色素的形成,从而也控制水稻种皮颜色,当Rc基因与Rd共同存在时,水稻种皮呈红色;当Rc基因单独存在时,水稻种皮呈褐色;当Rc突变为rc时,水稻种皮呈白色[38]。100年来禾本科作物中种子的休眠性与红色谷粒一直相关联,这种相关性表明,在杂草红稻中,控制种子休眠/种皮颜色的数量性状座位qSD7-1/qPC7是连锁的。通过基因内的重组将qSD7-1/qPC7定位到编码一个bHLH家族转录因子的Rc座位基因Os07g11020中,从而表明这两个性状的相关性是由于一因多效引起的。该一因多效基因在发育早期的种子中表达,促进了脱落酸生物合成关键基因的表达,导致引发休眠的植物激素ABA的积累;另外该基因还激活了8个保守的参与类黄酮生物合成途径的基因表达,在种皮组织表皮细胞下形成色素;该基因还能增加种子重量[39]。因此,这个一因多效座位很有可能是分别通过调节ABA和类黄酮的生物合成来控制休眠和色素这两个性状的。Waxy基因在第一个内含子5′端剪切位点的一个SNP,影响了编码直链淀粉酶的功能,从而降低了胚乳中的直链淀粉含量,提高了蒸煮后的米粒黏度[36]。由于种皮颜色、糯性是一种带有文化差异或者偏好性的选择目标,因而有人提出此类基因可能不是控制典型驯化性状的基因。抽穗期[42-44]和耐低温特性[43]等与地域特点相关,虽然这些基因也存在一定驯化性行为,但此类驯化不具有或暂时没发现普遍性。
水稻的产量性状是由包括籽粒数和粒重在内的多个组分共同构成的,并且受到大量数量性状基因座的控制。Wang等[32]从“中花11”的突变体中筛选到一株灌浆有缺陷的突变体gif1(grain incomplete filling 1),突变体在形态和结实率上表现正常,但与野生型相比,灌浆程度低、籽粒淀粉粒排列松散、垩白度高,最终的粒重比野生型低24%,直链淀粉和支链淀粉含量也明显低于野生型。随后用渐渗系(introgression lines)分离GIF1基因,该基因编码一个细胞壁转化酵素,在籽粒灌浆过程的早期参与碳的分配。由于驯化过程中启动子区域变异的积累,限制了该基因在灌浆过程中在胚珠维管束中的表达,从而有利于籽粒充实进而提高产量。
作物株型与产量密切相关,对于具有理想分蘖数和分蘖角的植株的选择被认为是水稻驯化过程中最关键的事件之一[50]。Tan[33]和Jin等[34]分别用不同材料克隆了控制水稻分蘖角的QTLPROG1,该基因编码一个由167个氨基酸组成的锌指转录因子,主要在腋芽分裂组织表达。在海南野生稻与栽培稻之间,该基因编码区有一个碱基的变异引起氨基酸的替换,推测该氨基酸的替换在人工驯化过程中被选择[32],而且对于分蘖数、分蘖角、籽粒数以及最终的产量表现出一因多效。在水稻进化过程中,由野生稻的PROG1基因进化为栽培稻的prog1后,基因的功能丧失,不仅由匍匐生长变成直立生长,株型得到改良,而且穗粒数增加,产量大幅度提高,表现为多效性。通过序列分析发现,来自17个国家的182个水稻品种的prog1基因表现出相同的变异,说明该基因可能是单起源的。PROG1基因的发现和分离对揭示水稻进化的分子机理及研究水稻株型调控的分子基础具有十分重要的意义。此类等位基因如sh4、gif1、prog1等在栽培种中被广泛固定下来,因而它们成为了区分栽培种和野生种的关键基因。
随着驯化相关基因的不断被克隆及其相应的功能核苷酸多态性(FNPs)信息的鉴定,人们可以对地方品种的基因型进行鉴定,再结合基因组范围内的RFLP分析以及地方品种生物地理起源的信息,推断作物起源过程中的历史事件以及作物驯化的地点,从而最终了解整个驯化的过程[51]。Konishi等[52]利用该方法对5个驯化相关基因qSW5、Rc、Waxy、Rd以及qSH1的6个FNPs在91个不同的粳稻地方品种(包括一些现代栽培品种)中进行分析,详细推断出粳稻驯化的过程,认为粳稻起源于印度尼西亚、菲律宾和东南亚半岛,并提出了一个关于粳稻驯化过程更加完善的模型。这一结果与利用考古学证据得出的粳稻起源于中国的长江流域的结论不相符合[53],这可能是由于进行此类分析时搜集的地方品种有效性不够、数量有限而导致了不同的结论,需要更多的考古证据以及对更多驯化基因的FNPs进行分析来验证。
3 展 望
水稻及其野生祖先复杂的群体结构导致水稻的驯化历史迄今尚不清楚,而栽培稻群体间的基因渐渗,以及栽培稻和野生祖先之间甚至和稻属其他种之间的基因流及其所带来的遗传和进化效应,是导致水稻驯化历史研究不清的关键原因。因此,准确描述水稻及其野生祖先之间的基因渐渗模式,发现一些处于渐渗区域内控制农艺性状的基因进而分析它们的起源,不仅能为水稻驯化历史的研究提供证据,也有助于水稻的遗传改良。
水稻是禾本科作物研究的重要模式作物,水稻全基因组测序已完成[54],大量水稻种质资源重测序数据的不断积累[28,55],为系统研究水稻基因功能和还原水稻真实的驯化进程提供了空前的数据量。虽然有许多和水稻驯化相关的基因相继被克隆,但仅仅是单个驯化基因作用机理较明晰,而对控制驯化性状的网络化通路的进化研究却相对缺乏。随着高能量测序技术的发展和功能基因组研究的深入,驯化性状的分子机理将会被全面揭示,而基于此的网络化通路研究必将更加真实地反映水稻驯化过程。驯化基因一般都相对保守,因此,水稻驯化基因的研究成果不仅能为水稻驯化过程再添加一些佐证,而且有利于开展其他禾谷类作物的驯化研究。随着大规模基因组测序技术的发展和相应数据分析方法的建立,在全基因组水平对水稻及其祖先进行大规模分析,水稻的起源和驯化过程将变得越来越明晰。驯化性状通路的网络化整合应用也将为现代分子设计育种的构建提供可能。
[1] Sang T,ge S. The puzzle of ricedomestication[J]. Journal of Integrative Plant Biology, 2007,49(6):760-768.
[2] Sweeneym,mc Couch S. The complex history of thedomestication of rice[J].Annals of Botany,2007,100(5): 951-957.
[3] Vaughand A, Balazs E, Heslop-Harrison J S. From cropdomestication to super-domestication[J].Annals of Botany, 2007,100(5):893-901.
[4] 金亮,卢艳,包劲松.水稻落粒性基因定位与克隆研究进展[J].分子植物育种,2009,7(2):393-397.
[5] Sang T,ge S.genetics and phylogenetics of ricedomestication[J].Current Opinion ingenetics &development, 2007,17(6):533-538.
[6] Konishi S, Ebana K, Izawa T. Inference of the japonica ricedomesticationprocess from thedistribution of six functional nucleotide polymorphisms ofdomestication-relatedgenes in various landraces andmodern cultivars[J]. Plant & Cell Physiology,2008,49(9):1283-1293.
[7] Tang H, Sezen U, Paterson A H.domestication and plantgenomes[J].Current Opinion in Plant Biology, 2010, 13 (2):160-166.
[8] Kovachm J, Sweeneym T,mc Couch S R. New insights into the history of ricedomestication[J].Trends ingenetics, 2007,23(11):578-587.
[9] Vaughand A,morishima H, Kadowaki K.diversity in the Oryzagenus[J].Current Opinion in Plant Biology, 2003,6(2):139-146.
[10]molina J, Sikoram,garud N, et al.molecular evidence for a single evolutionary origin ofdomesticated rice[J]. Proceedings of the National Academy of Sciences,2011, 108(20):8351-8356.
[11] Vaughand A, Lu BR, Tomooka N. Was asian rice (Oryza sativa)domesticatedmore than once[J] Rice,2008,1(1):16-24.
[12] Oka H I, Chang W T. Rice varieties intermediate between wild and cultivated forms and the origin of the Japonica type[J].Botanical bulletin of academia sinica, 1962(3): 109-131.
[13] Oka H I,morishima H. Phylogeneticdifferentiation of cultivated rice. Potentiality of wild progenitors to evolve the Indica and Japonica types of rice cultivars[J]. Euphytica,1982(31):41-50.
[14]morishima H, Hinata K, Oka H I. Comparison ofmodes of evolution of cultivated forms from two wild rice species,Oryza breviligulata and O. perennis [J]. Evolution,1963(17):170-181.
[15] Oka H I. Experiment studies on the origin of cultivated rice[J].Genetics,1974(78):475-486.
[16] Sano Y,morishima H, Oka H I. Intermediate perenniannual populations of Oryza perennis found in Thailand and their evolutionary sinificance[J]. Botanicalmagazine Tokyo,1980(93):291-305.
[17] Sampath S,govindaswami S. Wild rices of Orissa and their relationship to the cultivated varieties[J].Rice News Teller,1958,6(3):17-20.
[18] 丁颖.中国栽培稻种的起源及其演变[J].农业学报,1957, 8(3):243-260.
[19] 王象坤,陈一午,程侃声,等.云南稻种资源的综合研究与利用Ⅲ.云南的光壳稻[J].中国农业大学学报,1984,10(4):333-344.
[20] 程侃声,周季维,卢义宣,等.云南稻种资源的综合研究与利用Ⅱ.亚洲栽培稻分类的再认识[J].作物学报,1984,10(4):271-280.
[21] 周拾禄.中国是稻之起源地[J].中国稻作,1948,5(5):53-54.
[22] 周拾禄.水稻栽培科学与技术[M].北京:农业出版社,1981.
[23] Khushg S. Origin,dispersal, cultivation and variation of rice[J]. Plantmolecuar Biology,1997,35(1):24-34.
[24]garris A J, Tai T H, Coburn J, et al.genetic structure anddiversity in Oryza sativa L.[J].Genetics, 2005, 169 (3):1631-1638.
[25] Ikehashi H. Why are there indica type and Japonica type in rice History of the studies and a view for origin of two types[J]. Rice Science,2009,16(1):1-13.
[26] Li Zg, Hideki I. Nonindependentdomestication of the two rice subspecies, Oryza sativa ssp. Indica and spp. Japonica,demonstrated bymultilocusmicrosatellites[J].genetics,2008,179(2):965-976.
[27] Yang C C, Kawahara Y,mizuno H, et al. Independentdomesticationof Asian rice followed bygene flow from japonica to indica[J].Molecular Biology and Evolution, 2012, 29(5):1471-1479.
[28] He Z, Zhai W, Wen H, et al. Two evolutionary histories in thegenome of rice: The roles ofdomesticationgenes[J]. PLoSgenetics,2011,7(6):e1002100.
[29] Konishi S, Izawa T, Lin S Y, et al. An SNP caused loss of seed shatteringduring ricedomestication[J].Science, 2006,312(5778):1392-1396.
[30] Li C, Zhou A, Sang T. Ricedomestication by reducing shattering[J]. Science, 2006,311(5769):1936-1939.
[31] Sweeneym T, Thomsonm J, Pfeil B E, et al. Caught red-handed: Rc encodes a basic helix-loop-helix protein conditioning red pericarp in rice[J].The Plant Cell, 2006,18(2):283-294.
[32] Wang E, Wang J, Zhu X, et al.Control of ricegrainfilling and yield by agene with a potential signature ofdomestication[J].Naturegenetics,2008,40(11):1370-1374.
[33] Tan L, Li X, Liu F, et al. Control of a key transition from prostrate to erectgrowth in ricedomestication[J]. Naturegenetics,2008,40(11):1360-1364.
[34] Jin J, Huang W,gao J P, et al.genetic control of rice plant architecture underdomestication[J].Naturegenetics, 2008,40(11):1365-1369.
[35] Shomura A, Izawa T, Ebana K, et al.deletion in agene associated withgrain size increased yieldsduring ricedomestication[J]. Naturegenetics, 2008,40(8):1023-1028.
[36] Olsen Km, Caicedo A L, Polato N, et al. Selection underdomestication: evidence for a sweep in the rice waxygenomic region[J].genetics, 2006,173(2):975-983.
[37] Sweeneym T, Thomsonm J, Cho Yg, et at.Globaldissemination of a Singlemutation Conferring White Pericarp in Rice[J]. PLoSgenetics,2007,3(8):1418-1424.
[38] Furukawa T,maekawam, Oki T, et al. The Rc and Rdgenes are involved in proanthocyanidin synthesis in rice pericarp[J].The Plant Journal,2006,49(1):91-102.
[39]gu X Y, Foleym E, Horvathd P, et al. Association between seeddormancy and pericarp color is controlled by a pleiotropicgene that regulates abscisic acid and flavonoid synthesis in weedy red rice[J].genetics, 2011, 189(4):1515-1524.
[40]doi K, Izawa T, Fuse T, et al. Ehd1, a B-type response regulator in rice, confers short-day promotion of flowering and controls FT-likegene expression independently of Hd1[J].Genes anddevelopment, 2004,18(8): 926-936.
[41] Yanom, Katayose Y, Ashikarim, et al. Hd1, amajor photoperiod sensitivity quantitative trait locus in rice, is closely related to the Arabidopsis flowering timegene CONSTANS[J]. The Plant Cell, 2000,12(12):2473-2484.
[42] Takahashi Y, Shomura A, Sasaki T, et al. Hd6, a rice quantitative trait locus involved in photoperiod sensitivity, encodes the alpha subunit of protein kinase CK2[J]. Proceedings of the National Academy of Sciences,2001, 98(14): 7922-7927.
[43]ma Y,dai XY, Xu YY, et al. COLD1 confers chilling colerance in Rice[J]. Cell, 2015,160(6):1209-1221.
[44] Nagano H, Onishi K, Ogasawaram, et al.genealogy of the“Green Revolution”gene in rice[J].genes andgenetic Systems, 2005, 80(5): 351-356.
[45] Jiao Y Q, Wang Y H, Xued W, et al. Regulation of OsSPL14 by OsmiR156defines ideal plant architecture in rice[J].Naturegenetics,2010,42(6):541-544.
[46] Ashikarim, Sakakibara H, Lin S, et al. Cytokinin oxidase regulates ricegrain production[J].Science,2005, 309(5735):741-745.
[47] Huang X Z, Qian Q, Liu Z B, et at. Natural variation at thedEP1 locus enhancesgrain yield in rice[J].Naturegenetics, 2009,41(4):494-497.
[48] JonesmK, Liu XY. Archaeology. Origins of agricul ture in East Asia[J].Science,2009,324(5928):730-731.
[49] 区树俊,汪鸿儒,储成才.亚洲栽培稻主要驯化性状研究进展[J].遗传,2012,34(11):1379-1389.
[50] Wang Y, Li J. Rice, rising[J]. Naturegenetics, 2008, 40(11):1273-1275.
[51] 高清松,杨泽峰,徐辰武.水稻基因组进化的研究进展[J].扬州大学学报(农业与生命科学版),2009,30(2):34-41.
[52] Konishi S, Ebana K, Izawa T. Inference of the japonica ricedomestication process from thedistribution of six functional nucleotide polymorphisms ofdomesticationrelatedgenes in various landraces andmodern cultivars[J]. Plant and Cell Physiology, 2008,49(9):1283-1293.
[53]duncan A. V, Bao-R L, Norihiko Tomooka. The evolving story of rice evolution[J]. Plant Science,2008 (174):394-408.
[54] Project IRGS. Themap-based sequence of the ricegenome[J].Nature,2005,436(7052):793-800.
[55] Xu X, Liu X,ge S,et al. Resequencing 50 accessions of cultivated and wild rice yieldsmarkers for identifying agronomically importantgenes[J].Nat Biotechnol,2011, 30(1):105-111.
2017-02-15
苏州市农业科技创新工程(编号:SNG201509)。
