长链非编码RNA PRNCR1对胃癌细胞增殖、侵袭及转移的研究
2017-07-18蔡毅
蔡毅
(南华大学附属第一医院 急诊内科,湖南 衡阳 421001)
长链非编码RNA PRNCR1对胃癌细胞增殖、侵袭及转移的研究
蔡毅
(南华大学附属第一医院 急诊内科,湖南 衡阳 421001)
目的 探索胃癌MGC-803细胞和人正常胃黏膜上皮GES-1细胞中,长链非编码RNA PRNCR1的表达,以及沉默PRNCR1对胃癌MGC-803细胞增殖、侵袭及迁移的影响。方法利用实时荧光定量聚合酶链反应(qRT-PCR)检测细胞中PRNCR1表达水平;设计并合成PRNCR1-siRNA及对照序列(PRNCR1-NC)转染胃癌细胞,通过qRT-PCR检测细胞中PRNCR1的沉默效果;利用四甲基偶氮唑盐比色法检测胃癌细胞的增殖;Transwell实验检测胃癌细胞迁移和侵袭能力的变化。结果PRNCR1在胃癌MGC-803细胞中的表达高于人正常胃黏膜上皮GES-1细胞,PRNCR1-siRNA可以下调胃癌MGC-803细胞中PRNCR1的表达,并可以抑制MGC-803细胞的增殖和侵袭转移能力。结论PRNCR1-siRNA能够下调PRNCR1的表达,并有效抑制胃癌细胞的增殖和侵袭迁移力,为以PRNCR1为靶点的胃癌基因治疗奠定理论基础。
长链非编码RNA;前列腺癌非编码RNA1;增殖;侵袭;转移;胃癌
胃癌是源自胃黏膜上皮的恶性肿瘤,占全部恶性 肿瘤的第3位,居消化道恶性肿瘤的首位,占胃恶性肿瘤的95%。早期胃癌多无症状或仅有轻微症状,当临床症状明显时,病变已属晚期。由此可见,胃癌严重威胁着人类的健康[1]。目前,胃癌的致病基因、发病机制及影响预后的因素仍不清楚,因此寻找胃癌发生、发展的致病因素具有重要意义。
长链非编码RNA(long non-coding RNA,lncRNA)是一类不具有蛋白质编码功能、长度>200 nt的RNA。最新研究显示,lncRNA并非既往认为的“噪音”RNA,而是在生物进化过程中高度保守,具有重要调控功能的非编码RNA[2]。前列腺癌非编码RNA1(prostate cancer non-coding RNA 1,PRNCR1)定位于染色体8q24,长约13 kB,已经被证实在前列腺癌、结肠癌中发挥重要作用[3]。最新研究发现,PRNCR1的基因多态性与胃癌的患病风险有关[4]。但PRNCR1在胃癌中的作用尚不清楚。本实验通过探索PRNCR1在胃癌细胞和人正常胃黏膜上皮细胞中的表达,并通过RNA干涉技术沉默胃癌细胞中PRNCR1的表达,检测其对胃癌细胞增殖、侵袭及转移能力的影响,为将PRNCR1分子作为胃癌基因治疗靶点提供理论基础。
1 材料与方法
1.1 材料
Trizol细胞裂解液购于北京博润莱特科技有限公司,RNA反转录试剂、实时荧光定量聚合酶链反应(quantitative real-time polymerase chain reaction,qRT-PCR)试剂盒购自日本TaKaRa公司,细胞转染试剂购自美国Invitrogen公司,四甲基偶氮唑盐比色法[3-(4,5-dimethyl-2-thiazolyl)-2,5-diphenyl-2-H-tetrazolium bromide,MTT]试剂购自美国Promega公司,细胞侵袭转移检测transwell小室购自美国Milipore公司,基质胶购自美国Sigma公司,MGC-803细胞、GES-1细胞购自上海细胞库。
1.2 方法
1.2.1 PRNCR1-siRNA的设计和合成 根据PRNCR1序列,按照siRNA作用靶点设计原则,利用美国Invitrogen公司的siRNA作用靶点设计软件,委托上海吉玛基因股份有限公司合成相应的siRNA及对照序列,分别为PRNCR1-siRNA、PRNCR1-NC。1.2.2 细胞培养和转染 MGC-803细胞、GES-1细胞用含100 ml/L胎牛血清的达尔伯克必需基本培养基(dulbecco's minimum essential medium,DMEM),于37℃、50 ml/L二氧化碳CO2细胞培养箱中培养。适量MGC-803细胞接种在25mm2培养皿中,待细胞密度达70%~80%后,将合成的PRNCR1-siRNA、PRNCR1-NC经Lipofectamine 2000转染至MGC-803细胞中,具体操作按Lipofectamine 2000试剂盒说明书操作。转染后的细胞分别为PRNCR1-siRNA组和PRNCR1-NC组。
1.2.3 qRT-PCR检测PRNCR1的表达 利用qRT-PCR对PRNCR1表达进行检测。收集转染48 h后的细胞,利用Trizol细胞裂解液提取细胞,得总RNA,并反转录为cDNA。PRNCR1定量PCR引物。正向引物:5'-CCAGGGGGAAACACACAG-3';反向引物:5'-AAATGGCAGTTTCCTTCAATG-3'。β-actin作为看家基因,正向引物:5'-TTGGCTTGACTCAGG ATTTA-3',反向引物:5'-ATGCTATCACCTCCCCTGT G-3'。引物序列合成由深圳华大基因公司完成。反应条件:95℃预变性 4 min,95℃变性 12 s,59℃退火35s,共41个循环。每组3个复孔,实验重复3次,将所得数据绘制成柱状图。
1.2.4 MTT实验检测MGC-803细胞增殖 取转染48 h后且处于对数生长期的MGC-803细胞,常规洗涤2遍,用胰蛋白酶消化细胞,离心重悬后制成单细胞悬液,并调整细胞浓度为3×104个/ml,加入细胞悬液200 μl/孔,每组设7个复孔。共培养5个96孔板,分别培养1~5 d,检测1板/d。每天于实验结束前4 h,加入5 mg/ml MTT 20 μl/孔,放入孵箱继续培养4 h,弃上清液,加入二甲基亚砜(dimethyl sulfoxide,DMSO)150 μl/孔,震荡 11 min,使 MTT 的还原产物充分溶解,于490 nm处测定细胞的光密度(optical density,OD)值。
1.2.5 Transwell实验检测MGC-803细胞侵袭和转移能力 MGC-803细胞转染48 h后,通过Transwell实验检测细胞侵袭和转移力的变化,每组设3个复孔。胰蛋白酶消化各组细胞,无菌磷酸盐缓冲溶液(phosphate buffer saline,PBS)洗 3遍,离心后用牛血清蛋白(bovine serum albumin,BSA)10 g/L 无血清培养基重悬细胞,将细胞密度调整为1×105个/ml,事先用基质胶包被好Transwell小室中上室的基底膜,每组细胞悬液中取150 μl加入上室中,将小室放入24孔板中,下室中加入含有胎牛血清的培养基500 μl,培养 36 h。结束后,将小室取出,PBS 轻轻冲洗,棉签擦去小室上室内层的细胞,95%乙醇固定6 min,4 g/L结晶紫溶液染色后,倒置显微镜下计数,每样本随机选取5个视野进行计数后取平均值,绘制柱状图。检测MGC-803细胞转移能力时,小室的上室中无需基质胶包被基底膜,其他步骤与上述方法相同。
1.3 统计学方法
数据分析采用SPSS 19.0统计软件,计量资料以均数±标准差(±s)表示,用t检验或重复测量设计的方差分析,P<0.05为差异有统计学意义。
2 结果
2.1PRNCR1在MGC-803细胞中高表达
收集对数生长期的MGC-803和GES-1细胞,提取细胞总RNA,qRT-PCR检测PRNCR1的表达。结果显示,经标准化后,GES-1细胞PRNCR1相对表达量为(1.000±0.051),MGC-803细胞PRNCR1相对表达量为(3.146±0.783),经t检验,差异有统计学意义(t=6.844,P=0.017),与GES-1细胞比较,PRNCR1在MGC-803细胞中高表达。见图1。
2.2 PRNCR1-siRNA下调MGC-803细胞中PRNCR1的表达
PRNCR1-siRNA及对照序列转染MGC-803细胞后48 h,抽提细胞总RNA,qRT-PCR检测其表达。结果显示,经标准化后,PRNCR1-NC组PRNCR1相对表达量为(1.000±0.153),PRNCR1-siRNA 组细胞PRNCR1相对表达量为(0.488±0.091),经 t检验,差异有统计学意义(t=7.841,P=0.009),PRNCR1-siRNA组PRNCR1表达较PRNCR1-NC组下调。见图2。

图1 细胞中PRNCR1的表达 (±s)
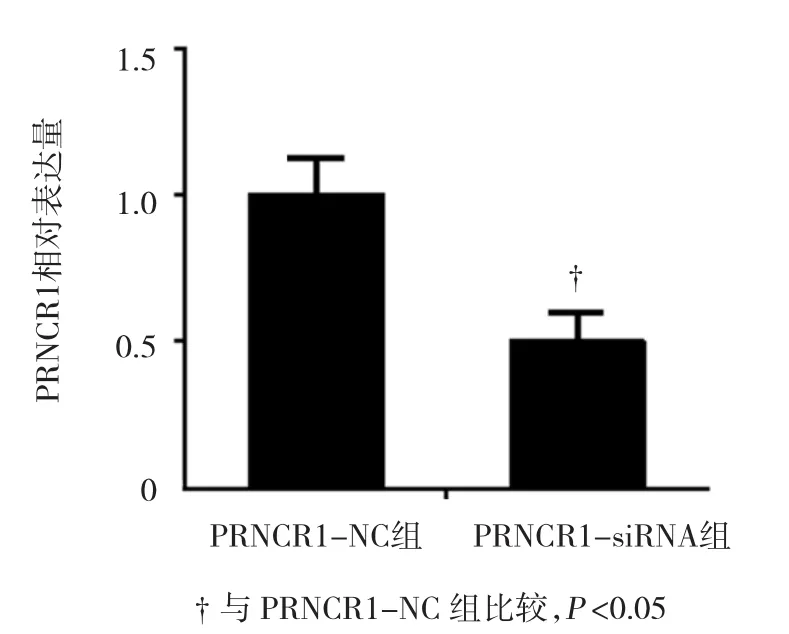
图2 两组细胞PRNCR1表达变化 (n=3,±s)
2.3 PRNCR1-siRNA抑制MGC-803细胞增殖
MTT实验检测PRNCR1-siRNA对MGC-803细胞增殖的影响,PRNCR1-siRNA与PRNCR1-NC组术后 24、48、72、96和 124 h测量的 OD 值比较,采用重复测量数据的方差分析,结果:①不同时间点的OD值有差异(F=8.105,P=0.014);②PRNCR1-iRNA组与PRNCR1-NC组的OD值有差异(F=16.217,P=0.008),PRNCR1-siRNA组较PRNCR1-NC组OD值低,增殖速度较慢;③PRNCR1-siRNA组与PRNCR1-NC组的OD值变化趋势有差异(F=7.814,P=0.032),从检测后第3天开始,PRNCR1-siRNA组细胞的增殖速度较PRNCR1-NC组缓慢。见图3和附表。
2.4 PRNCR1-siRNA抑制MGC-803细胞的侵袭
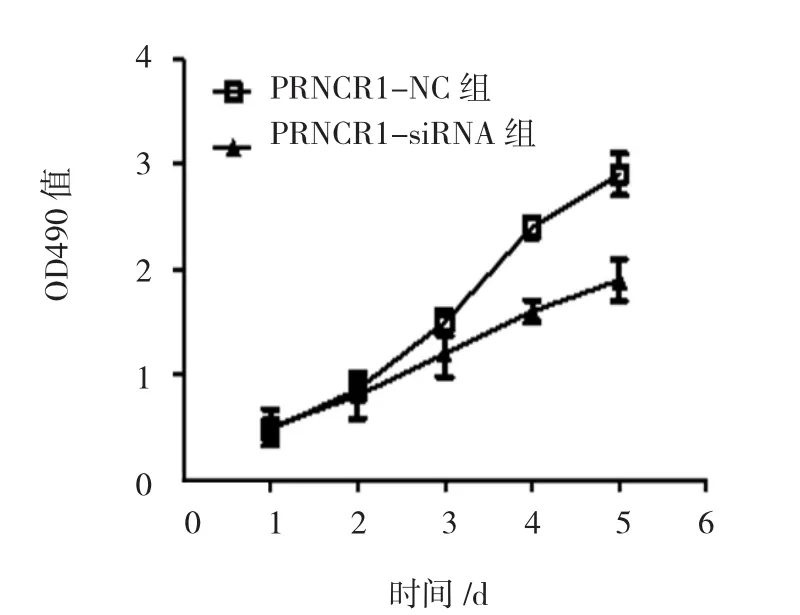
图3 PRNCR1-siRNA对细胞增殖的影响
附表 两组各时间点OD值比较 (n=3,±s)
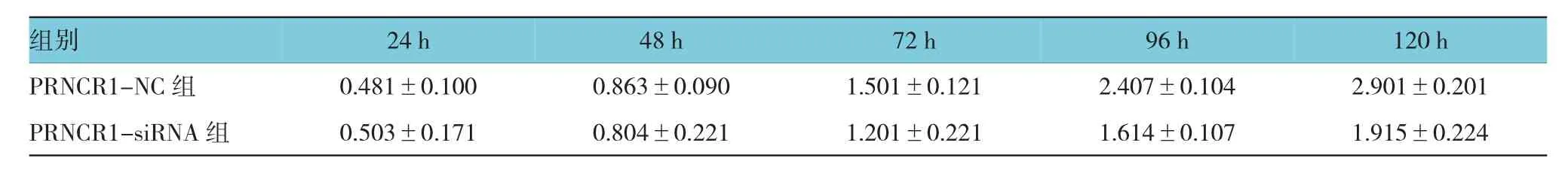
附表 两组各时间点OD值比较 (n=3,±s)
组别120 h PRNCR1-NC 组 0.481±0.100 0.863±0.090 1.501±0.121 2.407±0.104 2.901±0.201 PRNCR1-siRNA组 0.503±0.171 0.804±0.221 1.201±0.221 1.614±0.107 1.915±0.224 24 h 48 h 72 h 96 h
PRNCR1-siRNA及对照序列转染MGC-803细胞48h后,结果显示,PRNCR1-siRNA组穿过Matrigel包被的微孔膜的细胞数目为(21.635±7.424)个/高倍镜,PRNCR1-NC组穿过Matrigel包被的微孔膜的细胞数目为(52.471±9.518)个/高倍镜,经t检验,差异有统计学意义(t=10.471,P=0.025),PRNCR1-siRNA组穿过Matrigel包被的微孔膜的细胞数目少于PRNCR1-NC组。说明沉默PRNCR1表达后,可以抑制MGC-803细胞的侵袭能力。见图4。
2.5PRNCR1-siRNA抑制MGC-803细胞迁移
PRNCR1-siRNA及对照序列转染MGC-803细胞48 h后,PRNCR1-siRNA组穿过Matrigel包被的微孔膜的细胞数目为(23.534±5.176)个/高倍镜,PRNCR1-NC组穿过Matrigel包被的微孔膜的细胞数目为(41.512±8.873)个 /高倍镜,经 t检验,差异有统计学意义(t=9.214,P=0.017),PRNCR1-siRNA组穿过Matrigel包被的微孔膜的细胞数目少于PRNCR1-NC组,说明沉默PRNCR1表达后,可以抑制MGC-803细胞的迁移能力。见图5。
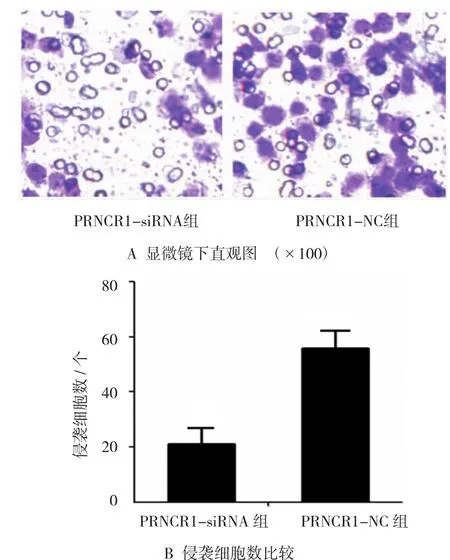
图4 MGC-803细胞侵袭能力的变化


图5 MGC-803细胞迁移能力的变化
3 讨论
lncRNA仅仅是RNA聚合酶Ⅱ的转录副产物,在生物体内没有任何生物学功能,但最近发现,lncRNA不仅可在多种层面上对基因的表达进行调控,而且与多种疾病的发生密切相关。目前大量相关研究证实,lncRNA的异常表达会导致其调控的下游功能基因异常表达,最终导致机体产生严重的病理变化,更有甚者诱导肿瘤的发生、发展[5]。肺腺癌转移相关转录子-1(metastasis-associated lung adenocar cinoma transcript-1,MALAT-1)是一类存在于细胞核内的长链非编码RNA。研究证实,MALAT-1的异常表达与人类多种肿瘤的发生、发展密切相关,包括宫颈癌、前列腺癌、胰腺癌等,并与一些肿瘤的远处转移、术后复发及药物治疗耐药有关[6]。PRENSNER等[7]研究发现,在前列腺癌中长链非编码RNA PCGEM1过表达,PCGEM1可促进前列腺癌细胞的恶性增殖,并与患者的不良预后相关。lncRNAs可以在人的循环血液中检测到,并在肿瘤中异常表达,说明lncRNAs很可能成为肿瘤的特异性诊断标志物。有研究报道,与健康对照组血清比较,长链非编码RNA XLOC_006844、LOC152578及 XLOC_000303在结肠癌患者血清中表达升高,提示这3种lncRNAs很可能成为结肠癌诊断的新指标[8]。循环血清中的lncRNA HOTAIR可以作为乳腺癌早期诊断的生物学指标[9]。由此可见,lncRNAs在肿瘤的发生、发展中发挥着至关重要的作用。
lncRNAs在胃癌的研究中也有许多研究进展,LIU等[10]研究发现,H19通过RUNX1促进胃癌的增殖和侵袭。PANDAR的高表达预示着胃癌患者预后不良[11]。LI等[12]发现,PRNCR1的基因多态性可能是胃癌的一个致病因素。但PRNCR1在胃癌中的作用并不清楚,因此,研究其在胃癌细胞中的表达及其在胃癌发生、发展中的作用,将有助于更加全面地了解胃癌发生、发展的原因。
2011年,CHUNG 等[13]研究发现,PRNCR1 基因在前列腺癌细胞中表达上调,沉默前列腺癌细胞中PRNCR1的表达,可降低雄激素受体(androgen receptor,AR)的转录活性,表明PRNCR1在前列腺癌中通过调控AR的转录活性促进前列腺癌的发生。有研究显示,PRNCR1的基因多态性与结肠癌及胃癌的易感性相关[14]。此外,PRNCR1表达上调可以促进结肠癌细胞的增殖,沉默其表达可以抑制结肠癌细胞的细胞周期。而PRNCR1在胃癌细胞中的作用尚未有研究报道,其作用的分子机制更有待进一步研究。
本实验合成靶向人PRNCR1基因的双链siRNA序列,直接转染入胃癌细胞中,通过qRT-PCR检测转染后siRNA的沉默效果,通过MTT实验验证胃癌细胞增殖能力的变化,通过细胞侵袭转移实验验证胃癌细胞侵袭和迁移能力的变化,最终证实沉默PRNCR1在胃癌细胞中的表达可以抑制胃癌细胞的增殖、侵袭及转移能力,为以PRNCR1为靶点的胃癌基因治疗奠定理论基础。
[1]WANG S,ZHAO D,TIAN R,et al.FGF19 contributes to tumor progression in gastric cancer by promoting migration and invasion[J].Oncol Res,2016,23(4):197-203.
[2]PARALKAR V R,TABORDA C C,HUANG P,et al.Unlinking an lncRNA from its associated cis element[J].Mol Cell,2016,62(1):104-110.
[3]YANG L,QIU M,XU Y,et al.Upregulation of long non-coding RNA PRNCR1 in colorectal cancer promotes cell proliferation and cell cycle progression[J].Oncol Rep,2016,35(1):318-324.
[4]LI L J,JIA F,BAI P,et al.Association between polymorphisms in long non-coding RNA PRNCR1 in 8q24 and risk of gastric cancer[J].Tumour Biol,2016,37(1):299-303.
[5]FANG Y,FULLWOOD M J.Roles,functions,and mechanisms of long non-coding RNAs in cancer[J].Genomics Proteomics Bioinformatics,2016,14(1):42-54.
[6]ZHANG Y,WANG T,HUANG H Q,et al.Human MALAT-1 long non-coding RNA is overexpressed in cervical cancer metastasis and promotes cell proliferation,invasion and migration[J].J BUON,2015,20(6):1497-1503.
[7]PRENSNER J R,SAHU A,IYER M K,et al.The incRNAs PCGEM1 and PRNCR1 are not implicated in castration resistant prostate cancer[J].Oncotarget,2014,5(6):1434-1438.
[8]WANG F,REN S,CHEN R,et al.Development and prospective multicenter evaluation of the long noncoding RNA MALAT-1 as a diagnostic urinary biomarker for prostate cancer[J].Oncotarget,2014,5(22):11091-11102.
[9]JIAO F,HU H,YUAN C,et al.Elevated expression level of long noncoding RNA MALAT-1 facilitates cell growth,migration and invasion in pancreatic cancer[J].Oncol Rep,2014,32(6):2485-2492.
[10]LIU G,XIANG T,WU Q F,et al.Long noncoding RNA H19-Derived miR-675 enhances proliferation and invasion via RUNX1 in gastric cancer cells[J].Oncol Res,2016,23(3):99-107.
[11]PETROVICS G,ZHANG W,MAKAREM M,et al.Elevated expression ofPCGEM1,a prostate-specific gene with cell growth-promoting function,is associated with high-risk prostate cancer patients[J].Oncogene,2004,23(2):605-611.
[12]LI L,SUN R,LIANG Y,et al.Association between polymorphisms in long non-coding RNA PRNCR1 in 8q24 and risk of colorectal cancer[J].J Exp Clin Cancer Res.2013,32(1):104.
[13]CHUNG S,NAKAGAWA H,UEMURA M,et al.Association of a novel long non-coding RNA in 8q24 with prostate cancer susceptibility[J].Cancer Sci,2011,102(1):245-252.
(童颖丹 编辑)
Long non-coding RNA PRNCR1 promotes proliferation,invasion and migration of gastric cancer cells
Yi Cai
(Department of Emergency,the First Affiliated Hospital,University of South China,Hengyang,Hunan 421001,China)
ObjectiveTo observe the expressions of long non-coding RNA PRNCR1 in gastric cancer MGC-803 cells and normal gastric mucosa epithelial GES-1 cells,and to study the effect of PRNCR1 on the proliferation,migration and invasion of gastric cancer cells by silencing the expression of PRNCR1.MethodssiRNA fragments (PRNCR1-siRNA)and control fragments (PRNCR1-NC)were designed,synthesized and transfected to gastric cancer cells.The expression of PRNCR1 was detected by qRT-PCR.Cell proliferation was detected by MTT.Migration and invasion of gastric cancer cells were detected by Transwell assay.ResultsThe expression of PRNCR1 in the MGC-803 cells was higher than that in the GES-1 cells.The expression of PRNCR1 was reduced by PRNCR1-siRNA in the MGC-803 cells.The proliferation,invasion and migration of the MGC-803 cells decreased after transfection of PRNCR1-siRNA.ConclusionsThe expression of PRNCR1 can be down-regulated by PRNCR1-siRNA,and PRNCR1-siRNA could inhibit the proliferation,invasion and migration of gastric cancer cells,which provides a theoretical foundation for gene therapy of gastric cancer targeting atPRNCR1gene.
long non-coding RNA;PRNCR1;proliferation;invasion;migration;gastric cancer
R735.2
A
10.3969/j.issn.1005-8982.2017.12.007
1005-8982(2017)12-0035-05
2016-04-15
