CRISPR/Cas9基因组编辑技术及其在草类植物中的应用
2017-07-01周美亮李金博孙占敏吴燕民兰州大学草地农业科技学院甘肃兰州7000中国农业科学院生物技术研究所北京100081深圳市铁汉生态环境股份有限公司广东深圳518040
王 丹,周美亮,李金博, 孙占敏,吴燕民(1.兰州大学草地农业科技学院,甘肃 兰州 7000; .中国农业科学院生物技术研究所,北京 100081;.深圳市铁汉生态环境股份有限公司,广东 深圳 518040)
植物生产层
CRISPR/Cas9基因组编辑技术及其在草类植物中的应用
王 丹1,2,周美亮2,李金博3, 孙占敏2,吴燕民1,2
(1.兰州大学草地农业科技学院,甘肃 兰州 730020; 2.中国农业科学院生物技术研究所,北京 100081;3.深圳市铁汉生态环境股份有限公司,广东 深圳 518040)
CRISPR/Cas9系统作为新一代基因组编辑技术,一经问世就受到广泛关注。CRISPR/Cas9系统具有对特定基因位点进行精确修饰(包括敲除、插入、替换等)的强大功能,同时具有操作简单、效率高、适应性广等技术优势,目前已在微生物、植物、动物以及人类基因治疗等整个生命科学领域得到应用。本文简要介绍了CRISPR/Cas9技术的发展历程、作用机理、技术优势、功能及应用领域,总结该技术在植物领域的基因组定向编辑中的研究进展。并提出了这项技术在草类植物基因功能分析、基因代谢调控途径与遗传改良等方面的应用前景,为相关领域的研究工作奠定了基础。
CRISPR/Cas9;基因组编辑;草类植物;定点敲除;基因功能;遗传改良
基因编辑(genome editing)是可以对基因组进行定点修饰的一种技术,是研究特定基因功能的基础。近年来,基因组编辑技术主要有锌指核酸酶(zinc finger nuclease, ZFN)技术和转录激活样效应因子核酸酶(transcription activator-like effector nucleases, TALEN)技术。这两种人工核酸酶的原理都是通过DNA结合蛋白与核酸内切酶FokⅠ切割结构域融合, 在靶位点将DNA切断。而CRISPR/Cas9 (clustered regularly interspaced short palindromic repeats/CRISPR-associated proteins)系统作为新一代基因组编辑技术,一经问世就受到人们的高度关注和极大反响[1]。CRISPR/Cas9系统是细菌和古细菌在长期演化过程中形成的一种抵御病毒侵袭的免疫防御系统,通过将入侵噬菌体和DNA的片段整合到CRISPR中,并利用相应的CRISPR RNAs(crRNAs)来指导同源序列的降解,对抗入侵的病毒及外源DNA[2],科学家们利用sgRNA识别特定序列引导Cas9蛋白切割原理,通过对CRISPR/Cas9系统改造,对特定基因位点进行敲除、插入和替换等精确编辑。因其操作简单、重组效率高、靶位点选择具有广泛性、同时可针对多个基因进行编辑、具有广泛的物种适应性及试验周期短、成本低等优势,受到研究者的青睐[3]。CRISPR/Cas9体系可进行探究基因功能,对目标生物的基因组进行特异性的基因编辑,应用领域十分广泛,几乎涉及到原核生物、动植物基因编辑与人类基因治疗等整个生物界[4-5]。CRISPR/Cas9体系目前已在多种植物中开展了基因组编辑相关研究,但在草业领域的研究应用尚处于起步阶段,利用CRISPR/Cas9系统可有效地开展基因功能研究,发掘优良基因,进行代谢调控研究,探明代谢调控机理,进行有针对性的基因敲除和优良基因的定点整合和多个优良农业性状的聚合,将为草业领域基因遗传改良研究带来突破性进展。
1 CRISPR/Cas9系统概述
1.1 CRISPR/Cas9系统的发展历程
1987年Ishino等[6]在对一种细菌的碱性磷酸酶基因进行研究时,发现一段串联的短回文重复结构。1993年,Mojica等[7]在极端耐盐的古细菌中发现一段具有30个碱基并被36个碱基隔开的重复回文序列,与已知的微生物的重复序列家族都不具有一致性,推测这些存在于古细菌中的重复序列结构可能具有重要功能[8]。截至2000年,先后在20余种不同的细菌和古细菌中,发现了结构组成与CRISPR类似的序列[9], 2002年这种结构正式被命名为成簇规律性间隔短回文重复,即CRISPR,并预测这种特殊的重复序列发挥着至关重要的生物学功能[10]。2005年,科学家发现一部分细菌的CRISPR中的间隔序列与细菌病毒基因组序列信息高度一致,并推测这一序列可能参与了应对噬菌体入侵的免疫防御[11]。2007年,嗜热链球菌CRISPR/Cas9系统直接参与细菌对噬菌体的免疫反应被证明[12]。2008年,验证CRISPR能直接干扰DNA阻止外源质粒在表皮葡萄球菌的转移[13],证明Cas9蛋白是抵抗病毒的基础[14]。2009年,揭示了CRISPR/Cas9系统是通过Cas9蛋白识别外源RNAs,然后进行干扰或切割阻止其表达,阻止病毒和其它原核生物入侵[15]。2011年,CRISPR/Cas9系统作用机制被阐述[16],同时推测Cas9、tracrRNA(trans-activating crRNA)和crRNA共同作用降解与crRNA互补的外源DNA分子[17]。2012年,Charpentier和Doudna两位科学家使用重组Cas9和体外转录的crRNA证实CRISPR/Cas9系统可以对特定的DNA位点进行切割,提出可利用CRISPR/Cas9系统进行基因组编辑[18]。2013年,验证CRISPR/Cas9系统对人和小鼠进行基因组编辑的可行性[19],并首次证实CRISPR/Cas9系统能够用于植物进行基因组定点编辑[20],并发表了CRISPR/Cas9系统能有效的进行基因定点沉默,可同时调控多个基因,该技术适用于基因组编辑、多种生物医学研究、基因组功能分析和微生物代谢工程等[21]。此后人们应用 CRISPR/Cas9技术在生命科学领域开展了大量的研究工作,其强大的功能和应用领域被逐步开拓。
1.2 CRISPR/Cas9的作用机理
CRISPR/Cas9系统是细菌和古细菌在长期演化过程中形成的一种适应性免疫防御系统,广泛存在于细菌和古细菌的基因组中[22]。CRISPR/Cas9系统的核心部分包括基因组(染色体或质粒)中的CRISPR阵列和Cas9蛋白,CRISPR阵列由一个前导序列(leader)和重复序列(repeats)及来源于病毒或质粒等外源DNA的间隔序列(spacer)相间组成,外源DNA的间隔序列前体(protospacer)侧翼有一段保守短序列,即间隔序列前体旁基序PAM(protospacer adjacent motif),是获取DNA片段所需的识别序列。CRISPR/Cas9与靶位点识别的特异性主要依赖于sgRNA与靠近PAM(NGG)处10~12 bp碱基的配对。Cas9蛋白往往与CRISPR位点的重复序列相连,是一类较大的多态性家族,包括一些核酸酶、解旋酶、聚合酶和RNA结合蛋白等,具有核酸相关的功能域。在CRISPR/Cas9系统中当噬菌体病毒或质粒入侵时,系统会将噬菌体的一段DNA序列重组并整合到CRISPR 5′端的两个重复序列之间,当病毒再次入侵时,系统将能够识别入侵者携带的外源核酸中的特殊片段,CRISPR系统转录生成 crRNA与系统编码tracrRNA结合形成向导RNA(single guide RNA, sgRNA),sgRNAs与Cas9蛋白结合形成蛋白复合物,该复合物特异性识别并整合入侵噬菌体的DNA序列后,Cas9蛋白具有核酸内切酶的活性,可将与crRNA互补的双链DNA切割,形成双链断裂,导致入侵噬菌体或质粒DNA降解,抵御入侵的病毒及外源DNA,提供免疫防御[23-25]。
科学家们依据CRISPR/Cas9系统在细菌体内的免疫防御原理,通过人工改造sgRNA 和Cas9蛋白,应用CRISPR/Cas9系统进行基因组编辑研究(图1),设计sgRNA序列依据识别靶基因序列(coding sequence, CDS) 区域达到精确定位功能,将设计好的sgRNA序列与Cas9蛋白构建表达载体,转染靶细胞,先进行基因组靶位点识别,CRISPR/Cas9系统与目标DNA结合后,直接参与目标DNA的解旋和对DNA的切割使双链断裂,然后激发生物体本身的修复机制,在修复过程中会产生碱基的缺失或插入,造成DNA的突变[26],结合人工设计修复模板,完成基因组的定点敲除、插入、替换引入外源基因等操作。随着研究的不断深入开展,该项技术将会更加高效地应用在与基因组编辑相关的研究中。
1.3 CRISPR/Cas9系统的优势
CRISPR/Cas9系统作为新一代的基因组编辑技术,表现出许多优越性:1) 实验操作简便、周期短、成本低,只需设计sgRNA就可以靶向不同的位点,针对基因的编码区构建出功能缺失突变体,开展基因转录和翻译水平的研究[27]。2) 系统靶向精确性更高,更具有可控性。通过改变sgRNA的序列实现对基因的特异性修饰,较短的sgRNA序列使CRISPR/Cas9的切割效率更高。可以通过对Cas9蛋白的修饰,让它不切断DNA双链,而只是切开单链,这样可以大大降低切开双链后带来的染色体变异风险[28-29]。3) 靶位点选择的广泛性,CRISPR/Cas9要求靶位点序列在生物基因组中广泛存在,使得该项技术几乎可以应用到所有物种进行基因组编辑[30]。4) CRISPR/Cas9系统可以同时对靶基因的不同位点进行编辑,也可以同时针对多个基因进行编辑[31],其在基因操作中的优势也比较明显。5) 应用CRISPR/Cas9系统进行基因修饰可遗传,具有直接获得纯合子的可能,可同时对两个位点切割,然后在单链DNA模板的介导下进行同源重组,在两个位点同时编辑[32],缩短试验周期。6) CRISPR/Cas9系统可获得没有筛选基因、报告基因等外源基因的转基因植物,外源插入基因可以在后代的分离中很容易与靶突变位点发生分离,从而获得无筛选基因的遗传改良作物[33],大大提高人们对转基因作物的接受程度。

图1 CRISPR/Cas9系统工作原理示意图Fig. 1 Schematic diagram describing the working principle of the CRISPR/Cas9 system
注:根据文献[4]和[25]整理而成。
Note: Modified by reference [4] and [25].
2 CRISPR/Cas9系统的功能及应用领域
CRISPR/Cas9系统可用于基因位点各种修饰方式,包括基因敲除、基因修复、同源重组、定点整合、大片段删除以及多基因同时敲除蛋白质定位、抑制或激活基因转录及表观基因组编辑等,随着研究的深入,更多的功能将会被开发利用[34]。CRISPR/Cas9作为RNA靶向的基因组定向编辑新技术,因其强大的功能,在生命科学的各个领域均能展开相关的应用研究。
2.1 微生物领域
CRISPR/Cas9系统是从细菌抗噬菌体病毒系统中演变而来,通过对CRISPR/Cas9系统有针对性地编辑微生物基因组靶向杀死野生型细胞,已成功应用于肺炎链球菌(Streptococcuspneumoniae)[35]和大肠杆菌(Escherichiacoli)[36],同时开展了酿酒酵母(Saccharomycescerevisiae)等位基因的置换[37],提高了乳酸杆菌(Lactobacillus)潜在应用价值研究[38],对病毒基因组展开全面系统研究[39],伴随CRISPR/Cas9系统的深入研究,该项技术在微生物领域的应用将全面展开。
2.2 动物领域
利用CRISPR/Cas9技术对基因组进行精确编辑,有效提高了研究效率,而且大大缩短了育种年限。目前CRISPR/Cas9介导的基因靶向修饰成功地应用于多种动物研究应用,在模式动物秀丽线虫(Caenorhabditiselegans)[40]、果蝇(Drosophilamelanogaster)[41]、斑马鱼(Daniorerio)[42]、爪蟾(Xenopuslaevis)[43]、小鼠(Musmusculus)[44]、大鼠(Rattusnorvegicus)[45]中采用Cas9-sgRNA技术实现靶基因的定点敲除和修饰。CRISPR/Cas9技术将构建的Cas9 mRNA/sgRNA注射受精卵,可以高效地获得基因敲除的猪(Susscrofa)[46],成功获得了肌肉生长抑制素MSTN(Myostatin)基因敲除的绵羊(Ovisaries)[47],并建立在活体动物细胞中观察特定基因座的方法[48]。此外在兔(Oryctolaguscuniculus)[49]、猴(Macacamulatta)[50]等动物中也获得了应用CRISPR/Cas9技术成功进行基因敲除的报道。
2.3 医学领域中应用基因治疗
当CRISPR/Cas9基因编辑技术诞生,人们就开始探讨这项技术应用于医学领域的研究,尝试应用同源介导的修复机制,将正确的序列引入细胞中替代突变的序列,进行基因修复,以此达到基因治疗的目的。应用CRISPR系统研究在活细胞中有效激活基因,促进对整个基因组的功能筛选以及特定疾病的基因鉴定[51]。应用CRISPR/Cas9技术构建小鼠癌症模型[52],人干细胞阿尔茨海默病模型[53],加速对病症的研究工作。此外,利用CRISPR/Cas9技术修复肝细胞中Fah突变基因[54],修复囊肿性纤维化跨膜传导受体基因[55]、肿瘤基因敲除[56]、遗传疾病缺陷基因的修复[57],在逆转录病毒和DNA病毒感染引起的疾病[58],应用性抗菌药物[59]等方面都开展了相关研究,并取得一定的进展。
2.4 植物领域
应用CRISPR/Cas9基因编辑技术在基因组的原位实现对基因的定点突变、定点整合、基因置换及基因修复等,是植物遗传改良和分子设计的理想途径。通过基因的定点突变与置换,获得目标性状的遗产改良,可以避免转基因表达的不可控性和食品安全顾虑,是一种比较理想和高效的作物遗传改良方法。通过CRISPR/Cas9能更加快速高效的实现植物性状的改良,对植物基因功能的研究与应用具有重要的意义。目前已经在包括模式植物拟南芥(Arabidopsisthaliana)、烟草(Nicotianatabacum),以及重要农作物水稻(Oryzasativa)、小麦(Triticamaestivum)等在内的多种植物中开展基因组定点编辑研究(表1)。
2.4.1 拟南芥 CRISPR/Cas9技术应用于模式植物拟南芥,开展了对绿色荧光蛋白报告基因GFP(green fluorescent protein)和乙醇脱氢酶基因ADH1(alcohol dehydrogenase 1)位点突变研究,通过对T2、T3代稳定遗传性检测表明:其突变效果稳定且符合孟德尔遗传定律[60-61]。利用CRISPR/Cas9技术对拟南芥赤霉素应答基因GAI(gibberellic acid insensitive),胁迫应答基因JAZ1(jasmonate-zim-domain protein1),油菜素内脂受体基因BRI1(brassinosteroid insensitive 1),镁螯合酶基因CHLI(magnesium chelatase subunit 1),种皮颜色调控基因TT4(transparent testa 4)等不同基因的12个靶位点进行编辑,在T2代中单个突变植物后代发现约22%的单株为纯合体[62],诱导拟南芥ADH1和TT4基因位点,结果均能有效诱导同源修复途径发生的突变[63],对卵细胞特异性启动子EC1.2(egg cell-specific gene)相关基因位点进行编辑研究,实现多基因的突变[64]。
2.4.2 烟草 CRISPR/Cas9系统在烟草中的应用,构建靶向番茄红素脱氢酶PDS(phytoene desaturase)基因位点的Cas9/sgRNA,采用RE-PCR方法分析,结果表明突变率为1.8%~2.4%[65]。通过CRISPR/Cas9系统敲除烟草的两个转录因子基因PDS和PDR6,插入或缺失频率约16.2%~20.3%[66],在靶向报告基因GUS(β-glucuronidase, GUS)、番茄红素脱氢酶NbPDS、戊烯基/二甲基烯丙基二磷酸合酶基因NbIspH等的编辑研究中,也证实了CRISPR/Cas9系统成功应用于烟草[67]。
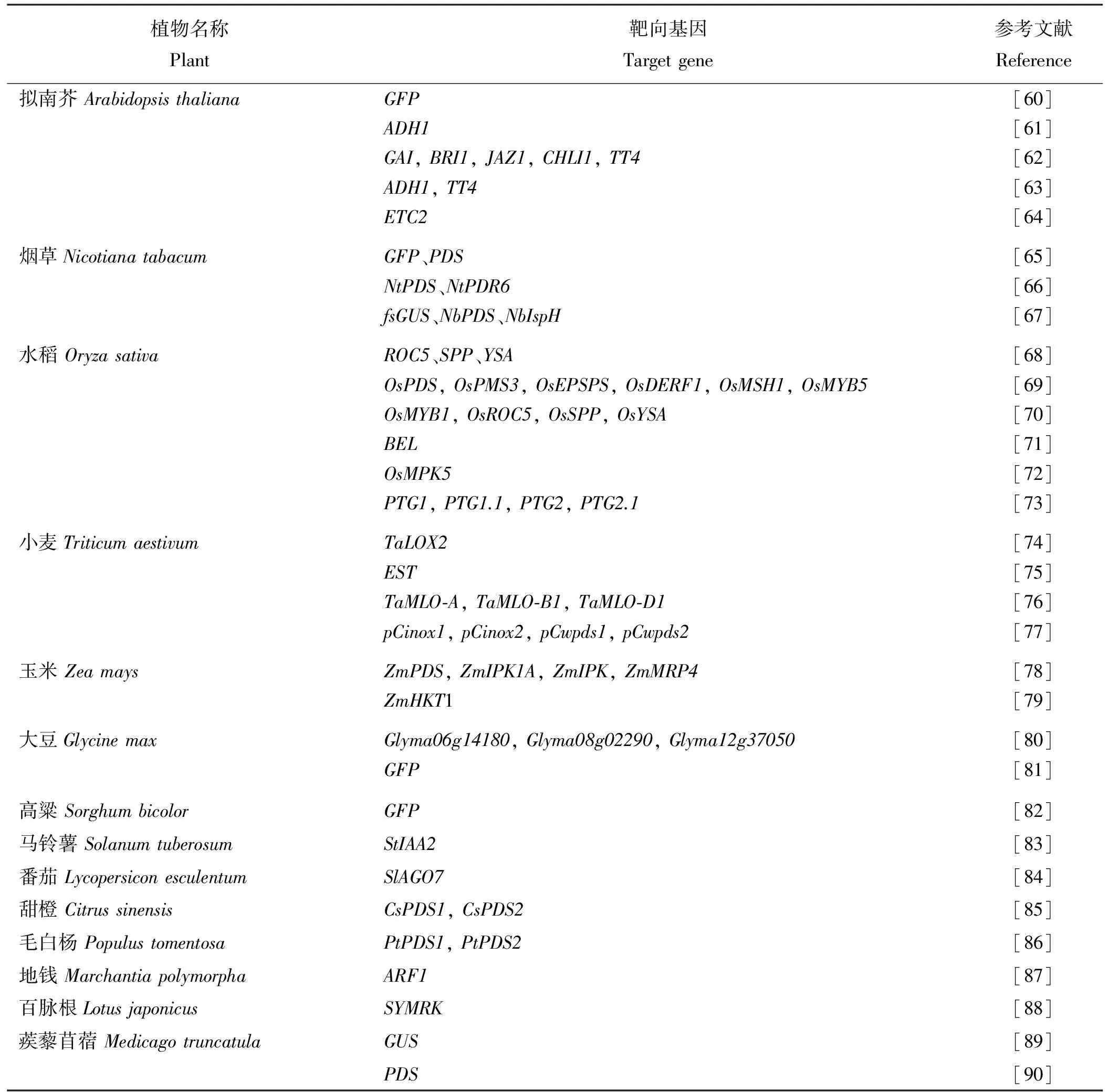
表1 CRISPR/Cas9系统在不同植物种中的应用研究Table 1 Studies on the application of CRISPR/Cas9 system in different plant species
2.4.3 水稻 利用CRISPR/Cas9系统在水稻中实现了抗性相关基因ROC5(rice outermost cell-specific gene 5)、叶绿体加工酶基因SPP(stromal processing peptidase)、叶绿体发育基因YSA(young seedling albino)[68]和周期蛋白依赖性激酶基因CDK(cyclin-dependent kinase)[69]等基因的定点突变。针对两个水稻亚种中的基因敲除标记基因OsPDS(phytoene desaturase gene)、OsPMS3(theOsPMS3 gene codes a noncoding RNA)、OsEPSPS(a key enzyme in the pathway for aromatic amino acid synthesis),转录因子OsDERF1、OsMYB5、OsMYB1和逆境应答相关基因OsMSH1(DNA mismatch repair protein)、OsROC5、OsSPP、OsYSA等10个靶标基因进行定点突变,获得转基因株系中突变率为44.4%,其中7.7%为纯合系[70]。针对水稻除草剂抗性基因BEL(bentazon sensitive lethal gene)设计3个不同gRNAs,结果表明CRISPR/Cas9系统突变效率在2%~16%,表型分析显示转基因植物对除草剂苯达松敏感[71]。通过构建3个与水稻胁迫应答相关基因OsMPK5不同位点配对的Cas9/gRNAs发现,gRNA与靶标DNA的配对位置是决定Cas9/gRNA打靶特异性的关键因素[72]。Xie等[73]构建多个tRNA-gRNA多顺反子PTG(polycistronic tRNA-gRNA gene) 基因,应用CRISPR/Cas9技术,获得了稳定的水稻转基因株系。
2.4.4 小麦 CRISPR/Cas9系统在小麦中的应用,靶向脂氧合酶基因TaLox2(lipoxygenase),获得稳定的基因突变株系[74],靶向EST(expressed sequence tags)基因,结果表明利用CRISPR/Cas9系统可减少小麦植酸含量[75],对MLO(mildew-resistance locus)基因位点进行突变,获得了抗白粉病的特性小麦[76],对报告基因inox(inositol oxygenase)和pds(phytoene desaturase)基因不同位点的研究表明CRISPR/Cas9系统可同时对一个基因不同位点及不同的两个基因进行编辑[77]。
2.4.5 玉米 应用CRISPR/Cas9系统,获得两个靶向植酸合成的关键酶基因ZmIPK(important phytic acid kinese)的玉米,突变率分别为16.4%和19.1%[78]。靶向高亲和性钾转运蛋白基因HKT(high-affinity potassium transporter),在玉米原生质体中瞬时表达和转基因玉米植株中稳定遗传的突变体[79]。
2.4.6 大豆 在应用CRISPR/Cas9系统对大豆进行靶向基因组编辑试验中,分别应用大豆GmU6和拟南芥AtU6启动子突变Glyma06g14180(predicted 11 U6 genes in soybean),Glyma08g02290和Glyma12g37050基因,结果表明,使用大豆GmU6启动子的打靶效率明显高于使用拟南芥AtU6启动子的打靶效率[80]。Jacobs等[81]构建了针对GFP的5′和3′的两个CRISPR/Cas9载体,结果表明,针对5′的效率远高于3′而且突变频率随着培养时间的延长而增加。
2.4.7 高粱 利用CRISPR/Cas9系统,构建应用Y158载体,包含U6启动子、靶向红色荧光蛋白基因DsRED2(red fluorescent protein)的5′端,针对高粱幼胚进行转化,结果获得稳定表达[82]。
2.4.8 马铃薯 通过构建含有U6启动子,sgRNA靶向编码IAA蛋白的StIAA2(encoding an Aux/IAA protein)基因的载体,利用CRISPR/Cas9系统敲除马铃薯StIAA2基因,获得突变体,在T1中发现纯合子,表明应用该系统可进行马铃薯基因编辑相关研究[83]。
2.4.9 番茄 Brooks等[84]建构了两个sgRNAs敲除番茄花器官发育相关基因SlAGO7,获得转基因突变植株,对转基因后代分析显示CRISPR/Cas9在番茄中诱导的突变能够稳定遗传给后代。
2.4.10 甜橙 CRISPR/Cas9在甜橙中应用突变PDS基因,检测突变频率在3.2%~3.9%,为今后利用该技术进行柑橘的品种改良奠定了基础[85]。
2.4.11 杨树 通过分析不同sgRNA结合毛白杨pds靶基因DNA序列后对突变效率的影响,其中3′端的碱基配对更为重要。利用该技术,获得多个杨树转录因子及结构基因的敲除突变体株系,为将来开展基因功能研究和杨树遗传改良奠定了基础[86]。
2.4.12 地钱 Sugano等[87]构建针对生长素正调控因子ARF1(auxin response factor)基因的sgRNA,转化地钱壳孢子,在T1代中检测突变率为11.1%,这些突变能够以胞芽无性生殖的方式稳定遗传给后代。
在开展基础研究的同时,也开展了针对CRISPR/Cas9系统的优化和提高效率等研究,Johnson等[91]通过设计12个不同的sgRNA,评估CRISPR/Cas9系统效率,探索优化设计方案,Lei等[92]提供了一个26种植物设计sgRNA和检测脱靶位点的开放的网络平台,这些都促进了该项技术在植物中的应用效率。
3 CRISPR/Cas9系统在草类植物中的应用前景
CRISPR/Cas9系统在草类植物基因功能、基因代谢及表达调控、遗传改良等研究中具有广阔的应用前景。
3.1 基因功能研究
应用CRISPR/Cas9系统进行基因组编辑研究,针对丰富的草类植物种质资源开展基因组学研究,可全面深入地挖掘基因功能,获得重要的遗传信息。Wang等[88]应用CRISPR/Cas9系统对豆科模式植物百脉根(Lotusjaponicas)共生固氮SNF(symbiotic nitrogen fixation)相关基因进行编辑研究,建构以SYMRK(symbiosis receptor-likekinase)基因为靶向的SYMRK-sgRNA,获得突变植株,加快了SNF相关基因功能的研究。草类植物资源丰富,生态适应性广泛,在各种极端生境中都有生长,应用CRISPR/Cas9技术将促进发掘一些在极端生境中生长的乡土草品种的特殊性状基因,如在沙漠中生长的草类的耐旱基因,在盐碱地生长的草类植物的耐盐碱基因,在高海拔环境生长的草类植物的耐寒、耐贫瘠基因等等。开展草类植物优良基因质源的收集,实现优良基因的高效利用。
3.2 基因代谢及表达调控研究
应用CRISPR/Cas9系统加速了科学家们探明基因代谢途径和转录调控机制的脚步,可获得多个基因定点敲除、插入等突变,实现基因组水平上的基因改造、转录调控与表观遗传调控[93]。Meng等[90]利用CRISPR/Cas9系统成功对豆科模式牧草蒺藜苜蓿(Medicagotruncatula)的PDS基因进行定点敲除,并在T0代得到10.35%的纯合敲除突变体,为苜蓿等豆科牧草的功能基因组学研究提供了新的研究工具。据此,可通过设计突变同一个代谢通路上的靶基因,通过进行不同基因功能缺失突变,建立功能基因的突变体库,促进草类植物代谢和基因调控机理研究。探明草类植物的营养代谢、激素调控、水分运输等与生长发育相关的代谢调控途径及生物和非生物逆境应答途径,对于促进草类植物的资源开发、栽培利用等都具有重要的意义。
3.3 遗传改良
目前,在草类植物遗传改良的研究中,已开展应用CRISPR/Cas9系统对百脉根共生固氮SNF相关基因进行编辑研究[88],对蒺藜苜蓿基因GUS组定向编辑研究[89],对PDS基因进行定点敲除研究[90]。CRISPR/Cas9系统应用在牧草和草坪草的遗传改良,能有针对性的在基因组水平上改造牧草和草坪草的遗传性状,提高育种的目的性和可操作性,用过设计目标结构产生可遗传变异,更加快速、高效的获得基因组靶位点的突变、特异性变异和定向基因修饰等,加速草业领域培育优良品种进程。根据草类植物的培育需求,应用CRISPR/Cas9系统使优良基因定点整合,针对草坪草改良抗旱、耐病、耐践踏、再生性强等性状的基因,针对牧草定向改良抗逆性强、产量高、营养丰富等基因性状,针对生态修复用途的草类植物可改良耐贫瘠、抗逆性强、耐盐碱、抗风、抗倒伏等一些特定需求的基因性状,同时探讨多个优良农业性状的聚合。使得遗传改良效率更高,使牧草和草坪草育种更具科学性和精确性。
应用CRISPR/Cas9系统必将加快现代牧草和草坪草遗传改良育种进程,成为提高产量、抗性和品质的一个重要契机,基因组编辑的研究快速发展,推动基因的功能研究和物种的改良,必将为草业领域基因功能基础理论研究和遗传改良应用带来广阔的应用前景,为草业的发展带来新的契机。
References:
[1] Lander E S.The heroes of CRISPR.Cell,2016,164(1-2):18-28.
[2] Makarova K S,Haft D H,Barrangou R,Brouns S J J,Charpentier E,Horvath P,Moineau S,Mojica F J M,Wolf Y I,Yakunin A F,Oost J V D,Koonin E V.Evolution and classification of the CRISPR-Cas systems.Nature Reviews Microbiology,2011,9(6):467-477.
[3] Pennisi E.The CRISPR craze.Science,2013,341:833-836.
[4] Sander J D,Joung J K.CRISPR-Cas systems for editing,regulating and targeting genomes.Nature Biotechnology,2014,32(4):347-355.
[5] Listed N.2013 runners-up genetic microsurgery for the masses.Science,2013,342:1434-1435.
[6] Ishino Y,Shinagawa H,Makino K,Amemura M,Nakata A.Nucleotide sequence of theiapgene,responsible for alkaline phosphatase isozyme conversion inEscherichiacoli,and identification of the gene product.Journal of Bacteriology,1987,169(12):5429-5433.
[7] Mojica F J M,Juez G,Rodriguez-Valera F.Transcription at different salinities of haloferax mediterranei seguences adjacent to partially modified pstl sites.Molecular Microbiology,1993,9:613-621.
[8] Mojica F J M,Ferrer C,Juez G,Rodriguez-Valera F.Long stretches of short tandem repeats are present in the largest replicons of the archaea haloferax mediterranei and haloferax volcanii and could be involved in replicon partitioning.Molecular Microbiology.1995,17:85-93.
[9] Mojica F J,Diez-Villase C,Soria E,Juez G.Biological significance of a family of regularly spaced repeats in the genomes of archaea,bacteria and mitochondria.Molecular Microbiology,2000,36(1):244-246.
[10] Jansen R,Embden J D,Gaastra W,Schouls L M.Identification of genes that are associated with DNA repeats in prokaryotes.Molecular Microbiology,2002,43(6):565-1575.
[11] Mojica F J,Diez-Villasenor C,Garcia-Martinez J,Soria E.Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic element.Journal of Molecular Evolution,2005,60(2):174-182.
[12] Barrangou R,Fremaux C,Deveau H,Richards M,Boyaval P,Moineau S,Romero D A,Horvath P.Crispr provides acquired resistance against viruses in prokaryotes.Science,2007,315:1709-1712.
[13] Marraffini L A,Sontheimer E J.CRISPR interference limits horizontal gene transfer in staphylococci by targeting DNA.Science,2008,322:1843-1845.
[14] Brouns S J,Jore M M,Lundgren M,Westra E R,Slijkhuis R J H,Snijdeers A L,Dichman M J,Makarova K S,Koonin E V,Oost J.Small CRISPR RNAs guide antiviral defense in prokaryotes.Science,2008,321:960-964.
[15] Hale C R,Zhao P,Olson S,Duff M O,Graveley B R,Wells L,Terms R M,Terms M P.RNA-guided RNA cleavage by a CRISPR RNA-Cas protein complex.Cell,2009,139(5):945-956.
[16] Makarova K S,Aravind L,Wolf Y I,Koonin E V.Unification of Cas protein families and a simple scenario for the origin and evolution of CRISPR-Cas systems.Biology Direct,2011,6:38.
[17] Deltcheva E,Chylinski K,Sharma C M,Gonzales K,Chao Y,Pirzada Z A,Eckert M R,Charpentier E.CRISPR RNA maturation bytrans-encoded small RNA and host factor RNase Ⅲ.Nature,2011,471:602-607.
[18] Jinek M,Chylinski K,Fonfara I,Hauer M,Doudna J A,Charpentier E.A programmable Dual-RNA guided DNA endonuclease in adaptive bacterial immunity.Science,2012,337:816-821.
[19] Cong L,Ran F A,Cox D,Lin S,Barretto R,Habib N,Hsu P D,Wu X B,Jiang W Y,Marraffini L A,Zhang F.Multiplex genome engineering using CRISPR/Cas systems.Science,2013,339:819-823.
[20] Shan Q W,Wang Y P,Li J,Gao C X.Targeted genome modification of crop plants using a CRISPR-Cas system.Nature Biotechnology,2013,31(8):686-688.
[21] Qi L S,Larson M H,Gilbert L A,Doudna J A,Weissman J S,Arkin A,Lim W A.Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression.Cell,2013,152:1173-1183.
[22] Terns M P,Terns R M.CRISPR-based adaptive immune systems.Current Opinion in Microbiology,2011,14(3):321-327.
[23] Doudna J A,Charpentier E.Genome editing:The new frontier of genome engineering with CRISPR-Cas9.Science,2014,346:1258096.
[24] Wiedenheft B,Sternberg S H,Doudna J A.RNA-guided genetic silencing systems in bacteria and archaea.Nature,2012,482:331-338.
[25] Samanta M K,Dey A,Gayen S.CRISPR/Cas9:An advanced tool for editing plant genomes.Transgenic Research,2016,25(5):561-573.
[26] Horvath P,Barrangou R.CRISPR/Cas,the immune system of bacteria and archaea.Science,2010,327:167-170.
[27] Hsu P D,Lander E S,Zhang F.Development and applications of CRISPR- Cas9 for genome engineering.Cell,2014,157(6):1262-1278.
[28] Li W,Teng F,Li T,Zhou Q.Simultaneous generation and germline transmission of multiple gene mutations in rat using CRISPR-Cas systems.Nature Biotechnology,2013,31(8):684-686.
[29] Wang H,Yang H,Shivalila C S,Dawlaty M M,Cheng A W,Zhang F,Jaenisch R.One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-Mediated genome engineering.Cell,2013,153(4):910-918.
[30] Jiang W Y,Bikard D,Cox D,Zhang F,Marraffini L A.RNA-guided editing of bacterial genomes using CRISPR-Cas systems.Nature Biotechnology,2013,31(3):233-239.
[31] Li J F,Norville J E,Aach J,McCormack M,Zhang D D,Bush J,Church G M,Sheen J.Multiplex and homologous recombination-mediated genome editing inArabidopsisandNicotianabenthamianausing guide RNA and Cas9.Nature Biotechnology,2013,3:688-691.
[32] Yang H,Wang H,Shivalila C S,Cheng A W,Shi L,Jaenisch R.One-step generation of mice carrying reporter and conditional alleles by CRISPR/Cas-mediated genome engineering.Cell,2013,154(6):1370-1379.
[33] Zhou H B,Liu B,Weeks D P,Spalding M H,Yang B.Large chromosomal deletions and heritable small genetic changes induced by CRISPR/Cas9 in rice.Nucleic Acids Research,2014,42(17):10903-10914.
[34] Shalem O,Sanjana N E,Hartenian E,Shi X,Scott D A,Mikkelsen T S,Heckl D,Ebert B L,Root D E,Doench J G,Zhang F.Genome-scale CRISPR-Cas9 knockout screening in human cells.Science,2014,343:84-87.
[35] Jiang W,Bikard D,Cox D,Zhang F,Marraffini L A.RNA-guided editing of bacterial genomes using CRISPR -Cas systems.Nature Biotechnology,2013,31(3):233-239.
[36] Jiang Y,Chen B,Duan C,Sun B B,Yang J J,Yang S.Multigene editing in theEscherichiacoligenome using the CRISPR -Cas9 system.Applied and Environmental Microbiology,2015,81(7):2506-2514.
[37] Dicarlo J E,Norville J E,Mali P,Rios X,Aach J,Church G M.Genome engineering inSaccharomycescerevisiaeusing CRISPR-Cas systems.Nucleic Acids Research,2013,41(7):4336-4343.
[38] Pijkeren J P,Britton R A.Precision genome engineering in lactic acid bacteria.Microbial Cell Factories,2014,13(1):1-10.
[39] Yuen K S,Chan C P,Wong N H,Ho C H,Ho T H,Lei T,Deng W,Tsao S W,Chen H L,Kok K H,Jin D Y.CRISPR/Cas9 -mediated genome editing of Epstein-Barr virus in human cells.Journal of General Virology,2015,96:626-636.
[40] Friedland A E,Tzur Y B,Esvelt K M,Colaiacovo M P,Church G M,Calarco J A.Heritable genome editing inC.elegansvia a CRISPR-Cas9 system.Nature Methods,2013,10(8):741-743.
[41] Bassett A R,Tibbit C,Ponting C P,Liu J L.Highly efficient targeted mutagenesis ofDrosophilawith the CRISPR/Cas9 system.Cell Reports,2013,4(1):220-228.
[42] Hwang W Y,Fu Y F,Reyon D,Maeder M L,Tsai S Q,Sander J D,Peterson R T,Yeh J R J,Joung J K.Efficient genome editing in zebrafish using a CRISPR-Cas system.Nature Biotechnology,2013,31(3):227-229.
[43] Nakayama T,Fish M B,Fisher M,Oomen-Hajagos J,Thomsen G H,Grainger R M.Simple and efficient CRISPR/Cas9-mediated targeted mutagenesis inXenopustropicalis.Genesis,2013,51:835-843.
[44] Koike-Yusa H,Li Y,Tan E P,Velasco-Herrera M D C,Yusa K.Genome-wide recessive genetic screening in mammalian cells with a lentiviral CRISPR-guide RNA library.Nature Biotechnology,2014,32:267-273.
[45] Shao Y J,Guan Y T,Wang L,Qiu Z G,Liu M Z,Chen Y T,Wu L J,Li Y M,Ma X Y,Liu M Y,Li D L.CRISPR/Cas-mediated genome editing in the rat via direct injection of one-cell embryos.Nature Protocols,2014,9(10):2493-2512.
[46] Hai T,Teng F,Guo R,Li W,Zhou Q.One-step generation of knockout pigs by zygote injection of CRISPR/Cas system.Cell Research,2014,24:372-375.
[47] Han H B,Ma Y H,Wang T,Lian L,Tian X Z,Hu R,Deng S L,Li K P,Wang F,Li N,Liu G S,Zhao Y F,Lian Z G.One-step generation of myostatin gene knockout sheep via the CRISPR/Cas9 system.Frontiers of Agricultural Science and Engineering,2014,1(1):2-5.
[48] Chen B,Gilbert L A,Cimini B A,Schnitzbauer J,Zhang W,Li G W,Park J,Blackbum E H,Weissman J S,Qi L S,Huang B.Dynamic imaging of genomic loci in living human cells by an optimized CRISPR/Cas system.Cell,2013,155:1479-1491.
[49] Yan Q M,Zhang Q J,Yang H Q,Zou Q J,Tang C C,Fan N N,Lai L G.Generation of multi-gene knockout rabbits using the Cas9/gRNA system.Cell Regeneration,2014,3(1):12.
[50] Liu Y Y,Shen B,Cui Y Q,Chen Y C,Wang J Y,Wang L,Kang Y,Zhao X Y,Si W,Li W,Xiang A P,Zhou Z M,Li T Q,Tan T,Pu X Q,Wang F,Ji S H,Zhou Q,Huang X X,Ji W Z,Sha J H.Generation of gene-modified cynomolgus monkey via Cas9/RNA-mediated gene targeting in one-cell embryos.Cell,2014,156:836-843.
[51] Konermann S,Brigham M D,Trevino A E,Joung J,Abudayyeh O O,Barcena C,Hsu P D,Habib N,Gootenberg J S,Nishimasu H,Nureki O,Zhang F.Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex.Nature,2014,517:583-588.
[52] Platt R J,Chen S D,Zhou Y,Yim M J,Swiech L,Kempton H R,Dahlman J E,Parnas O,Eisenhaure T M,Jovanovic M,Graham D B,Jhunjhunwala S,Heidenreich M,Xavier R J,Langer R,Anderson D G,Hacohen N,Regev A,Feng G P,Sharp P A,Zhang F.CRISPR-Cas9 knockin mice for genome editing and cancer modeling.Cell,2014,159(2):440-455.
[53] Paquet D,Kwart D,Chen A,Sproul A,Jacob S,Teo S,Olsen K M,Gregg A,Noggle S,Tessier-Lavigne Marc.Efficient introduction of specific homozygous and heterozygous mutations using CRISPR/Cas9.Nature,2016,533:125.
[54] Yin H,Xue W,Chen S D,Bogorad R L,Benedetti E,Grompe M,Koteliansky V,Sharp P A,Jacks T,Anderson D G.Genome editing with Cas9 in adult mice corrects a disease mutation and phenotype.Nature Biotechnology,2014,32(6):551-553.
[55] Schwank G,Koo B K,Sasselli V,Dekkers J F,Heo I,Demircan T,Sasaki N,Boymans S,Cuppen E,Ent C K,Nieuwenhuis E E S,Beekman J M,Clevers H.Functional repair of CFTR by CRISPR/Cas9 in intestinal stem cell organoids of cystic fibrosis patients.Cell Stem Cell,2013,13(6):653-658.
[56] Xue W,Chen S D,Yin H,Tammela T,Papagiannakopoulos T,Joshi N S,Cai W X,Yang G,Bronson R,Crowley D G,Zhang F,Anderson D G,Sharp P A,Jacks T.CRISPR-mediated direct mutation of cancer genes in the mouse liver.Nature,2014,514:380-384.
[57] Ousterout D G Kabadi A M,Thakore P I,Majoros W H,Reddy T E,Gersbach C A.Multiplex CRISPR /Cas9-based genome editing for correction of dystrophin mutations that cause duchenne muscular dystrophy.Nature Communications,2015,6:6244.
[58] Liao H K,Gu Y,Diaz A,Marlett J,Takahashi Y,Li M,Suzuki K,Xu R,Hishida T,Chang C J,Esteban C R,Young J,Belmonte J C L.Use of the CRISPR/Cas9 system as an intracellular defense against HIV-1 infection in human cells.Nature Communications,2015,6:6413.
[59] Bikard D,Euler C W,Jiang W Y,Nussenzweig P M,Goldberg G W,Duportet X,Fischetti V A,Marraffini L A.Exploiting CRISPR-Cas nucleases to produce sequence-specific antimicrobials.Nature Biotechnology,2014,32(11):1146-1150.
[60] Jiang W,Yang B,Weeks D P.Efficient CRISPR/Cas9-mediated gene editing inArabidopsisthalianaand inheritance of modified genes in the T2and T3generations.PloS One,2014,9(6):e99225.
[61] Schiml S,Fauser F,Puchta H.The CRISPR/Cas system can be used as nuclease for in planta gene targeting and as paired nickases for directed mutagenesis inArabidopsisresultingin heritable progeny.The Plant Journal,2014,80(6):1139-1150.
[62] Feng Z Y,Mao Y F,Xu N F,Zhang B T,Wei P L,Yang D L,Wang Z,Zhang Z J,Zheng R,Yang L,Zeng L,Liu X D,Zhu J K.Multigeneration analysis reveals the inheritance,specificity,and patterns of CRISPR/Cas-induced gene modifications in Arabidopsis.Proceedings of the National Academy of Sciences,2014,111(12):4632-4637.
[63] Fauser F,Schiml S,Puchta H.Both CRISPR/Cas-based nucleases and nickases can be used efficiently for genome engineering inArabidopsisthaliana.The Plant Journal,2014,79(2):348-359.
[64] Wang Z P,Xing H L,Dong L,Zhang H Y,Han C Y,Wang X C,Chen Q J.Egg cell-specific promoter-controlled CRISPR/Cas9 efficiently generates homozygous mutants for multiple target gene inArabidopsisin a single generation.Genome Biology,2015,16:144.
[65] Nekrasov V,Staskawicz B,Weigel D,Jones J D G,Kamoun S.Targeted mutagenesis in the model plant nicotiana benthamiana using Cas9 RNA-guided endonuclease.Nature Biotechnology,2013,31:691-693.
[66] Gao J P,Wang G H,Ma S Y,Xie X D,Wu X W,Zhang X T,Wu Y Q,Zhao P,Xia Q Y.CRISPR/Cas9-mediated targeted mutagenesis inNicotianatabacum.Plant Molecular Biology,2015,87(1):99-110.
[67] Yin K Q,Han T,Liu G,Chen T Y,Wang Y,Yu A Y,Liu Y.A geminivirus-based guide RNA delivery system for CRISPR/Cas9 mediated plant genome editing.Science Reports,2015,5:14926.
[68] Feng Z Y,Zhang B T,Ding W N,Liu X D,Yang D L,Wei P L,Cao F Q,Zhu S H,Zhang F,Mao Y F,Zhu J,K.Efficient genome editing in plants using a CRISPR/Cas system.Cell Research,2013,23(10):1229-1232.
[69] Endo M,Mikami M,Toki S.Multigene knockout utilizing offtarget mutations of the CRISPR /Cas9 system in rice.Plant Cell Physioloy,2015,56(1):41-47.
[70] Zhang H,Zhang J S,Wei P L,Zhang B T,Gou F,Feng Z Y,Mao Y F,Yang L,Zhang H,Xu N F,Zhu J K.The CRISPR/Cas9 system produces specific and homozygous targeted gene editing in rice in one generation.Plant Biotechnology Journal,2014,12(6):797-807.
[71] Xu R F,Li H,Qin R Y,Wang L,Li L,Wei P C,Yang J B.Gene targeting using the agrobacterium tumefaciens-mediated CRISPR-Cas system in rice.Rice,2014,7(1):5-8.
[72] Xie K,Yang Y.RNA-guided genome editing in plants using a CRISPR-Cas system.Molecular Plant,2013,6(6):1975-1983.
[73] Xie K,Minkenberg B,Yang Y N.Boosting CRISPR/Cas9 multiplex editing capability with the endogenous tRNA-processing system.Proceeding of the National Academy Sciences USA,2015,112(11):3570-3575.
[74] Shan Q W,Wang Y P,Li J,Zhang Y,Chen K L,Liang Z,Zhang K,Liu J X,Jeffxi J Z,Qiu J L,Gao C X.Genome editing in rice and wheat using the CRISPR/Cas system.Nature Protocols,2014,9(10):2395-2410.
[75] Sharma S,Upadhyay S K.Functional characterization of expressed sequence tags of bread wheat (Triticumaestivum) and analysis of CRISPR binding sites for targeted genome editing.American Journal of Bioinformatics Research,2014,4(1):11-22.
[76] Wang Y P,Cheng X,Shan Q W,Zhang Y,Liu J X,Gao C X,Qiu J L.Simultaneous editing of three homoeoalleles in hexaiploid bread wheat confers heritable resistance to powdery mildew.Nature Biotechnology,2014,32(9):947-951.
[77] Upadhyay S K,Kumar J,Alok A,Tuli R.RNA-Guided genome editing for target gene mutations in wheat.G3:Genes/Genomes/Genetics,2013,3:2233-2238.
[78] Liang Z,Zhang K,Chen K L,Gao C X.Targeted mutagenesis inZeamaysusing TALENs and the CRISPR/Cas system.Journal of Genetics and Genomics,2014,41(2):63-68.
[79] Xing H L,Dong L,Wang Z P,Zhang H Y,Han C Y,Liu B,Wang X C,Chen Q J.CRISPR/Cas9 toolkit for multiplex genome editing in plants.BMC Plant Biology,2014,14:327.
[80] Sun X J,Hu Z,Chen R,Jiang Q Y,Song G H,Zhang H,Xi Y J.Targeted mutagenesis in soybean using the CRISPR-Cas9 system.Scientific Reports,2015,5:10342.
[81] Jacobs T B,Lafyeete P R,Schmitz R J,Parrott W A.Targeted genome modifications in soybean with CRISPR/Cas9.BMC Biotechnology,2015,15:16-25.
[82] Jiang W,Zhou H,Bi H,Fromm M,Yang B,Weeks D P.Demonstration of CRISPR/Cas9/sgRNA-mediated targeted gene modification in Arabidopsis,tobacco,sorghum and rice.Nucleic Acids Research,2013,41(20):e188.
[83] Wang S H,Zhang S B,Wang W X,Xiong X Y,Meng F R,Cui X.Efficient targeted mutagenesis in potato by the CRISPR/Cas9 system.Plant Cell Report,2015,34(9):1473-1476.
[84] Brooks C,Nekrasov V,Lippman Z B,Eck J V.Efficient gene editing in tomato in the first generation using the clustered regularly interspaced short palindromic repeats/CRISPR-associated 9 system.Plant physiology,2014,166(3):1292-1297.
[85] Jia H,Wang N.Targeted genome editing of sweet orange using Cas9/sgRNA.PLoS One,2014,9(4):e93806.
[86] 刘婷婷,范迪,冉玲玉,姜渊忠,刘瑞,罗克明.应用 CRISPR/Cas9 技术在杨树中高效敲除多个靶基因.遗传.2015,3(10):1044-1052. Liu T T,Fan D,Ran LY,Jiang Y Z,Liu R,Luo K M.Highly efficient CRISPR/Cas9-mediated targeted mutagenesis of multiple genes inPopulus.Hereditas,2015,37(10):1044-1052.(in Chinese)
[87] Sugano S S,Shirakawa M,Takagi J,Matsuda Y,Shimada T,Hara-Nishimura I,Kohchi T.CRISPR/Cas9-mediated targeted mutagenesis in the liverwortMarchantiapolymorphaL.Plant and Cell Physiology,2014,55(3):457-481.
[88] Wang L X,Wang L L,Tan Q,Fan Q L,Zhu H,Hong Z L,Zhang Z M,Duanmu D Q.Efficient inactivation of symbiotic nitrogen fixation related genes inLotusjaponicasusing CRISPR-Cas9.Frontiers in Plant Science,2016,DOI:10.3389/fpls.2016.01333.
[89] Michno J M,Wang X B,Liu J Q,Curtin S J,Kono T J Y,Stupar R M.CRISPR/Cas mutagenesis of soybean andMedicagotruncatulausing a new web-tool and a modified Cas9 enzyme.GM Crops & Food Biotechnology in Agriculture and the Food Chain,2015,6(4):243-252.
[90] Meng Y Y,Hou Y L,Wang H,Ji R H,Liu B,Wen J Q,Niu L F,Lin H.Targeted mutagenesis by CRISPR/Cas9 system in the model legumeMedicagotruncatula.Plant Cell Reports,2017,36:371-374.
[91] Johnson R A,Gurevich V,Filler S,Samach A,Levy A A.Comparative assessments of CRISPR-Cas nucleases cleavage efficiency in planta.Plant Molecular Biology,2015,87:143-156.
[92] Lei Y,Lu L,Liu H Y,Li S,Xing F,Chen L L.CRISPR-P:A web tool for synthetic single-guide RNA design of CRISPR-system in plants.Molecular Plant,2014,7(3):1494-1496.
[93] Zhang D D,Li Z X,Li J F.Targeted gene manipulation in plants using the CRISPR/Cas technology.Journal of Genetics and Genomics,2016,43(5):251-262.
(责任编辑 王芳)
CRISPR/Cas9 system and its prospective application in grass
Wang Dan1,2, Zhou Mei-liang2, Li Jin-bo3, Sun Zhan-min2, Wu Yan-min1, 2
(1.College of Pastoral Agriculture Science and Technology, Lanzhou University, Lanzhou 730020, China;2.Biotechnology Research Institute, Chinese Academy of Agricultural Sciences (CAAS), Beijing 100081, China;3.Shenzhen Techand Landscape Co., Ltd. Shenzhen 518040, China)
The CRISPR/Cas9 system is a new, precise genome editing technology, which has
wide attention from the scientific community globally. CRISPR/Cas9 is widely utilised for studying gene functions, because it enables precise modification of specific gene sites, including knockout, insertion, and substitution. CRISPR/Cas9 has many advantages, including convenience, precision, efficiency, and a wide selection of target sites. Presently, this technology is applied in all the field of life sciences, including microorganisms, plants, animals, and human gene therapy. The present article briefly introduces the course of development of CRISPR/Cas9 technology, mechanism of action, technical advantage, functions, and its applications. It summarizes the progresses in targetted plant genome editing, and discusses the potential applications of this process in research on grass species, including studies on functional genomics, gene metabolism and regulation, and genetic improvement, which provides a useful reference for the later researches and application of this field.
CRISPR/Cas9 system; genome editing; grass; targeted mutagenesis; gene function research; genetic improvement
Wu Yan-min E-mail:wuyanmin@caas.cn
2016-07-20 接受日期:2016-11-01
国家自然科学基金项目(31372361);国家重点基础研究发展“973”项目(2014CB138701);广东省省级科技计划项目(2015B090904008)
王丹(1982-),女,辽宁本溪人,在读博士生,研究方向为植物逆境生理与分子生物学。 E-mail:wangdd0310@163.com
吴燕民(1955-),男,陕西子长人,研究员,博士,研究方向为植物分子生物学与基因工程。E-mail:wuyanmin@caas.cn
10.11829/j.issn.1001-0629.2016-0396
S540.32;Q943.2
A
1001-0629(2017)06-1204-11
王丹,周美亮,李金博,孙占敏,吴燕民.CRISPR/Cas9基因组编辑技术及其在草类植物中的应用.草业科学,2017,34(6):1204-1214.
Wang D,Zhou M L,Li J B,Sun Z M,Wu Y M.CRISPR/Cas9 system and its prospective application in grass.Pratacultural Science,2017,34(6):1204-1214.
