玉米大斑病菌StSNF1的基因组定位、蛋白质结构预测及表达分析
2017-07-01何世道杨瑞秀苑德鹏孙艳秋高增贵
何世道,杨瑞秀,刘 博,苑德鹏,姚 远,孙艳秋,高增贵
(沈阳农业大学 植物免疫所,辽宁 沈阳 110866)
玉米大斑病菌StSNF1的基因组定位、蛋白质结构预测及表达分析
何世道,杨瑞秀,刘 博,苑德鹏,姚 远,孙艳秋,高增贵
(沈阳农业大学 植物免疫所,辽宁 沈阳 110866)
为了明确StSNF1在玉米大斑病菌基因组中的位置,解析该基因编码蛋白的结构特征,探究该基因在侵染寄主的不同时期、分生孢子萌发侵染过程中以及不同碳源培养下的表达情况。结果表明,该基因ID号为008026214,全长3 046 bp,位于scaffold_17负链的97 793-100 838位置。StSNF1与玉米圆斑病菌SNF1同源关系较近,由877个氨基酸残基编码而成。StSNF1蛋白具有氮端的蛋白激酶结构域、氮端的碳代谢产物去阻遏蛋白激酶结构域和碳端的激酶相关结构域。在蛋白激酶结构域内,具有ATP结合位点和丝氨酸/苏氨酸活性位点。Real-time PCR结果表明,StSNF1在侵染后期高表达,72 h表达量最高;StSNF1在分生孢子萌发24 h时(即侵入丝形成时期)表达量最高;StSNF1在蔗糖为单一碳源的培养基中表达量最高,果胶培养基次之。综上所述,StSNF1与侵染寄主和碳源利用密切相关,在侵染后期发挥作用,利用寄主细胞内的非发酵型碳源进行次级侵染。
玉米大斑病菌;SNF1;蛋白激酶;表达分析
玉米大斑病是全世界玉米种植区的常见病害,它是由凸脐蠕孢菌(Exserohilumturcicum(Pass.) Leonard & Suggs)引起的一种玉米叶部病害[1-2]。在我国东北、华北北部、西北和南方山区冷凉玉米产区发病比较严重[3]。玉米大斑病流行可使玉米减产30%,而种植感病品种产量损失可达50%之多[4],给玉米生产带来严重威胁。
目前,关于玉米大斑病菌致病因子研究主要集中在细胞信号传导途径类、细胞壁降解酶类、氧化还原酶类、黑色素类以及毒素等方面,其中细胞信号传导途径包括cAMP-PKA系统和MAPK通路等。cAMP-PKA系统中5′-AMP的浓度直接调控AMPK(AMP-activated protein kinase)的活性[5]。SNF1p(蔗糖非发酵蛋白激酶,Sucrose nonfermenting 1 protein kinase),属于AMPK家族,它是碳源代谢去阻遏过程中的关键酶,首次是在酿酒酵母中被发现,在真核生物中普遍存在且高度保守[6-7]。
碳分解代谢物阻遏(Carbon catabolite repression,CCR)是微生物中普遍存在的一种机制,微生物通常优先利用简单的发酵碳源(如葡萄糖),该碳源的代谢产物会对其他非发酵碳源相关基因的表达或相关蛋白的活性产生抑制作用[8-9]。在酵母中,酵母菌优先利用葡萄糖,当葡萄糖含量充足时,SNF1p活性将会受到抑制;当葡萄糖匮乏时,酵母则会利用非发酵碳源维持自身新陈代谢,在此过程中SNF1p起到了关键作用。此外,酵母ΔSNF1无法利用蔗糖,在乙醇或甘油为单一碳源的培养基上生长时均受到抑制[10]。
SNF1 在植物病原真菌中的功能已有报道,主要集中在碳源利用、致病性和产孢等方面。植物病原菌中,玉米圆斑病菌(Cochlioboluscarbonum)、尖孢镰刀菌(Fusariumoxysporum)、禾谷镰刀菌(Fusariumgraminearum)、稻瘟病菌(Magnaportheoryzae)、大丽轮枝菌(Verticilliumdahliae)、指状青霉菌(Penicilliumdigitatum)等SNF1突变株在利用单一碳源方面存在严重缺陷,但不同真菌的SNF1突变株在利用碳源方面所产生的缺陷存在差异[11]。如ΔFoSNF1在利用果胶方面与野生型无明显差异,但ΔPdSNF1在只含有果胶的培养基上生长却严重受阻。玉米圆斑病菌、尖孢镰刀菌、禾谷镰刀菌、大丽轮枝菌、指状青霉菌中,SNF1通过调控细胞壁降解酶相关基因来影响致病性,以上几种SNF1突变株致病性均下降。此外ΔMoSNF1分生孢子的萌发和附着孢的形成受阻,ΔPdSNF1在培养和侵染过程中产孢量下降[12-17]。
本研究利用生物信息学技术,分析StSNF1在基因组中的位置、该基因编码蛋白的理化性质以及结构特征,并利用Real-time PCR技术对玉米大斑病菌侵染寄主过程中、分生孢子萌发侵染过程中以及不同碳源培养下StSNF1的表达模式进行初步探究,为进一步明确StSNF1的生物学功能奠定基础。
1 材料和方法
1.1 试验材料
1.1.1 供试菌株 玉米大斑病菌(Setosphaeriaturcica)野生型菌株SYY1303由沈阳农业大学植物保护学院植物免疫所玉米病害实验室保存。
1.1.2 主要试剂 RNA提取试剂盒(MiniBEST Universal RNA Extraction Kit)、反转录试剂盒(PrimeScriptTMRT Reagent Kit With gDNA Eraser)、Real-time PCR试剂盒(SYBR®Premix Ex TaqTMⅡ)均购于TaKaRa公司。
1.1.3 供试培养基 PDA培养基:马铃薯200 g,葡萄糖20 g,琼脂15 g,加水定容至1 L。水琼脂培养基:琼脂12 g,加水定容至1 L。改良Fries培养基:蔗糖30 g,酒石酸胺5 g,NaCl 0.1 g,CaCl20.1 g,KH2PO41 g,NH4NO31 g,MgSO40.5 g,酵母浸膏0.5 g,琼脂12 g,加水定容至1 L。
1.2 试验方法
1.2.1StSNF1的生物信息学分析 通过Blast搜索S.turcica基因组数据库(JGI,http://genome.jgi.doe.gov/Settu1),确定StSNF1在基因组中的精确位置。利用软件MEGA 4.0构建系统发育树。利用在线软件ProtParam(http://www.expasy.org/tools/protparam.html)分析核酸及氨基酸序列、蛋白质理化性质。利用在线软件InterProScan(http://www.ebi.ac.uk/Tools/pfa/iprscan/)对StSNF1蛋白的功能域进行预测。分别利用SOMPA和PHYRE2软件对StSNF1蛋白的二级结构和三维结构进行分析与预测[18-20]。
1.2.2 玉米大斑病菌侵染过程中StSNF1的表达分析 将玉米大斑病菌菌株SYY1303培养于PDA平板上,26 ℃黑暗培养7 d。用直径为1 cm的打孔器打取菌饼,用镊子将菌柄接种至玉米叶片上,保湿24~48 h。分别在接种后0,6,12,24,36,48,72,96,120 h,剪取菌饼下方的玉米叶片80~100 mg,液氮速冻样品,-80 ℃保存备用。利用Snapgene软件设计Real-time PCR引物,以18S rRNA作为内参基因[21],引物序列如下:StSNF1-F:5′-TCTTGAAACAG
GAAGCAGTGTG-3′,StSNF1-R:5′-CCAACTAACCCG
TACTCAGGTA-3′,18S-F:5′-GGCATCAGTATTCAGGTTGTC-3′,18S-R:5′-GTTAAGACTACGACGGTATC-3′,引物均由上海生工生物工程有限公司合成。采用TaKaRa MiniBEST Universal RNA Extraction Kit提取玉米大斑病菌不同侵染时期样品的RNA。采用PrimeScriptTMRT Reagent Kit With gDNA Eraser试剂盒进行cDNA的合成。采用美国伯乐公司CFX96荧光定量PCR仪,以不同侵染时期的cDNA为模板,SYBR®Premix Ex TaqTMⅡ(Tli RNaseH Plus)试剂盒进行Real-time PCR扩增。25 μL PCR体系中包含:2×SYBR®Premix ExTaqTM12.5 μL,StSNF1基因上下游引物各1 μL,模板2 μL,加双蒸水至25 μL。Real-time PCR程序:95 ℃,30 s;95 ℃,5 s,60 ℃,30 s,40个循环;95 ℃,5 s;60 ℃,30 s。每个样品设置4个技术性重复,3个生物学重复。
1.2.3 玉米大斑病菌分生孢子萌发侵染过程中StSNF1的表达分析 将玉米大斑病菌菌株SYY1303培养于PDA水琼脂平板上,26 ℃黑暗培养,分别收集6,12,24 h的分生孢子,采用TaKaRa MiniBEST Universal RNA Extraction Kit提取玉米大斑病菌分生孢子萌发不同时期样品的RNA。采用PrimeScriptTMRT Reagent Kit With gDNA Eraser试剂盒进行cDNA的合成。采用Real-time PCR方法检测不同阶段的表达规律,方法同1.2.2。
1.2.4StSNF1在不同碳源培养下的表达分析 将玉米大斑病菌菌株SYY1303培养于改良的固体Fries培养基,其中将碳源换成1%的葡萄糖、蔗糖、果糖、果胶和甘油,26 ℃黑暗培养7 d,收集菌丝提取RNA,反转录成cDNA,采用Real-time PCR方法检测StSNF1的表达情况,方法同1.2.2。
2 结果与分析
2.1StSNF1的生物信息学分析
2.1.1StSNF1在基因组中的定位 以前期扩增到的StSNF1序列为探针序列,利用BlastN搜索玉米大斑基因组数据库,基因ID号为008026214,基因全长3 046 bp,scaffold_17负链的97 793-100 838位置。
2.1.2 系统发育分析 利用MEGA 4.0软件对StSNF1与玉米圆斑病菌、指状青霉菌、核盘菌、尖孢镰刀菌、红褐肉座菌和稻瘟病菌的SNF1核苷酸序列进行系统发育与分析。由图1可知,StSNF1与CcSNF1同源关系较近。

图1 StSNF1系统发育树
2.1.3 理化性质分析 利用ProtParam软件对StSNF1蛋白理化性质进行分析,该蛋白质含有877个氨基酸残基,分子量为97 941.49,理论等电点为8.29,预测该蛋白质在280 nm处的吸光度为0.871~0.873,不稳定系数为53.24,脂肪系数为74.41,蛋白质的平均疏水性为-0.638。
2.1.4 保守功能域分析 通过InterProScan在线软件对StSNF1蛋白质序列进行分析,结果发现,该蛋白质的N端第62-317个氨基酸残基位置具有1个蛋白激酶结构域,第359-406个氨基酸残基具有1个泛素相关结构域(UBA)、碳代谢产物去阻遏蛋白激酶结构域,而C端具有5个激酶相关结构域1(KA1),分别是第473-495,538-589,575-688,743-784,842-875个氨基酸残基(图2)。进一步分析发现,在蛋白激酶结构域内,第71-94位置具有蛋白激酶ATP结合位点,第183-195位置具有丝氨酸/苏氨酸活性位点。
2.1.5 StSNF1蛋白结构域分析及高级结构预测 StSNF1蛋白的二级结构是由SOMPA软件分析得到,发现该蛋白中无规则卷曲的含量为48.23%,由423个氨基酸残基构成,α-螺旋的含量为27.59%,β-折叠含量较少,占15.85%,β-转角含量最少,仅占8.32%(图3)。
利用PHYRE2在线软件预测并分析StSNF1蛋白的三维结构,第63-318个氨基酸残基所构成的StSNF1蛋白部分的三维结构模型如图4所示。该蛋白的三维结构包括一个C端域和一个N端域,C端域由2个α-螺旋和6个β-折叠组成,N端域由7个α-螺旋和2个β-折叠组成,两者之间由形成裂缝的铰链连接,符合二级结构预测结果。利用SAVES在线软件对StSNF1蛋白的三维结构模型进行评估,并利用拉氏图(The ramachandran diagram)鉴定构象的合理性。96.8%的残基位点位于构象的核心区域,2.9%的残基位点位于构象的允许区域,不合理残基占0.3%;3D-1D score≥0.2占95.70%。综上所述,在StSNF1蛋白的三维模型中,绝大多数的氨基酸残基构成的二面角是合理的,空间结构稳定,该模型具有合理性。
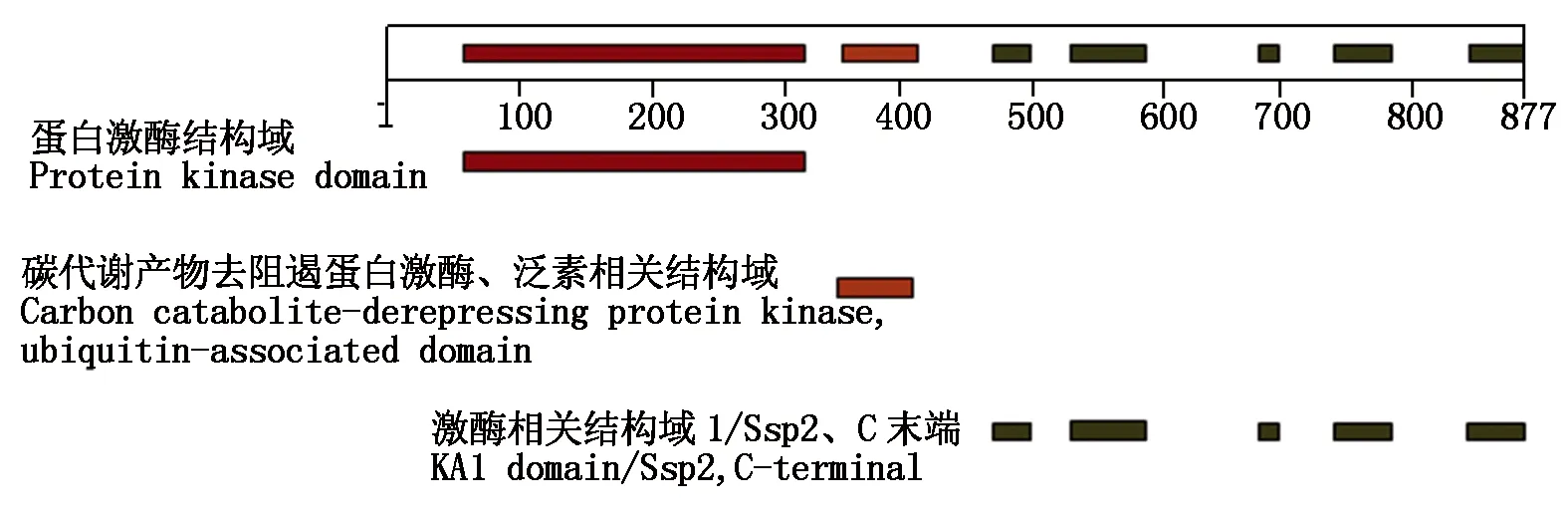
图2 SNF1蛋白质的保守结构
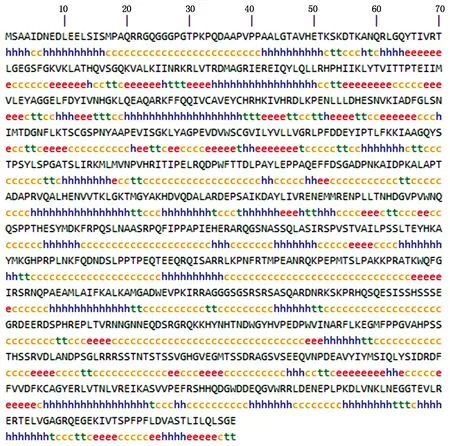
h.α-螺旋;e.β-折叠;t.β-转角;c.无规则卷曲。
2.2 玉米大斑病菌不同侵染时期StSNF1的表达规律
利用Real-time PCR检测玉米大斑病菌在不同侵染时期的表达情况,如图5所示,随着侵染时间增加,StSNF1表达量逐渐增加,其中72 h时表达量最大,96 h时有所下调,120 h时表达量再次上调。
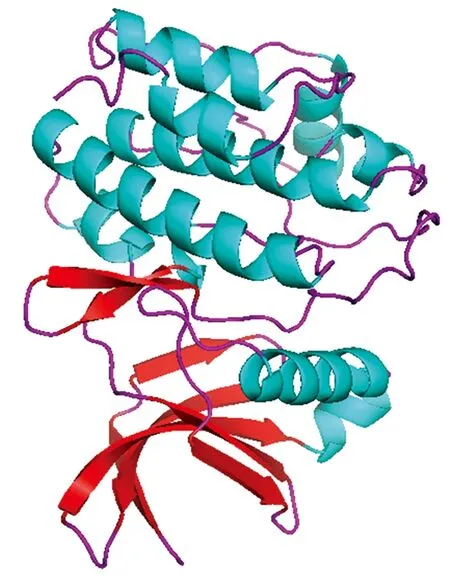
图4 预测的StSNF1蛋白三维结构
2.3 玉米大斑病菌分生孢子侵染生长过程中StSNF1的表达规律
利用Real-time PCR检测StSNF1在分生孢子侵染生长不同阶段的表达规律,如图6所示,StSNF1在分生孢子诱导萌发6 h(即附着胞形成时期)表达量最低,12 h(即附着胞成熟时期)表达量随之升高,24 h(即侵入丝形成时期)表达量达到最大。
2.4StSNF1在不同碳源培养下的表达分析
利用Real-time PCR检测StSNF1在不同碳源培养条件下的表达情况,如图7所示,StSNF1在蔗糖
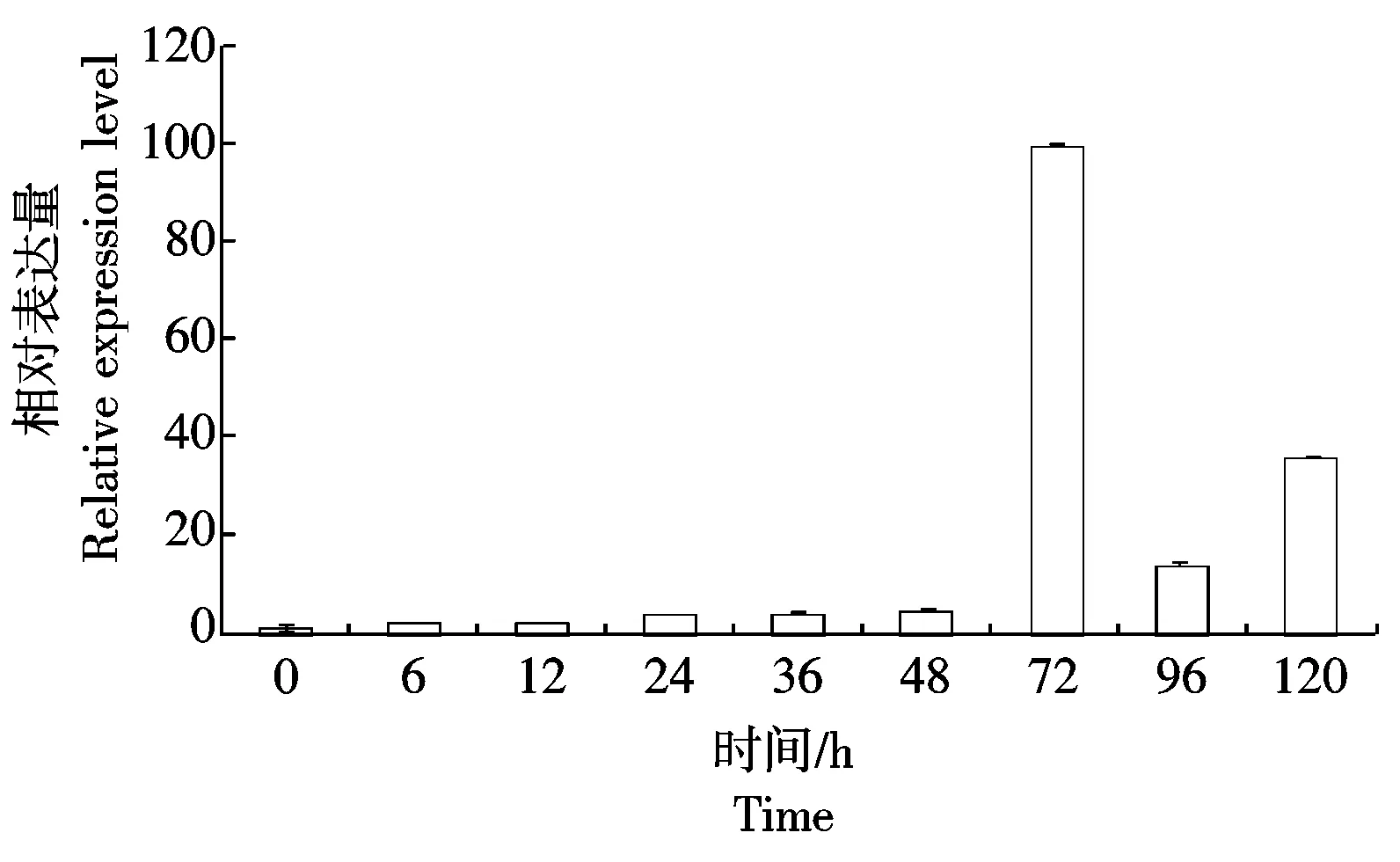
图 5 玉米大斑病菌不同侵染时期StSNF1的表达量
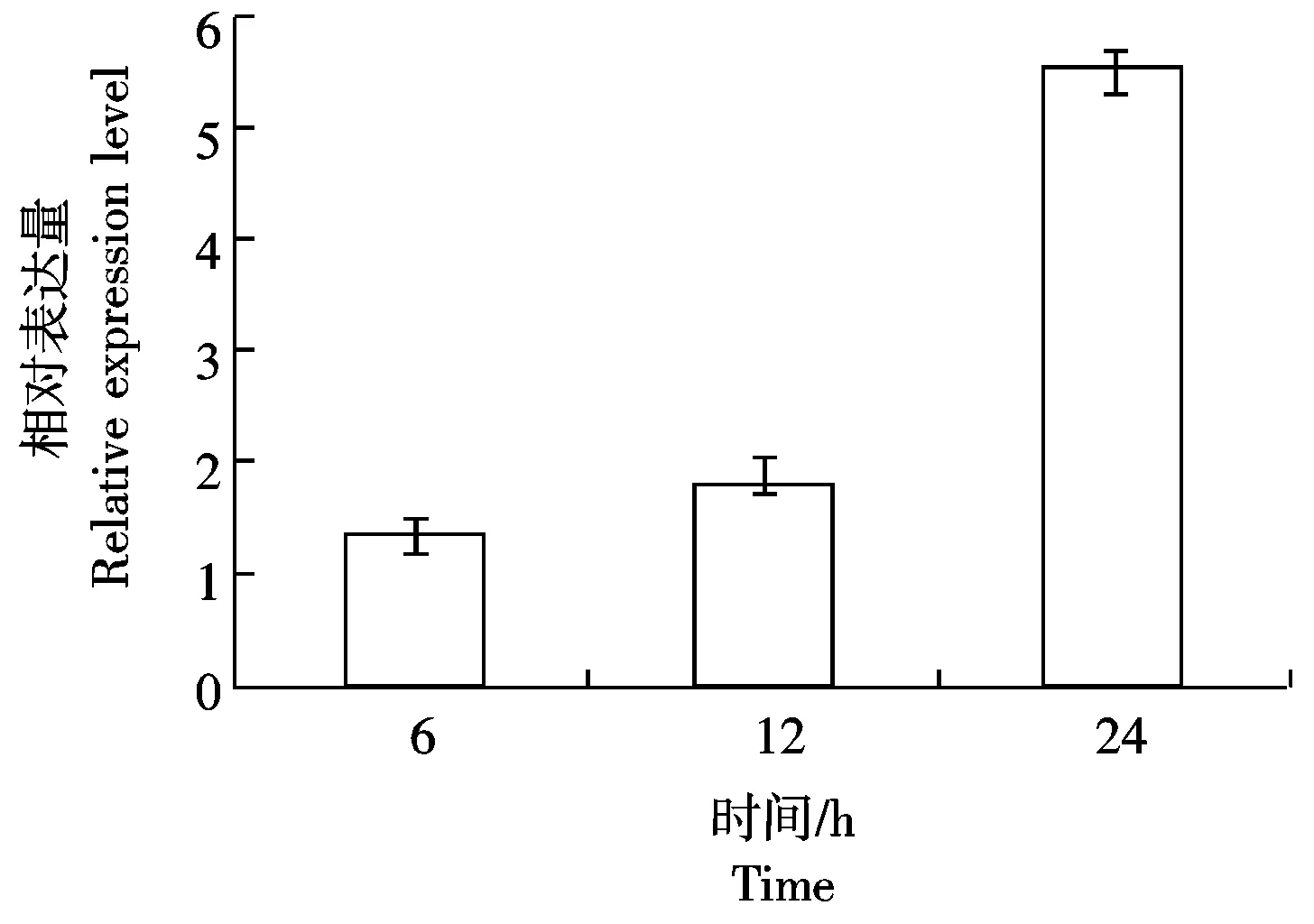
图 6 玉米大斑病菌分生孢子侵染生长过程中StSNF1的表达量
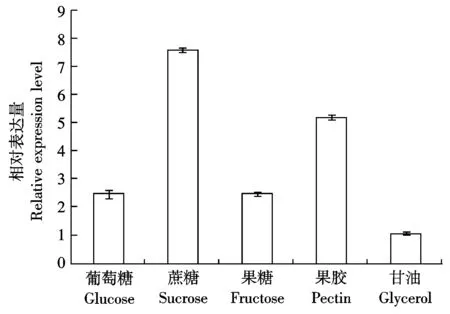
图7 玉米大斑病菌不同碳源培养下StSNF1的表达量
培养基中表达量最高,其次是果胶培养基,在甘油培养基中表达量最低。
3 讨论
本研究运用生物信息学技术,确定了StSNF1位于玉米大斑病菌基因组scaffold_17负链的97 793-100 838位置,全长为3 046 bp。通过同源性分析,该基因与CcSNF1同源关系较近。研究表明,SNF1p(蔗糖非发酵蛋白激酶,Sucrose nonfermenting 1 protein kinase)是AMPK在酿酒酵母中的同源蛋白,并且在真核生物中普遍存在且高度保守[6-7],该激酶的活性受到环化腺苷酸(cAMP)信号通路中5′-AMP的调控[22]。本研究分析了StSNF1蛋白的保守功能域,发现该蛋白具有典型的蛋白激酶结构域、碳代谢产物去阻遏蛋白激酶结构域和KA1(Kinase associated domain 1)结构域[23-24]。在蛋白激酶结构域内,具有ATP结合位点和丝氨酸/苏氨酸活性位点,这与曾晓清在稻瘟病菌和刘一贤在香蕉枯萎病菌中的蛋白结构研究结果相同[25-26],因此,StSNF1蛋白是典型的丝氨酸/苏氨酸蛋白激酶家族成员。
植物病原真菌中SNF1的功能已有报道,Tonukari等[12]通过研究发现,C.carbonum的SNF1基因与细胞壁降解酶和毒力有关,CcSNF1突变株致病性下降。Lee等[15]研究发现,在Gibberellazeae中SNF1可以调控细胞壁水解酶的活性,GzSNF1突变株对大麦的致病性下降,细胞壁降解酶相关基因表达下调。本研究首次在S.turcica侵染寄主过程中对StSNF1的表达情况进行探究,发现该基因在接种后72 h的表达量达到最大,唐琳等[27]研究表明,S.turcica细胞壁降解酶基因均在接种后期上调表达,这与本试验中StSNF1表达的时间节点相一致,由此推测StSNF1可能参与调控细胞壁降解酶基因的表达。Yi等[14]研究发现,SNF1对于M.oryzae孢子的萌发有重要作用。本研究发现,S.turcica分生孢子萌发侵染过程中StSNF1的表达量逐步上调,24 h时(侵入丝形成时期)达到最大,由此可以确定StSNF1与分生孢子萌发侵染有关。Tzima等[16]研究发现,V.dahliael中SNF1与碳源代谢密切相关,VdSNF1突变株无法利用果胶。本研究发现,StSNF1在蔗糖和果胶培养基上的表达量是在葡萄糖培养基上表达量的2~3倍,由此可以确定StSNF1与碳源利用有关。综上所述,StSNF1在侵染后期表达,该基因通过调控细胞壁降解酶的活性侵染寄主,利用寄主细胞内的非发酵型碳源进行次级侵染。
本研究通过Real-time PCR技术对StSNF1的表达情况进行了初步分析,想深入了解该基因的功能还需要获得StSNF1突变株,通过比较野生型与突变株在碳源利用、菌体发育以及致病性等方面的差异来明确其功能。
[1] 张秀霞,高增贵,周晓锟,等.东北地区玉米大斑病菌生理分化研究[J].华北农学报,2012,27(3):227-230.
[2] Tang L, Gao Z G, Yao Y, et al. Identification and genetic diversity of formae speciales ofSetosphaeriaturcicain China[J]. Plant Disease, 2015, 99(4): 482-487.
[3] 董金皋.农业植物病理学(北方本) [M].北京:中国农业出版社,2001.
[4] Perkins J M, Hooker A L. Reactions of eighty-four sources of chlorotic lesion resistance in corn to three biotypes ofHelminthosporiumturcicum[J]. Plant Diseases, 1981, 65(6):502.
[5] Carling D, Zammit V A, Hardie D G. A common bicyclic protein kinase cascade inactivates the regulatory enzymes of fatty acid and cholesterol biosynthesis[J]. FEBS Letters, 1987, 223(2): 217-222.
[6] Kayikciö, Nielsen J. Glucose repression inSaccharomycescerevisiae[J]. Fems Yeast Research, 2015, 15(6):68.
[7] Celenza J L C M. Cloning and genetic mapping ofSNF1, a gene required for expression of glucose-repressible genes inSaccharomycescerevisiae[J]. Molecular &Cellular Biology, 1984, 4(1): 49-53.
[8] Chulkin A M, Vavilova E A, Benevolenskii S V. Mutational analysis of carbon catabolite repression in filamentous fungusPenicilliumcanescens[J]. Molecular Biology, 2011, 45(5):804.
[9] 吴 艳,顾 阳,任 聪,等.微生物分解代谢物控制蛋白CcpA的研究进展[J].生命科学,2011(9):882-890.
[10] Hedbacker K, Carlson M. Snf1/ampk pathways in yeast[J]. Frontiers in Bioscience, 2011, 13(7):2408-2420.
[11] 张天园.柑橘绿霉病菌转录因子PacC、Crz1及蛋白激酶SNF1的功能分析[D].杭州:浙江大学,2013.
[12] Tonukari N J, Scott-craig J S, Walton J D. TheCochlioboluscarbonumSNF1 gene is required for cell wall-degrading enzyme expression and virulence on maize[J]. The Plant Cell, 2000, 12(2): 237-248.
[13] Ospina-giraldo M D, Mullins E, Kang S. Loss of function of theFusariumoxysporumSNF1 gene reduces virulence on cabbage andArabidopsis[J]. Current Genetics, 2003, 44(1): 49-57.
[14] Yi M, Park J H, Ahn J H, et al. MoSNF1 regulates sporulation and pathogenicity in the rice blast fungusMagnaportheoryzae[J]. Fungal Genetics and Biology,2008, 45(8): 1172-1181.
[15] Lee S H, Lee J, Lee S, et al. GzSNF1 is required for normal sexual and asexual development in the ascomyceteGibberellazeae[J]. Eukaryotic Cell, 2009, 8(1): 116-127.
[16] Tzima A K, Paplomatas E J, Rauyaree P, et al. VdSNF1, the sucrose nonfermenting protein kinase gene ofVerticilliumdahliae, is required for virulence and expression of genes involved in cell-wall degradation[J]. Molecular Plant-microbe Interactions, 2011, 24(1): 129-142.
[17] Zhang Tianyuan, Sun Xuepeng, Xu Qian, et al.PdSNF1, a sucrose non-fermenting protein kinase gene, is required forPenicilliumdigitatumconidiation and virulence[J]. Applied Microbiology and Biotechnology, 2013, 97(12): 5433-5445.
[18] 刘微微,郭丽婕,贾 慧,等.玉米大斑病菌交配型基因克隆及生物信息学分析[J].华北农学报,2014(5):10-15.
[19] 张长志,李 坡,谷守芹,等.玉米大斑病菌转录因子StSte1 的表达特性分析及其调控基因的筛选[J].中国农业科学,2013,46(8):1603-1609.
[20] 巩校东,王 玥,张 盼,等.玉米大斑病菌MAPK基因StIME2的基因组定位、蛋白质结构预测及表达分析[J].中国农业科学,2015,48(13):2549-2558.
[21] SHEN Shen, HAO Zhimin, GU Shouqin, et al. The catalytic subunit of cAMP-dependent protein kinase A StPKA-c contributes to conidiation and early invasion in the phytopathogenic fungusSetosphaeriaturcica[J]. FEMS Microbiology Letters, 2013, 343(2): 135-144.
[22] 陈 雷.AMP激活的蛋白质激酶(AMPK)调控机制的研究[D].北京:清华大学,2010.
[23] Jiao Rubin, Postnikoff S, Harkness T A, et al. The SNF1 kinase ubiquitin-associated domain restrains its activation, activity, and the yeast life span[J]. The Journal of Biological Chemistry, 2015, 290(25): 15393-15404.
[24] Cho Y S, Yoo J, Park S, et al. The structures of the kinase domain and UBA domain of MPK38 suggest the activation mechanism for kinase activity[J]. Acta Crystallographica, 2014, 70(2):514-21.
[25] 曾晓清.稻瘟病菌SNF1途径和细胞自噬蛋白MoAtg14的功能分析[D].杭州:浙江大学,2014.
[26] 刘一贤,周端咏,毛 超,等.香蕉枯萎病菌SNF1基因的克隆及生物信息学分析[J].广东农业科学,2012,39(21):160-164.
[27] 唐 琳,高增贵,杨瑞秀,等.凸脐蠕孢菌玉米、高粱专化型的细胞壁降解酶活性及基因表达分析[J].植物保护学报,2015,42(6):935-941.
Analysis of the Genomic Location, Protein Structure Prediction and Expression ofStSNF1 inSetosphaeriaturcica
HE Shidao, YANG Ruixiu, LIU Bo, YUAN Depeng, YAO Yuan, SUN Yanqiu, GAO Zenggui
(Institute of Plant Immunology, Shenyang Agricultural University, Shenyang 110866, China)
Sucrose nonfermenting 1 protein kinase is the key enzyme in the process of carbon source metabolic derepression. For this, the location ofStSNF1 in genome, the protein structural characteristics of StSNF1, the expression level ofStSNF1 at different infection stages in maize, different stages in the conidial invasive growth ofSetosphaeriaturcicaand under the different carbon sources were studied. The results showed that the Gene ID number was 008026214, length was 3 046 bp, located between 97 793 and 100 838 in the antisense strand of scaffold_17 in the genome ofS.turcica. The StSNF1 protein was more closely related toCochlioboluscarbonumSNF1, and encoded by 877 amino acid residues. StSNF1 showed characteristic conserved domains of protein kinase, carbon catabolite-derepressing protein kinase at N-terminal and a kinase associated domain 1 at C-terminal. An ATP binding site and a serine/threonine active site were found in the protein kinase domain. The results of Real-time PCR indicated that the expression ofStSNF1 was significantly up-regulated in late stage of infection, especially at 72 h. The expression trends ofStSNF1 was up-regulated during the conidia germination.The highest expression ofStSNF1 was found in the medium that sucrose as the sole carbon source followed by pectin. All the results suggestedStSNF1 was closely related to the infection and the carbon source utilization. Moreover, in the late stage of infection, the intracellular non-fermentable carbon sources of host were used in secondary infection.
Setosphaeriaturcica;SNF1;Protein kinase;Expression analysis
2017-04-20
国家重点研发计划课题(2016YFD0300704)
何世道(1991-),男,辽宁营口人,在读硕士,主要从事玉米病害研究。
高增贵(1966-),男,内蒙古准格尔人,研究员,博士,主要从事玉米病害和蔬菜病害生物防治等研究。
S513.03;Q78
A
1000-7091(2017)03-0085-06
10.7668/hbnxb.2017.03.013
