绿豆SSR—PCR反应体系的建立与优化
2017-06-30赵雅楠王颖张东杰
赵雅楠++王颖++张东杰
摘要:为探究影响绿豆SSR-PCR反应的因素,建立绿豆SSR-PCR最佳反应体系,为绿豆遗传多样性分析、品种鉴定等提供参考。采用正交设计与单因素试验联合分析法,从Mg2+、Taq酶、引物、dNTPs及模板DNA 5个因素对绿豆SSR-PCR反应体系进行优化分析。5个因素中,Mg2+对SSR-PCR结果影响最大。SSR-PCR反应最佳体系为 20 μL 体系中含0.8 mmol/L Mg2+、1.25 U Taq酶、1.4 μmol/L引物、1.8 mmol/L dNTPs、25 ng模板DNA。该绿豆 SSR-PCR反应体系的建立为进一步利用SSR分子标记技术研究绿豆资源奠定了基础。
关键词:绿豆;SSR-PCR;正交设计;单因素试验;分子标记技术
中图分类号: S643.401文献标志码: A文章编号:1002-1302(2017)08-0023-03
绿豆[Vigna radiata(Linn.)Wilczek]是豇豆属(Vigna)亚洲豇豆亚属(Ceratotropis)的主要栽培豆种,是我国的原产作物,有2 000多年的栽培历史,目前的主产区集中在内蒙古、吉林、黑龙江等地,种植面积和产量均居世界前列。绿豆种子富含淀粉、蛋白质、膳食纤维、矿物质及维生素等营养素和低聚糖、黄酮等功能成分[1],同时还是传统的药食同源食材,可清毒解暑、降血糖、抗肿瘤等,被誉为粮食中的“绿色珍珠”“济世粮谷”。然而在长期种植过程中,由于绿豆优良亲本材料的频繁交流,导致品种退化、遗传变异水平降低、新品育种缓慢,使绿豆在国内外市场的发展面临严峻考验[2]。
近些年,DNA分子标记技术不断发展,为作物育种、遗传多样性分析、种子纯度鉴定等提供了新途径。简单重复序列(simple sequence repeats,简称SSR)是广泛分布于植物基因组中的以1~6个串联核苷酸为重复单位的DNA序列,作为第二代分子标记技术的典型代表,SSR因多态性丰富、重复性好、标记共显性等特点[3],在大豆、玉米、水稻等作物的遗传图谱构建、遗传多样性分析、QTL定位等研究中应用广泛[4-6],而目前国内基于SSR技术对绿豆的研究较少,与一些大宗作物相比,绿豆基因组研究还处于落后阶段,特别是针对绿豆SSR-PCR体系建立及优化的研究鲜有报道。本研究利用正交设计与单因素试验联合分析法对影响绿豆SSR-PCR反应体系的5个因素(Mg2+、Taq酶、引物、dNTPs、模板DNA)进行优化,旨在为绿豆SSR标记研究提供参考。
1材料与方法
1.1试验材料
以绿豆C0633为试材,试材模板DNA由北京市农林科学院作物研究所提供。以来源于NCBI的引物VJ31122B(正向:5′-TCACAAAGGGAGGGAAGAGA-3′;反向:5′-CCCCAGGTTGGTTGGTTGGA-3′)为目标引物,由生工生物工程(上海)股份有限公司合成。植物基因组DNA提取试剂盒、Mg2+、10×buffer、dNTPs、Taq DNA聚合酶等均购于天根生化科技(北京)有限公司。
1.2试验方法
1.2.1绿豆基因组DNA提取以新鲜绿豆嫩叶为材料,利用Plant Genomic DNA Kit提取绿豆基因组DNA,用1%琼脂糖凝胶电泳检测提取质量,利用TBD-3000核酸蛋白检测仪分析DNA纯度与浓度,以确定是否满足后续试验要求,检测合格后稀释成30 ng/μL,置于-20 ℃冰箱保存。
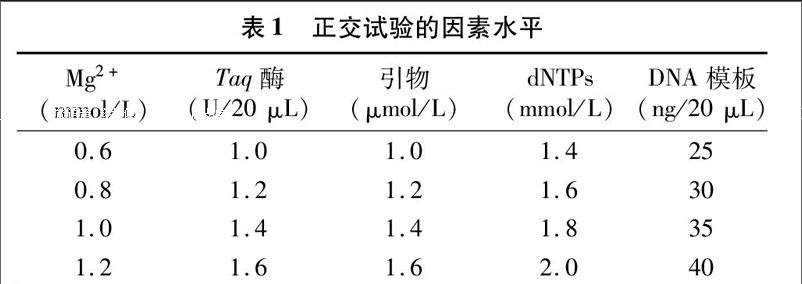
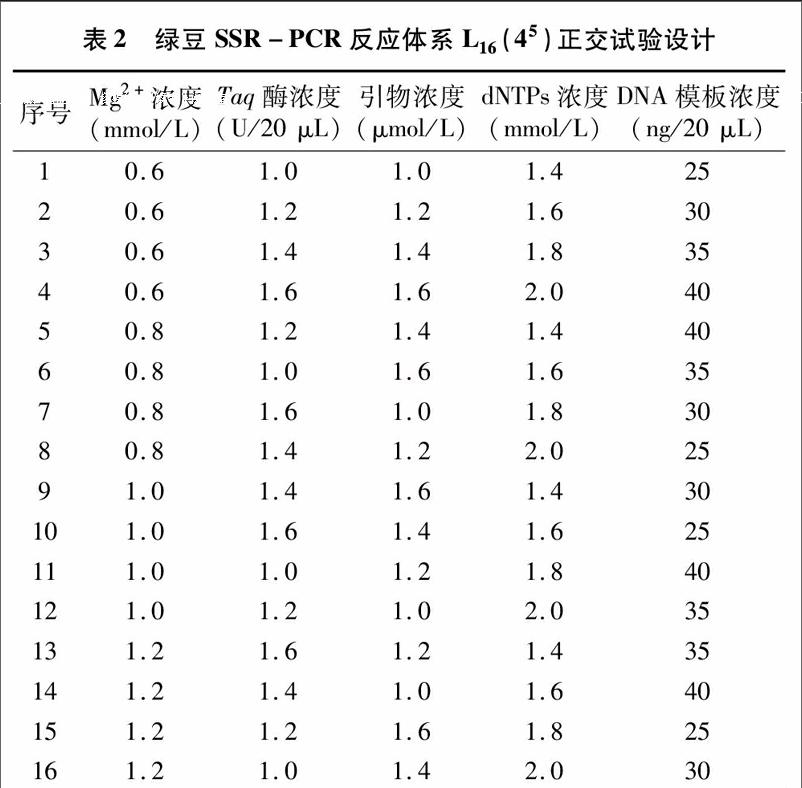
1.2.2正交设计优化采用L16(45)正交设计对Mg2+、Taq酶、引物、dNTPs及模板DNA进行优化。总体系为20 μL,除上述变化因素外,每组体系还含有2 μL 10×PCR buffer,其他用ddH2O补齐,每个处理重复2次(表1、表2)。
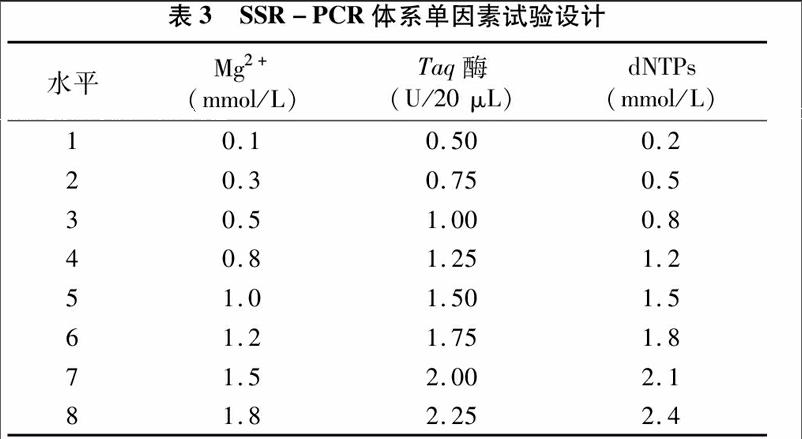
1.2.3单因素试验设计在正交设计优化的基础上,对影响SSR-PCR反应体系的部分因素进行单因素分析,进一步优化SSR-PCR体系。每个因素设置8个水平,每个梯度处理重复2次(表3),总体系中包含2 μL 10×PCR buffer,其他影响因素浓度参照正交设计中的最优组合条件,用ddH2O补齐至20 μL。
1.2.4SSR-PCR扩增及检测PCR反应在东胜龙ETC-811 PCR仪上进行,参数为94 ℃预变性5 min,40个循环(94 ℃ 变性30 s,54 ℃退火30 s,72 ℃延伸30 s),72 ℃延伸
8 min,4 ℃下保存。以Low MW DNA Marker-A作对照,利用2%琼脂糖凝胶电泳检测PCR扩增产物,在紫外凝胶成像系统下观察拍照。
2结果与分析
2.1正交试验结果分析
由图1可知,16个处理因Mg2+、Taq酶、引物、dNTPs及模板DNA濃度的不同而导致扩增效果存在明显差异。其中第4组处理未扩增出条带,11、12、13组条带较弱,第3、6、10组处理效果较好,条带清晰、明亮,未出现明显拖尾、弥散现象。参照贺石林的统计方法[7],对2次正交试验结果进行独立打分统计,根据条带强弱、背景清晰程度等进行评价,最优处理得16分,最差处理得1分,各体系得分分别为9、11,10、10,9、7,1、1,9、10,15、16,10、8,9、9,3、2,16、15,4、3,2、2,4、3,7、7,9、8,7、11。根据2次评分求出各因素不同水平评分之和T、平均分K以及极差R。在假设不考虑各因素间交互作用的情况下,R值越大,表示该因素对反应体系的影响越大,K越大,则表示反应水平越好[8]。由表4可知,5个因素对 SSR-PCR 反应影响顺序依次为Mg2+>Taq酶>dNTPs>引物>DNA模板。
2.2单因素试验设计优化绿豆SSR-PCR体系
2.2.1Mg2+对SSR-PCR反应的影响Mg2+是影响SSR-PCR反应的重要因素之一,通过调控Taq酶活性影响PCR反应结果[9]。由图2可知,单因素优化试验设置的Mg2+浓度梯度范围内均能扩增出条带,但清晰度、特异性及背景颜色存在差异。Mg2+浓度在0.1~0.5 mmol/L时,扩增条带较弱、辨识度较低;浓度为0.8~1.0 mmol/L时,扩增条带清晰、明亮;当浓度大于1.0 mmol/L时,扩增产物量减少,条带现弥散状。故最佳反应体系中的Mg2+浓度确定为0.8 mmol/L,与正交优化试验结果一致。
2.2.2Taq酶对SSR-PCR反应的影响由图3可知,在一定范围内,随着Taq酶用量的增加,扩增条带逐渐清晰,但达
到一定浓度后,Taq酶用量即成为扩增效果的负相关因素。Taq酶浓度为0.5 U/20 μL时,未扩增出条带;浓度为0.75~1.00 U/20 μL时,扩增条带较弱且稳定性较差,结果不可靠;当Taq酶浓度为1.25 U/20 μL时,条带清晰、重复性好;而当浓度大于1.25 U/20 μL时,非特异性扩增条带增多,并出现引物二聚体。综合上述分析,最佳反应体系中Taq酶用量为1.25 U/20 μL,与正交优化试验结果相比,Taq酶浓度在最佳组合10与最佳水平之间。
2.2.3dNTPs对SSR-PCR的影响dNTPs是SSR-PCR反应的重要底物,由图4可知,当dNTPs浓度为0.2~0.8 mmol/L,条带较暗且有引物二聚体出现;当浓度为1.2、1.5 mmol/L时,引物二聚体消失,条带逐渐明亮清晰;当浓度达到1.8 mmol/L时,扩增效果最佳。随着dNTPs浓度继续增加,扩增效果差异不大,因此,从节约材料角度考虑,选择 1.8 mmol/L 为dNTPs的最佳反应浓度。
3讨论
SSR是以PCR反应为基础的分子标记技术,分析结果受PCR扩增效果影响,而PCR反应的扩增情况受诸多因素控制,且不同作物对PCR反应的敏感程度及要求也各不相同,因此建立和优化SSR-PCR反应体系是十分必要的。本研究采用正交设计与单因素试验综合分析法,对影响绿豆SSR-PCR反应体系的Mg2+、dNTPs、Taq酶、引物及模板DNA进行优化,该法充分利用了正交设计的整齐可比性、均衡分散性、可伸缩及效应明确的优点,既解决了单因素设计得到的各因素最优水平不一定是最佳组合的矛盾,又避免了试验量大、周期长等问题,使SSR-PCR的建立和优化更加有效、严谨。但须指出,对正交优化结果进行直观分析存在一定主观成分,因此在接下来的研究中应增加重复试验次数,多次评判以减少打分误差,也可以借助软件对结果进行分析。同时本研究在正交试验中的Mg2+浓度、酶含量、dNTPs浓度都在单因素优化试验的最佳范围内,但结果并不完全吻合,这可能是因为正交试验设置的水平梯度间隔较大,也可能是各因素之间存在一定的交互作用,试验得到的最佳浓度不同。此外,从试验结果还可以看出,SSR-PCR反应体系中对因素浓度的要求并不十分精确,即在一定范围内均可得到清晰、稳定的条带。结合考虑扩增效果及节约原料等因素,最终得到的最佳体系为(20 μL):0.8 mmol/L Mg2+、1.25 U Taq酶、1.4 μmol/L引物、1.8 mmol/L dNTPs、25 ng模板DNA。
根据方差分析可知,Mg2+对SSR-PCR反应体系影响最大,其次是Taq酶,然后依次是dNTPs、引物,模板DNA对体系影响最小。Mg2+是Taq DNA聚合酶的辅助因子,能够影响酶活性,同时还会与dNTPs产生拮抗作用[10],对PCR扩增效果的影响主要表现在PCR扩增产物的特异性及产量方面。Taq酶是Mg2+的依赖性酶,浓度过低会导致活性不够,PCR反应不充分,扩增产物产量少,浓度过高不仅会浪费原料,还会降低PCR反应的特异性,导致扩增条带难以辨识[11]。dNTPs是PCR反应的重要原料,浓度过高会增加非特异性扩增,浓度过低则可能消耗过快,造成单链化,最终导致扩增失败。引物与模板结合的特异性及稳定性直接影响PCR扩增产率。引物浓度过低,会降低产物产量,浓度过高则会与模板DNA结合形成二聚体,造成背景弥散、颜色加深[12]。同时,本研究发现模板DNA对SSR-PCR反应的影响最小,这可能与SSR标记的特点有关,即少量模板即可满足反应的需求,
对浓度和数量要求不严。另外,有学者指出,任何形式的DNA和RNA都能够满足作为底物的要求,在大多数的PCR过程中,对DNA模板浓度要求很宽泛,在某适宜浓度范围内即可,本研究结果与其基本吻合。
参考文献:
[1]王兰芬,武晶,景蕊莲,等. 绿豆种质资源芽期抗旱性鉴定[J]. 植物遗传资源学报,2014,15(3):498-503.
[2]刘慧. 我国绿豆生产现状和发展前景[J]. 农业展望,2012,8(6):36-39.
[3]Reddy R N,Madhusudhana R,Mohan S M,et al. Characterization,development and mapping of unigene-derived microsatellite markers in sorghum [ Sorghum bicolor (L.) Moench][J]. Molecular Breeding,2012,29(3):543-564.
[4]柏锡,梁爽,邢雪莹. 干旱、高温胁迫下大豆SSR标記与产量性状关联分析[J]. 东北农业大学学报, 2015,46(10):15 - 22.
[5]王贺,姜敏,刘欣芳,等. 玉米抗大斑病Ht基因有效SSR标记的筛选[J]. 玉米科学,2011,19(1):48-51,55.
[6]Singh S,Gautam R K,Singh R K,et al. Diversity analysis of Sesbania acculeata by using transferable rice SSR markers[J]. Crop Research,2010,39(1/2/3):127-131.
[7]贺石林. 中医科研设计与统计方法[M]. 长沙:湖南科学技术出版社,1989.
[8]王韦,肖松,吴洪娥,等. 月季ISSR-PCR体系的建立[J]. 种子,2015,34(7):33-36.
[9]程江,郑常祥. 水稻SSR-PCR的反应体系优化及RIL亲本间多态性引物筛选[J]. 贵州农业科学,2013(4):10-13.
[10]糜亚男,张水寒,蔡媛,等. 杜仲SSR-PCR反应体系的优化[J]. 中国实验方剂学杂志,2015(2):1-6.
[11]梁玉琴,李芳东,傅建敏,等. 正交设计优化柿属植物SSR-PCR反应体系[J]. 经济林研究,2011,29(4):17-22.
[12]徐璕,张合平. 南方红豆杉SSR-PCR扩增体系的建立[J]. 广西林业科学,2015,44(1):54-58.
