万寿菊类胡萝卜素裂解双加氧酶基因CCD1克隆与表达分析
2017-06-15刘晓丛曾丽刘国锋彭勇政陶懿伟张邀月王梦茹
刘晓丛,曾丽,2,刘国锋,彭勇政,陶懿伟,张邀月,王梦茹
(1上海交通大学农业与生物学院,上海 200240;2农业部都市农业(南方)重点实验室,上海 200240;3华中农业大学园艺林学学院/园艺植物生物学教育部重点实验室,武汉 430070)
万寿菊类胡萝卜素裂解双加氧酶基因CCD1克隆与表达分析
刘晓丛1,曾丽1,2,刘国锋3,彭勇政1,陶懿伟1,张邀月1,王梦茹1
(1上海交通大学农业与生物学院,上海 200240;2农业部都市农业(南方)重点实验室,上海 200240;3华中农业大学园艺林学学院/园艺植物生物学教育部重点实验室,武汉 430070)
【目的】克隆万寿菊(Tagetes erecta L.‘Scarletade’)类胡萝卜素裂解双加氧酶基因CCD1(TeCCD1),分析其序列特征和表达特性,为阐明其在类胡萝卜素降解途径中生物学功能及进一步探讨万寿菊花色形成机理提供理论基础。【方法】依据万寿菊花蕾转录组数据,利用同源序列比对结果设计引物,结合RT-PCR技术克隆获得万寿菊CCD1 cDNA全长,分析其序列特征;利用Real-time PCR分析舌状花未开花蕾、半开花蕾、开放的头状花序和完全开放的头状花序4个不同发育时期的基因表达特性。【结果】克隆获得万寿菊CCD1(GenBank登录号:KX557488)的cDNA全长序列为1 746 bp,编码区长度1 626 bp,编码541个氨基酸。蛋白质分析表明TeCCD1为不稳定蛋白,不含信号肽,属RPE65超家族(登录号:PF03055),包含CCD家族保守结构域,主要定位于细胞质。万寿菊CCD1核酸序列与除虫菊CCD1同源性最高,为89%;氨基酸序列分析表明万寿菊CCD1与除虫菊CCD1同源性高达93%,与其他19个不同种属的CCD1同源性在75%—83%,说明TeCCD1是高度保守的基因;系统进化树分析显示TeCCD1的进化基本符合植物分类学的进化规律,并具有明显的种属特征,万寿菊与菊科同源基因亲缘关系最近。Real-time PCR分析表明TeCCD1在舌状花发育过程中均有表达,随舌状花的开放逐渐升高,S4期达到最大值。【结论】克隆获得万寿菊舌状花的CCD1,是典型的CCD家族成员,为高度保守的基因,主要定位于细胞质,万寿菊舌状花颜色变浅可能与CCD1表达量增加导致类胡萝卜素降解有关。
万寿菊;类胡萝卜素裂解双加氧酶基因;花色;基因表达
0 引言
【研究意义】万寿菊(Tagetes erecta L.)为菊科万寿菊属的高附加值经济花卉,根据其用途可分为观赏万寿菊和色素万寿菊,观赏万寿菊是重要的花坛花卉之一,色素万寿菊是提取叶黄素(Lutein)的优质植物源材料[1]。万寿菊广泛用于园林绿化、食品添加剂、医药、化妆品、保健品、生物农药及饲料添加剂等方面[2-4],中国是全世界万寿菊种植面积最大的国家之一,在云南、山东诸城、吉林、山西及内蒙赤峰等地均有大面积种植,并取得了良好的经济和社会效益。研究万寿菊类胡萝卜素代谢途径中裂解双加氧酶基因CCD1,为阐明万寿菊花色机理提供候选基因和理论依据。【前人研究进展】类胡萝卜素是植物重要的光合色素之一,作为可吸收光的辅助色素镶嵌于有色体和叶绿体膜中,是植物光合作用和植物呈色色素的重要组成部分。1950年,PORTER和LINCOLN[5]首次提出植物类胡萝卜素的合成途径,1997年TAN等[6]在玉米种子中第一次鉴定出类胡萝卜素裂解双加氧酶,为类胡萝卜素积累的研究提供了新方向,YUAN 等[7]系统总结了园艺作物类胡萝卜素合成与降解途径,进一步完善了园艺植物类胡萝卜素合成和降解的代谢途径。目前催化类胡萝卜素合成代谢途径的相关核心基因和酶已确定,而降解代谢途径相关酶基因在拟南芥、烟草、枇杷、菊科、牡丹、甜瓜、黄瓜、甜橙、番红花、黄桃、葡萄、桂花、杜鹃花及百合等多种不同科属植物中也已被克隆与研究[8-21],生物信息学分析发现参与拟南芥类胡萝卜素降解代谢途径的关键酶基因有9个,统称为类胡萝卜素裂解双加氧酶基因,英文简称CCDs,其中5个命名为NCED,与ABA合成有关,包括NCED2、NCED3、NCED5、 NCED6 及NCED9;4个命名为CCD,与β-柠乌素、β-紫罗酮及独脚金内酯等合成有关,包括CCD1、CCD4、CCD7及CCD8[22]。在几种CCD中,CCD1主要催化裂解类胡萝卜素9-10和9’-10’双键,具广泛的底物特异性,且是唯一功能定位于细胞质的类胡萝卜素裂解双加氧酶,前人多通过CCD1研究植物类胡萝卜素积累、花色变化及香气物质的产生,杨永霞等[9]研究CCD1在烟草中的表达,花中表达量高于其茎、叶及根。HAN 等[19]研究表明在2个桂花品种中CCD1和CCD4两个基因过表达导致其花瓣中α-胡萝卜素和β-胡萝卜素几乎无积累。AULDRIDGE等[23]敲除CCD1导致拟南芥成熟种子中类胡萝卜素含量显著增加。另外还发现CCD1影响矮牵牛、葡萄、枸杞及番茄等果实色泽和香气的产生及变化[24-25]。【本研究切入点】前人已通过同源克隆的方法获得了万寿菊舌状花中类胡萝卜素生物合成途径中各种关键酶基因[26],MOEHS[27]、王国兰[28]和林登贵[29]等研究了叶黄素合成关键酶基因LCY,当LCYe表达量高,LCYb表达低时,则导致α-胡萝卜素含量较高,叶黄素含量低,推测LCYe和LCYb可能是万寿菊类胡萝卜素合成途径关键基因。张嫔[30]研究了根癌农杆菌介导的万寿菊PSY遗传转化体系的影响因素。但目前有关万寿菊降解代谢途径关键酶基因方面的研究尚未见报道。【拟解决的关键问题】拟通过同源克隆得到万寿菊CCD1序列,进行生物信息学分析,并与NCBI中已发表的近源物种相关基因做系统进化分析,结合Real-time PCR技术检测该基因在万寿菊舌状花4个不同发育时期的表达量,初步推测万寿菊舌状花不同发育时期花色变化与类胡萝卜素裂解双加氧酶之间的关系,为阐明其在类胡萝卜素降解途径中生物学功能及进一步探讨万寿菊花色机理提供候选基因和理论参考。
1 材料与方法
试验于 2015年在上海交通大学农业与生物学院进行。
1.1 材料
1.1.1 植物材料 ‘猩红色’万寿菊(Tagetes erecta L. ‘Scarletade’)(花色深橙色),种子购自美国泛美种子公司(PanAmerican Seed),种植于试验田。参照DEL VILLAR-MARTÍNEZ等[26]方法,2015年7—9月分别采集万寿菊头状花序4个不同花发育时期的材料(图1):S1期,未开花蕾;S2期,半开花蕾;S3期,开放的头状花序;S4期,完全开放的头状花序,立即冷冻于液氮中,保存于-80℃的冰箱中备用。
1.1.2 主要试剂和试剂盒 胶回收试剂盒购自爱思进生物技术(杭州)有限公司(AXYGEN);RNA提取试剂盒、Trans2K DNA Marker、反转录试剂盒及ClontechRace试剂盒等购自北京TransGen生物技术有限公司(Transgen);大肠杆菌感受态细胞trans5α、PMD18-T载体、普通Taq酶及dNTP购自宝生物工程有限公司(TaKaRa);引物和样品测序由上海生工生物工程技术有限公司完成。
1.2 试验方法
1.2.1 RNA提取及单链cDNA的合成 取4个不同发育时期的万寿菊头状花序外缘第2—3层舌状花,去掉基部,选用舌状花中上部约0.1 g,混合,按照TransGen公司Transzol Up Plus RNA Kit试剂盒步骤提取RNA,NanoDrop2000测其浓度及纯度,凝胶电泳检测RNA质量。反转录总RNA为2 μg,按照TaKaRa公司的TaKaRa RNA PCR Kit (AMV) Ver. 3.0使用说明构建反应体系。

图1 万寿菊‘Scarletade’品种舌状花发育时期Fig. 1 Ray floret development of ‘Scarletade’
1.2.2 引物设计及 cDNA全长序列的克隆 通过DNAMAN对万寿菊花蕾转录组数据(由华中农业大学何燕红副教授提供)中查找到的目的基因与 NCBI中已报道的CCD1序列进行比对,初步判断序列编码起始终止位点,并利用软件Premier primer 5.0设计引物。TeCCD1全长克隆 PCR引物为 CCD1-F:TTCTATCTCCACACACACCAACTCT,CCD1-R:TAATTCTGGCATCCCATATACTCAT;PCR程序为94℃预变性5 min;94℃ 30 s,退火温度为60℃ 30 s,72℃ 2 min,35个循环;72℃ 10 min。扩增产物在1%琼脂糖凝胶进行电泳,采用凝胶成像分析系统拍照分析。扩增产物按照 TransGen公司 EasyPure Quick Gel Extraction Kit胶回收试剂盒说明书回收,回收目的片段连接pMD18-T载体,置于连接仪16℃过夜,连接产物热激转化感受态大肠杆菌trans5α,涂布于含100 mg·mL-1氨苄青霉素LB固体培养基,过夜筛选阳性克隆,阳性克隆送上海生工生物工程有限公司测序。
1.2.3 基因的生物信息学分析 通过NCBI上Blast工具验证测序结果,确认获得目的基因序列,利用DNAMAN Translation工具将测序得到的核酸序列翻译成氨基酸序列,得到编码的蛋白质的氨基酸组成。结合ProtParam、TMpred、NPS@中的DPM、SignalP 4.1 Server、ProtScale(亲/疏水性)、NCBI、和 SOPMA等网站或在线软件工具对目的基因所编码蛋白质的一级结构、二级结构、理化性质及生理功能进行分析和预测;利用DNAMAN比对不同物种间同源基因核酸序列和氨基酸序列,并采用Mega 6.0构建Neighbor Joining进化树(1 000 bootstraps)。
1.2.4 基因表达分析 分别取S1、S2、S3及S4 4个不同时期的万寿菊舌状花,取材方法同1.2.1,提取RNA,每时期3个生物学重复,反转录为cDNA。根据克隆所得基因序列设计荧光定量 PCR引物,TeCCD1荧光定量引物为CCD1-F:CAGCACCTTTC ATTTCAT,CCD1-R:CAATCTCCGCCTCAGTAG;内参基因 EF1α(基因登录号:EF413022.1)引物为E-F:TGATTACGGGTACATCCCAAGC;E-R:TGACACCAAGAGTGAAAGCAAGA;实时荧光定量PCR反应体系为20 µL:2×SYBR Premix Ex Taq 10 µL、cDNA模版1 µL、正反向引物各0.4 µL、RNase free ddH2O 8.2 µL。每个样品设置3个重复。反应在Bio-Rad CFX Connect TM Real-Time System 上进行。反应条件为95℃ 30 s;95℃ 15 s,60℃ 30 s,72℃ 30 s,40个循环。根据扩增曲线确定每个基因相应的 Ct值,以 EF1α为内参,相对量采用 2-ΔΔCt方法计算[31]。
2 结果
2.1 基因克隆及理化性质分析
利用特异性引物进行基因扩增,得到TeCCD1目的条带(图2);测序后DNAMAN和ProtParam分析表明TeCCD1全长序列为1 746 bp,编码区长度1 626 bp,推测其编码541个氨基酸(图3),分子量60 953.9 Da,等电点 5.79,脂肪族指数为 29.10,不稳定系数为 40.25,表明其为不稳定蛋白,GenBank登录号KX557488。
利用 TMpred分析预测 TeCCD1跨膜区域为136—156;通过ProtScale预测TeCCD1中氨基酸的疏水性/亲水性,第149位氨基酸疏水性最大,为2.0,第 410位氨基酸亲水性最大,为-2.567,总亲水性系数为0.706,表明TeCCD1为疏水性蛋白。
用SignalP 4.1 Server对TeCCD1蛋白进行分析并预测信号肽,TeCCD1预测结果为‘NO’,表明不含信号肽。
采用 PSORT WWW Server中 PSORT ⅡPrediction工具确定其细胞定位,TeCCD1出现在细胞质、细胞核、囊泡分泌系统、细胞骨架和线粒体的可能性分别为65.2%、21.7%、4.3%、4.3%和4.3%,表明TeCCD1可能主要定位于细胞质。
利用NCBI中CDD分析其功能保守域,表明TeCCD1属于RPE65超家族,为包含CCD家族类胡萝卜素裂解氧化酶特殊保守结构域的一类超家族。
利用NPS@中的DPM方法预测CCD1编码蛋白质的二级结构,α螺旋、β折叠、无规则卷曲及延伸链二级结构类型的氨基酸残基数占氨基酸总数的比例分别为30.50%、4.80%、53.42%和11.28%。
图2 万寿菊CCD1克隆电泳图Fig. 2 The electrophoresis results of TeCCD1 gene products
2.2 不同物种同源序列比对及系统进化分析
NCBI上下载已报道的 CCD1核酸序列,利用DNAMAN进行序列比对,万寿菊CCD1核苷酸序列与除虫菊CCD1同源性最高,可达89%,与麻疯树、葡萄、苹果、胡萝卜、杨树及梅花CCD1的同源性均为79%。利用DNAMAN对19个不同种属CCD1氨基酸序列进行比对,图4结果表明与万寿菊同源性最高的为除虫菊,可高达93%,与其他种属植物同源性在75%—83%,表明在长期的进化过程中TeCCD1是高度保守的基因。
图5表明TeCCD1的进化基本符合植物分类学的进化规律,并具有明显的种属特征。TeCCD1进化树存在双子叶植物和单子叶植物两个分支,水稻、玉米与同属于单子叶植物的番红花CCD1聚于单子叶进化分支;而双子叶植物中万寿菊与同属菊科的除虫菊同源基因亲缘关系最近,其次是蔷薇科的玫瑰、草莓、梅花以及杨柳科的杨树。将 19个不同种属的 CCD1与玫瑰、桂花及拟南芥的CCD4氨基酸序列比对发现,CCD1与CCD4蛋白同属于CCD家族,具同源性,但两者又分属于不同进化分支,表明克隆出的目的基因为CCD1,与CCD4在进化上有明显差异。

图3 万寿菊CCD1核苷酸序列及其编码的氨基酸序列Fig. 3 Nucleotide sequence and deduced amino acid sequence of TeCCD1 cDNA
2.3 TeCCD1表达特性
万寿菊舌状花S1、S2、S3及S4 4个时期颜色分别为黄绿色、淡黄色、深橙色、浅橙色,S3期颜色最深。图6表明万寿菊舌状花发育4个时期CCD1均有表达,呈先低后高的变化趋势,且各个时期存在显著差异,S4 期TeCCD1相对表达量显著高于其他3个时期,S1期最低,S2与S3期差异不显著;S2和S3期均为S1期的30倍左右,S4期为S2和S3期的3倍左右,表明TeCCD1相对表达量高低可能与万寿菊花色变化有关。
3 讨论
TeCCD1与除虫菊CCD1同源性最高,与其他物种已知CCD1同源性也较高,均在75%—83%,表明该基因在进化过程中具有高保守性[32]。PSORT ⅡPrediction预测 TeCCD1在细胞内功能定位于细胞质的概率最大,表明TeCCD1可能主要定位在细胞质,这与赵军林[33]和高军平[34]等研究结果一致。
进化树分析结果表明TeCCD1的进化基本符合植物分类学的进化规律,并具有明显的种属特征,存在双子叶植物和单子叶植物两个进化分支和种属进化特征,王晓庆等[12]和韦艳萍等[35]分别在牡丹和菊科植物中研究发现CCD家族成员基因进化树存在进化差异和种属特征,与本试验结果基本一致。
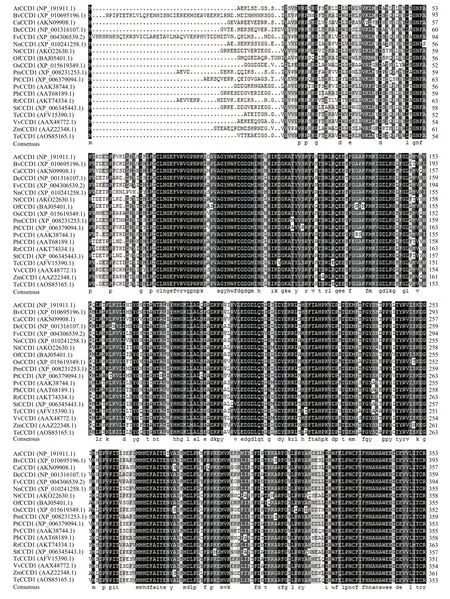

图4 万寿菊CCD1与其他植物CCD1蛋白质同源性比对Fig. 4 Alignment of the deduced CCD1 proteins of African marigold and other species
前人研究结果表明植物花色变化取决于所含色素种类及含量,万寿菊花色主要与所含类胡萝卜素种类及含量有关,而色素种类及含量又受合成与降解相关酶基因调控。色素种类及含量是决定花色形成的最主要的因子,如万寿菊、金盏菊、黄色蔷薇、百合等植物花色主要与类胡萝卜素种类及含量有关,花色范围从黄色至深橙色[36]。DEL VILLAR-MARTÍNEZ等[26]研究发现万寿菊舌状花初期花色为白绿色,含有叶绿素和类胡萝卜素,随花朵开放,花色变为淡黄色或黄色,舌状花仅有类胡萝卜素。本研究发现万寿菊‘Scarletade’品种舌状花4个时期颜色分别为黄绿色、淡黄色、深橙色和浅橙色,S3期颜色最深,林登贵[29]通过HPLC检测了万寿菊‘Scarletade’品种的叶黄素、α-胡萝卜素、β-胡萝卜素、玉米黄素、番茄红素 5种类胡萝卜素的总含量,在S1期最低,S2与S3期无显著差异,S4期显著低于S2与S3期,结合其结果,表明万寿菊‘Scarletade’品种花色与类胡萝卜素种类及含量有关。
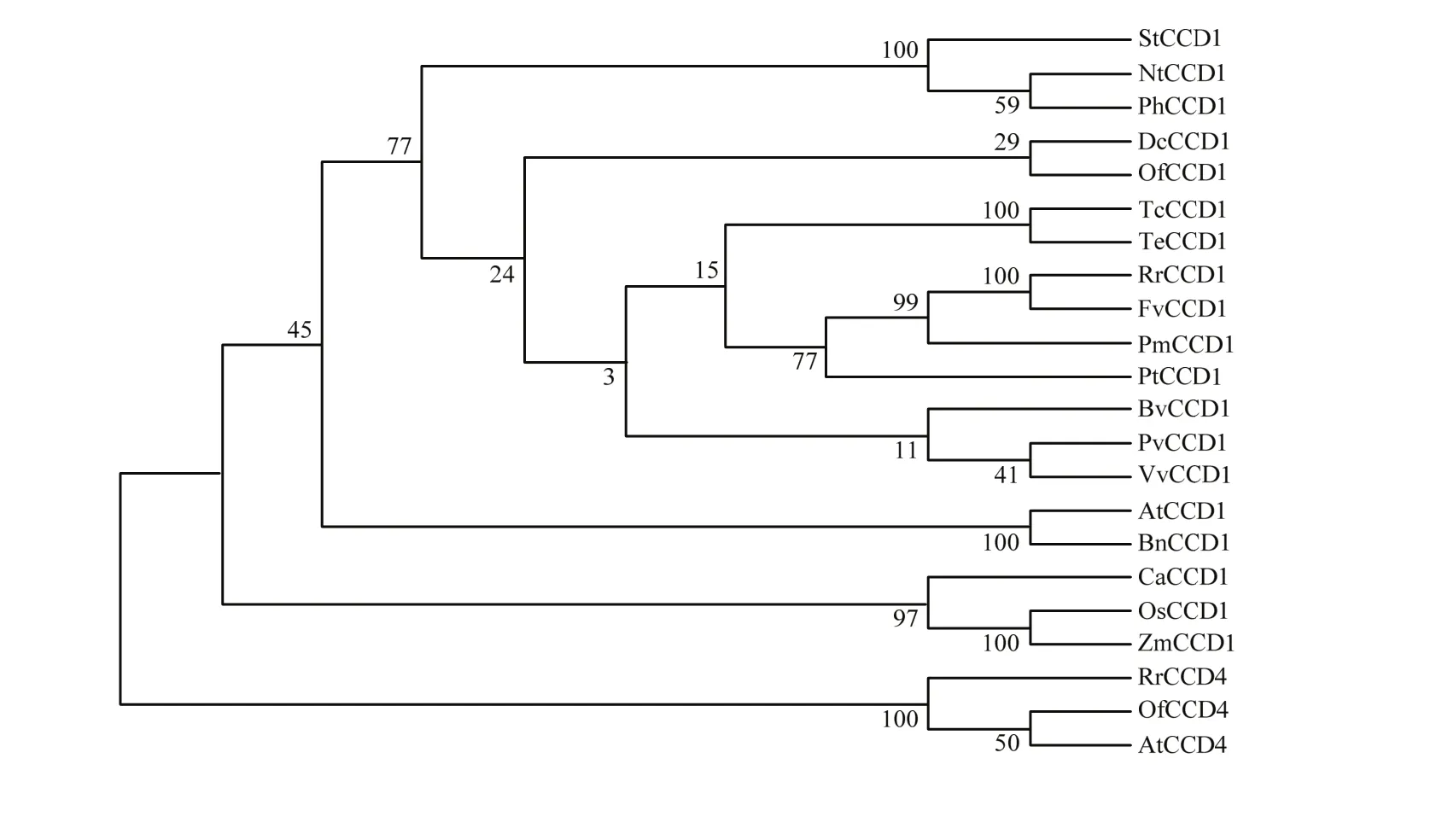
图5 CCD1系统进化分析Fig. 5 Phylogenetic analysis of CCD1
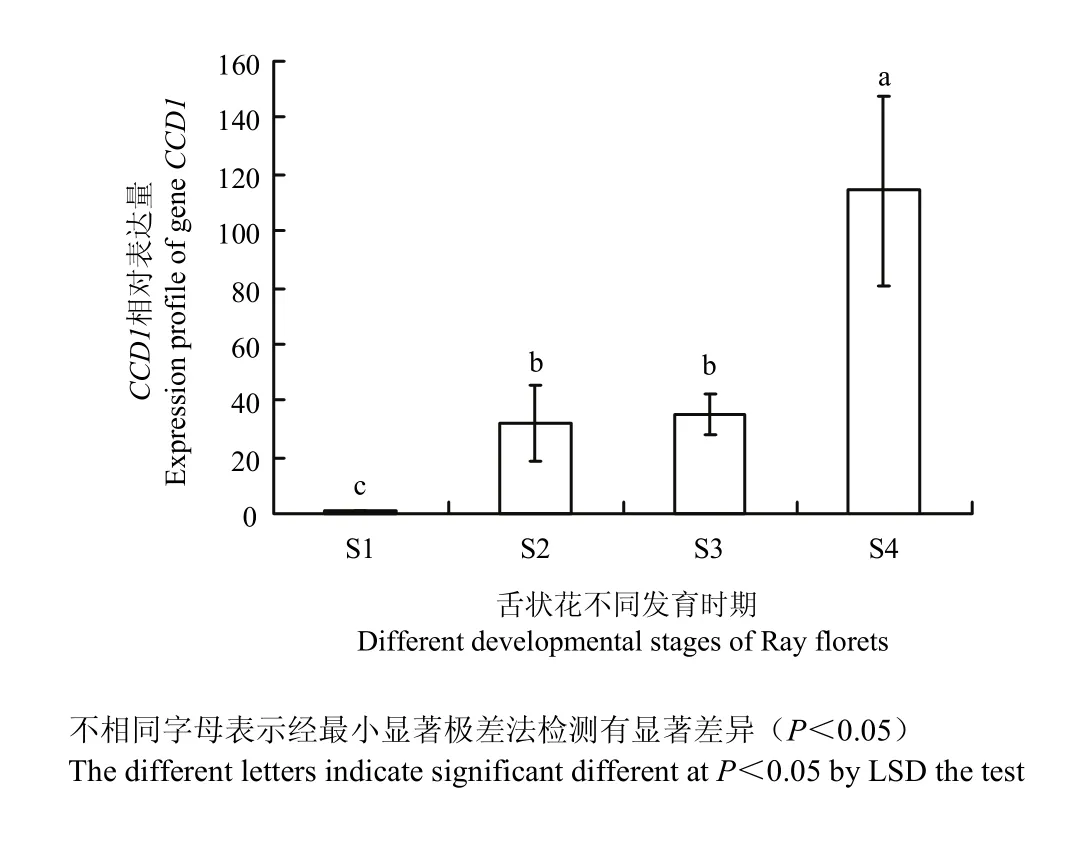
图 6 不同发育时期舌状花类胡萝卜素裂解双加氧酶TeCCD1相对表达量Fig. 6 Expression profile of CCD1 in ray floret of ‘Scarletade’at different developmental stages
本研究通过Real-time PCR检测TeCCD1在万寿菊舌状花4个不同发育时期表达量,结果表明TeCCD1 在4个不同发育时期均有表达,表达量随舌状花的开放逐渐升高。有研究标明5种类胡萝卜素总含量的变化趋势为‘低-高-低’[29],与本研究结果TeCCD1 S1期表达量较低,S4期较高的变化趋势基本一致。这可能是由于万寿菊S1期舌状花颜色偏绿色,其中含有较多的叶绿素而非类胡萝卜素,可被分解的底物少,TeCCD1表达水平较低;而其他 3个时期主要色素为类胡萝卜素,S4期TeCCD1高表达可能导致类胡萝卜素降解,表现为舌状花外观颜色变浅。在甜瓜、矮牵牛、番茄、枸杞、菌根和拟南芥等植物中的研究也发现 CCD1表达量与类胡萝卜素含量变化有关,周莉[13]研究发现桔色果肉甜瓜中检测到成熟期前类胡萝卜素总量持续升高,CCD1表达量也增加,但衰老期CCD1表达量最高,类胡萝卜素却明显下降,同时在白绿色果肉甜瓜中CCD1整体表达趋势均较低;SIMKIN等[24,37]及 TIAN等[25]发现在矮牵牛、番茄和枸杞中CCD1表达量降低可以减少类胡萝卜素降解为可挥发香气物质 β-紫罗兰酮;AULDRIDGE等[23]在拟南芥成熟种子中敲除 CCD1导致类胡萝卜素合成增加;FLOSS等[38]在Medicago truncatula菌根中RNA干涉CCD1,导致菌根内积累大量C27脱辅基类胡萝卜素,白色菌根变成黄橙色;IBDAH等[39]为验证CCD1功能,在Escherichia coli菌株中过表达CmCCD1,导致类胡萝卜素降解,橙黄色菌落变为浅白色;因此,推测万寿菊舌状花颜色变浅可能与TeCCD1表达量增加,导致类胡萝卜素降解有关。
万寿菊花色变化与基因表达量之间的关系有待进一步研究,舌状花颜色变浅可能是由于CCD1与一个或多个CCD家族基因或其他降解相关基因相互协同或拮抗作用。除了结构基因影响类胡萝卜素积累外,光照、外源激素及转录因子表达等也会影响类胡萝卜素含量[40-43],万寿菊舌状花不同发育时期花色变化与类胡萝卜素降解途径中起关键作用的酶基因及其他影响因子的作用机制有待进一步阐明。
4 结论
本研究克隆获得万寿菊舌状花类胡萝卜素裂解双加氧酶基因 TeCCD1;其编码的蛋白主要位于细胞质;蛋白质分析发现TeCCD1不含信号肽,有跨膜结构,为疏水性蛋白,属RPE65超家族,具类胡萝卜素裂解双加氧酶家族共同的保守结构域特点。同源性分析表明万寿菊CCD1为高度保守的基因;TeCCD1的进化基本符合植物分类学的进化规律,并具有明显的种属特征。TeCCD1在舌状花发育 4个时期的表达量差异显著,S4期达到最大值,与花色变化趋势一致,表明万寿菊舌状花颜色变浅可能与TeCCD1表达量增加,从而导致类胡萝卜素降解有关。
[1] DEINEKA V I, SOROKOPUDOV V N, DEINEKA L A,YU M T. Flowers of marigold (Tagetes) species as a source of xanthophylls. Pharmaceutical Chemistry Journal, 2007, 41(10): 540-542.
[2] TSAO R, WANG M C, DENG Z Y. Lutein: separation, antioxidant activity, and potential health benefits. Acs Symposium, 2007, 956: 352-372.
[3] STRINGHAM J M, BOVIER E R, WONG J C, HAMMOND B R. The influence of dietary lutein and zeaxanthin on visual performance. Journal of Food Science, 2010, 75(1): R24-R29.
[4] BERMAN J, ZORRILLA-LÓPEZ U, FARRÉG, C ZHU C F, SANDMANN G, TWYMAN R M, CAPELL T, CHRISTOU P. Nutritionally important carotenoids as consumer products. Phytochemistry Reviews, 2015, 14(5): 727-743.
[5] PORTER J W, LINCOLN R E. Lycopersicon selections containing a high content of carotenes and colourless polyenes. II. The mechanism of carotene biosynthesis. Archives of Biochemistry and Biophysics 1950, 27: 390-395.
[6] TAN B C, SCHWARTZ S H, ZEEVAART J A, MCCARTY D R. Genetic control of abscisic acid biosynthesis in maize. Proceedings of the National Academy of Sciences USA, 1997, 94(22): 12235-12240.
[7] YUAN H, ZHANG J X, NAGESWARAN D, LI L. Carotenoid metabolism and regulation in horticultural crops. Horticulture Research, 2015, 2: 15036.
[8] TAN B C, JOSEPH L M , DENG W T, LIU L J, LI Q B, KENNETH C, MCCARTY D R. Molecular characterization of the Arabidopsis 9-cis epoxycarotenoid dioxygenase gene family. The Plant Journal, 2003, 35(1): 44-56.
[9] 杨永霞, 牛志强, 张松涛, 崔红, 刘国顺. 烟草类胡萝卜素降解关键基因 CCD1的克隆与表达分析. 中国烟草学报, 2014, 20(1): 84-89. YANG Y X, NIU Z Q, ZHANG S T, CUI H, LIU G H. Cloning and expression of gene CCD1 critical to cleavage of tobacco carotenoid. Acta Tabacaria Sinica, 2014, 20(1): 84-89. (in Chinese)
[10] 梁乘榜. 枇杷果实品质分析及 CCD1基因克隆与表达研究[D]. 扬州: 扬州大学, 2013. LIANG C B. Quality analysis and CCD1 gene cloning and expression of loquat (Eriobotrya japonica) fruit [D]. Yangzhou: Yangzhou University, 2013. (in Chinese)
[11] OHMIYA A, KISHIMOTO S, AIDA R, YOSHIOKA S, SUMITOMO K. Carotenoid cleavage dioxygenase (CmCCD4a) contributes to white color formation in chrysanthemum petals. Plant Physiology, 2006, 142(3): 1193-1201.
[12] 王晓庆, 张超, 王彦杰, 董丽. 牡丹NCED基因的克隆和表达分析.园艺学报, 2012, 39(10): 2033-2044. WANG X Q, ZHANG C, WANG Y J, DONG L. Isolation and expression of 9-cis epoxycarotenoid dioxygenase gene in tree peony. Acta Horticulturae Sinica, 2012, 39(10): 2033-2044. (in Chinese)
[13] 周莉. 不同变种甜瓜果实香气, 颜色及相关的 CmCCD1基因表达的研究[D]. 天津: 天津大学, 2013. ZHOU L. Analysis of aroma components, color and related CmCCD1 expression in different varities of melon (Cucumis melo L.) fruit [D]. Tianjin: Tianjin University, 2013. (in Chinese)
[14] 徐庆华, 胡宝忠, 李凤兰, 金峰, 王多佳. 黄瓜 CsCCD7基因的克隆及表达研究. 热带亚热带植物学报, 2011, 19(4): 365-373. XU Q H, HU B Z, LI F L , JIN F, WANG D J. Cloning of CsCCD7 and its expression in Cucumis sativus L. Journal of Tropical and Subtropical Botany, 2011, 19(4): 365-373. (in Chinese)
[15] 曾文芳. 甜橙八氢番茄红素合成酶基因及其启动子的克隆与功能分析[D]. 武汉: 华中农业大学, 2012. ZENG W F. Identification and functional characterization of promoters and gens of phytoene synthase from sweet orange [D]. Wuhan: Huazhong Agricultural University, 2012. (in Chinese)
[16] RUBIO-MORAGA A, RAMBLA J L, FERNÁNDEZ-DE-CARMEN A, TRAPEROMOZOS A, AHRAZEM O, ORZAEZ D, GRANELL A, GOMEZ-GOMEZ L. New target carotenoids for CCD4 enzymes are revealed with the characterization of a novel stress-induced carotenoid cleavage dioxygenase gene from Crocus sativus. Plant MolecularBiology, 2014, 86: 555-569.
[17] ADAMI M, FRANCESCHI P D, BRANDI F, LIVERANI A, GIOVANNINI D, ROSATI C, DONDINI L, TARTARINI S. Identifying a carotenoid cleavage dioxygenase (ccd4) gene controlling yellow/white fruit flesh color of peach. Plant Molecular Biology Reporter, 2013, 31(5): 1166-1175.
[18] 温可睿. 葡萄类胡萝卜素裂解双加氧酶基因克隆及功能鉴定[D].哈尔滨: 东北林业大学, 2012. WEN K R. Clone and function identification of carotenoid cleavage dioxygenase from Vitis vinifera [D]. Haerbin: Northeast Forestry University, 2012. (in Chinese)
[19] HAN Y J, WANG X H, CHEN W C, DONG M F, YUAN W J, LIU X, SHANG F D. Differential expression of carotenoid-related genes determines diversified carotenoid coloration in flower petal of Osmanthus fragrans. Tree Genetics & Genomes, 2014, 10(2): 329-338.
[20] URESHINO K J, NAKAYAMA M, MIYAJIMA I. Contribution made by the carotenoid cleavage dioxygenase 4 gene to yellow colour fade in azalea petals. Euphytica, 2016, 207: 401-417.
[21] HAI N T L, MASUDA J I, MIYAJIMA I, NGUYEN T Q, MOJTAHEDI N, HIRAMATSU M, KIM J H, OKUBO H,. Involvement of carotenoid cleavage dioxygenase 4 gene in Tepal color change in Lilium brownii var. Colchesteri. Japanse of Society for Horticultural Science, 2012, 81(4): 366-373.
[22] AULDRIDGE M E, MCCARTY D R, KLEE H J. Plant carotenoid cleavage oxygenases and their apocarotenoid products. Current Opinion in Plant Biology, 2006, 9(3): 315-321.
[23] AULDRIDGE M E, BLOCK A, VOGEL J T, DABNEYSMITH C, MILA I, BOUZAYEN M, MAGALLANES-LUNDBACK M, DELLAPENNA D, MCCARTY D R, KLEE H J. Characterization of three members of the Arabidopsis carotenoid cleavage dioxygenase family demonstrates the divergent roles of this multifunctional enzyme family. The Plant Journal, 2006(45): 982-993.
[24] SIMKIN A J, UNDERWOOD B A, AULDRIDGE M, LOUCAS H M, SHIBUYA K, SCHMELZ E, CLARK D G, KLEE H J. Circadian regulation of the PhCCD1 carotenoid cleavage dioxygenase controls emission of β-Ionone a fragrance volatile of petunia flowers. Plant Physiology, 2004, 136(3): 3504-3514.
[25] TIAN X W, JI J, WANG G, JIN C, GUAN C F, WU G X. Molecular cloning and characterization of a novel carotenoid cleavage dioxygenase 1 from Lycium chinense. Biotechnology and Applied Biochemistry, 2015, 62(6): 772-779.
[26] DEL VILLAR-MARTÍNEZ A A, GARCÍASAUCEDO P A, CARABEZTREJO A, CRUZHERNÁNDEZ A, PAREDESLOPEZA O. Carotenogenic gene expression and ultrastructural changes during development in marigold. Journal of Plant Physiology, 2005, 162(9): 1046-1056.
[27] MOEHS C P, TIAN L, OSTERYOUNG K W, DELLAPENNA D. Analysis of carotenoid biosynthetic gene expression during marigold petal development. Plant Molecular Biology, 2001, 45(3): 281-293.
[28] 王国兰, 程曦, 任君安, 乔伟, 罗昌, 吴忠义, 黄丛林, 康宗利, 张秀海. 色素万寿菊中叶黄素合成相关基因的表达分析. 分子植物育种, 2012, 10(1): 67-72. WANG G L, CHENG X, REN J A, QIAO W, LUO C, WU Z Y, HUANG C L, KANG Z L, ZHANG X H. Expression analysis of lutein biosynthetic genes in pigment marigold. Molecular Plant Breeding, 2012, 10(1): 67-72. (in Chinese)
[29] 林登贵. 万寿菊花色差异形成机理分析[D]. 上海: 上海交通大学, 2014. LIN D G. Dissecting the mechanism of marigold flowers color formation [D]. Shanghai: Shanghai Jiao Tong University, 2014. (in Chinese)
[30] 张嫔. 万寿菊属植物染色体核型分析及万寿菊 Psy基因遗传转化体系影响因素的研究[D]. 上海: 上海交通大学, 2012. ZHANG P. The study of karyotype on genus Tagetes L. and factors in the genetic transformation system of Psy gene for Tagetes erecta L. [D]. Shanghai: Shanghai Jiao Tong University, 2012. (in Chinese)
[31] LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-delta delta C(T)) method. Methods, 2001, 25: 402-408.
[32] 彭公信. 基因进化保守性和变异性比较分析[D]. 石家庄: 河北科技大学, 2012. PENG G X. Genetic evolution conservative variability and comparative analysis [D]. Shijiazhuang: Hebei University of Science & Technology, 2012. (in Chinese)
[33] 赵军林, 于喜艳, 王秀峰. 橙色果肉甜瓜β-胡萝卜素积累的分子机理. 山东农业科学, 2014, 46(5): 7-11, 30. ZHAO J L, YU X Y, WANG X F. Molecular mechanism of β-carotene accumulation in orange-fleshed Muskmelon. Shangdong Agricultural Sciences, 2014, 46(5): 7-11, 30. (in Chinese)
[34] 高军平. 烟草类胡萝卜素加氧酶基因的克隆与功能研究[D]. 重庆:重庆大学, 2013. GAO J P. Clonging and functional analysis of carotenoid cleavage dioxygenase gene in Nicotiana tabacum [D]. Chongqing: Chongqing University, 2013. (in Chinese)
[35] 韦艳萍, 刘云飞, 许桂梅, 万红建, 李志邈, 叶青静, 王荣青, 阮美颖, 姚祝平, 周国治, 杨悦俭. 菊分支 NCED 酶分子特征及进化分析. 核农学报, 2015, 29(2): 278-289. WEI Y P, LIU Y F, XU G M, WAN H J, LI Z M, YE Q J, WANG R Q, RUAN M Y, YAO Z P, ZHOU G Z, YANG Y J. Molecular characteristics and phylogenetic analysis of NCED enzyme in Chrysanthemum. Journal of Nuclear Agricultural Sciences, 2015, 29(2): 278-289. (in Chinese)
[36] 安田齐, 傅玉兰, 译. 花色的生理生物化学. 北京: 中国林业出版社, 1989: 310-318. AN T Q, FU Y L. Physiology and Biochemistry of Flower Color. Beijing: China Forestry Publishing House, 1989: 310-318. (in Chinese)
[37] SIMKIN A J, SCHWARTZ S H, AULDRIDGE M, TAYLOR M G, KLEE H J. The tomato carotenoid cleavage dioxygenase 1 genes contribute to the formation of the flavor volatiles β-ionone, pseudoionone and gerany lacetone. Plant Journal, 2004, 40: 882-892.
[38] FLOSS D S, SCHLIEMANN W, SCHMIDT J, STRACK D, WALTER M H. RNA interference-mediated repression of MtCCD1 in mycorrhizal roots of Medicago truncatula causes accumulation of C27 apocarotenoids, shedding light on the functional role of CCD1. Plant Physiology, 2008, 148(3): 1267-1282.
[39] IBDAH M, AZULAY Y, PORTNOY V, WASSERMAN B, BAR E, MEIR A, BURGER Y, HIRSCHBERG J, SCHAFFER A A, KATZIR N, TADMOR Y, LEWINSOHN E. Functional characterization of CmCCD1, a carotenoid cleavage dioxygenase from melon. Phytochemistry, 2006, 67: 1579-1589.
[40] LEE W L, HUANG J Z, CHEN L C, TSAI C C, CHEN F C. Developmental and LED light source modulation of carotenogenic gene expression in oncidium gower ramsey flowers. Plant Molecular Biology Reporter, 2013, 31(6): 1433-1445.
[41] ALFEREZ F, POZO L V, ROUSEFF R R, BURNS J K. Modification of carotenoid levels by abscission agents and expression of carotenoid biosynthetic genes in ‘Valencia’ sweet orange. Journal of Agricultural and Food Chemistry, 2013, 61(12): 3082-3089.
[42] BU J W, NI Z D, AISIKAER G, JIANG Z H, KHAN Z U, MOU W S, YING T J. Postharvest ultraviolet-c irradiation suppressed Psy1 and Lcy-beta expression and altered color phenotype in tomato (Solanum lycopersicum) fruit. Postharvest Biology and Technology, 2014, 89: 1-6.
[43] KARLOVA R, ROSIN F M , BUSSCHER-LANGE J, PARAPUNOVA V , DO P T, FERNIE A R, FRASER P D, BAXTER C, ANGENENT G,DE MAAGD R A . Transcriptome and metabolite profiling show that APETALA2a is a major regulator of tomato fruit ripening. Plant Cell, 2011, 23(3): 923-941.
(责任编辑 赵伶俐)
Cloning and Expression Analysis of Carotenoid Cleavage Dioxygenase 1 (CCD1) Gene in Tagetes erecta L.
LIU XiaoCong1, ZENG Li1,2, LIU GuoFeng3, PENG YongZheng1, TAO YiWei1, ZHANG YaoYue1, WANG MengRu1
(1School of Agriculture and Biology, Shanghai Jiaotong University, Shanghai 200240;2Key Laboratory of Urban Agriculture (South) Ministry of Agriculture, Shanghai 200240;3College of Horticulture and Forestry Science, Huazhong Agricultural University/Key Laboratory of Horticultural Plant Biology of Ministry of Education, Wuhan 430070)
【Objective】Carotenoid Cleavage Dioxygenase 1 gene of Tagetes erecta L. ‘Scarletade’ (TeCCD1) was cloned for bioinformatics and gene expression analysis, which can help clarifying its biological functions in carotenoid degradation pathway and providing a theoretical foundation to further clarify the mechanism of African marigold flower color formation.【Method】According to the transcriptome of African marigold flower bud, the full-length cDNA of TeCCD1 had been obtained, and gene expression profile of ray florets at developmental stages of closed bud, semi-open bud, open flower and fully open flower was studied by Real-time PCR.【Result】The full-length sequence of CCD1 cDNA obtained from African marigold is 1 746 bp (GenBank accession number: KX557488), with a coding region length of 1 626 bp, putatively encoding 541 amino acids. Protein analysis indicated that TeCCD1 is an unstable protein and has no signal peptide, which belongs to the RPE65 superfamily (GenBank accession number is PF03055) having the same conserved domain of CCD family, and it is mainly located in the cytoplasm. CCD1nucleic acid sequence of African marigold is 89% homologous to that of Pyrethrum. Amino acid sequence analysis suggested that CCD1 of African marigold is 93% homologous to that of Pyrethrum, and 75%-83% homologous to that of 19 different species, indicating that TeCCD1 is highly conserved gene. Phylogenetic analysis showed that the evolution of TeCCD1 is basically in accordance with the evolution law of plant taxonomy and has obvious characteristics of species, which has a closest relationship with that of the species in Compositae. The results of Real-time PCR demonstrated that expression of TeCCD1 increased along with the development of ray florets and reached the maximum value at S4 stage.【Conclusion】CCD1 homolog was cloned in Tagetes erecta L. ‘Scarletade’ identified to be a typical member of the CCD family, which is a highly conserved gene located in the cytoplasm. The color fading of ray florets during the late development phase is possibly caused by the increase of expression of TeCCD1, which contributes to a decrease in carotenoid content.
Tagetes erecta L.; carotenoid cleavage dioxygenase 1 (CCD1); flower color; gene expression
2016-10-19;接受日期:2017-01-18
教育部“创新团队发展计划”(IRT13065)
联系方式:刘晓丛,E-mail:lxc05012013@163.com。通信作者曾丽,E-mail:zljs@sjtu.edu.cn。通信作者刘国锋,E-mail:gfliu@mail.hzau.edu.cn
