巴西橡胶树PEPRK基因家族成员的鉴定和表达分析
2017-05-30肖小虎隋金蕾戚继艳方永军周斌辉唐朝荣
肖小虎 隋金蕾 戚继艳 方永军 周斌辉 唐朝荣

摘 要 PEP羧化酶激酶相关激酶(PEP carboxylase kinase-related kinases,PEPRKs)是CDPK-SnRK超家族的一员,目前对其生理功能尚不清楚。本研究以拟南芥和杨树的PEPRK序列作为探针,对橡胶树和其他5种植物(木薯、水稻、杨树、蓖麻和拟南芥)的基因组和转录组数据进行全面搜索,共鉴定得到25个PEPRK基因家族成员,其中包括5个橡胶树PEPRK基因,命名为HbPEPRK1-5。序列分析发现橡胶树PEPRK和其他植物类似,在序列中部含有一个相对比较保守的激酶结构域,而两端的同源性相对较低。在基因结构和进化方面,PEPRK主要分为2个大的分支,相同分支的成员在结构域和内含子分布上更为相似,其中橡胶树和木薯PEPRK在进化上具有较近的亲缘关系。在基因的表达方面,HbPEPRK1主要在稳定期的叶片中表达,HbPEPRK2和HbPEPRK5在树叶、树皮和胶乳中均有表达,HbPEPRK4主要在种子中表达;另外,研究发现橡胶树PEPRK家族成员的表达还与叶片发育、乙烯利刺激、真菌侵染和低温胁迫具有一定的相关性。
关键词 巴西橡胶树;PEPRK;基因家族;结构与进化;表达分析
中图分类号 S794.1 文献标识码 A
Identification and Expression of PEPRK Genes
from Hevea brasliensis
XIAO Xiaohu1, SUI Jinlei1,2, QI Jiyan1, FANG Yongjun1,
ZHOU Binhui1,2, TANG Chaorong1*
1 Rubber Research Institute, CATAS, Danzhou, Hainan 571737, China
2 College of Agronomy, Hainan University, Haikou, Hainan 570228, China
Abstract Phosphoenolpyruvate carboxylase kinase-related kinase is a member of CDPK-SnRK superfamily. Little is known about its functions. By searching the transcriptome and genome databases, we have identified 25 PEPRK genes from Hevea brasliensis and five other plant species, including five members from H. brasliensis, which were named HbPEPRK1 to 5. Sequence analysis revealed that all PEPRK genes comprised one protein kinase domain. The plant PEPRK clustered into two major groups in phylogenetic tree, the members in the same group shared a similar introns and domains distribution. H. brasliensis PEPRKs were more closely related to PEPRKs of Manihot esculenta. The results of expression analysis indicated that HbPEPRK1 was mainly expressed in mature leaves, HbPEPRK2 and HbPEPRK5 were expressed in leaves, bark and latex, HbPEPRK4 was mainly expressed in seeds. In addition, the expression of H. brasliensis PEPRK genes was related to leaf development, ethephon stimulation, C. cassiicola infection and low temperature.
Key words Hevea brasiliensis; PEPRK; gene family; structure and evolution; expression analysis
doi 10.3969/j.issn.1000-2561.2017.01.013
在植物中,CDPK-SnRK超家族包含7种丝氨酸-苏氨酸蛋白蛋白激酶:钙依赖蛋白激酶(Ca2+-dependent protein kinases,CDPKs),CDPK相关蛋白激酶(CDPK-related kinases,CRKs),磷酸烯醇式丙酮酸羧激酶(phosphoenolpyruvate carboxylase kinases,PPCKs),PEP羧化酶激酶相关激酶(PEP carboxylase kinase-related kinases,PEPRKs),钙调素依赖性蛋白激酶(calmodulin-dependent protein kinases,CaMKs),钙/钙调素依赖型蛋白激酶(calcium and calmodulin-dependent protein kinases,CCaMKs)和SnRKs蛋白激酶[1]。PEPRKs是与CDPK高度同源和密切相关的一类丝/苏蛋白激酶,仅存在于植物体内,关于其功能研究报道较少[1-3]。在橡胶树中,李超燕等[4]根据EST序列利用RACE从橡胶树中克隆得到一個PEPRK基因,命名为HbPEPRK1,并利用荧光定量PCR技术对其表达进行了初步分析。本研究以已发表[1,3]PEPRK蛋白氨基酸序列为探针,对橡胶树、木薯、蓖麻、拟南芥、杨树和水稻6种植物的全基因组数据进行全面搜索,鉴定得到25个PEPRK基因家族成员,其中橡胶树中有5个。笔者们利用相关软件和数据库,对橡胶树PEPRK基因家族成员的基因结构、进化和表达水平进行了系统分析。研究结果将有助于本课题组深入了解橡胶树PEPRK的生物学功能。
1 材料与方法
1.1 材料
本实验室Solexa测序所用的巴西橡胶树(Hevea brasiliensis)热研7-33-97材料,除了根来自于组培苗外,其他组织(包括胶乳、树皮、树叶、种子、雌花和雄花)均来自中国热带农业科学院试验场3队的正常割胶橡胶树(开割2 a以上),根来自于中国热带农业科学院橡胶研究所种质资源圃华玉伟老师赠送的热研7-33-9组培苗;不同发育时期的橡胶树叶片来自于中国热带农业科学院橡胶所种质资源圃,为一年生的热研7-33-97嫁接苗;乙烯利处理的材料主要来自于海南省儋州市中国热带农业科学院试验场3队,品系为热研7-33-9,正常开割树(3天一刀,不涂乙烯利刺激),用1.5%乙烯利在不同时间处理橡胶树,相应4个时间点处理为0、3、12、24 h。
网上测序数据来自NCBI SRA数据库(http://www.ncbi.nlm.nih.gov/sra),包括不同组织(PRJNA201084),真菌侵染(Corynespora cassiicola tolerance,PRJNA179126)、高低温干旱胁迫(PRJNA182078)和乙烯利处理(PRJNA182079),详细信息参见数据库中实验说明。
1.2 方法
1.2.1 基因家族成员的鉴定与序列分析 为了全面鉴定橡胶树、拟南芥、杨树、水稻、木薯和蓖麻的PEPRK基因家族成员,本课题组从NCBI(http://www.ncbi.nlm.nih.gov/)和phytozome(https://phytozome.jgi.doe.gov/pz/portal.html)数据库下载了5种植物的基因组和转录组数据。以拟南芥和杨树的PEPRK序列为探针,对6种植物的基因组和转录组进行搜索,得到候选PEPRK基因家族成员,利用InterProScan(http://www.ebi.ac.uk/Tools/pfa/iprscan/)对候选基因的结构域进行分析。再利用EMBOSS数据库的在线软件Pepstats(http://www.ebi.ac.uk/Tools/seqstats/emboss_ pepstats/)对基因的等电点和分子量进行批量分析。
1.2.2 基因结构与进化分析 利用在线软件GSDS2.0(http://gsds.cbi.pku.edu.cn/)对橡胶树和其他5种植物PEPRK基因家族成员的外显子/内含子组织结构进行分析。利用MEGA 6.0软件对橡胶树和其他5种植物的PEPRK氨基酸序列一起构建系统发育树,采用Neighbor-Joining方法进行分子系统学分析,进行1 000次bootstrap统计学检验。
1.2.3 基因的表达模式分析 利用本实验室和NCBI的solexa转录组数据对橡胶树PEPRK基因家族成员的表达模式进行分析[5]。首先,将NCBI的SRA数据库(http://www.ncbi.nlm.nih.gov/sra/)中橡胶树相关的solexa转录组数据下载到本地服务器,去除低质量序列,利用程序RSEM进行表达分析[6]。
2 结果与分析
2.1 PEPRK基因的鉴定与序列分析
通过本地BLAST搜索各植物转录组和基因组数据,从橡胶树、木薯、蓖麻、拟南芥、杨树和水稻6种植物中共鉴定得到25个PEPRK(表1),其中橡胶树5个,命名为HbPEPRK1-5。通过序列分析发现,橡胶树PEPRK蛋白为371~522个氨基酸,分子量为42~57 ku,等电点在5.0~8.8之间,内含子数目为2~3个。氨基酸序列多序列比对发现,橡胶树PEPRK蛋白都含有蛋白激酶保守结构域(图1)。
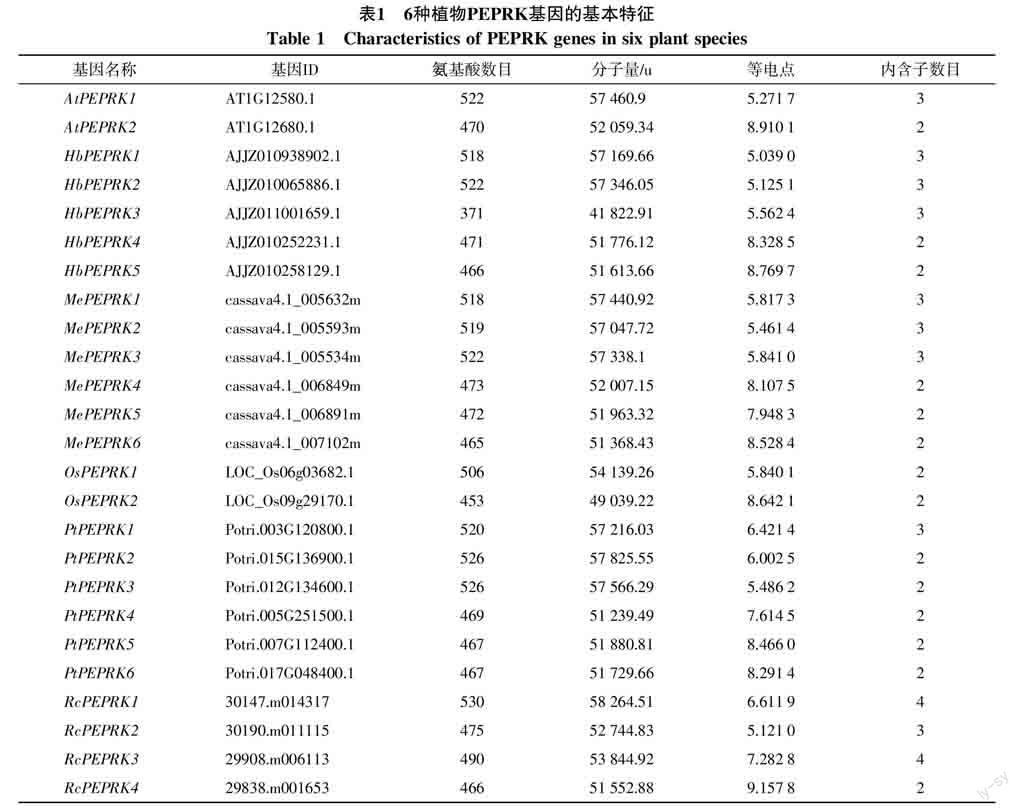
2.2 基因结构和进化分析
基因外显子/内含子组织结构和进化分析发现(图2),6种植物PPCK家族成员在进化上分为2个较大的分支。在第一个分支,除了RcPEPRK1含有4个内含子,OsPEPRK1、PtPEPRK2和PtPEPRK3含有2个内含子,其他成员均只含有3个内含子;第二个分支中,除了RcPEPRK3含有4个内含子,其他成员均只含有2个内含子,与第一个分支相比第二个分支各成员第一个外显子要长,蛋白激酶结构域的位置更居中一些。在进化上,橡胶树和木薯、蓖麻的PEPRK聚在一起,具有较近的亲缘关系。另外,部分家族成员成对聚在一起,如HbPEPRK2和HbPEPRK3、PtPEPRK2和PtPEPRK3、PtPEPRK5和PtPEPRK6,说明这些成员之间具有更高的同源性,但部分基因对的表达却存在明显差异,如HbPEPRK2和HbPEPRK3(图3和图4)。
2.3 橡胶树PEPRK基因家族成员的表达分析
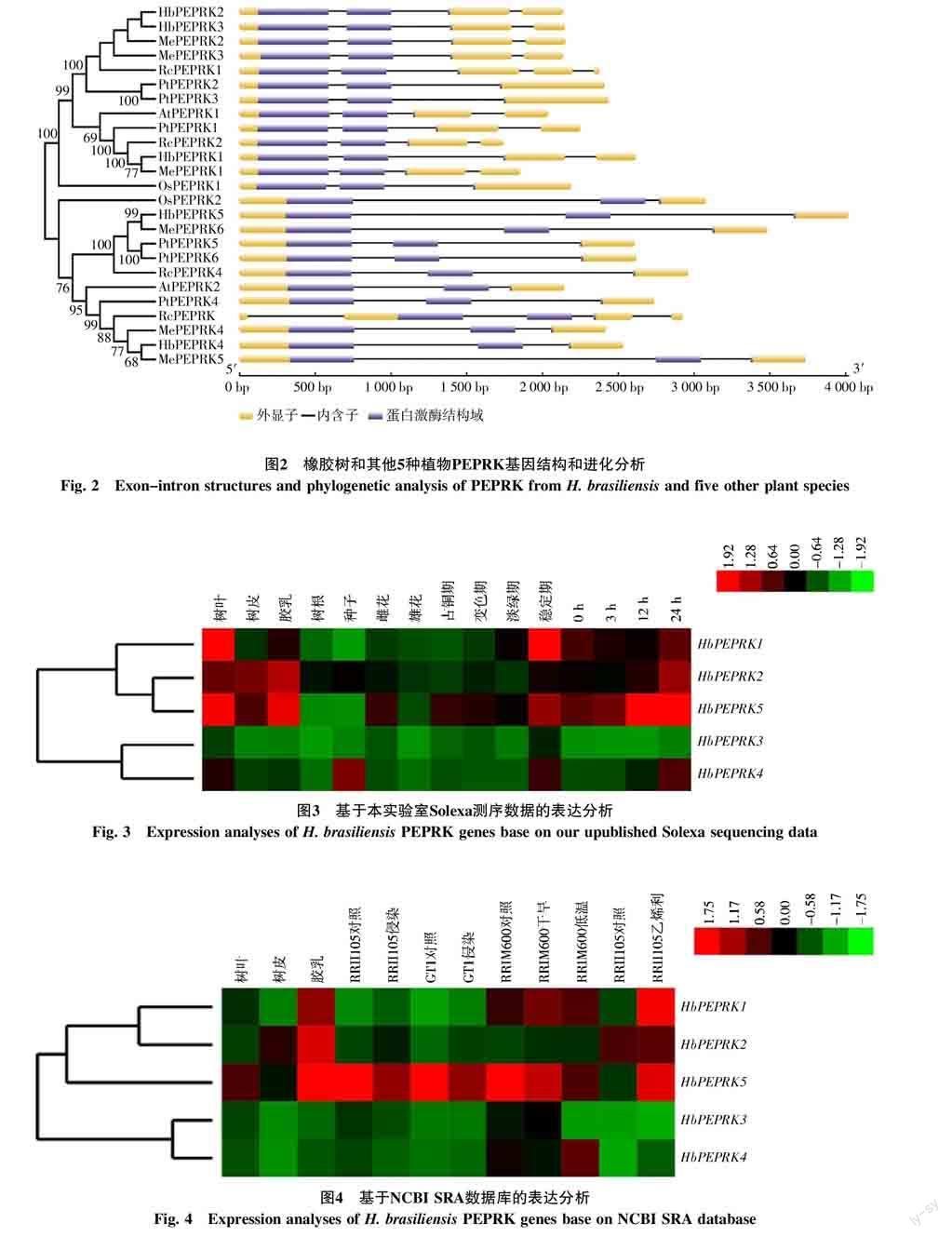
本实验室Solexa转录组数据的分析结果表明(图3),HbPEPRK1主要在叶片和胶乳中表达,并且在叶片发育过程中表达丰度明显上升,稳定期达到最高;HbPEPRK2主要在树叶、树皮和胶乳中均有表达,在其他组织中的表达丰度偏低,乙烯利刺激后表达丰度明显升高,24 h后达到最高;HbPEPRK5主要在叶片和胶乳中表达,在树皮和花中也有表达,但表达丰度相对较低,乙烯利刺激后表达丰度明显升高;另外HbPEPRK4主要在种子中表达,在稳定期叶片中也有表达;HbPEPRK3在各组织中表达都较低或不表达。
本课题组还利用NCBI的SRA数据库中的转录组数据对橡胶树PEPRK的表达进行了分析(图4)。分析结果表明,HbPEPRK1主要在胶乳和叶片中表達,干旱和乙烯利刺激都会不同程度的诱导HbPEPRK1表达;HbPEPRK2主要在胶乳中表达,C. cassiicola侵染会一定程度上诱导其表达;HbPEPRK5主要在胶乳中表达,C. cassiicola侵染和低温处理会降低其表达,而乙烯利刺激会诱导其表达;另外HbPEPRK3在各组织中表达都比较低,低温处理会降低其表达;HbPEPRK4同样在各组织中表达都较低,而低温和乙烯利处理会诱导其表达。
从以上2部分实验数据的结果来看,部分家族成员的表达具有普遍性,如HbPEPRK2在各组织都表达;有些成员在个各组织中表达都很低或不表达,如HbPEPRK3;还有一些成员组织特异性表达,如HbPEPRK4主要在种子中表达,在其他组织的表达都很低。另外,通过2部分数据的比较来看,大部分数据结果是一致的,这说明本实验结果的可靠性,也有些结果有些差异,如HbPEPRK1在树叶和胶乳中的表达,2部分数据结果存在一定的差异,这可能和材料的种类或采集时间有一定的关系,需要后续实验进一步验证。
3 讨论
PEPRK是CDPK-SnRK超家族的一員,与CDPK有较高的同源性,但对其功能的研究相对CDPK来说要少很多。已有研究表明,CDPK在植物生长发育和逆境胁迫应答等方面起着重要作用[7-10],PEPRK是否具有类似功能目前尚不清楚。在橡胶树中,李超燕等[4]利用RACE克隆得到一个PEPRK基因,通过比对发现该基因和本研究的HbPEPRK5为同一基因。本研究利用高通量测序数据表达分析的结果和李超燕等[4]的结果基本一致,均为在胶乳中表达丰度较高,但乙烯利刺激的结果却稍有不同,本研究中在0~24 h HbPEPRK5的表达有明显的递增趋势,而李超燕等[4]的结果是在3 h上升到最高然后下降。在本研究中,HbPEPRK1主要在稳定期的叶片中表达,HbPEPRK2和HbPEPRK5在树叶、树皮和胶乳中均有表达,HbPEPRK4主要在种子中表达;乙烯利刺激刺激后所有家族成员的表达都表现出一定的上升趋势,说明橡胶树PEPRK基因可能参与橡胶树乙烯信号传导相关反应。另外,研究发现橡胶树PEPRK还与叶片发育、真菌侵染和低温胁迫具有一定的相关性。本实验室的高通量数据已成功应用于橡胶树基因组数据分析以及橡胶树蔗糖合成酶基因家族相关分析,相关结果发表在Nature Plants[11]和Febs Journal[5],这进一步证实了本文数据结果的可靠性。本研究首次从橡胶树、木薯和蓖麻全基因组中鉴定出PEPRK所有基因家族成员,并利用相关软件和数据库对橡胶树PEPRK各家族成员的基因结构、进化和表达模式进行了全面系统的分析,研究结果将为进一步研究PEPRK在橡胶树生长发育和逆境胁迫应答等方面的功能打下基础。
参考文献
[1] Hrabak E M, Chan C W M, Michael G, et al. The Arabidopsis CDPK-SnRK superfamily of protein kinases[J]. Plant Physiology, 2003, 132(2): 666-680.
[2] Singh U M, Chandra M, Shankhdhar S C, et al. Transcriptome wide identification and validation of calcium sensor gene family in the developing spikes of finger millet genotypes for elucidating its role in grain calcium accumulation[J]. Plos One, 2014, 9(8): e103 963-103 963.
[3] Zuo R, Hu R B, Chai G H, et al. Genome-wide identification, classification, and expression analysis of CDPK and its closely related gene families in poplar(Populus trichocarpa)[J]. Molecular Biology Reports, 2012, 40(3): 2 645-2 662.
[4] 李超燕, 朱家红, 徐 靖, 等. 巴西橡胶树HbPEPRK1的克隆和表达分析[J]. 基因组学与应用生物学, 2015, 34(10): 2 181-2 186.
[5] Xiao X H, Tang C R, Fang Y J, et al. Structure and expression profile of the sucrose synthase gene family in the rubber tree: indicative of roles in stress response and sucrose utilization in the laticifers[J]. Febs Journal, 2014, 281(1): 291-305.
[6] Li B, Dewey C N. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome[J]. BMC Bioinformatics, 2011, 12(1): 93-99.
[7] Kong X P, Lv W, Jiang S S, et al. Genome-wide identification and expression analysis of calcium-dependent protein kinase in maize[J]. Bmc Genomics, 2013, 14(1): 447-450.
[8] Ray S, Agarwal P, Arora R, et al. Expression analysis of calcium-dependent protein kinase gene family during reproductive development and abiotic stress conditions in rice (Oryza sativa L. ssp. indica)[J]. Molecular Genetics & Genomics, 2007, 278(5): 493-505.
[9] Zhang H F, Liu W Z, Zhang Y P, et al. Identification, expression and interaction analyses of calcium-dependent protein kinase (CPK)genes in canola(Brassica napus L.)[J]. Bmc Genomics, 2014, 15(1): 211.
[10] Zuo R, Hu R B, Chai G H, et al. Genome-wide identification, classification, and expression analysis of CDPK and its closely related gene families in poplar(Populus trichocarpa)[J]. Molecular Biology Reports, 2012, 40(3): 2 645-2 662.
[11] Tang C R, Meng Y, Fang Y J, et al. The rubber tree genome reveals new insights into rubber production and species adaptation[J]. Nature Plants, 2016, 2(6): 16 073.
