巴西蕉AP2/ERF超家族全基因组分析
2017-05-30侯晓婉胡伟徐碧玉金志强张鲁斌鹿志伟
侯晓婉 胡伟 徐碧玉 金志强 张鲁斌 鹿志伟
摘 要 香蕉作为热带和亚热带发展中国家最重要的粮食和经济作物,其产量和质量受到低温、干旱、高盐等非生物逆境胁迫的严重影响。基于香蕉A基因组测序数据,利用生物信息学技术和方法,对巴西蕉中调控植物非生物逆境胁迫应答方面发挥重要作用的AP2/ERF超家族基因全基因组进行系统分析。结果表明:共获得119个巴西蕉AP2/ERF超家族基因,并将其划分成ERF(100)、RAV(15)和soloist(4)家族,其中ERF家族又被划分为10个亚家族,每个亚家族基因具有相似的保守motif和基因结构。此研究结果不仅为巴西蕉应答非生物逆境脅迫下AP2/ERF超家族基因功能和响应机制的研究、香蕉品种抗逆改良奠定理论基础,而且也为不同物种的AP2/ERF超家族基因的系统发育和进化研究提供方法和理论依据。
关键词 香蕉;AP2/ERF;全基因组分析
中图分类号 S668.1 文献标识码 A
Abstract Banana(Musaceae, Musa)is one of the most important foods and commercial crops in tropical and subtropical developing countries. The abiotic stresses, such as freezing, drought and salt, influence heavily the yield and quality of banana. A genome-wide analysis and expression profiles analysis on AP2/ERF super gene family of banana, which play an important role in responding to abiotic stresses, were conducted based on banana A genome sequencing. The main results showed that 119 AP2/ERF genes from banana A genome were divided into three families of ERF(100), RAV(15)and soloist(4). ERF family were classified again into ten subfamilies which were named fromⅠto Ⅹ. The same subfamily had same intro-exon structure and conserved motif domain. Taken together, the methods and theories in this paper could be used to study the development and evolution of gene coming AP2/ERF super family in other species. This study could identify some abiotic stress-responsive candidate MaERF genes, which would lay a solid foundation for genetic improvement of banana cultivars in the future.
Key words banana(Musaceae, Musa); AP2/ERF; genome-wide analysis
doi 10.3969/j.issn.1000-2561.2017.02.019
1961年Simmonds等[1]将含尖叶蕉性状的基因称为A基因,将含有长梗蕉性状的基因称为B基因,按其基因型,并参照染色体数将由原始的野生尖叶蕉(Musa acuminate Colla)和长梗蕉(Musa balibisiana)种内或者种间杂交之后逐渐进化而成的香蕉品种分为AA、AAA、AAAA、AAB、AAAB、AABB、AB、ABBBB、BBB等组,而主要香蕉栽培品种巴西蕉、大蕉和粉蕉均为三倍体,其基因型分别为AAA、ABB和AAB[1-3],特别是巴西蕉,因其具有较高的商业价值,很受收购商和蕉农的欢迎,普遍被大面积种植。然而香蕉是多年生常绿大型草本单子叶植物,对低温、干旱、高盐等非生物逆境胁迫异常敏感,非生物逆境胁迫的发生严重影响其产量和质量。
AP2/ERF(APETELLA2/Ethylene Responsive Element Binding Factor)超家族是植物最大的转录因子之一,其至少包含1个AP2/ERF结构域。基于AP2/ERF结构域的数目和是否出现其他DNA结合区域,将其划分为AP2、ERF、RAV和soloist家族。其中AP2家族基因包含2个重复的AP2/ERF结构域;ERF家族基因仅包含1个AP2/ERF结构域;RAV家族基因包含1个AP2/ERF结构域和1个B3 DNA结合区域[4-5]。AP2/ERF超家族在植株花器官的发育、细胞增生、次生代谢、激素信号响应及生物和非生物胁迫应答中发挥重要的作用。Wan等[6]研究报导花生AhERF019在转基因拟南芥中过表达的增强了植株对干旱、高温和高盐的耐受性。荷花LcERF054和麻疯树JcERF1的表达被高盐诱导,增强了转基因植株对高盐的耐受性[7-8]。芝麻的AP2si16和水稻的OsERF71改善了芝麻和水稻对干旱的耐受性[9-10]。ERF家族基因在植株响应外界生物和非生物胁迫方面分别发挥着不同的功能。前人对拟南芥中ERF各家族基因的功能进行研究,结果发现ERF家族的GroupIII亚家族基因在植株响应低温、高盐和干旱胁迫方面发挥重要作用;GroupVII和GroupIX亚家族基因在植株对病害的耐受性方面发挥重要作用[4,11]。
对植物基因家族的全基因组进行分析,识别出的分类组和假定功能motif将对研究每个家族内基因的生物功能有很大作用。自2006~2014年,已先后对拟南芥(Arabidopsis thaliana)、水稻(Oryza sativa L.)、大豆(Giycinemax L.)、玉米(Zea mays L.)、杨树(Populus trichocarpa)、 高粱(Sorghum bicolor L.)、葡萄(Vitis vinifera L.)、苹果(Malus domestica)、中国大白菜(Brassica rapa ssp. pekinensis)、荷花(Lotus corniculatus)、黃瓜(Cucumbers)、马铃薯(Potato)、苜蓿(Medicago truncatula)等许多物种的AP2/ERF超家族进行了全基因组分析[4-5,7,12-15]。近年来,香蕉基因组的研究也越来越深入,2012年就完成了A基因组的测序工作[16],2016年Lakhwani等[17]从香蕉A基因组数据库中筛选了小果野芭蕉(M. acuminata)和野蕉(M. balbisiana)的AP2/ERF超家族基因,从基因进化角度对其进行了整体分析,但是基因型为AAA的巴西蕉AP2/ERF超家族基因的家族分类和结构分析还未见报道。
本研究根据香蕉A基因组数据库中,基因型为AAA巴西蕉完整、非冗余的AP2/ERF超家族基因全基因组数据,对其进行了系统的分析,将为巴西蕉应答非生物逆境胁迫下AP2/ERF超家族功能基因的挖掘和鉴定,及后期巴西蕉胁迫响应机制和抗逆品种改良的研究奠定理论基础,也将为不同物种AP2/ERF超家族基因的系统发育和进化研究提供方法和理论依据。
1 材料与方法
1.1 巴西蕉AP2/ERF超家族基因的识别和系统发育树的构建
从香蕉A基因组数据库(http://banana-genome.cirad.fr/)中筛选基因型为AAA的巴西蕉AP2/ERF超家族DNA和蛋白序列[16]。另外,以已知的AP2/ERF超家族基因结构特征为基础,运用CDD和PFAM数据库[18-19],通过保守结构域的识别进一步对AP2/ERF超家族基因进行家族划分,将包含2个重复AP2/ERF结构域的基因划分为AP2家族,将仅包含1个AP2/ERF结构域的基因划分为ERF家族,而将包含1个AP2/ERF结构域和1个B3 DNA结合区域的基因划分为RAV家族。此外,AP2/ERF结构域发生高度分化但与其他物种中ERF基因有较高相似性的基因被归为soloist家族[4-5,15,17]。从Rice Genome Annotation Project[20]中获得水稻ERF家族氨基酸序列。用BLAST分析所有水稻的ERF蛋白,深入识别香蕉数据库中的ERF家族蛋白。基于巴西蕉和水稻中识别的ERF进行多序列比对,运用Clustal X 2.0和MEGA 5.0构建系统发育树。
1.2 巴西蕉AP2/ERF超家族基因结构和motif分析
在香蕉A基因组数据库中下载基因型为AAA的巴西蕉AP2/ERF超家族基因的全基因组和CDS序列。用GSDS2.0软件对香蕉AP2/ERF超家族全部基因结构进行分析[21];用MEME软件对香蕉AP2/ERF超家族全部基因的氨基酸motif组件进行分析[22],并基于InterProScan数据对识别的motif做进一步的诠释[23]。
2 结果与分析
2.1 巴西蕉ERF家族基因的识别和进化分析
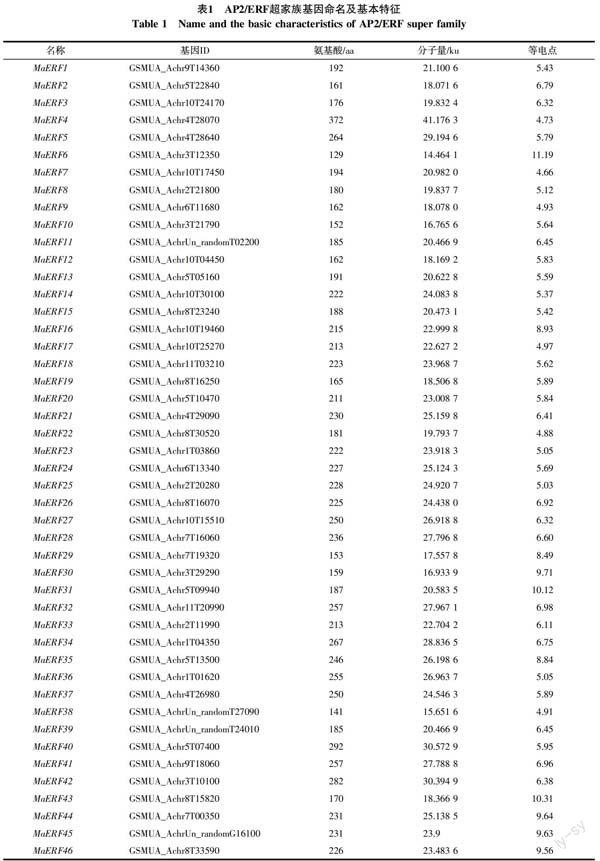
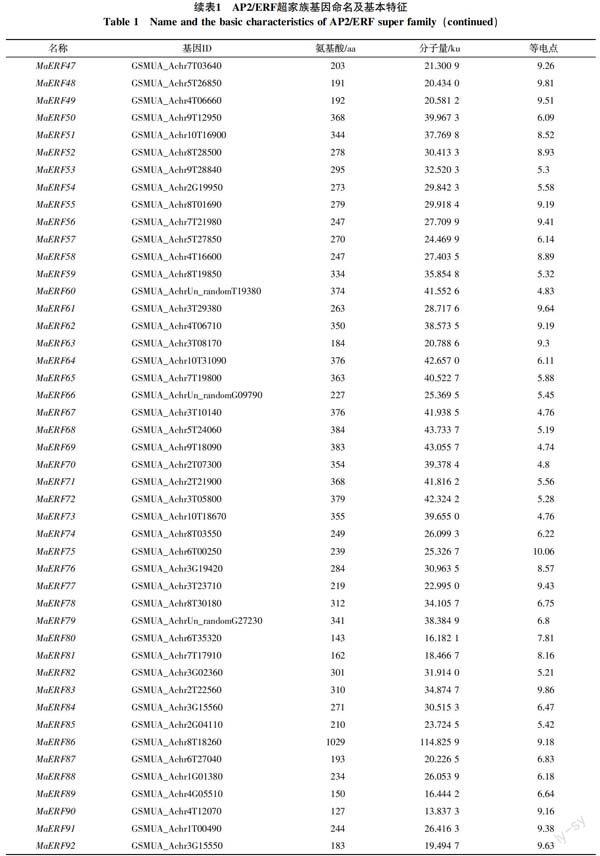
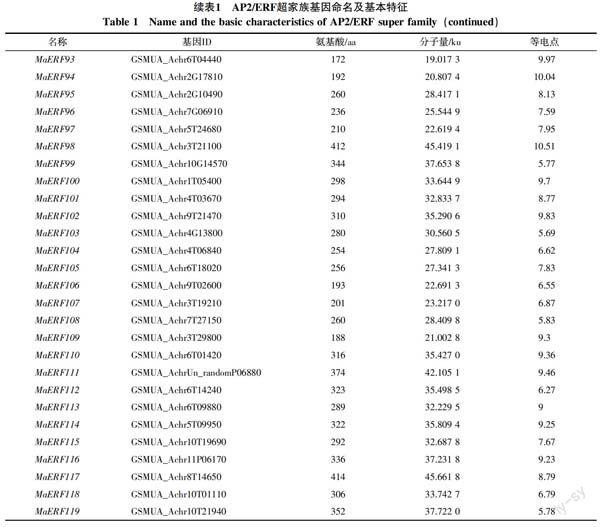
从香蕉A基因组数据库中共筛选出基因型为AAA巴西蕉的非冗余、完整的AP2/ERF基因119个,依据拟南芥中MYB、WRKY、bZIP和bHLH转录因子的命名方法[24-26],从MaERF1~MaERF119对其命名。ExPASY分析结果表明,119个假定的AP2/ERF超家族基因编码的蛋白全长为127~1 029 aa,相关分子量为13.837 3~114.825 9 ku,等电点从4.66到11.19(表1)。
对119个基因进行保守区域检测和多重序列分析,结合AP2/ERF超家族分类标准,分别将100个基因归为ERF家族,15个基因归类为RAV家族;4个AP2/ERF结构域发生高度分化,但与其他物种中ERF基因有较高相似性的基因归为soloist家族;因没有包含2个重复AP2/ERF结构域的基因,所以AAA基因型巴西蕉中未划分出AP2家族。
用香蕉和水稻ERF家族基因编码的AP2/ERF结构域的氨基酸序列进行了多序列比对,并构建无根系统发育树(图1)。结果表明,基于序列比对的相似性,香蕉100个ERF家族基因又被划分成10亚组,即GroupⅠ~GroupⅩ。GroupⅠ亚家族包含MaERF50,-51,-52;GroupⅡ包含MaERF1,-7~-17,-22;GroupⅢ包含MaERF18~-21,-23~-27,-32~-39;Group Ⅳ包含MaERF40,-41,-42;Group Ⅴ包含7个基因,分别为MaERF2~-6,-28,-29;MaERF53~-59,-99,-112,-118和-119属于GroupⅥ;GroupⅦ亚家族中有MaERF60和MaERF63~-73;GroupⅧ包含MaERF基因-30,-31,
43~-49,-74~-82;MaERF84,-85和MaERF87~-97属于亚家族GroupⅨ;GroupⅩ亚家族有3个基因-MaERF103、MaERF104和MaERF105。根据水稻GroupⅤ和GroupⅩ的划分,又将香蕉GroupⅤ中的MaERF4-6划分为Ⅴb,剩余的GroupⅤ亚家族基因划分为Ⅴa;香蕉GroupⅩ中的MaERF105被划分为Ⅹa,而Ⅹb有基因MaERF103和MaERF104。
2.2 巴西蕉AP2/ERF家族基因结构分析
基于巴西蕉119个AP2/ERF超家族基因AP2/ERF保守结构域氨基酸序列同源比对,依据其相似性建立了一个无根的系统进化树,然后对基因内含子和外显子结构进行比较分析(图2)。结果显示,所有的巴西蕉AP2/ERF超家族基因也被划分为3个大的家族:ERF、RAV和Soloist,其中ERF家族又被划分成10个亚组,分别命名为Ⅰ~Ⅹ,并且每个亚家族中各基因具有相似的内含子-外显子结构,如GroupⅠ、GroupⅡ、GroupⅣ和GroupⅨ均只包含1个外显子;RAV和Soloist家族中的基因大多含有2个外显子。每个亚组中相似的外显子数目说明了他们之间较近的进化关系和相似的功能。这一结果和图1的进化结果一致,进一步证实了AP2/ERF超家族分类的准确性。系统发育树和基因结构分析说明它们之间有较近的进化关系,清晰的展示了巴西蕉ERF家族基因每个分支早期的进化关系。
2.3 巴西蕉AP2/ERF家族基因保守motif分析
通过MEME软件分析,从119个香蕉AP2/ERF超家族基因中搜索出15个保守motif,并运用InterPro 资料对其进一步诠释(图3,表2)。15个motif中含有2个功能区域,分别为AP2/ERF 结构域和DNA-binding结构域,其中AP2/ERF 结构域在motif 1和motif 2中,而DNA-binding结构域在motif 1、4、5和6中。含有AP2/ERF结构域的motif 1和motif 2除了在Soloist家族的MaERF106、MaERF107、MaERF108和MaERF109基因中未被诠释外,剩余的所有AP2/ERF超家族基因中均诠释有AP2/ERF结构域;在RAV家族基因中还诠释出了DNA-binding结构域。
另外,每个ERF亚组中基本含有相同的保守motif组件。如GroupⅠ均含有motif 1、2和10;GroupⅣ中基因均含有motif 1、2、3、10和14。保守Motif的分析更进一步的证明了香蕉ERF家族进化和分类关系的准确性,并且每个亚组含有相同motif预示着同一亚组基因有着相似的功能。
3 讨论
近年来,拟南芥、水稻、大豆、葡萄等物种AP2/ERF家族基因的全基因组分析已被完成,并且该领域的研究也越来越被重视,仅2014年就报导了玉米、马铃薯和黄瓜3种植物AP2/ERF的全基因组分析的结果,2016年又对小果野芭蕉(M. acuminata)、长梗蕉(M. balbisiana)和苜蓿的AP2/ERF超家族基因全基因组分析进行了报导[7,12,15,17]。本研究对基因型为AAA的巴西蕉AP2/ERF超基因家族进行了分析,共识别出巴西蕉119个全长、非冗余的AP2/ERF超家族基因,又将其划分成3个家族,分别为ERF(100个)、RAV(15个)和soloist(4个)家族。这一分类结果与大白菜、水稻、黄瓜、玉米、荷花等植株AP2/ERF超家族基因划分结果不同,以上植株均是将AP2/ERF超家族基因划分为AP2、ERF、RAV和soloist 4个家族。此外,这一分类结果也与同一物种的小果野芭蕉和长梗蕉的不同,2016年Lakhwani等[17]将小果野芭蕉和长梗蕉的AP2/ERF超家族基因划分为AP2、ERF、RAV和soloist共4个家族,其中AP2家族有2个重复的AP2/ERF结构域,ERF家族仅含有1个AP2/ERF结构域。巴西蕉是野生尖叶蕉和长梗蕉种内或者种间杂交之后逐渐进化而来的3倍体新品种,其不存在包含2个AP2/ERF结构域的AP2家族基因,可能是在进化过程中1个AP2/ERF结构域消失的原因。因此,根据AP2/ERF结构域的数目和是否出现其他DNA结构域特征,将本研究识别出的119个AAA基因型巴西蕉AP2/ERF超家族基因划分为ERF、RAV和soloist是正确的,这一结果对于后续AAA基因型巴西蕉AP2/ERF超家族基因的筛选和功能研究具有一定的指导意义。
一般而言,转录因子DNA结合域外的区域包含重要的功能区域,参与转录启动、蛋白之间互作和核定位。因此,植物转录因子大家族的一个亚组内成员的功能区域,即氨基酸序列motif通常是保守的,如MYB、WRKY、NAC、Dof、GATA和GRAS[24,27-29]。一個亚组中蛋白的motif可能有相似的功能,有些可能在转录调控中发挥重要作用[4]。本研究依据水稻ERF家族和巴西蕉100个ERF家族基因编码的AP2/ERF保守结构域氨基酸序列的比对结果,又将ERF家族划分为10个亚组,即GroupⅠ~GroupⅩ。对于AP2/ERF超家族中ERF家族的划分,近几年不同物种中的报导均一致,都被划分为10个亚组,也与香蕉进化的原始种小果野芭蕉和长梗蕉的划分结果一致[17]。对巴西蕉ERF家族基因结构和保守功能motif分析发现,聚类到同一亚组中的ERF基因具有相似的基因结构和相似的motif组件。这一结果也符合前人对其他植物ERF家族全基因组分析后得出的结论[4-5,7,15]。因此,划分到同一亚组的ERF基因具有较近的进化关系,并且可能在巴西蕉响应外界生物和非生物胁迫方面具有相似的功能。
综上所述,本研究对基因型为AAA的栽培种巴西蕉的AP2/ERF超家族进行了系统的分析,将获得的119个巴西蕉AP2/ERF超家族基因划分成ERF、RAV和soloist共3个家族,其中ERF家族又被划分为10个亚家族,每个亚家族基因具有相似的保守motif和基因结构。对巴西蕉AP2/ERF超家族基因的研究为不同物种基因家族的系统发育和进化研究提供了方法和理论依据,更有助于巴西蕉响应非生物逆境胁迫下AP2/ERF超家族功能基因的挖掘和鉴定,对于后期非生物逆境胁迫响应机制的研究、香蕉品种抗逆改良奠定了基础。
參考文献
[1] Simmonds N W. Megasporogenesis and female fertility in 3 edible triploid bananas[J]. Journal of Genetics, 1961, 57(2-3): 269.
[2] 吴坤林. 香蕉的生物学特性及其组织培养技术[J]. 生物学通报, 2006, 41(10): 5-8.
[3] 程晓培. 香蕉MADS-box转录因子在胁迫条件下的表达分析[D]. 海口: 海南大学, 2013.
[4] Nakano T, Suzuki K, Fujimura T, et al. Genome-wide analysis of the ERF gene family in Arabidopsis and rice[J]. Plant Physiology, 2006, 140(2): 411-432.
[5] Song X M, Li Y, Hou X L. Genome-wide analysis of the AP2/ERF transcription factor superfamily in Chinese cabbage(Brassica rapa ssp. pekinensis)[J]. BMC Genomics, 2013, 14(1): 1-15.
[6] Wan L Y, Wu Y S, Huang J Q, et al. Identif ication of ERF genes in peanuts and functional analysis bof AhERF008 and AhERF019 in abiotic stress response[J]. Funct Integr Genomics, 2014, 14(3): 467-477.
[7] Sun Z M, Zhou M L, Xiao X G, et al. Genome-wide analysis of AP2/ERF family genes from Lotus corniculatus shows LcERF054 enhances salt tolerance[J]. Functional & Integrative Genomics, 2014, 14(3): 453-466.
[8] Yang H, Yu C, Yan J, et al. Overexpression of the Jatropha curcas JcERF1 gene coding an AP2/ERF-Type transcription factor increases tolerance to salt in transgenic tobacco[J]. Biochemistry(Moscow), 2014, 79(11): 1 226-1 236.
[9] Dossa K, Wei X, Li D H, et al. Insight into the AP2/ERF transcription factor superfamily in sesame and expression profiling of DREB subfamily under drought stress[J]. BMC Plant Biology, 2016, 16(1): 171-186.
[10] Lee D K, Jung H, Jang G, et al. Overexpression of the OsERF71 transcription factor alters rice root structure and drought resistance[J]. Plant Physiology Preview, 2016, 172(1): 379.
[11] Magome H, Yamaguchi S, Hanada A, et al. dwarf and delayed-flowering, a novel Arabidopsis mutant deficient in gibberellin biosynthesis because of overexpression of a putative AP2 transcription factor[J]. Plant J, 2014, 37(4): 720-729
[12] Du H W, Huang M, Zhang Z X, et al. Genome-wide analysis of the AP2/ERF gene family in maize waterlogging stress response[J]. Euphytica, 2014, 198(1): 115-126.
[13] Zhuang J, Cai B, Peng R H, et al. Genome-wide analysis of the AP2/ERF gene family in Populus trichocarpa[J]. Biochem Biophys Res Commun, 2008, 371(3): 468-474.
[14] Zhuang J, Peng R H, Cheng Z M, et al. Genome-wide analysis of the putative AP2/ERF family genes in Vitis vinifera[J]. Scientia Horticulturae, 2009, 123(1): 73-81.
[15] Hu L F, Liu S Q. Genome-wide identification and phylogenetic analysis of the ERF gene family in cucumbers[J]. Genetics and Molecular Biology, 2011, 34(4): 624-633.
[16] D'Hont A, Denoeud F, Aury J M, et al. The banana(Musa acuminata)genome and the evolution of monocotyledonous plants[J]. Nature, 2012, 488(1): 213-217.
[17] Lakhwani D, Pandey A, Dhar Y V, et al. Genome-wide analysis of the AP2/ERF family in Musa species reveals divergence and neofunctionalisation during evolution[J]. Scientific Report, 2016, 6: 18 878.
[18] Marchler-Bauer A, Derbyshire M K, Gonzales N R, et al. CDD: NCBI's conserved domain database[J]. Nucleic Acids Res, 2015, 43(1): D222-D226.
[19] Finn R D, Bateman A, Clements J, et al. The Pfam protein families database[J]. Nucleic Acids Res, 2014, 42(1): D222-D230.
[20] Kawahara Y, de la Bastide M, Hamilton J P, et al. Improvement of the Oryza sativa Nipponbare reference genome using next generation sequence and optical map data[J]. Rice, 2013, 6(1): 1-10.
[21] Hu B, Jin J, Guo A Y, et al. GSDS 2.0: An upgraded gene feature visualization server[J]. Bioinformatics, 2015, 31(7): 1 296-1 297.
[22] Brown P, Baxter L, Hickman R, et al. MEME-LaB: Motif analysis in clusters[J]. Bioinformatics, 2013, 29(9): 1 696-1 697.
[23] Mulder N, Apweiler R. InterPro and InterProScan: Tools for protein sequence classification and comparison[J]. Methods Mol Biol, 2007, 396(1): 59-70.
[24] Eulgem T, Rushton P J, Robatzek S, et al. The WRKY superfamily of plant transcription factors[J]. Trends Plant Science, 2000, 5(5): 199-206.
[25] Jakoby M, Weisshaar B, Droge-Laser W, et al. bZIP transcription factors in Arabidopsis[J]. Trends in Plant Science, 2002, 7(3): 106-111.
[26] Heim M A, Jakoby M, Werber M, et al. The basic helix-loop-helix transcription factor family in plants: a genome-wide study of protein structure and functional diversity[J]. Molecular Biology and Evolution, 2003, 20(5): 735-747.
[27] Ooka H, Satoh K, Doi K, et al. Comprehensive analysis of NAC family genes in Oryza sativa and Arabidopsis thaliana[J]. DNA Res, 2003, 10(2): 239-247.
[28] Tian C, Wan P, Sun S, et al. Genome-wide analysis of the GRAS gene family in rice and Arabidopsis[J]. Plant Molecular Biology, 2004, 54(3): 519-532.
[29] Gupta S, Malviya N, Kushwaha H, et al. Insights into structural and functional diversity of Dof(DNA binding with one finger)transcription factor[J]. Planta, 2015, 1(7): 549-562.
