玉米叶片叶绿素含量的全基因组关联性分析
2017-05-19滕守振汪海梁海生辛红佳李圣彦郎志宏
滕守振汪海梁海生辛红佳李圣彦郎志宏
(1. 西南科技大学生命科学与工程学院,绵阳 621010;2. 中国农业科学院生物技术研究所,北京 100081)
玉米叶片叶绿素含量的全基因组关联性分析
滕守振1,2汪海2梁海生1,2辛红佳2李圣彦2郎志宏2
(1. 西南科技大学生命科学与工程学院,绵阳 621010;2. 中国农业科学院生物技术研究所,北京 100081)
植物叶片的叶绿素含量与叶片的光合作用效率和产量潜力紧密相关,因此是作物的一个重要生理指标。但是,目前已克隆的控制叶绿素含量的基因大多来自拟南芥和水稻,尚不清楚玉米自然群体中哪些基因控制叶片叶绿素含量的变异。本研究发现玉米苗期第一片叶片的叶绿素含量和吐丝期穗位叶的叶绿素含量高度相关,且与后者相比具有更高的遗传力。进一步分析了287份玉米自交系的第一片叶的叶绿素含量,利用558 269个单核苷酸多态性分子标记进行全基因组关联性分析,获得9个显著关联SNP位点和16个候选基因。通过候选基因的序列分析和功能预测,发现两个可能和叶绿素含量相关的基因,包括拟南芥Tic22基因的同源基因和水稻衰老相关基因SAG12的同源基因。
玉米;叶绿素值;GWAS
玉米(Zea mays)是当今世界最重要的粮食作物之一,主要应用于食品,饲料和工业生产。玉米的自然群体具有丰富的表型变异和基因型变异,这就为研究玉米基因型与表型的多样性关系提供了极大的便利[1]。确定控制重要农艺性状的自然等位变异,不仅有助于解析重要农艺性状的遗传基础,而且可以为分子标记辅助育种提供有效的基因资源和分子标记[2],因此具有重要的理论意义和应用价值。近年来,关联分析成为解析复杂数量性状遗传基础的一个重要手段[3]。关联分析以自然群体为材料,以连锁不平衡为基础[4],将目标性状的遗传变异与遗传多态性相结合,可直接鉴定出与表型变异相关的基因[5]。
叶绿素是绿色植物进行光合作用的基础物质,是植物叶片的主要光合色素,是研究玉米生长特性、生理变化的重要指标[6,7]。高等植物包含两种类型的叶绿素,即叶绿素a和叶绿素b。叶绿素a存在于反应中心复合体上,但是叶绿素b仅存在于天线复合体上,主要起保护聚光叶绿素结合蛋白的作用。另外,叶绿素及其派生物对光极其敏感,在光照下很容易产生氧自由基,所以植物生长期间需要精确地调节叶绿素的代谢[8]。目前已经克隆的控制叶绿素含量的基因大部分来自水稻和拟南芥。已有文献报道,与低产水稻相比,高产水稻拥有更高的叶绿素含量和光合作用速率[9]。在水稻(Oryza sativa)中,Gc基因过量表达时,叶绿素b和叶绿素的含量分别上升了100%和25%[10],其上位基因DE1的突变会导致该表型更加明显[11]。过表达植物磺肽素基因(OSpsk3)的转基因水稻比野生型的叶绿素含量提高了2.3倍[12]。近期有研究表明,Ghd7基因通过下调叶绿素合成基因降低了叶绿素的含量[13]。在拟南芥中,MYC2/3/4和ANAC019/055/072可以通过直接上调叶绿素分解代谢基因来降低叶绿素含量[14];NAP-AAO3[15]和 ABF1、ABF2、ABF3[16]可以通过调节ABA的方式间接调节叶绿素的含量;而STAY-GREEN1基因则通过与代谢酶结合的方式缓解叶绿素的降解[17];vpp1 基因产物则通过抵制氧自由基的方式,保护叶绿体膜,从而阻止叶绿素的进一步降解[18]。此外,一些基因通过调节衰老间接控制叶绿素的含量。在烟草和番茄中,异戊烯转移酶基因(ipt)的过量表达会提高细胞分裂素的含量,延缓衰老以及衰老期间叶绿素的降解[19];小麦(Triticum aestivum)Tackx4基因编码细胞分裂素氧化酶,Tackx4基因的过表达会降低细胞分裂素的含量并加速衰老,从而加剧叶绿素的降解[20]。由此可见,叶绿素含量是大量基因和多种机制相互作用的共同结果。
在玉米中已有研究表明叶片的叶绿素含量是决定玉米的光合作用效率以及产量的重要因素[21,22]。本研究测量了温室条件下三叶期自交系玉米第一片叶子的叶绿素含量,也测量了田间生长的自交系玉米在成熟期穗位叶的叶绿素含量,发现二者有正相关的关系,且前者的遗传力更高。因此,本研究进一步以 287 份玉米自交系为材料,使用SPAD-502 plus来测量玉米第一片叶子的叶绿素值,通过全基因组关联性分析,解析与玉米叶片叶绿素含量显著相关的单核苷酸多态性分子标记位点,并对候选基因进行分析和功能预测,旨在为发掘控制叶绿素含量的自然变异提供参考。
1 材料与方法
1.1 材料
287份玉米自然群体来自严建兵教授实验室;SPAD-502 plus购自柯尼卡美能达公司。
1.2 方法
1.2.1 玉米的种植 共使用287份自交系玉米,每个自交系种植15棵,每5棵玉米种在一个小盆里。种植深度为3 cm,然后在上面覆上一层与花盆等高的营养土,插上标签牌,放入托盘中,待种植完成后统一浇水。
1.2.2 玉米的培养 玉米的生长条件为:28℃,16/8 h光照/黑暗,光照强度为100-150 mEm-2S-1,湿度40%-60%。
1.2.3 玉米叶绿素的测量 对于生长至第3片叶与第2片叶等长的玉米幼苗,取下其第一片叶,使用仪器SPAD-502 plus测量第一片叶子的中部。对于田间生长的玉米自交系,在玉米植株开始吐丝当天,测量穗位叶的中部。对每片叶子测量5-10个数据,取平均值记为该叶子的叶绿素值。
1.2.4 数据处理 利用R软件(版本号3.3.1)计算出每个自交系叶绿素的平均值,使用Tassel 5(版本号5.2.31)的混合线性模型[23],进行叶绿素含量与单核苷酸多态性标记位点的关联分析。Pearson相关系数的计算和正态分布检验分别采用R语言stats软件包的cor函数和shapiro.test函数(https://www. r-project.org/)。
2 结果
2.1 玉米苗期第一片叶与吐丝期穗位叶叶绿素含量的相关性分析和遗传力分析
本研究首先测量了103份自交系玉米三叶期第一片叶的叶绿素含量,以及相同玉米材料田间种植条件下吐丝期穗位叶的叶绿素含量,并对两个性状进行相关性分析和遗传力分析。如图1所示,第一片叶和穗位叶的叶绿素含量具有明显的线性关系,且相关性较高(Pearson相关系数r=0.74)。此外,如表1所示,与穗位叶的叶绿素含量相比,第一片叶的叶绿素含量具有更高的遗传力。这说明在温室条件下,玉米第一片叶的叶绿素含量比田间条件下穗位叶的叶绿素含量受环境影响更小。由于全基因组关联分析需要精确测量高遗传力的表型,因此本研究进一步对玉米第一片叶的叶绿素含量进行分析。
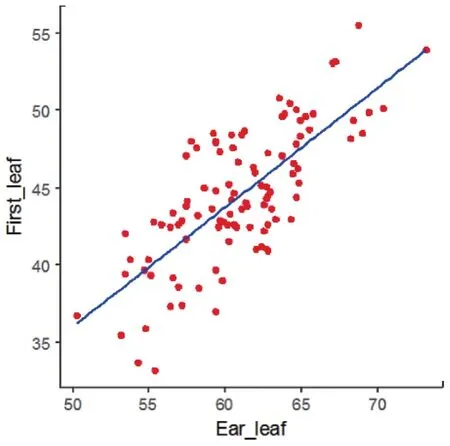
图1 第一片叶子与穗位叶叶绿素含量的相关性分析

表1 第一片叶子与穗位叶叶绿素含量的遗传力分析
2.2 玉米第一片叶的叶绿素含量在自然群体中的多样性
玉米的叶绿素含量容易受到环境的影响,所以本次实验是在可控的温室中进行。本研究共测量了287份自交系玉米的叶绿素含量,结果(图2)表明叶绿素含量值符合正态分布,适合作全基因组关联分析。在这些自交系中,5个叶绿素值最低的自交系是BT1、CIMBL37、CML282、CIMBL117和By4944。5个叶绿素值最高的自交系是RO8、CIMBL137、GEMS64、GEMS52和GEMS44。这些自交系可以用来进一步研究第一片叶叶绿素值的生理效应,也可以作为亲本构建群体,进一步开发分子育种所需的标记。这287份自交系可以分为TST、NSS、SS和混合型群体,分析各个亚群体的叶绿素值发现,SS玉米第一片叶子的叶绿素值平均比NSS高,而NSS玉米的叶绿素值平均比TST高。
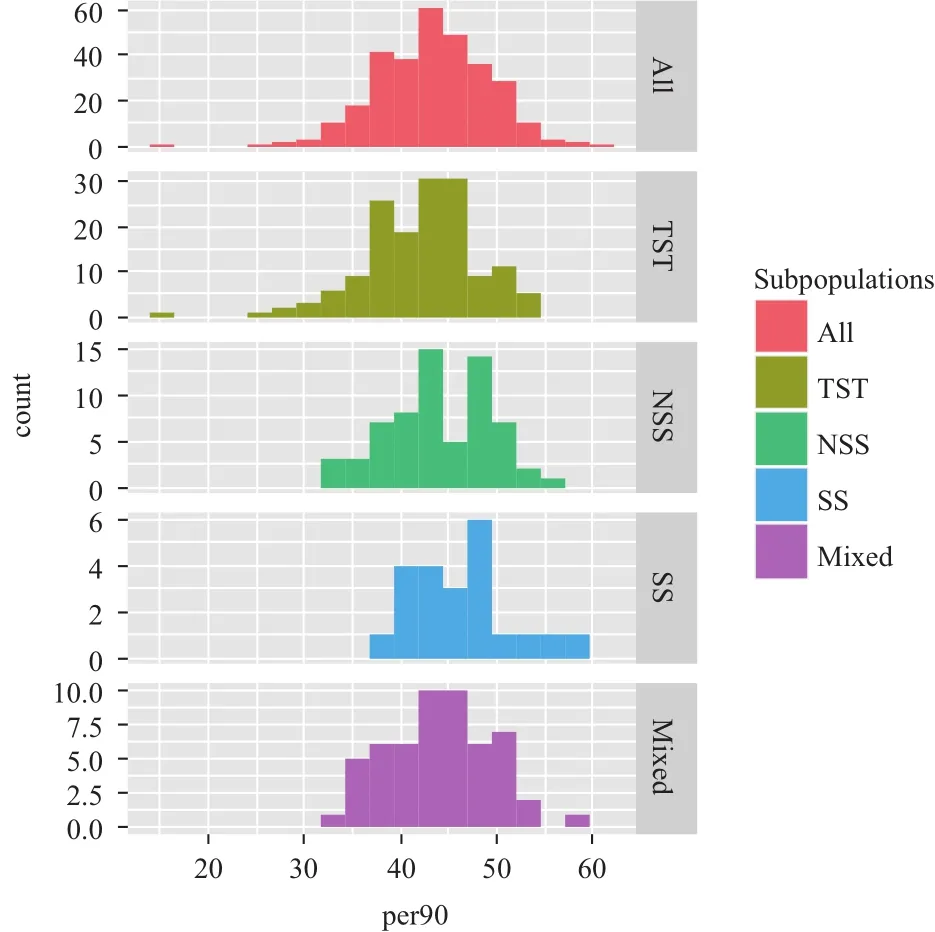
图2 不同亚群自然群体的叶绿素值
2.3 玉米第一片叶叶绿素含量的全基因组关联分析
使用混合线性模型进行全基因组关联分析。如表2和图3所示,具有统计学显著意义的SNP标记位点有9个(P≤9.09×10-8,P< 0.05/n,n=使用的标记总数),其中有5个SNP位点表现为极其显著(P≤1.82×10-8,P< 0.01/n)。这些SNP位点大部分分布在10号染色体上,在3号和5号染色体上也有分布。
以B73基因组序列为参考,在显著关联位点上下游6E4 bp寻找候选基因,最终得到16个候选基因。对这16个候选基因进行同源分析和功能注释,结果如表3所示。

表2 和叶绿素含量显著相关的SNP位点

图3 叶绿素含量全基因组关联分析的曼哈顿图

表3 16个候选基因的基本信息
在这些候选基因中,GRMZM2G097959编码Tic22蛋白。在拟南芥中Tic22蛋白具有将叶绿体蛋白从叶绿体间隙输入到叶绿体基质的功能[24],该蛋白基因的敲除会导致拟南芥的叶绿体变小,而且突变体叶绿素含量仅为野生型叶绿素含量的一半[25]。GRMZM2G363926编码一个半胱氨酸蛋白酶。在水稻中,该基因的同源基因SAG12负调控细胞的程序性死亡,SAG12基因的突变导致水稻受到环境胁迫时,细胞死亡率显著升高[26]。这两个候选基因和显著关联SNP在基因组上的相对位置如图4所示。GRMZM2G097959位于chr3.S_2451902上游1kb处,GRMZM2G363926在chr10.S_100431934、chr10.S_100431952、chr10.S_100431969、chr10.S_100432171下游约50 kb处。

图4 候选基因GRMZM2G097959和GRMZM2G363926与显著关联SNP在基因组上的相对位置
3 讨论
已有研究表明,玉米叶片的叶绿素含量是叶片光合作用速率的一个重要决定因素。在本研究中,利用287份玉米自交系分析了玉米自然群体中叶绿素含量,发现叶绿素含量在玉米自然群体中具有高度的多态性。本研究获得了9个和叶绿素含量显著关联的SNP位点,以及16个候选基因,但是这些基因的功能目前尚不清楚。功能注释表明这些候选基因中至少有两个基因可能控制着叶绿素含量。
一个候选基因是GRMZM2G097959。GRMZM2-G097959在拟南芥中的同源基因编码Tic22蛋白,该蛋白位于叶绿体内膜上[24],主要在早期生长阶段表达,具有将叶绿体蛋白从叶绿体间隙输入到叶绿体基质的功能。该基因的敲除导致拟南芥在早期生长阶段叶片的变化非常明显,主要表现为叶绿体变小,而且叶绿素含量仅为野生型叶绿素含量的50%。但是随着生长时间的延长,突变体与野生型的叶绿素含量趋于一致[25]。我们推测玉米的GRMZM2G097959基因可能在玉米中也具有相同或相似的生化和生理功能。另一个候选基因是GRMZM2G363926。该基因在拟南芥中的同源基因是衰老相关基因12(SAG12),编码木瓜蛋白酶型的半胱氨酸蛋白酶,该基因只在衰老过程中诱导表达,但是它的突变并没有影响到拟南芥的表型[27]。但是在水稻中的研究发现,水稻SAG12基因的突变并不会影响正常条件下生长的水稻,当水稻受到环境胁迫时,该基因突变会导致水稻细胞死亡率显著升高。因此,水稻SAG12基因在胁迫条件下负调控细胞程序性死亡[26]。在下一步研究中,可以利用具有不同单倍型候选基因的玉米品种构建双亲群体,通过连锁分析验证关联分析的结果,并利用基因敲除等技术手段验证候选基因的功能。
4 结论
本研究通过全基因组关联分析,解析了玉米第一片叶叶绿素含量的遗传结构。获得9个与叶绿素含量显著关联的SNP位点以及16个候选基因。对候选基因的序列分析和功能预测表明其中至少有两个基因可能控制着玉米自然群体叶绿素含量的变异。
[1]Yan JB, Warburton M, Crouch J. Association mapping for enhancing maize(Zea mays L. )genetic improvement[J]. Crop Science, 2011, 51(2):433-449.
[2]Zhang X, Zhang H, Li LJ, et al. Characterizing the population structure and genetic diversity of maize breeding germplasm in Southwest China using genome-wide SNP markers[J]. BMC Genomics, 2016, 17(1):697.
[3]Jannink J, Bink MC, Jansen RC. Using complex plant pedigrees to map valuable genes[J]. Trends in Plant Science, 2001, 6(8):337-342.
[4]Zondervan KT, Cardon LR. The complex interplay among factors that influence allelic association[J]. Nature Reviews Genetics, 2004, 5(2):89-100.
[5]李玮瑜, 张斌, 张嘉楠, 等 . 利用关联分析发掘小麦自然群体旗叶叶绿素含量的优异等位变异[J]. 作物学报, 2012, 38(6):962-970.
[6]刘贞琦, 刘振业, 马达鹏, 等 . 水稻叶绿素含量及其与光合速率关系的研究[J]. 作物学报, 1984, 10(1):57-62.
[7]左宝玉, 李世仪, 匡廷云, 等 . 玉米不同层次叶片叶绿体的超微结构和叶绿素含量变化[J]. 作物学报, 1987, 13(3):213-218.
[8]Masuda T, Fujita YC. Regulation and evolution of chlorophyll metabolism[J]. Photochemical & Photobiological Sciences, 2008, 7(7):1131-1149.
[9]Peng SB, Gurdevs K, Parminder V, et al. Progress in ideotype breeding to increase rice yield potential[J]. Field Crops Research, 2008, 108(1):32-38.
[10]Wang F, Wang G, Li X, et al. Heredity, physiology and mapping of a chlorophyll content gene of rice(Oryza sativa L. )[J]. Journal of Plant Physiology, 2008, 165(3):324-330.
[11]Huang JL, Qin F, Zang GC, et al. Mutation of OsDET1 increases chlorophyll content in rice[J]. Plant Science, 2013, 210:241-249.
[12]Huang JY, Wang YF, Yang JS. Over-expression of OsPSK3 increases chlorophyll content of leaves in rice[J]. Hereditas, 2010, 32(12):1281-1289.
[13]Wang QX, Xie WB, Xing HK, et al. Genetic architecture of natural variation in rice chlorophyll content revealed by a genome-wide association study[J]. Molecular Plant, 2015, 8(6):946-957.
[14]Zhu X, Chen J, Xie Z, et al. Jasmonic acid promotes degreening via MYC2/3/4- and ANAC019/055/072-mediated regulation of major chlorophyll catabolic genes[J]. Plant Journal, 2015, 84(3):597-610.
[15]Yang JD, Worley E, Udvardi M. A NAP-AAO3 regulatory module promotes chlorophyll degradation via ABA biosynthesis in Arabidopsis leaves[J]. Plant Cell, 2014, 26(12):4862-4874.
[16]Gao S, Gao J, Zhu X, et al. ABF2, ABF3 and ABF4 promote ABA-mediated chlorophyll degradation and leaf senescence by transcriptional activation of chlorophyll catabolic genes and senescence-associated genes in Arabidopsis[J]. Molecular Plant, 2016, 9(9):1272-1285.
[17]Sakuraba Y, Park SY, Kim YS, et al. Arabidopsis STAY-GREEN2 is a negative regulator of chlorophyll degradation during leaf senescence[J]. Molecular Plant, 2014, 7(8):1288-1302.
[18]Zhang LG, Kusaba M, Tanaka A, et al. Protection of chloroplast membranes by VIPP1 rescues aberrant seedling development in Arabidopsis nyc1 mutant[J]. Frontiers in Plant Science, 2016, 7(73):533.
[19] Ma QH, Liu YC. Expression of isopentenyl transferase gene(ipt)in leaf and stem delayed leaf senescence without affecting root growth[J]. Plant Cell Reports, 2009, 28(11):1759-1765.
[20] Chang C, Lu J, Zhang HP, et al. Copy number variation of cytokinin oxidase gene Tackx4 associated with grain weight and chlorophyll content of flag leaf in common wheat[J]. PLoS One, 2015, 10(12):e0145790.
[21]Gitelson AA, Peng Y. Efficiency of chlorophyll in gross primary productivity:A proof of concept and application in crops[J]. Journal of Plant Physiology, 2016, 201:101-110.
[22]Wagle P, Zhang Y, Jin C, et al. Comparison of solar-induced chlorophyll fluorescence, light-use efficiency, and process-based GPP models in maize[J]. Ecological Applications, 2016, 26(4):1211-1222.
[23]Bradbury PJ, Zhang Z, Kroon DE, et al. TASSEL:Software for association mapping of complex traits in diverse samples[J]. Bioinformatics, 2007, 23:2633-2635.
[24]Kasmati AR, Töpel M, Khan NZ, et al. Evolutionary, molecular and genetic analyses of Tic22 homologues in Arabidopsis thaliana chloroplasts[J]. PLoS One, 2013, 8(5):e63863.
[25] Rudolf M, Machettira AB, Groβ LE, et al. In vivo function of Tic22, a protein import component of the intermembrane space of chloroplasts[J]. Molecular Plant, 2013, 6(3):817-829.
[26]Singh S, Singh A, Nandi AK. The rice OsSAG12-2 gene codes for a functional protease that negatively regulates stress-induced cell death[J]. Journal of Biosciences, 2016, 41(3):1-9.
[27]Otegui MS, Yoo-Sun N, Martínez DE, et al. Senescence-associated vacuoles with intense proteolytic activity develop in leaves of Arabidopsis and soybean[J]. Plant Journal, 2005, 41(6):831-844.
(责任编辑 李楠)
Genome-wide Association Study of Chlorophyll Content in Maize Leaves
TENG Shou-zhen1,2WANG Hai2LIANG Hai-sheng1,2XIN Hong-jia2LI Sheng-yan2LANG Zhi-hong2
(1. School of Life Science and Engineering,Southwest Science and Technology University,Mianyang 621010;2. Biotechnology Research Institute,Chinese Academy of Agricultural Sciences,Beijing 100081)
The chlorophyll content of plant leaves is closely related to photosynthetic efficiency and yield potential,thus it is an important physiological indicator of all crop species. However,currently most cloned genes controlling chlorophyll content are from Arabidopsis and rice,and the genetic basis of natural variation in chlorophyll content in maize is still unclear. In this research it was discovered that the chlorophyll content of the first leaves at the seedling stage was highly correlated with that of ear leaves at silking stage,and the former had a higher heritability than the latter. This study further analyzed the chlorophyll contents of first leaves from 287 maize inbred lines. Genome-wide association study using 558 269 SNP markers revealed 9 SNPs significantly associated with the measured trait,leading to the discovery of 16 candidate genes. Two genes potentially controlling chlorophyll content in maize were identified by sequence analysis and functional annotation of these candidate genes:a homolog of the Arabidopsis Tic22 and a homolog of rice SAG12 relating to aging.
maize;chlorophyll content;GWAS
10.13560/j.cnki.biotech.bull.1985.2017.04.013
2016-12-07
国家自然科学基金项目(31501325),国家重点研发计划(2016YFD0100405)
滕守振,男,硕士,研究方向:生物化学与分子生物学;E-mail:tengshouzhen_91@sina.cn
郎志宏,女,研究员,研究方向:玉米功能基因组学;E-mail:langzhihong@caas.cn
