基于同位素标记定量分析烟碱诱导SH-SY5Y细胞的差异表达蛋白
2017-05-19朱贝贝李翔宇陈欢王红娟侯宏卫胡清源
朱贝贝 李翔宇 陈欢 王红娟 侯宏卫 胡清源
(国家烟草质量监督检验中心,郑州 450001)
基于同位素标记定量分析烟碱诱导SH-SY5Y细胞的差异表达蛋白
朱贝贝 李翔宇 陈欢 王红娟 侯宏卫 胡清源
(国家烟草质量监督检验中心,郑州 450001)
以烟碱诱导后的人神经母瘤细胞株为材料,分析烟碱处理后细胞蛋白表达谱的变化,应用iTRAQ标记结合nano-LC-Q-TOF MS技术,鉴定烟碱诱导神经元细胞的差异表达蛋白质并对其进行生物信息学分析。结果显示,烟碱处理组与对照组相比共有132个蛋白差异表达(|ratio|≥1.5,P<0.05),差异表达的蛋白主要参与基因表达、MAPK级联反应、GTP调节信号传导和神经营养因子TRK受体信号传导等生物学过程。其中烟碱可能通过改变蛋白酶体调节神经细胞的平衡性而产生烟碱依赖,MAPK级联反应和泛素化-蛋白酶体通路在这一过程中发挥重要作用。
差异蛋白组学;iTRAQ;烟碱;SH-SY5Y
与酒精、吗啡、大麻、可卡因等致瘾性药物相似,反复抽吸含有烟碱的卷烟,也能导致成瘾。卷烟烟气中,烟碱是引起习惯性抽烟的主要成分,也是吸烟致瘾的主要原因[1,2]。但是,烟碱成瘾相关的分子机制尚不清楚,需要对烟碱成瘾相关分子机制展开进一步研究。研究表明,反复接触致瘾性药物可以诱导脑部特定区域蛋白质表达水平的改变,这种表达的改变将介导个体神经元和神经通路的改变,进而导致成瘾[3,4]。已有研究表明烟碱可以诱导大鼠前额皮质、杏仁核、伏隔核和纹状体区域发动蛋白、层粘连蛋白受体,醛缩酶、α-烯醇酶1和N-马来亚酰胺融合蛋白显著差异表达[5,6]。因此,研究成瘾蛋白质表达水平的变化,可以为研究成瘾相关的分子机制提供重要的数据支撑和理论解释。
目前各种分子技术已被用来研究与药物成瘾相关蛋白质表达的变化。蛋白组学技术可以整体获得药物成瘾过程中蛋白质组的动态变化,为更好的理解药物成瘾的分子机制提供强有力的工具[7]。细胞模型可以简化模型的复杂程度,观察单一类型细胞蛋白质的变化,鉴定一些在成瘾中发挥重要作用的低丰度蛋白,而且实验条件可控,如温度、烟碱浓度和暴露时间[8],烟碱诱导蛋白质分子水平地改变更容易检测。
因此,本研究使用人神经母瘤细胞(SH-SY5Y)作为体外研究烟碱成瘾相关蛋白质表达的细胞系。以同位素相对标记与绝对定量(iTRAQ)蛋白组学技术通过nao-LC-Q-TOF分离鉴定烟碱诱导组和对照组蛋白表达的差异,最终,通过生物信息学分析差异表达蛋白质的细胞组成、生物过程、生物功能和参与的相关信号通路(图1),结果将有助于理解调控烟碱成瘾相关的生物过程。
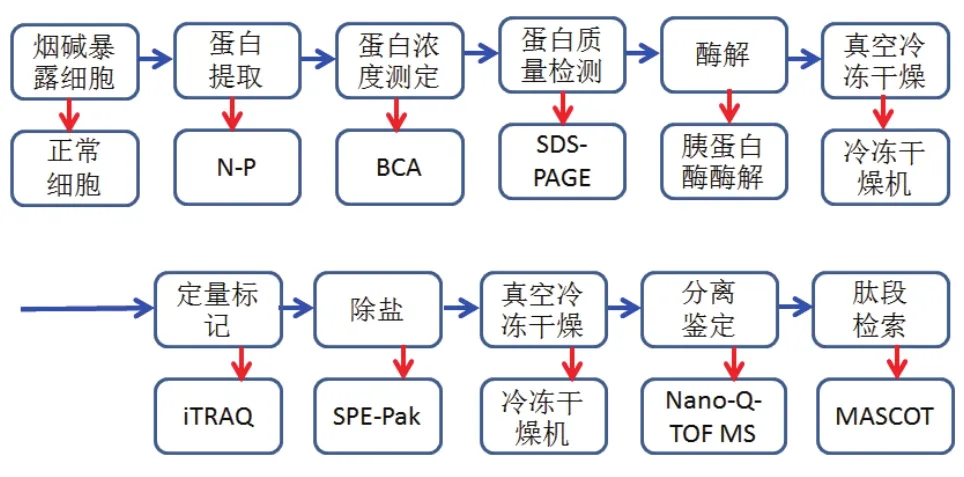
图1 烟碱诱导SH-SY5Y细胞蛋白质差异表达的分析流程
1 材料与方法
1.1 材料
材料:SH-SY5Y(美国 ACTT细胞库)。
试剂:DMEM培养基和青链霉素(美国Hyclone公司);优级胎牛血清(美国Gibco公司);PBS,二硫苏糖醇(DTT),碘乙酰胺(IAA),四乙铵(TEAB)(美国Sigma公司);乙腈(ACN)、尿素、甲酸(FA)、质谱级胰酶和N-PER神经蛋白提取液(Promega公司);iTRAQ试剂盒(美国AB Sciex公司);仪器:Nano-LC9(Thermo)-Q-TOF MS(德国Bruker),多功能酶标仪(美国Molecular Devices),真空冷冻干燥机(德国CHRIST)。
1.2 方法
1.2.1 细胞培养和收集 细胞接种后,在37℃、5% CO2条件下培养,使用含有10%血清和1%双抗的DMEM培养基,当细胞的融合度达到80%时随机分为实验组和对照组各3瓶分别在有无1 mmol/L的烟碱培养基中培养1 h后,酶解消化收集,使用磷酸缓冲盐溶液(PBS)清洗后加入蛋白提取液,BCA法蛋白定量。
1.2.2 蛋白定量标记 分别取100 μg细胞全蛋白每组做2次重复实验,定容到30 μL;加3倍体积(0.01 mol/L DTT、8 mol/L尿素、0.1 mol/L Tris-HCL(pH8.5))的还原剂,37℃水浴1 h;加入与还原剂等体积的烷基化试剂(0.05 mol/L IAA、8 mol/L尿素、0.1 mol/L Tris-HCL(pH8.5))室温避光20 min;将蛋白溶液移入10 kD的超滤管中离心40 min(12 000×g,4℃),将未反应液体全部离心;向每个超滤管中加入150 μL的0.3 mol/L TEAB离心40 min(12 000×g,4℃),将未反应的液体全部离心下去,重复3次;然后按20∶1比例加入胰蛋白酶37℃酶解16 h;将酶切好的样品室温下离心10 min(12 000×g,4℃),加100 μL的0.2 mol/L TEAB, 室 温 离 心20 min(12 000×g),重复上述步骤2次;酶解消化后的肽段溶液离心于收集管底;冷冻干燥后,向其中加入100 μL 0.2 mol/L TEAB 使其浓度为1 μg/μL充分混匀,向4管对应iTRAQ标签(分子量分别为114、115、116和117的标签)中加入200 μL乙醇,115、116标记对照组,114、117标记实验组;室温反应2 h,加400 μL色谱水终止反应30 min;混合后经seppark C18柱除盐,冻干。
1.2.3 肽段液相分离 将冻干后的样品用0.1%甲酸水溶液复溶上到Trap柱(100 μm×2 cm,C18,5 μm),由流动相A(0.1%甲酸水)和B(0.1%甲酸乙腈)经梯度(表1)洗脱到分析柱(75 μm×50 cm,C18,2 μm)。
1.2.4 质谱鉴定 液相色谱分离后进入Q-TOF MS(Bruker)鉴定。采用CaptiveSpray离子源,在正离子模式下,毛细管电压为4 500 V,干燥气体流速为4.0 L/min,干燥温度为180℃,固定循环扫描时间为3 s,质谱全扫描质量范围50-2 200 m/z,二级质谱全扫描质量范围50-2 200 m/z,碰撞能量范围为23-65 eV进行信号采集。
1.2.5 数据库检索 使用Mascot(Version 2.4.1)搜索引擎对Uniprot-human数据库搜库。搜库参数:最大漏切位点数2;半胱氨酸脲甲基化固定修饰、蛋氨酸可变氧化修饰、N端乙酰化可变修饰;定量:iTRAQ 4标;肽端允许误差:20 ppm;MS/MS允许误差:0.1 Da,显著阈值:P<0.05。
1.2.6 定量蛋白质组学和生物信息学分析 利用iTRAQ定量的比值(代表实验组/对照组平均值表达量比值的4个ratio值的平均值)筛选出暴露组和对照组的差异表达的蛋白。使用DAVID对差异蛋白质进行生物过程,细胞组成,生物功能进行分析,KEGG进行生物通路分析。搜索STRING数据库并构建差异蛋白互作模型。

表1 流动相梯度
2 结果
2.1 蛋白质组鉴定和iTRAQ定量分析
对1 mmol/L烟碱暴露1 h的SH-SY5Y进行iTRAQ差异蛋白组学分析,在烟碱暴露组和实验组中共鉴定到1 316种蛋白质,其中1 273种蛋白有定量信息,iTRAQ标记效率为96.7%,表明标记效率良好。对照组两次重复标记定量蛋白比值95%的置信区间分布在(1.091,1.147);实验组两次重复标记定量蛋白比值95%的置信区间分布在(1.099,1.142),表明重复性好,实验结果可靠。由于体系偏差的存在,为减少假阳性的结果,至少选择有两个定量肽段、|比值|≥1.5倍且单样本t检验P<0.05的蛋白为差异蛋白,共筛选出132种差异蛋白。
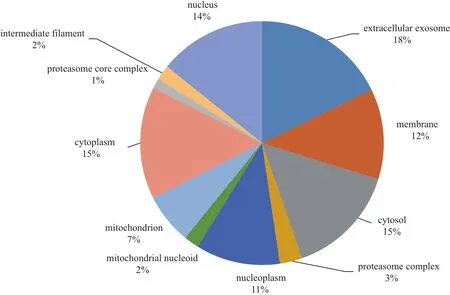
图2 差异蛋白亚细胞定位
2.2 差异蛋白功能分类分析
使用DAVID软件依据Gene Ontology条目对132个差异蛋白进行GO分析,这些蛋白质显著富集了58种生物学过程,30种细胞组成和22种分子功能。显著富集的细胞位置主要包括胞外体(45.54%),细胞质(43.56%),核(43.56%),膜(36.63%),线粒体(20.79%)和蛋白酶体复合物(8.91%)等(图2)。根据分子功能注释(图3)显著富集的分子功能主要为结合蛋白质和活性蛋白质。根据生物过程注释(图4),主要的生物过程为小分子代谢(27.7%),基因表达(25.7%),细胞氮化合物代谢过程(16.8%)和病毒过程(15.8%),此外还有GTP调节信号传导(12.8%),MAPKK激活(6.9%),MAPK级联(6.9%)和神经营养因子TRK信号通路(6.9%)等生物过程(图4)。

图3 差异蛋白分子功能分析

图4 差异蛋白生物过程分析
2.3 差异蛋白信号通路分析
对上述132种显著差异表达的蛋白质进行KEGG分析,参与的生物通路主要包括代谢途径,泛素化-蛋白酶体通路,精氨酸和脯氨酸代谢,系统性红斑狼仓和氨酰tRNA生物合成。此外,还有与细胞外粘附,连接相关的通路,和细胞内质网蛋白质加工,谷胱甘肽代谢,RNA转运,肌动蛋白细胞骨架,钙信号通路和cGMP-PKG信号通路,及神经疾病相关的酒精成瘾和亨廷顿病相关的通路。
2.4 差异蛋白互作分析
对上述132显著差异的蛋白质进行STRING蛋白质互作分析,117个蛋白质均与STRING数据库中的蛋白质匹配(图5)。该网络以(图5红色圈出蛋白)蛋白酶体家族、核糖体蛋白家族、真核翻译起始因子、氨酰t-RNA合成酶和乙醛脱氢酶18家族A1(表2)等蛋白质为中心,向四周扩散,在烟碱诱导的神经元细胞蛋白质的表达中发挥重要作用。

表2 网络互作中心蛋白
3 讨论
根据生物信息学分析的结果,位于互作网图交汇点的差异表达蛋白包括蛋白酶体家族,核糖体蛋白家族,真核翻译起始因子,氨酰-tRNA合成酶和乙醛脱氢酶18家族A1。差异表达的蛋白酶体蛋白主要包括PSMB1、PSMB2、PSMB5、PSMD2、PSMD12、PSMC6和PSMA1,这与Kane等[11]发现烟碱诱导大鼠前额皮质中蛋白酶体显著差异表达结果相似。这些差异表达的蛋白酶体蛋白主要参与MAPK级联生物过程和泛素-蛋白酶体通路。烟碱通过α7nAChR乙酰胆碱受体诱导MAPK级联反应[12-14],能够进一步激活其下游的长时程增强效应和长期记忆[15,16];泛素化-蛋白酶体通路中,蛋白酶体能够识别并清除泛素化的蛋白,维持烟碱诱导生物体产生蛋白质的稳态[11,17],长期的烟碱暴露将导致神经元突触可塑性[18]。差异表达的核糖体蛋白主要包括L27A、L8、L31、S23和S5,这与烟碱暴露小鼠皮层神经元的研究中发现一致[19]。差异表达的核糖体蛋白和酰胺酰t-RNA合成酶主要参与蛋白质的合成,这可能是由于蛋白合成上游的信号级联反应如蛋白激酶C和PI3K信号通路被烟碱抑制[19]。在安非他命急性诱导小鼠大脑区域的研究中,氨酰t-RNA合成酶基因同样存在差异表达,影响安非他命依赖[20]。因此,酰胺酰t-RNA合成酶对烟碱依赖的形成影响值得进一步的研究。值得注意的是,本研究发现的ALDH18A1显著差异表达,尚未见相关报道。多项研究指出,乙醛与烟碱共同暴露增强大鼠的烟碱依赖程度[21],由于ALDH18A1是乙醛氧化为乙酸反应的催化剂, ALDH18A1可能在烟碱和乙醛的协同依赖中发挥重要作用。

图5 差异蛋白互作网络分析
在神经退行性疾病的通路中,差异蛋白的表达包括超氧化物歧化酶(SOD1)、电压依赖阴离子通道1(VDAC1)和电压依赖阴离子通道3(VDAC3)。研究表明,烟碱对突触前后信号调节与神经退行性疾病亨廷顿病,帕金森综合症显著相关[22,23]。SOD1过表达在神经退行性疾病中具有保护作用,影响生物体氧化防御系统[24,25];VDAC1和VDAC3蛋白家族位于线粒体外膜上,VDAC缺失小鼠的学习能力和突触可塑性都有所减弱[26];另外,VDAC蛋白通过偶联乙酰胆碱受体,影响烟碱与烟碱乙酰胆碱受体的结合[27]。目前,烟碱对SOD1、VDAC1和VDAC3蛋白表达的影响还未见报道。
4 结论
本研究通过iTRAQ蛋白组学技术初步鉴定了烟碱诱导神经元蛋白质的差异表达,共鉴定筛选出132个差异表达蛋白,差异表达的蛋白质中蛋白酶体、真核翻译起始因子、氨酰-tRNA合成酶核糖体蛋白、ALDH18A1位于蛋白质互作的中心,在烟碱诱导神经元蛋白质的表达中发挥重要作用。但烟碱诱导差异表达的蛋白在神经元中的作用仍需要进一步验证,这些差异表达的蛋白在烟碱依赖的形成中如何发挥作用的,仍需要动物成瘾模型进行进一步的评估。
[1]Subramaniyan M, Dani JA. Dopaminergic and cholinergic learning mechanisms in nicotine addiction[J]. Annals of the New York Academy of Sciences, 2015, 1349(1):46-63.
[2]Prochaska JJ, Benowitz NL. The Past, present, and future of nicotine addiction therapy[J]. Annual Review of Medicine, 2015, 67(1):411-420.
[3]Wong CC, Mill J, Fernandes C. Drugs and addiction:an introduction to epigenetics[J]. Addiction, 2011, 106(3):480-490.
[4]Renthal W, Nestler EJ. Chromatin regulation in drug addiction and depression[J]. Dialogues in Clinical Neuroscience, 2009, 11(3):257-268.
[5]Hwang YY, Li MD. Proteins differentially expressed in response to nicotine in five rat brain regions:Identification using a 2-DE/MS-based proteomics approach[J]. Proteomics, 2006, 6(10):3138-3153.
[6]Yeom M, Shim I, Lee HJ, et al. Proteomic analysis of nicotineassociated protein expression in the striatum of repeated nicotinetreated rats[J]. Biochem Biophys Res Commun, 2005, 326(2):321-328.
[7]Hemby SE. Cocainomics:new insights into the molecular basis of cocaine addiction[J]. Journal of Neuroimmune Pharmacology, 2010, 5(1):70-82.
[8]Bodzon-Kulakowska A, Suder P, Mak P, et al. Proteomic analysis of striatal neuronal cell cultures after morphine administration[J]. J Sep Sci, 2009, 32(8):1200-1210.
[9]Benowitz NL. Nicotine addiction[J]. The New England Journal of Medicine, 2010, 362(24):311-631.
[10]Dajas-Bailador F, Wonnacott S. Nicotinic acetylcholine receptors and the regulation of neuronal signalling[J]. Trends in Pharmacological Sciences, 2004, 25(6):317-324.
[11]Kane JK, Konu Ö, Ma JZ, et al. Nicotine coregulates multiple pathways involved in protein modification/degradation in rat brain[J], Molecular Brain Research. 2004, 132(2):181-191.
[12]Arora K, Cheng J, Nichols RA. Nicotinic acetylcholine receptors sensitize a MAPK-linked toxicity pathway on prolonged exposure to β-Amyloid[J]. Journal of Biological Chemistry, 2015, 290(35):21409-21420.
[13]Schaal C, Chellappan SP. Nicotine-mediated cell proliferation and tumor progression in smoking-related cancers[J]. Molecular Cancer Research, 2014, 12(1):14-23.
[14]Tang K, Wu H, Mahata SK, et al. A crucial role for the mitogenactivated protein kinase pathway in nicotinic cholinergic signaling to secretory protein transcription in pheochromocytoma cells[J]. Molecular Pharmacology, 1998, 54(1):59-69.
[15]Picciotto MR. Nicotine-mediated activation of signal transduction pathways[J]. Novartis Foundation Symposium, 2008, 275(2):83-95.
[16]Lykhmus O, Mishra N, Koval L, et al. Molecular mechanisms regulating LPS-induced inflammation in the brain[J]. Frontiers in Molecular Neuroscience, 2016, 19(9):1-13.
[17]Li MD, Kane JK, Wang J, et al. Time-dependent changes in transcriptional profiles within five rat brain regions in responseto nicotine treatment[J]. Brain Research Molecular Brain Research, 2004, 132(2):168-180.
[18]Ehlers MD. Activity level controls postsynaptic composition and signaling via the ubiquitin-proteasome system[J]. Nature Neuroscience, 2003, 6(3):231-242.
[19]Wang J, Gutala R, Sun D, et al. Regulation of platelet-derived growth factor signaling pathway by ethanol, nicotine, or both in mouse cortical neurons[J]. Alcoholism Clinical & Experimental Research, 2007, 31(3):357-375.
[20]Sokolov BP, Polesskaya OO, Uhl GR. Mouse brain gene expression changes after acute and chronic amphetamine[J]. Journal of Neurochemistry, 2003, 84(2):244-252.
[21] Belluzzi JD, Wang R, Leslie FM. Acetaldehyde enhances acquisition of nicotine self-administration in adolescent rats[J]. Neuropsychopharmacology, 2005, 30(4):705-712.
[22]Quik M, Perez XA, Bordia T. Nicotine as a potential neuroprotective agent for Parkinson's disease[J]. Movement Disorders, 2012, 27(8):947-957.
[23]Dineley KT. Nicotinic acetylcholine receptors in Alzheimer’s and Parkinson’s disease[M]. Springer New York, 2014:383-415.
[24]Song G, Nesil T, Cao J, et al. Nicotine mediates expression of genes related to antioxidant capacity and oxidative stress response in HIV-1 transgenic rat brain[J]. Journal of Neurovirology, 2015, 22(1):114-124.
[25]Lin C, Yona JM, Lee BJ, et al. Antiteratogenic effects of β-Carotene in cultured mouse embryos exposed to nicotine[J]. Evidencebased Complementary and Alternative Medicine, 2013, 2013(2):70-75.
[26]王红霞, 何家田, 刘炳玉, 等. 电压依赖性阴离子通道蛋白1和G蛋白N端乙酰化的纳升电喷雾串联质谱分析[C]. 中国蛋白质组学学术大会, 2005.
[27]Lykhmus O, Gergalova G, Koval L, et al. Mitochondria express several nicotinic acetylcholine receptor subtypes to control various pathways of apoptosis induction[J]. International Journal of Biochemistry & Cell Biology, 2014, 53(7):246-252.
(责任编辑 李楠)
iTRAQ-based Quantitative Proteomic Analyses of Differentially Expressed Proteins in Nicotine-induced SH-SY5Y Cells
ZHU Bei-bei LI Xiang-yu CHEN Huan WANG Hong-juan HOU Hong-wei HU Qing-yuan
(China National Tobacco Quality Supervision & Test Centre,Zhengzhou 450001)
SH-SY5Y,induced by nicotine,was used to seek the effects of nicotine exposure on the alteration of proteins expression profile. The differentially expressed proteins in nicotine-induced neuronal cells were identified using the iTRAQ and nano-LC-Q-TOF MS proteomics,and their bioinformatics was also analyzed. The results showed that the levels of 132 proteins were differentially altered in response to nicotine(| ratio | ≥ 1.5,P<0.05),including proteins involved in gene expression,MAPK cascade,GTP signal transduction and neurotrophin TRK receptor signaling. Together,our data suggested that the nicotine might elicit dependence by altering the neuronal balance of proteasome subunits,with the MAPK cascade and ubiquitin-proteasome pathway playing a pivotal role.
differential proteomics;iTRAQ;nicotine;neuronal SH-SY5Y
10.13560/j.cnki.biotech.bull.1985.2017.04.012
2016-10-19
中国烟草总公司科技重点项目(110201402037)
朱贝贝,女,硕士研究生,研究方向:吸烟与健康及生物标志物;E-mail:beibeizhus@126.com
侯宏卫,男,研究员,研究方向:吸烟与健康及生物标志物;E-mail:qsfctc@163.com胡清源,男,研究员,研究方向:烟草化学研究及烟草质量监督检验;E-mail:huqy1965@163.com
