禽流感病毒HA蛋白与人受体的分子对接
2017-05-10邓迎春柳青黄强
邓迎春 柳青 黄强
(复旦大学生命科学学院,上海200438)
禽流感病毒HA蛋白与人受体的分子对接
邓迎春 柳青 黄强*
(复旦大学生命科学学院,上海200438)
血凝素(hemagg lutinin,HA)是位于禽流感病毒表面的糖蛋白。在病毒感染过程中,HA与禽类宿主细胞表面受体结合,介导病毒膜与宿主核内体膜的融合,在传染过程中发挥关键作用。自然界中的禽流感病毒处于不断演化之中,其HA的禽受体结合位点常常发生氨基酸变异。因此,当HA变异体与人受体结合能力较强时,禽流感病毒往往会发生跨种传播而感染人。为预防禽流感的跨种传播,人们迫切需要发展大规模快速检测或预测HA变异体与人受体结合亲和力的方法,以评估各种新发禽流感病毒的跨种传播能力,提前筛选出有潜在危险的病毒株。针对此问题,本研究以H7N9亚型的HA蛋白H7为研究对象,发展了一种运用分子对接的计算方法,预测HA变异体与人受体的结合亲和力。该方法的计算结果表明,H7与人受体的结合亲和力普遍弱于有较强传染人能力的H1,说明H7N9亚型病毒的跨种传播能力普遍较弱;但是,计算分析也揭示,部分新发的H7N9毒株的HA有强的人受体结合亲和力,提示在自然演化过程中,H7N9病毒有可能演化出具有较强的感染人能力的新毒株,这与2013年禽流感疫情的实际发生情况相一致。因此,本文所发展的计算方法可用于快速预测新发禽流感病毒HA与人受体的结合亲和力,为新发禽流感病毒的跨种传播风险评估提供理论依据。
禽流感病毒;跨种传播;受体结合特异性;分子对接;结合自由能
1 引言
禽流感是一种由禽流感病毒引起的鸟禽类传染病。野生水禽是禽流感病毒的自然宿主,禽流感主要在禽类中流行。但是,当禽流感病毒的基因组发生一定变异后,病毒会发生跨种传播而感染人,甚至引发人类流感大流行。例如,1918年H1N1西班牙流感、1957年H2N2亚洲流感、1968年H3N2香港流感、1977年H1N1墨西哥流感和2009年H1N1猪流感1-5。其中最严重的1918年流感造成的死亡人数达五千万6。因此,对禽流感病毒跨种传播机制的研究,有助于更好地对禽流感进行防控。
血凝素(HA)蛋白是禽流感病毒表面的一种糖蛋白。其在病毒感染宿主细胞过程中发挥关键作用:与宿主细胞表面糖蛋白末端的特异性糖链结合,介导病毒膜与宿主核内体膜的融合7。HA蛋白是由HA 1链和HA 2链组成的三聚体。HA蛋白可被分为两个结构域(图1):远离病毒外膜的球状结构域和靠近外膜的茎状结构域。受体结合域位于球状结构域的顶端。它状如浅口袋,边缘是3个二级结构单元:130环(130-loop)、190螺旋(190-helix)、220环(220-loop)(H3亚型氨基酸序号);底部是4个保守氨基酸(Y98,W 153,H183,Y195,H3亚型氨基酸序号)7。
HA蛋白结合域一般与禽受体a2,3唾液酸(a2,3 term inal SIA,SAa2,3Gal)结合。但是,当HA蛋白基因组发生变异后,HA可能与人受体a2,6唾液酸(a2,6 term inal SIA,SAa2,6Gal)结合。病毒因此发生跨种传播而感染人。HA蛋白受体结合特异性的转换是病毒产生人适应性的关键特性之一7。HA蛋白受体结合特异性极大地影响禽流感病毒的宿主范围8。HA受体结合特异性从禽受体转为人受体,是出现致疫情病毒的先决条件9。HA蛋白一两个氨基酸置换突变就足以导致这种转换9,10。H1的E190D/G225D突变,H2/H3的Q226L/G228S突变,H7N9的Q226L/I、G186V突变都导致了其受体结合特异性的转换9,11-14。突变可能使HA蛋白与人受体之间相互作用发生改变。突变后人受体糖环能与H1第190位D和225位D之间形成氢键15,16。突变后H2/H3第226位L创造一个疏水的环境使HA与带C6疏水原子的人受体结合;第228位S与人受体末端SA1形成氢键17,18。
图1 HA蛋白和人受体类似物结构图Fig.1 Structuresof HA and LSTc(a)HA(PDB code:4JTV);(b)human-like receptoranalogue(LSTc)
目前人们主要通过实验方法和理论计算方法来检测禽流感病毒HA与人受体的相互作用。实验方法能较为准确地分析病毒株与人受体的结合能力19,20。但是,自然界中的禽流感病毒多样性丰富,变异频率高,不断有新的毒株产生,使用实验方法分析每一毒株的结合能力有一定难度。所以,能进行大规模快速预测的理论计算方法是实验手段的有力补充。通过预测方法的前期计算筛选,可以有效地缩小需进一步实验分析的病毒株数目。而且,随着二代测序的应用,人们可以快速地获得HA蛋白的氨基酸序列。所以,是否可以发展一种从氨基酸序列出发构建HA三维结构模型,快速预测HA蛋白与人受体结合能力的理论计算方法,以便人们可以有效地发现危险毒株?针对此问题,本文发展了一种计算预测方法,通过分子对接来计算H7亚型病毒的HA与LSTc的相互作用,预测病毒HA与人受体的结合能力,分析其与人受体的结合特异性,以快速地预测新发禽流感病毒感染人的潜力。
2 计算方法
2.1 分子对接系统的构建
本文选择了H1和H7禽流感病毒亚型的HA蛋白作为研究对象。我们从蛋白质结构数据库(Protein Data Bank)下载了H1和部分H7的HA蛋白晶体结构,对HA蛋白晶体结构进行去水去杂原子处理,对只有单亚基的晶体结构运用PyMOL对称性命令构建三聚体,对亚基个数超过3的晶体结构删除多余的亚基,获得HA蛋白的标准三聚体模型;从GISAID数据库(http://platform.gisaid.org/)和GenBank数据库(http://www.ncbi.nlm.nih.gov/genbank/)下载了部分H7N9亚型HA蛋白的氨基酸序列,然后通过同源建模方法21构建HA蛋白的三维结构模型。共获得12个H1晶体结构、37种H7N9毒株HA蛋白的三维结构模型、2种H7N9毒株HA蛋白晶体结构和1种H7野生毒株晶体结构(见Supporting Information表S1)。
为构建精细准确的对接系统,本研究的H1,以H1与LSTc的复合物晶体结构4JTV为参考;从4JTV中分离出C链的人受体类似物LSTc,作为后续构建对接系统的配体,其余H1蛋白通过PyMOL软件被align到4JTV的坐标体系中,另存H1蛋白于4JTV坐标体系中,因此配体LSTc位于蛋白质受体结合域。以此方法构建H1亚型的分子对接系统。H7以A/Anhui/01/2013(PDB code:4BSE)为参考构建分子对接系统。
2.2 分子对接
AutoDock程序在生物分子复合物的结构和功能分析及分子设计方面被广泛地用以预测22-24。本研究使用了AutoDock4.224进行分子对接研究。以禽流感病毒的HA蛋白为受体,LSTc为配体构建分子对接系统。由于在建模配体-蛋白相互作用中,水的表现和角色非常重要25,加水对接能明显地提高对接的能量估计精度和对接性能25,我们在对接中给配体加了水,模拟配体分子在水环境中与受体结合的过程。对接参数设置为,配体可旋转键12个;将LSTc中唾液酸C8原子坐标设为格子中心;格子大小设置为X,Y,Z:80,80,80;对接次数为2000,每个蛋白对接模拟2000次,其它参数为系统默认。对接结束后对于每个蛋白我们将获得配体的2000个不同构象与HA蛋白的结合自由能。结合构象和结合自由能将作为后续处理和分析的依据。分子对接流程见图2。
3 结果与讨论
3.1 最低结合自由能分析
本文选择的H1毒株与人受体的结合能力都很强15-17,26-28。以3UBE15为例,它是引发2009年H1N1流感的毒株A/California/04/2009的晶体结构。H1N1曾导致多次全球疫情大流行,死亡人数众多。它在人与人之间传播的效率证明2009H1N1完全适应了在人与人之间传播29,30。糖微阵列研究和ELISA实验表明人型H1N1只与SAa2,6Gal结合,几乎不与SAa2,3Gal结合15,具有很强的人受体偏好性。本文选择的39个H7N9毒株是从Lam等31的文章附录文件中选择的,我们尽量在附录中选择传人的毒株来做分子对接。野生型H7毒株A/ turkey/Italy/214845/2002(H7N3)(PDB code:4BSI)13不感染人。

图2 HA蛋白变异体组的分子对接流程图Fig.2 M olecu lar dock ing flow chart of HA variants
我们将39种H7N9对接后的最低结合自由能列于表1。我们发现39种H7N9毒株的最低结合自由能在-53.3至-43.8 kJ·mol-1之间。较高结合自由能的毒株有A/Nanjing/5/2013,A/Jiangsu/09/2013等,较低结合自由能的毒株有A/Guangdong/05/ 2013,A/Hong Kong/734/2014等。最高能量毒株为A/Nanjing/5/2013,其最低结合自由能为-43.8 kJ· mol-1,最低能量毒株是A/Guangdong/05/2013,其最低结合自由能为-53.3 kJ·mol-1。H1N1亚型A/ California/04/2009(3UBE)毒株的最低结合自由能为-51.2 kJ·mol-1。而39种H7N9毒株中,有12/39约31%的毒株最低结合自由能大于-46.1 kJ· mol-1,大部分25/39约64%的毒株的最低结合自由能在-50.2至-46.1 kJ·mol-1之间,仅有2/39约5%小于-50.2 kJ·mol-1。H7N9病毒的HA与人受体的结合亲和力普遍弱于有较强传染人能力的H1N1,说明H7N9亚型病毒的跨种传播能力普遍较弱。但是有些H7N9中毒株的最低结合自由能接近H1N1,表明H7N9在进化过程中可能会演化出感染人能力较强的毒株,值得注意。在文献14中确认的8种致死毒株中,6/8约75%的最低结合自由能小于等于-46.1 kJ·mol-1(表2)。野生型H7的最低结合自由能较高,为-46.7 kJ·mol-1。
3.2 构象分类
由于人受体末端糖环SA1与HA结合域底部保守氨基酸153W之间有范德华力7。而糖环GlcNAc3一端连接着宿主糖蛋白,不太可能与153W靠得太近。所以合理构象中HA蛋白的153W残基离SA1距离近,离GlcNAc3远。我们计算了HA 153W残基CH2原子与SA1糖环C5原子的距离D_SIA,以及HA 153W残基CH2原子与GlcNAc3糖环C1原子的距离D_NAG。以前者为横坐标,后者为纵坐标,遍历AutoDock结果中所有可能构象,以能量最低的构象颜色为红,能量最高的构象颜色为蓝,能量介于两者之间的构象按能量高低做渐变色图,见图3(a)。经计算,A/California/ 04/2009 HA和LSTc复合物晶体结构3UBE中D_SIA约为0.481 nm,D_NAG约为1.088 nm。因此我们舍弃了D_NAG小于0.8 nm的构象,这部分构象结合方向为无效结合方向。我们将这部分构象称为无效构象,将D_NAG大于等于0.8 nm的构象称为有效构象。文中后续处理对象皆为有效构象。以有效构象中结合自由能最低的构象为参考,计算其余有效构象与最低能量构象的均方根偏差(RMSD)。然后以每个构象的结合自由能为纵坐标,以每个构象与最低能量构象的RMSD为横坐标,以最低能量颜色为红,最高能量颜色为蓝,中间能量构象颜色在红和蓝之间渐变,作HA蛋白对接的能量分布散点图(energy distribution scatter diagram),见图3(b)。靠近最低结合自由能构象的点出现得越多,表明对接中出现低结合自由能的概率越大,此HA蛋白与人受体越容易结合,亲和力越强。我们看到H1与人受体的结合能力强,其有很多构象在结合自由能和RMSD方面有接近能量最低的构象的趋势。图3(c)列举了部分趋势上接近最低能量的构象。能量小于-41.9 kJ· mol-1,RMSD小于0.4 nm的构象被列出。可以看到结合的可能构象有很多。

表1 H 7与LSTc的最低结合自由能Tab le1 Lowestbinding freeenergy of HA and LSTc

表2 8种致死的H7N9毒株HA的最低结合自由能Table2 The lowestbinding freeenergy of HA from 8 fatalH7N9 isolates
依据对接后的结合自由能和构象,我们对H7亚型的每一个HA蛋白作了对接能量分布散点图。我们发现39种H7N9毒株中,文献14中确定致死的8种毒株,有7种的能量变化和H1N1类似,分子对接预测正确率约为87.5%(图4)。A/Shanghai/1/ 2013和A/Anhui/1/2013是H7N9刚爆发时的毒株,已有结晶结构。其中对A/Anhui/1/2013毒株的研究发现其HA与人受体的结合力比野生型H7强,可能是通过Q226L和G186V突变获得了两个大的疏水残基;前一个突变与1957年的H2和1968年的H3的疫情病毒一样;H7获得了典型大流行禽流感毒株的一些受体结合特征13。A/Anhui/1/2013毒株被WHO推荐为候选的疫苗病毒32。研究发现A/Anhui/ 1/13 HA与人受体有一定的结合能力,如果再发生G228S单基因突变,能极大地提高HA与人受体的结合能力33。毒株A/Shanghai/1/2013能量分布亦与H1N1类似,其虽保留了226Gln,与人受体亦有结合能力20。一种致死毒株A/Jiangsu/01/2013的对接结果能量分布与H1N1类似度相对小,可能是由于HA三维结构建模存在偏差。

图3 构象分类Fig.3 Conform ational classification(a)all conformations,D_SIA is the distancebetween HA and SA 1, D_NAG is the distance between HA and GlcNAc3;(b)reasonable conformations;(c)binding posewhoseΔG<-41.9 kJ·mol-1and root-mean-square deviation(RMSD)<0.4 nm.color online

图4 8种致死的H 7N9毒株HA的对接能量分布图Fig.4 Energy distribution scatter diagram of HA from 8 fatalH 7N9 isolates*Thepattern of thepicturesis similar to H1 except(f).
剩余的31种H7N9毒株中,有些确定是从病人体内提取的样本,有些没有查询到相关文献。31种毒株的对接能量分布有些与H1N1能量分布类似,有些不类似。其中A/Zhejiang/22/2013是病人毒株,其HA也发生了Q226L置换突变34(图5(b))。把A/Anhui/02/2013与A/Anhui/01/2013和A/Anhui/ 03/2013比较发现(图5(a)):其中A/Anhui/01/2013和A/Anhui/03/201314是致死的,对接能量分布与H1N1类似并且能量偏低;而A/Anhui/02/2013不致死,其对接能量分布与H1N1类似,但结合自由能较高。说明有些H7N9毒株与人受体的对接能量分布与H1N1类似和接近,H7N9在演化过程中有可能产生与人受体具有较高亲和力的毒株。
根据世界卫生组织的报告,自2013年以后,H7N9禽流感病毒导致571人感染,212人死亡32。H7N9虽然获得了典型大流行禽流感毒株的一些受体结合特征,但是受体结合特异性依然是禽受体偏好,限制了H7N9在人与人之间传播13。A/ Shanghai/1/2013能与人受体结合,但依然偏好禽受体20。H7N9 HA与人受体的结合力与典型大流行H1毒株相比仍存在差距,与典型大流行毒株的高效气溶胶传播相比,该差距影响了H7N9通过气溶胶有效传播33。
3.3 亲和力-特异性图
由于对接后LSTc有很多可能构象,没有唯一最优构象,所以我们定义了一个特异性函数S来进行统计分析HA蛋白与人受体的亲和力。根据范特霍夫等温公式ΔG=ΔG0+RT ln J(其中ΔG为吉布斯自由能,ΔG0为同一温度和标准压强下的吉布斯自由能,R为气体常数,T为热力学温度),等温等压条件下,当反应达到平衡时,ΔG为0,J变为平衡常数。于是有ΔG0=-RT ln K,平衡常数K=exp(-ΔG0/RT)。我们用有效构象中最低结合自由能构象的平衡常数,除以所有有效构象的平衡常数之和,表示此HA蛋白最低构象出现的概率,代表此HA蛋白与人受体的结合特异性:
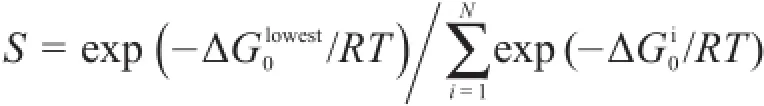
依据对接后的结合自由能和构象,我们用上述方法计算出每一个HA蛋白与人受体的特异性,与最低结合自由能作图,H1、H7N9和野生型H7分别被标记成绿色、红色和蓝色,得到图6。由图6可以看出,禽流感病毒HA变异体组与LSTc结合能力的整体趋势是图中箭头所指方向。HA蛋白与人受体LSTc的结合自由能越低,同时特异性越高,说明HA蛋白与人受体结合时最低能量构象出现的概率就越高,由此推断HA与人受体的亲和力越强。从趋势上看,H7N9与人受体的亲和力普遍低于具有较高感染人能力的H1N1,说明H7N9传染人的能力较弱;但有些H7N9毒株与人受体的结合力接近H1N1,说明H7N9在演化过程中可能会演化出具有较高感染人能力的毒株,这与2013年H7N9禽流感疫情的实际发生情况一致。Shi等20用H1N1 A/California/04/2009作为人受体特异性对照,野生型H5N1A/Anhui/1/2005作为禽受体特异性对照,研究H7N9 A/Anhui/1/2013、H7N9 A/ Shanghai/1/2013的受体结合特异性,发现H7N9A/ Anhui/1/2013和H7N9 A/Shanghai/1/2013既可以与禽受体也可以与人受体结合,但是二者与禽受体结合的特异性更强。野生型H7用蓝色标出,特异性明显较低。

图6 亲和力-特异性(S)图Fig.6 A ffinity-specificity(S)figuregreen points:H1;red points:H7N9;bluepoint:w ild type H7. coloronline
本文预测的是H7N9亚型与人受体结合的整体趋势,只有大规模计算所呈现的整体趋势才具有统计意义。针对具体毒株,亦需要精确建模和多次运算以保证结果的可靠性。由于二代测序技术的成熟应用,发展一种从氨基酸序列建三维模型着手,通过分子对接计算HA蛋白与LSTc的结合力,预测新发流感感染人能力的方法十分有意义。本预测系统可以应用于新发禽流感病毒感染人能力的快速预测。不过,对建模精度加以改善,并在对接前逐一检查每个建模模型,将可以增加预测的精确性和准确性。综上,本文为快速评估预测禽流感病毒感染人的能力提供了新方法新思路。
4 结论
禽流感病毒多样性丰富,变异频率高,不断有新的毒株产生。理论计算方法可以高通量大规模计算,是实验方法的有力补充。随着二代测序的成熟应用,发展一种通过氨基酸序列建HA蛋白三维结构模型,然后通过高通量计算快速预测禽流感病毒HA蛋白与人受体结合能力的方法,提前筛选出危险毒株,有利于优先针对危险毒株安排禽流感防控措施,防止禽流感病毒的跨种传播。本文通过分子对接方法,对禽流感HA蛋白-人受体的相互作用进行计算预测。我们首先对氨基酸序列进行同源建模,获取HA蛋白的三维结构模型;然后通过对接后的能量分布和特异性分析,对H7蛋白各变异体与LSTc的亲和力进行定量计算和分析比较。结果显示,H7N9与人受体的亲和力普遍弱于具有较高感染人能力的H1N1;但有些H7N9毒株与人受体的亲和力较强,说明H7N9演化过程中可能会产生感染人能力较强的毒株,与2013年H7N9禽流感疫情发生情况一致。所以本文的计算预测方法能够预测HA蛋白变异体与人受体的相互作用,快速预测新发禽流感病毒感染人的风险,为非人宿主禽流感病毒的风险评估提供理论依据。
致谢:感谢复旦大学生命科学学院黄强教授实验室的袁慧同学对全文进行了仔细修改,感谢汤洪海同学、王国栋同学、钱嘉强同学提出的宝贵意见。
Supporting In formation:The structuresof HA formolecular docking havebeen included.This information is available freeof charge via the internetatwww.whxb.pku.edu.cn.
(1)Taubenberger,J.K.;M orens,D.M.Rev.Sci.Tech.2009,28(1), 187.doi:10.20506/rst.28.1.1879
(2)dos Reis,M.;Hay,A.J.;Goldstein,R.A.J.Mol.Evol.2009,69 (4),333.doi:10.1007/s00239-009-9282-x
(3)Neumann,G.;Noda,T.;Kawaoka,Y.Nature 2009,459(7249), 931.doi:10.1038/nature08157
(4)Smith,G.J.;Vijaykrishna,D.;Bahl,J.;Lycett,S.J.;Worobey, M.;Pybus,O.G.;Ma,S.K.;Cheung,C.L.;Raghwani,J.; Bhatt,S.;Peiris,J.S.M.;Guan,Y.;Rambaut,A.Nature 2009, 459(7250),1122.doi:10.1038/nature08182
(5)Nakajima,K.;Desselberger,U.;Palese,P.Nature 1978,274, 334.doi:10.1038/274334a0
(6)Johnson,N.P.;Mueller,J.Bull.Hist.Med.2002,76(1),105. doi:10.1353/bhm.2002.0022
(7)Skehel,J.J.;W iley,D.C.Annu.Rev.Biochem.2000,69,531. doi:10.1146/annurev.biochem.69.1.531
(8)M atrosovich,M.;Stech,J.;K lenk,H.D.Rev.Sci.Tech.Off.Int. Epiz.2009,28(1),203.doi:10.20506/rst.28.1.1870
(9)Matrosovich,M.;Tuzikov,A.;Bovin,N.;Gambaryan,A.; K limov,A.;Castrucci,M.R.;Donatelli,I.;Kawaoka,Y.J.Virol. 2000,74(18),8502.doi:10.1128/JVI.74.18.8502-8512.2000
(10)Tumpey,T.M.;Maines,T.R.;Van Hoeven,N.;Glaser,L.; Solorzano,A.;Pappas,C.;Cox,N.J.;Swayne,D.E.;Palese,P.; Katz,J.M.;Garcia-Sastre,A.Science 2007,315,655. doi:10.1126/science.1136212
(11)Connor,R.J.;Kawaoka,Y.;Webster,R.G.;Paulson,J.C.; Virology 1994,205,17.doi:10.1006/viro.1994.1615
(12)Stevens,J.;Blixt,O.;Tumpey,T.M.;Taubenberger,J.K.; Paulson,J.C.;Wilson,I.A.Science 2006,312,404. doi:10.1126/science.1124513
(13)Xiong,X.L.;Martin,S.R.;Haire,L.F.;Wharton,S.A.; Daniels,R.S.;Bennett,M.S.;M cCauley,J.W.;Collins,P.J.; Walker,P.A.;Skehel,J.J.;Gamblin,S.J.Nature2013,499, 496.doi:10.1038/nature12372
(14)Wang,D.;Yang,L.;Gao,R.;Zhang,X.;Tan,Y.;Wu,A.;Zhu, W.;Zhou,J.;Zou,S.;Li,X.Y.;Sun,Y.;Zhang,Y.;Liu,Y.;Liu, T.;Xiong,Y.;Xu,J.;Chen,L.;Weng,Y.;Qi,X.;Guo,J.;Li,X. D.;Dong,J.;Huang,W.;Zhang,Y.;Dong,L.;Zhao,X.;Liu,L.; Lu,J.;Lan,Y.;Wei,H.;Xin,L.;Chen,Y.;Xu,C.;Chen,T.; Zhu,Y.;Jiang,T.;Feng,Z.;Yang,W.;Wang,Y.;Zhu,H.;Guan, Y.;Gao,G.F.;Li,D.;Han,J.;Wang,S.;Wu,G.;Shu,Y.Euro. Surveill.2014,19(25).doi:10.2807/1560-7917. ES2014.19.25.20836
(15)Xu,R.;M cbride,R.;Nycholat,C.M.;Paulson,J.C.;W ilson,I. A.J.Virol.2012,86(2),982.doi:10.1128/JVI.06322-11
(16)Zhang,W.;Shi,Y.;Qi,J.;Gao,F.;Li,Q.;Fan,Z.;Yan,J.;Gao, G.F.J.Virol.2013,87(10),5949.doi:10.1128/JVI.00545-13
(17)Liu,J.;Stevens,D.J.;Haire,L.F.;Walker,P.A.;Coombs,P.J.; Russell,R.J.;Gamblin,S.J.;Skehel,J.J.Proc.Natl.Acad.Sci. U.S.A.2009,106(40),17175.doi:10.1073/pnas.0906849106
(18)Eisen,M.B.;Sabesan,S.;Skehel,J.J.;Wiley,D.C.Virology 1997,232(1),19.doi:10.1006/viro.1997.8526
(19)Liao,H.Y.;Hsu,C.H.;Wang,S.C.;Liang,C.H.;Yen,H.Y.; Su,C.Y.;Chen,C.H.;Jan,J.T.;Ren,C.T.;Chen,C.H.; Cheng,T.J.;Wu,C.Y.;Wong,C.H.J.Am.Chem.Soc.2010, 132(42),14849.doi:10.1021/ja104657b
(20)Shi,Y.;Zhang,W.;Wang,F.;Qi,J.;Wu,Y.;Song,H.;Gao,F.; Bi,Y.;Zhang,Y.;Fan,Z.;Qin,C.;Sun,H.;Liu,J.;Haywood,J.; Liu,W.;Gong,W.;Wang,D.;Shu,Y.;Wang,Y.;Yan,J.;Gao, G.F.Science 2013,342(6155),243.doi:10.1126/ science.1242917
(21)Deng,Y.L.;Yu,L.;Huang,Q.Acta Phys.-Chim.Sin.2016,32 (9),2355.[邓玉玲,余璐,黄强.物理化学学报,2016,32 (9),2355.]doi:10.3866/PKU.WHXB201605171
(22)Goodsell,D.S.;Olson,A.J.Proteins:Struct.Funct.Genet. 1990,8(3),195.doi:10.1002/prot.340080302
(23)Huey,R.;Morris,G.M.;Olson,A.J.;Goodsell,D.S.J. Comput.Chem.2007,28(6),1145.doi:10.1002/jcc.20634
(24)Morris,G.M.;Huey,R.;Lindstrom,W.;Sanner,M.F.;Belew, R.K.;Goodsell,D.S.;Olson,A.J.J.Comput.Chem.2009,30 (16),2785.doi:10.1002/jcc.21256
(25)Forli,S.;Olson,A.J.J.Med.Chem.2012,55(2),623. doi:10.1021/jm2005145
(26)Gamblin,S.J.;Haire,L.F.;Russell,R.J.;Stevens,D.J.;Xiao, B.;Ha,Y.;Vasisht,N.;Steinhauer,D.A.;Daniels,R.S.;Elliot, A.;Wiley,D.C.;Skehel,J.J.Science 2004,303(5665),1838. doi:10.1126/science.1093155
(27)Xu,R.;Ekiert,D.C.;K rause,J.C.;Hai,R.;Crowe,J.E.,Jr.; Wilson,I.A.Science2010,328(5976),357.doi:10.1126/ science.1186430
(28)Yang,H.;Carney,P.;Stevens,J.PLoSCurr.2010,2,RRN1152. doi:10.1371/currents.RRN1152
(29)Fraser,C.;Donnelly,C.A.;Cauchemez,S.;Hanage,W.P.;Van Kerkhove,M.D.;Hollingsworth,T.D.;Griffin,J.;Baggaley,R. F.;Jenkins,H.E.;Lyons,E.J.;Jombart,T.;H insley,W.R.; Grassly,N.C.;Balloux,F.;Ghani,A.C.;Ferguson,N.M.; Rambaut,A.;Pybus,O.G.;Lopez-Gatell,H.;A lpuche-A randa, C.M.;Chapela,I.B.;Zavala,E.P.;Guevara,D.M.;Checchi, F.;Garcia,E.;Hugonnet,S.;Roth,C.Science2009,324(5934), 1557.doi:10.1126/science.1176062
(30)Munayco,C.V.;Gomez,J.;Laguna-Torres,V.A.;Arrasco,J.; Kochel,T.J.;Fiestas,V.;Garcia,J.;Perez,J.;Torres,I.;Condori, F.;Nishiura,H.;Chowell,G.Euro.Surveill.2009,14(32),6.
(31)Lam,T.T.;Zhou,B.;Wang,J.;Chai,Y.;Shen,Y.;Chen,X.; Ma,C.;Hong,W.;Chen,Y.;Zhang,Y.;Duan,L.;Chen,P.; Jiang,J.;Zhang,Y.;Li,L.;Poon,L.L.;Webby,R.J.;Smith,D. K.;Leung,G.M.;Peiris,J.S.;Holmes,E.C.;Guan,Y.;Zhu,H. Nature 2015,522(7554),102.doi:10.1038/nature14348
(32)http://www.who.int/influenza/human_animal_interface/ influenza_h7n9/RiskAssessment_H7N9_23Feb20115.pdf?ua=1 (33)Tharakaraman,K.;Jayaraman,A.;Raman,R.;Viswanathan,K.; Stebbins,N.W.;Johnson,D.;Shriver,Z.;Sasisekharan,V.; Sasisekharan,R.Cell2013,153(7),1486.doi:10.1016/j. cell.2013.05.034
(34)Ding,H.;Chen,Y.;Yu,Z.;Horby,P.W;Wang,F.;Hu,J.;Yang, X.;Mao,H.;Qin,S.;Chai,C.;Liu,S.;Chen,E.;Yu,H.BMC Infect.Dis.2014,14,698.doi:10.1186/s12879-014-0698-6
Molecular Docking of Human-Like Receptor to Hemagglutinins of Avian Influenza A Viruses
DENG Ying-Chun LIU Qing HUANG Qiang*
(School ofLife Sciences,Fudan University,Shanghai200438,P.R.China)
Hemagglutinin(HA)is a glycopro tein located on the surface of the avian influenza A viruses.HA plays a key ro le in the in fection process,binding to receptors on the hostcellsurface andmediating the fusion between vira land hostendosomalmembranes.In nature,in fluenza A virus undergoes continuous variation, particularly the am ino acid sequence at the receptorbinding site ofHA.When the binding ability ofHA variants towards human receptors becomes strong,influenza A virus can infecthumans.To prevent the influenza A virus from infecting humans,proper assessments of the infectious risk posed are urgently needed.Screening ofhigh risk virus strains by ana lyzing the binding ability ofHA variants forhuman receptors through a high-throughput method would be particularly usefu l.In this study,we used H7(a subtype ofHA)as a subjectand deve loped a molecu lar docking based theo retica l calculation m ethod to evaluate the a ffinity of HA variants for human receptors.The results showed that the binding affinity ofH7 forhuman receptors is lower than thatofH1,which shows a strong ability to infecthumans.This resultsuggests thatstrains of the H7 subtype are generally weakly in fectious in humans.Nevertheless,the ca lcu lation resu lts also showed that some new ly-found virus strainsof the H7N9 subtype have a high binding affinity forhuman receptors,suggesting that the H7N9 subtypem ight include strainsw ith a high risk for infecting humans.These results are consistentw ith the actua loccurrence of the 2013 H7N 9 epidem ic.Ourme thod may be used to rapidly pred ict the a ffinity of HA for human recep tors and provides a theoreticalbasis for the risk assessmentof the infectiousness of influenza Avirus toward humans.
In fluenza A virus;Interspecies transm ission;Recep tor-specificity;Molecular docking; Binding free energy
O 641
10.3866/PKU.WHXB201612052
www.whxb.pku.edu.cn
Received:October28,2016;Revised:December2,2016;Published online:December5,2016.
*Corresponding author.Email:huangqiang@fudan.edu.cn;Tel:+86-21-51630589.
The projectwassupported by the NationalNaturalScience Foundation of China(91430112,31671386)and SpecialProgram forApplied Research on Super Computation of the NSFC-Guangdong Joint Fund(the Second Phase),China.
国家自然科学基金(91430112,31671386)和NSFC-广东联合基金(第二期)超级计算科学应用研究专项和国家超级计算广州中心支持资助项目©Editorialoffice of Acta Physico-Chim ica Sinica
