玉米PLDs基因家族生物信息学分析
2017-04-25韩雨徐晶宇李佐同赵长江贺林杨克军谷英男赵莹闫博巍
韩雨,徐晶宇,李佐同,赵长江,贺林,杨克军,谷英男,赵莹,闫博巍
(黑龙江八一农垦大学农学院,大庆 163319)
玉米PLDs基因家族生物信息学分析
韩雨,徐晶宇,李佐同,赵长江,贺林,杨克军,谷英男,赵莹,闫博巍
(黑龙江八一农垦大学农学院,大庆 163319)
磷脂酶D(PLD)为植物中重要的一种水解酶,PLD在逆境下不仅参与细胞膜的老化过程和种子萌发的过程,还作为一个信号物质响应脱落酸的形成。为了进一步明确玉米PLDs基因家族的功能,通过对玉米PLDs基因家族基本信息的预测,结构域的预测,染色体定位,基因结构的分析,蛋白系统进化树分析以及盐碱胁迫下的ZmPLDs进行转录组分析,共鉴定出ZmPLDs15个,分布在α、β、ε、ζ四个亚家族中,且预测基因主要功能为响应逆境信号和ABA诱导的气孔开闭过程。通过对ZmPLDs生物信息学分析进一步明确了ZmPLDs基因家族的生物学功能。
玉米;ZmPLDs;生物信息学
在细胞的生物过程中磷脂酶(PLD)是普遍存在于真核细胞中的酶。PLD有不同的反应类型,其中主要的两种分别是动物存在于哺乳类动物中的糖基磷脂酰激酶的蛋白(GPI-PLD)和卵磷脂蛋白(PCPLD)。两种显著的PC-PLD基因在动物中已经被鉴定,他们参与了细胞信号转导和气孔的呼吸。酵母(accharo-myces cerevisiae)中仅有PC-PLD基因家族中基因被鉴定。
植物中PLD也得到了广泛的研究,特别是PLD的活性在植物中得到了首次研究[1]。 PLD基因在蓖麻中首次得到克隆[2-3]。逆境环境下PLD不仅在膜的老化和种子萌发的过程中起到重要的作用,也作为一个信号物质响应脱落酸的合成。在药理学中已经证明异元三聚体G-蛋白在PLD合成过程中起到了重要的作用[4]。最近的一项研究表明PLDα基因对烟草生长发育起到了重要的作用[5]。PLD形成二酰甘油和磷脂酸(PA),他们是植物重要的信号分子,对于植物的生长发育起到至关重要的作用[6-7]。至今已有20多个PLD基因从植物中克隆出来。多种亚家族被发现使得PLD的研究复杂化。被子植物中PLD分为三个主要的亚家族,他们是C2-PLD,PXPH-PLD和SP-PLD。C2-PLD的主要结构是N端有一个C2结构域,在中间和C端各有一个HKD结构域;其主要作用是将可溶性蛋白定位到细胞膜上,这一过程需要与Ga2+结合[8];PXPH-PLD的主要结构为在N端有PX和PH结构域,在C端有HKD结构域,其主要作用为参与聚磷酸肌醇信号转导过程[9]。SP-PLD在N端只有一个信号肽,没有其他的结构。亚组中的亚组α、β、γ、δ、ε属于 C2-PLD,ζ属于 PXPH-PLD,φ属于SP-PLD。
PLD基因家族在拟南芥和水稻中研究的全面,但在玉米中的研究仍不多见。通过对玉米PLD基因家族进行生物信息学分析能够进一步明确玉米中PLD基因的功能。
1 材料与方法
1.1 ZmPLDs基因家族基因基本信息
利用已知的拟南芥PLD基因家族中的12个基因搜索 NCBI数据库(http://www.ncbi.nlm.nih.gov/)和玉米基因组数据库(http://www.maizegdb.org/)进行比对;与 Phytozome(https://phytozome.jgi.doe.gov/)中玉米数据库中的基因进行比对,获得相应的Pfam号,去除冗余基因。利用NCBI和SMART(http:// smart.embl-heidelberg.de/)对非冗余的蛋白进行保守结构域分析。在玉米数据库中获得ZmPLDs基因的登录号,编码区长度,氨基酸数量。利用Expasy软件(http://web.expasy.org/protparam/)获得基因的分子式、分子量、跨膜螺旋、等电点、亲水系数。利用HMMTOP(http://www.enzim.hu/hmmtop/)、Wolfpsort(http://wolfpsort.org/)、Signal P 4.1 Server(http:// www.cbs.dtu.dk/services/SignalP/)对蛋白的跨膜螺旋、亚细胞定位、信号肽进行分析。
1.2 玉米PLD蛋白进化分析
利用MEGA6.0对蛋白的进化关系进行分析。
1.3 ZmPLDs基因结构及染色体定位
利用GSDS对ZmPLDs基因结构进行分析,通过玉米数据库MaizeGDB获得染色体位置,利用Map Inspect对定位图进行绘制。
1.4 蛋白结构域分析
采用 Inter Pro Scan 5(http://www.ebi.ac.uk/ Tools/pfa/iprscan5/)进行蛋白结构域的分析,应用程序选择HMMPfam、HMMSmart和Super Family。
1.5 玉米幼苗根系转录组分析
取100 mM NaHCO3处理0 h、12 h、24 h、72 h根系样品按Trizol试剂盒 (天根)进行总RNA提取,取500 ng进行1%琼脂糖电泳检测RNA并进行RNA文库构建,运用Hiseq2 000(美国Illumnia公司)进行高通量测序。差异转录本表达量计算采用FPKM计算度量指标(FPKM-Fragments Per Kilobase of transcript per Million fragments mapped), 利用Cuffnorm程序计算样本表达量,取log2在2倍以上且满足P value<0.05基因定义为差异基因。通过Blast2-GO软件对 Cufflinks组装得到的所有 unigenes与NCBI的nr蛋白质序列数据库比对,对unigenes进行GO注释和分类。
2 结果与分析
2.1 ZmPLDs基因鉴定和命名
根据比对结果获得基因序列,提交Pfam数据库进行验证,获得含有PLD基因特有结构域的非冗余蛋白序列15个,根据结构域对基因进行命名,分别为ZmPLDa1~7;ZmPLDβ1~4;ZmPLDε1~3;ZmPLDζ1。由表 1可知,ZmPLDs基因编码序列长度介于 672(ZmPLDζ1)~3 147(ZmPLDβ2)之间,氨基酸个数介于223~1 048之间,蛋白分子量介于 43.712 8 KDa(ZmPLDβ1)~116.478 KDa(ZmPLDβ1)之间,平均蛋白分子量为 80.910 KDa, 均为亲水性蛋白,ZmPLDα1、ZmPLDβ1、ZmPLDβ4呈碱性,其他基因呈酸性。ZmPLDs均不含跨膜螺旋结构且均不含有信号肽。
定位于叶绿体中的基因有:ZmPLDa1~6,Zm-PLDβ1,ZmPLDβ3~4,ZmPLDε1~2,ZmPLDζ1;定位于线粒体中的基因有:ZmPLDβ1、ZmPLDβ4;定位于细胞核中的基因有:ZmPLDα6、ZmPLDβ3、ZmPLDε2;定位于细胞质中的基因有:ZmPLDα2、ZmPLDα3、Zm-PLDα5、ZmPLDα6、ZmPLDα7、ZmPLDβ2、ZmPLDβ3、ZmPLDε1、ZmPLDε3、ZmPLDζ1。
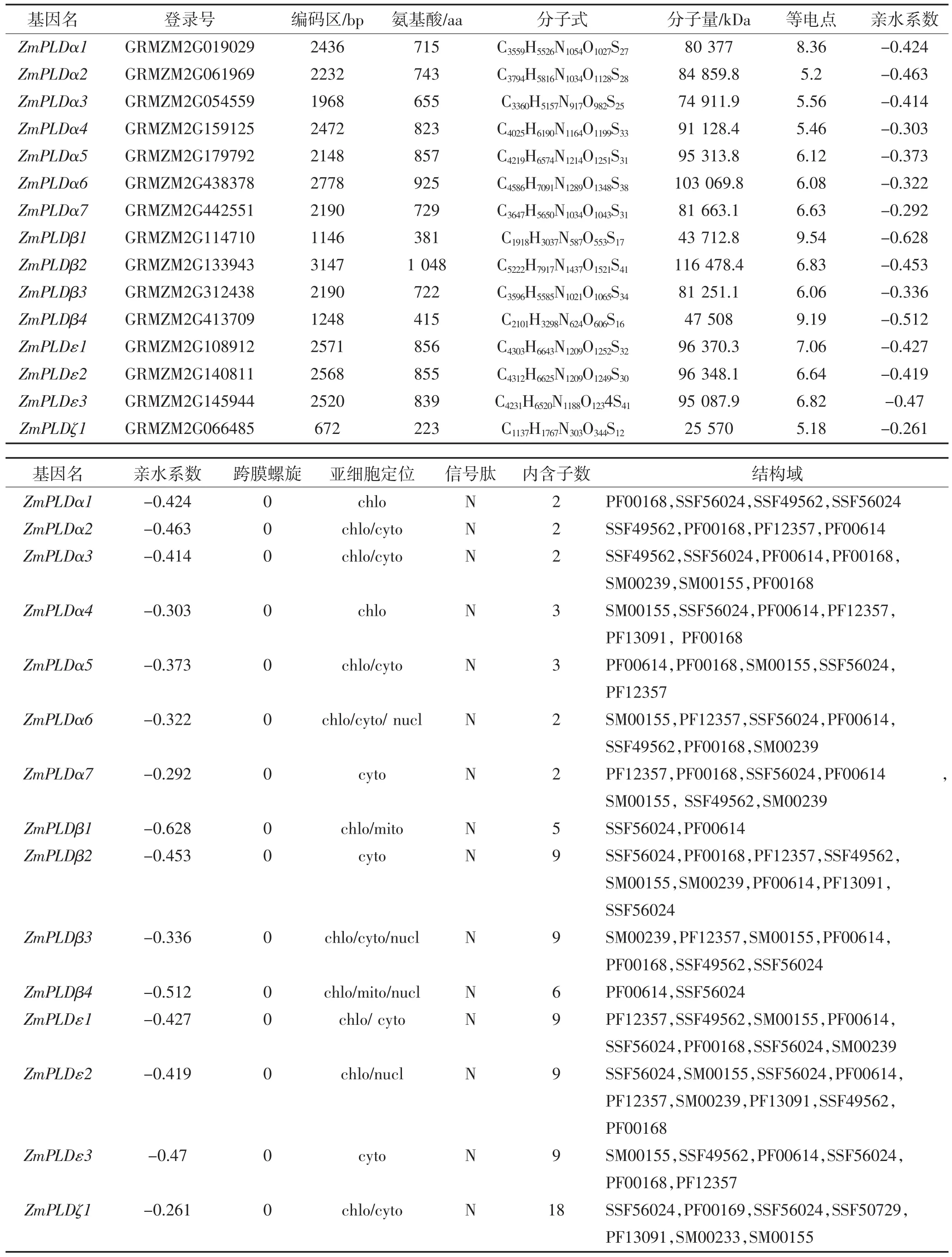
表1 ZmPLDs基因的特征Table 1 Characters of ZmPLDs genes
2.2 ZmPLDs蛋白结构域分析
由Inter ProScan分析发现可知(表1),大部分基因含有PLD基因家族中的C2(PF00168、SSF56024、SM00239)结构域和PLD活性相关结构域(PF00614、SM00155、PF12357)。含有PF00168的基因有Zm-PLDα1~7、ZmPLDβ2、ZmPLDβ3、ZmPLDε1、ZmPLDε2、ZmPLDε3;全部的基因都含有SSF56024结构域,含有 SSF49562结构域的基因有 ZmPLDα1~7、Zm-PLDβ2~3、ZmPLDε1~3;含有PF12357结构域的基因有 ZmPLDα2~7、ZmPLDβ2~3、ZmPLDε1~2;含有PF00614结构域的基因有:ZmPLDα2~7,ZmPLDβ1~4,ZmPLDε1~3,ZmPLDζ1;含有SM00239结构域的有 :ZmPLDα3、ZmPLDα6、ZmPLDα7,ZmPLDβ2 ~3,ZmPLDε1~3;含有SM00155结构域的有:ZmPLDα3~7,ZmPLDβ2、ZmPLDε1~3、ZmPLDζ1;含有PF13091 PLD相关结构域的有:ZmPLDβ2、ZmPLDε2、Zm-PLDζ1;ZmPLDζ1特有 PH型结构域 PF00169、SSF50729、SM00233。
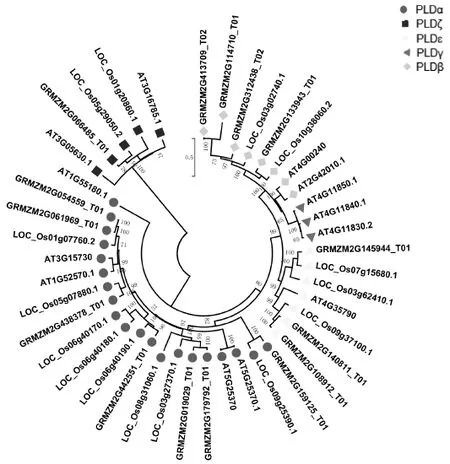
图1 玉米、水稻、拟南芥PLDs蛋白进化树Fig.1 Phylogenetic analysis of PLDs from maize,rice and arabidopsis
2.3 ZmPLDs蛋白进化分析
通过邻接法将拟南芥、水稻中所有PLDs与Zm-PLDs基因进行蛋白聚类分析,拟南芥、水稻的基因情况均已鉴定。可知玉米PLDs主要分布在PLDα、PLDβ、PLDε、PLDζ四个亚家族中,其中PLDα包含7个ZmPLDs蛋白,与5个拟南芥蛋白、8个水稻蛋白同组,PLDβ包含4个ZmPLDs,与2个拟南芥蛋白、2个水稻蛋白同组,PLDγ只有拟南芥,PLDε中包含3个ZmPLDs蛋白,与1个拟南芥蛋白、3个水稻蛋白同组。PLDζ中包含1个ZmPLDs蛋白,与2个拟南芥蛋白,2个水稻蛋白同组。由进化分析可知,玉米PLD蛋白与水稻亲缘关系更近。
2.4 ZmPLDs基因结构分析
对玉米PLDs基因结构进行分析(图2),可将ZmPLDs分为两个组,第一组为PLDα,其他亚家族为第二组。PLDa中所有基因的外显子个数均为2~4个,个数为3个的居多(ZmPLDα1、ZmPLDα2、ZmPLDα3、ZmPLDα6、ZmPLDα7),第二组中除ZmPLDζ1外其余基因外显子个数为5~10个,ZmPLDζ1外显子个数多达15个。第一组基因长度多集中在10 kb以内,其中ZmPLDα1基因长度最短,为3 kb左右,而第二组基因长度多集中于12 kb左右,ZmPLDζ1基因长度最长,为46 kb左右。
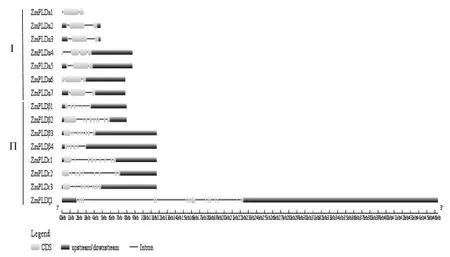
图2 ZmPLDs基因结构Fig.2 Gene structure of ZmPLDs gene family
2.5 染色体定位分析
对ZmPLDs进行染色体定位分析(图3)可知,ZmPLDs基因分布在玉米10条染色体中的1~9号染色体上。染色体3、4、7、8上都只有1个ZmPLDs基因,6号和9号染色体上有2个ZmPLDs基因,1号和2号染色体上有3个ZmPLDs基因。多数基因位于染色体的两端,远离中心粒,ZmPLDα5、ZmPLDβ3距离较近,靠近1号染色体中心位置,ZmPLDα1、ZmPLDα7距离较近,靠近9号染色体的中心粒。
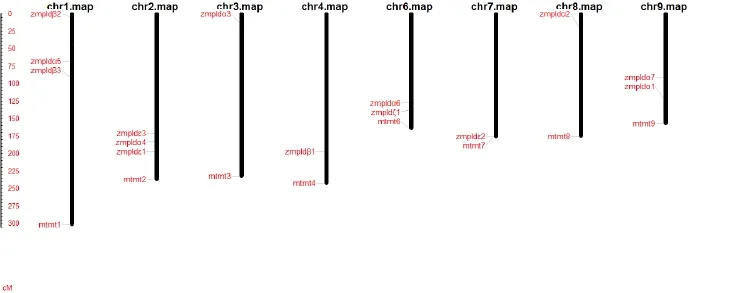
图3 ZmPLDs染色体定位Fig.3 Chromosomal distributions of ZmPLDs genes in the maize genome
2.6 ZmPLDs转录组测序分析
结合转录测序分析可知,盐碱胁迫不同时间点可将ZmPLDs分为四大组,第一组为12 h、24 h、72 h表达量均上调的基因上调,第二大组为12 h、24 h、72 h表达量均下调的基因,第三大组为表达量在12 h、24 h、72 h呈上调-下调-上调趋势,第四大组为表达量在12 h、24 h、72 h呈下调-上升-下调趋势。第一组中包括的基因有:ZmPLDα2、ZmPLDα6、Zm-PLDβ2、ZmPLDζ1;第二组包含ZmPLDα7、ZmPLDβ3、ZmPLDε1、ZmPLDα3、ZmPLDα4;第三大组包含Zm-PLDε2、ZmPLDε3;第四大组包含ZmPLDα1、ZmPLDα5。转录组数据没有覆盖到ZmPLDβ1、ZmPLDβ4。
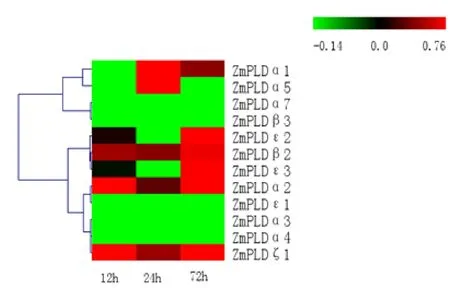
图4 玉米根系转录组测序分析Fig.4 Transcriptome sequencing of roots of maize seedlings
3 结果与讨论
PLD是普遍存在于植物中的磷脂酶基因家族,他能够水解磷脂、磷脂酰胆碱、磷脂酰乙醇胺,生成磷脂酸[11],也在信号转导、膜运输、细胞骨架形成、膜降解等多种生物过程中起到重要的作用。多种作物PLDs已被研究,在拟南芥中也得到广泛研究。但在玉米中PLDs还没有得到研究。玉米为C4植物[12],通过对ZmPLDs进行结构、功能上的分析,为C4植物中的PLDs功能进行进一步的鉴定。
实验共鉴定ZmPLDs基因15个,他们分别分布在α、β、ε、ζ四个亚家族中。拟南芥中PLDα、PLDβ[13]、PLDγ、PLDζ功能已被鉴定[14-17]。PLDα为PLDs基因家族中最常见的一类亚家族,本实验中鉴定出7个PLDα基因,可见PLDα亚家族在玉米中也是最常见的一类亚家族。由他们的蛋白结构域相似,ZmPLDα2、ZmPLDα3的亲缘关系较近,ZmPLDα1、ZmPLDα5的亲缘关系较近。PLDα的主要结构域为C2结构域,这也是PLDs基因家族中的标志性结构域。在拟南芥的PLDα亚家族的四个基因也十分相似,在C端含有ATG氨基酸序列,在N端含有TAA氨基酸序列。ZmPLDαs中外显子的数量在2~4之间,在拟南芥PLDαs的四个基因中外显子的个数均为4个[18],由此可见ZmPLDαs与AtPLDαs具有相似的功能区域。PLDαs的活性是由GPA1蛋白控制的,GPA1调节由ABA诱导的气孔开闭过程[19],因此表明PLDαs在ABA诱导的气孔开闭过程中起重要作用[20]。但是AtPLDα1功能缺失并没有抑制ABA诱导的气孔开闭过程[21],因此证明PLDαs并不是唯一调控此途径的基因亚家族,其他的亚家族也有可能参与调控此过程。ZmPLDαs功能区与拟南芥具有高度的相似性,因此可推断ZmPLDαs也有可能在ABA诱导的气孔开闭过程中起作用。干旱胁迫下AtPLDαs转录组测序表达量上调,证明干旱胁迫下PLDαs调控气孔开闭过程,保证植物正常生命活动,由此可见逆境胁迫下PLDαs起到重要作用。实验对盐碱胁迫不同时间点玉米根系进行转录组测序分析,ZmPLDα2、ZmPLDα6在12 h、24 h、72 h均为上调,表明在盐碱胁迫下基因参与植物的逆境调节途径;表明ZmPLDαs在盐碱胁迫下也起到重要的作用。ZmPLDα3、ZmPLDα4、ZmPLDα7这三个基因为适应逆境对植物的损伤因此表达量上调;ZmPLDα1、ZmPLDα5的变化趋势为下调-上调-下调,在逆境胁迫下参与玉米体内代谢平衡。ZmPLDαs在盐碱胁迫下变化显著,表明ZmPLDαs参与玉米体内逆境调节。此外,ZmPLDαs在玉米的1号、3号6号8号9号染色体上均有分布,可见ZmPLDαs对于调节玉米生长发育至关重要。
实验鉴定出4个ZmPLDβs基因,他们的主要结构域为C2结构域(PF00168)和PLD活性相关结构域(PF00614)。有研究表明AtPLDβs依赖于多磷酸肌醇途径、Ga2+的形成途径、并且能够调节植物体内的PH。PLDβs的活性与脂类基质囊泡中4,5-二磷酸磷脂酰肌醇(PIP2)途径中的磷脂酰肌醇(PIP),PI,PG,PS,PE,PA有关。PIP能够调节PLDβs的活性[22]。由此可见PLDβ主要存在于细胞质中。ZmPLDβ2、Zm-PLDβ3定位于细胞质中,可知ZmPLDβ2、ZmPLDβ3在ZmPLDβs中起主要作用。蛋白进化分析也表明ZmPLDβ2和ZmPLDβ3的亲缘关系更近。ZmPLDβs的外显子数量多于ZmPLDαs,可见ZmPLDβs的功能更为复杂。通过调节Ga2+浓度来验证PLDs对PIP2途径的影响,结果表明PLDβs对PIP2途径的影响多于PLDαs。由此可见ZmPLDs也可能参与PIP2途径。盐碱胁迫下ZmPLDβ2表达上调,ZmPLDβ3表达下调,达到显著变化,可知盐碱逆境下ZmPLDβs也参与调控植物体内平衡。
实验中鉴定出ZmPLDεs为3个,他们主要定位在叶绿体和细胞质中。ZmPLDεs外显子的数量为5~10个,定位在玉米2号、7号染色体上。ZmPLDεs共有的蛋白功能区是C2结构域(SM00239)和PLD活性相关结构域(PF00614)。有研究表明ZmPLDεs参与植物体内ROS调控作用,ROS是植物氧化还原过程中的副产物,植物通过启动体内的ROS防御系统来缓解逆境对植物造成的伤害[23]。由此可知Zm-PLDεs也参与植物体内的ROS调控。转录组数据得知ZmPLDεs变化显著,可见ZmPLDεs参与植物的逆境调节。但ZmPLDεs的变化趋势不一致,由此可以判断ZmPLDεs不是单一的调控因子,是由于植物体内环境变化还是外界因素干扰导致基因表达量发生变化需要通过实验进一步探讨。
ZmPLDζ仅一个,且含有PH结构域(PF00169)。PH结构域也是PLDs特有的结构域。ZmPLDζ1的基因长度达到46kb且外显子数多达14个,可见Zm-PLDζ1功能复杂。ZmPLDζ1位于6号染色体上且远离中心粒。有研究表明PLDζs在植物的根系中有较高的表达量[24]。PLDζs也参与拟南芥体内ABA保卫细胞信号转导过程,且PLDζs转录水平高于PLDαs[25]。盐碱胁迫下ZmPLDζ1的转录水平为上调,可见Zm-PLDζ1能够调节植物对逆境胁迫下的响应。
[1] Hanahan D J,Chaikoff I L.A new phospholipide-splitting enzyme specific for the ester linkage between the nitrogenous base and the phosphoric acid grouping[J].J Biol Chem,1947,169:699-705.
[2] Wang X,Xu L,Zheng L.Cloning and expression of phosphati-dylcholine-hydrolyzing phospholipase D from Ricinus communis L[J].J Biol Chem,1994,269:20312-20317.
[3] Chalifa-Caspi V,Eli Y.Liscovitch M.Kinetic analysis in mixed micelles of partially purified rat brain phospholipase D activity and its activation by phosphatidylinositol 4,5-bisphosphate [J].NeurochemicalResearch,1998,23(23):589-99.
[4] Munnik T,Irvine RF,Musgrave A.Phospholipid signaling in plants[J].Biochim Biophys Acta,1998,1389:222-272.
[5] Lein W,Saalbach G.Cloning and direct G-protein regulation of phospholipase D from tobacco[J].Biochim Biophys Acta,2001,1530:172-183.
[6] Chapman K D.Emerging physiological roles for N-acylphos-phatidylethanolamine metabolism in plants:signal transduc-tion and membrane protection[J].Chem Phys Lipids,2000,108:221-229.
[7] Munnik T.Phosphatidic acid:an emerging plant lipid second messenger[J].Trends Plant Sci,2001(6):227-233.
[8] Dushyanthan P,Mitchell JBO.L/D Protein Ligand Database(PLD):additional understanding of the nature and specificity of protein-ligand complexes[J].Bioinformatics,2003,19(14):1856-1857.
[9] Wenhua Z,Cunxi W,Chunbo Q,et al.The oleate-stimulated phospholipase D,PLD delta,and phosphatidic acid decrease H2O2-induced cell death in Arabidopsis[J]. Plant Cell,2003,15(10):2285-2295.
[10] Wang X.Multiple forms of phospholipase D in plants:the gene family,catalytic and regulatory proper-ties,and cellular functions[J].Prog Lipid Res,2000,39:109-149.
[11] Qin W,Pappan K,Wang X.Molecular heterogene-ity of phospholipase D(PLD):cloning of PLD and regulation of plant PLD and bypolyphosphoi-nositides and calcium[J]. J Biol Chem,1997,272:28267-28273.
[12] 王振.玉米不育系回交转育中利用SSR标记进行背景选择效果的研究[D].大庆:黑龙江八一农垦大学,2015.
[13] Wang X.Multiple forms of phospholipase D in plants:the gene family,catalytic and regulatory properties,andcellular functions[J].Prog Lipid Res,2000,39:109-149.
[14] Pappan K,Austin-Brown S,Chapman KD,et al.Substrate selectivities and lipid modulation of plant phospholipase[J].Arch Biochem Bio phys,1998,353:131-140.
[15] 孙盼盼,吴委林,宫赫阳,等.中国玉米LSC杂种优势群种质与美国BSSS种质间配合力分析[J].延边大学农学学报,2014(1):44.
[16] Pappan K,Zheng S,Wang X.Identification and characterization of a novel plant phospholipase D that requires polyphosphoinositides and submicromolar calcium for activity in Arabidopsis[J].J Biol Chem,1997,272:7048-7054.
[17] Ueki J,Morioka S,Komari T,et al.Purification and characterization of phospholipase D(PLD)from rice(Oryza sativa L.)and cloning of cDNA for PLD from rice and maize(Zea mays L.)[J].Plant&Cell Physiology,1995,36(5):41-69.
[18] Zhi L J,Chen Z Q.The Arabidopsis Phospholipase D Family.Characterization of a Calcium-Independent and Phosphatidylcholine-Selective PLDζ1 with Distinct Regulatory Domains[J].Plant Physiology,2002,128(3):535-537.
[19] Zhao J,Wang X(2004)Arabidopsis phospholipase Da1 interacts with the heterotrimeric G-protein a-subunit through a motif analogous to the DRY motif in G-proteincoupled receptors[J].J Biol Chem,279:1794-1800.
[20] Mishra G,Zhang W,Deng F,Zhao J,Wang X.A bifurcating pathway directs abscisic acid effects on stomatal closure and opening in Arabidopsis[J].Science,2006,312:264-266.
[21] Siegel R S,Xue S,Murata Y,et al.Calcium elevation-dependent and attenuated resting calcium-dependent abscisic acid induction of stomatal closure and abscisic acid-induced enhancement of calcium sensitivities of S-type anion and inward rectifying K channels in Arabidopsis guard cells[J].Plant J,2009,59:207-220.
[22] Qin W,Pappan K,Wang X.Molecular heterogeneity of phospholipase D(PLD).Cloning of PLD gamma and regulation of plant PLDgamma[J].Journal of Biological Chemistry,1997,272(45):28267-28273.
[23] Francesco P,Nathalie B,Mark K,et al.Arabidopsis phospholipase Dδ is involved in basal defense and nonhost resistance to powdery mildew fungi.[J].Plant Physiology,2013,163(2):896-906.
[24] Li M,Qin C,Welti R,et al.Double Knockouts of Phospholipases Dζ1 and Dζ2 in Arabidopsis Affect Root E-longation during Phosphate-Limited Growth But Do Not Affect Root Hair Patterning[J].PlantPhysiology,2006,140(2):761-770.
[25] Misugi U,Takeshi K,Eiji O,et al.Cooperative function of PLDδ and PLDα1 in abscisic acid-induced stomatal closure in Arabidopsis[J].Plant Physiology,2012,159(1):450-460.
Genome-wide Analysis of the ZmPLDs Gene Family in Maize(Zea mays)
Han Yu,Xu Jingyu,Li Zuotong,Zhao Changjiang,He Lin,Yang Kejun,Gu Yingnan,Zhao Ying,Yan Bowei
(College of Agronomy,Hei Longjiang Bayi Agriculture University,Daqing 163319)
Phospholipase D(PLD)was one of the most important hydrolase of plants,PLD was not only involved in the aging process of the cell membrane and the germination process of seeds under the stress environment,but also formed in response to ABA as a signal substance.In order to further clarify the function of ZmPLDs gene family,to predict the ZmPLDs basic information of maize,domain,the chromosomal localization,gene structure,the system protein evolution,and transcriptome analysis of ZmPLDs under salinity stress,15 ZmPLDs genes were identified,which distributed in α、β、ε、ζ subfamily.The main functions of the genes were the prediction signal in response to stress and ABA-induced stomatal closure.The ZmPLDs gene family biological function of maize could be clarified furtherly.
maize;ZmPLDs;bioinformatics
Q78
A
1002-2090(2017)02-0013-08
10.3969/j.issn.1002-2090.2017.02.003
2016-03-12
教育部博士学科点博导类专项科研基金(201222305110001);黑龙江省自然科学基金项目(C201324);黑龙江八一农垦大学研究生创新项目(YJSCX2014-Y05)。
韩雨(1991-),女,黑龙江八一农垦大学农学院2013级硕士研究生。
杨克军,男,教授,博士研究生导师,E-mail:byndykj@163.com。
