trnH—psbA序列作为DNA条形码在苍耳属杂草鉴定中的应用
2017-04-14袁俊杰魏霜马华龙阳张娜陈文杨
袁俊杰 魏霜 马华 龙阳 张娜 陈文 杨卓瑜


摘要:以trnH-psbA序列作为DNA条形码,对从国外截获的9种苍耳属杂草及1种国内苍耳进行物种鉴定研究。采用DNeasyPlantMiniKit试剂盒进行总DNA的提取,应用通用引物对其trnH-psbA基因进行PCR扩增,测序得到10种苍耳属杂草的trnH-psbA序列,并利用MEGA7.0软件进行比对分析构建系统进化树。结果显示:苍耳属杂草trnH-psbA基因序列共有543个位点,其中有53个变异位点,31个变异信息位点,22个单突变位点,30个碱基缺失;种间遗传距离为0~0.093,种内无遗传差异;进化树显示10个苍耳物种均处于独立分支,能够利用该条形码对苍耳属杂草进行区分鉴定。
关键词:trnH-psbA序列;DNA条形码;苍耳属;杂草鉴定
中图分类号:S451文献标志码:A文章编号:1003-935X(2016)02-0001-06
苍耳属(Xanthium)杂草是菊科的一类重要恶性杂草,该属杂草幼苗具有毒性,威胁牲畜健康,成熟总苞具刺和喙,易刺伤牲畜及人类,且入侵性极强,一旦传入、定植,将严重影响我国人畜健康和环境安全[1-2],因此,防止苍耳属杂草的入侵具有重要意义。在进出口货物中,苍耳属杂草种类繁多,近似种相似性较高,鉴定困难;且种子在运输过程中总苞表面坚硬的刺以及密布的柔毛等重要鉴定特征会发生较大磨损,给形态学鉴定带来很大的难度[3-4]。因此,建立稳定且准确的分子生物学分类方法具有重要意义。
DNA条形码(DNAbarcoding)技术是利用1段至几段标准的、易扩增的、种间差异显著大于种内差异的DNA片段来鉴别物种的新技术[5-7],是传统植物分类与鉴定的强有力的补充。张伟等在建立检疫性杂草银毛龙葵的DNA条形码检测技术中,使用来自不同基因组的3个DNA片段(tunS-trnG、ndhF、waxy)建立快速检测方法,并在此基础上探讨这3个基因作为茄属条形码辅助片段的可能性[8];陈冬美分析了毒麦属种内和种间rDNAITS区序列的差异来快速分辨鉴定6个近似种,结果与形态学分类基本一致[9];Gao等针对菊科494属2315种3490个样品进行最佳条形码筛选,提出以ITS2作为条形码,鉴定效率达76.4%[10]。以上研究均表明DNA条形码技术已经运用在杂草鉴定上,具有一定的可行性,对口岸快速鉴定杂草籽具有重要的意义。
在口岸鉴定中须将杂草籽鉴定至种,属于精确的物种鉴定,然而,传统的形态学鉴定方法需要很强的专业知识和经验,受鉴定人主观意识影响较大,所以应该在形态学初步鉴定的基础上(鉴定至科属水平较容易),探索分子层面鉴定物种的可能性。DNA条形码技术能够在较短时间内建成易被利用的应用系统,使标本鉴定过程实现自动化和标准化,突破对经验的过度依赖,在物种分类和鉴定方面展示出强大的生命力。trnH-psbA片段是进化速率最快的叶绿体间隔区之一,片段两端存在75bp的保守序列,便于设计通用引物。该片段引物通用性较好,扩增成功率较高,并且平均长度较短(大多数在450bp左右),有利于对降解材料扩增[11]。本研究以trnH-psbA序列作为DNA条形码,对10种苍耳属杂草进行物种区分鉴定研究,旨在为口岸杂草鉴定工作提出新思路。
1材料与方法
1.1试验材料
试验所用的苍耳属杂草种子样本是从近5年的进境粮谷(美国黄大豆、巴西黄大豆、乌克兰玉米、澳大利亚油菜籽、加拿大油菜籽、美国高粱等)中截获的,具体情况如表1所示,经中国检验检疫科学研究院鉴定,凭证标本保存于湛江出入境检验检疫局。
1.2试验方法
1.2.1DNA提取
采用改良后的DNeasyPlantMiniKit试剂盒法提取DNA,得到200μLDNA样品,于-20℃冰箱保存。
1.2.2PCR扩增和电泳检测
引物序列为trnH-psbAf-5′GTTATGCATGAACGTAATGCTC-3′;trnH-psbAr-5′CGCGCATGGTGGATTCACAATCC-3′。PCR反应使用宝生物工程(大连)有限公司的rTaq酶进行扩增,反应采用50μL体系:3μLDNA模板,5μL10×PCRbuffer,4μLdNTPs,0.25μLrTaq,各1μL正反引物,补充ddH2O至50μL。trnH-psbA的PCR条件:95℃4min;94℃30s,52℃1min,72℃1min,35个循环;72℃10min;4℃冷却。扩增产物用1%琼脂糖凝胶电泳检测、凝胶成像分析系统分析。
1.2.3测序及拼接
使用ABI3730XL测序仪(AppliedBiosystemsCo.,USA)对PCR产物进行双向测序。利用序列拼接软件CodonCodeAlignerV3.7.1对测序峰图文件进行剪切拼接,去除引物区及低质量序列。使用Clustalt和MEGA7.0软件对拼接后的序列进行比对分析并构建进化树。
2结果与分析
2.1PCR结果
从图1可看出,10种苍耳属杂草的trnH-psbA序列扩增条带单一清晰,无特异性条带及拖尾现象,与marker对比可知,该扩增产物大小接近500bp,与预期大小一致。登陆NCBI,将拼接后的序列进行BLAST检测,结果证明10种苍耳属杂草的trnH-psbA序列扩增及测序成功。
2.2碱基构成分析
利用MEGA7.0软件对10种苍耳属杂草进行ClustalW校对,校对后的序列繼续用MEGA7.0进行分子进化遗传分析。从表2可以看出,苍耳属杂草trnH-psbA序列的G、A、C、T碱基的变化范围依次减小,分别为1.3%、1%、1%、0.9%。其中,G碱基含量最高的是刺苍耳,为15.1%,最低的是北美苍耳、球果苍耳、西方苍耳、宾州苍耳、沃式苍耳,均为13.8%;A碱基含量最高的是北美苍耳、球果苍耳、西方苍耳、宾州苍耳、沃式苍耳,均为33.3%,最低的是苍耳,为32.3%;C碱基含量最高的是柱果苍耳、意大利苍耳、河岸苍耳,为12.8%,最低的是刺苍耳,为11.8%;T碱基含量最高的是柱果苍耳、意大利苍耳、河岸苍耳,均为40.8%,最低的是刺苍耳,为39.9%。
2.3基因位点分析
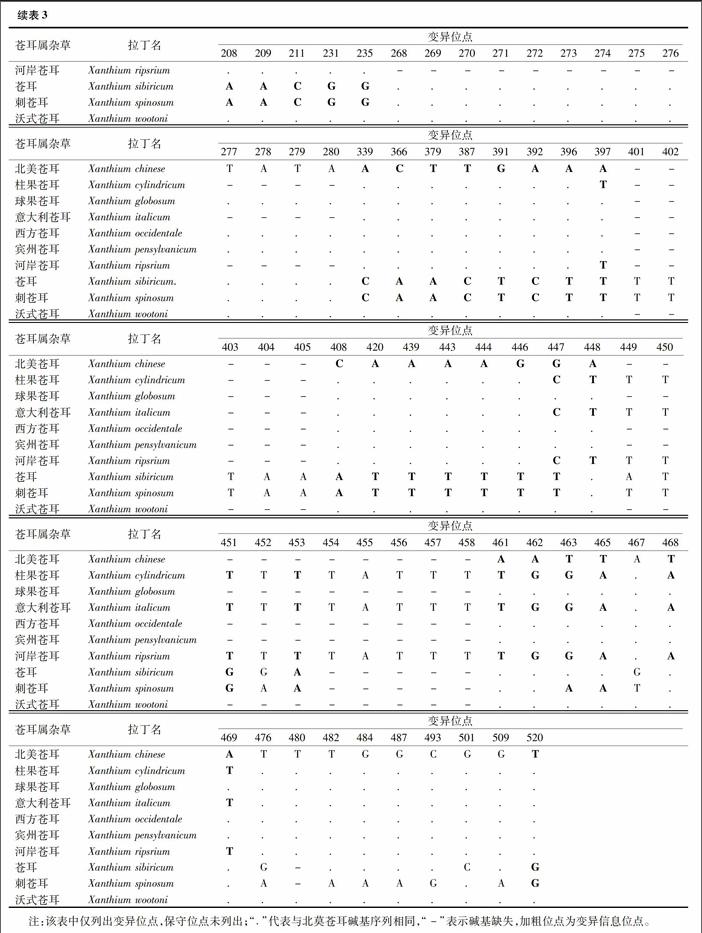
由MEGA7.0测算可知,苍耳属杂草trnH-psbA序列共有543个位点,其中490个保守位点、53个变异位点、31个变异信息位点、22个单突变位点,30个碱基缺失(表3)。
2.4遗传距离分析
同一地区样本的不同个体序列完全相同,种内遗传距离为0。利用MEGA7.0软件,采用Tajima-Nei模型计算种间遗传距离(表4),苍耳属杂草trnH-psbA序列的遗传距离最高值为0.093,为刺苍耳与柱果苍耳、意大利苍耳、河岸苍耳的遗传距离;最小值为0.000,为北美苍耳与球果苍耳、西方苍耳、宾州苍耳、沃式苍耳的遗传距离。这说明虽然北美苍耳与球果苍耳、西方苍耳、宾州苍耳、沃式苍耳从序列上看,于449~458位点存在片段缺失差异,但在Kimura2-parameter模型中被忽略不计,所以从遗传距离上无法区分。
2.5系统进化发育树
利用MEGA7.0软件,采用Neighbor-Joining方法构建系统发育树(图2),苍耳、刺苍耳被分为一支;柱果苍耳、意大利苍耳、河岸苍耳被分为一支;北美苍耳、球果苍耳、宾州苍耳、沃式苍耳、西方苍耳被分为一支;对其进行Bootstrap检验,其分组支持率为100%,说明这3支苍耳属杂草在系统进化发育中是相互独立的。柱果苍耳、意大利苍耳、河岸苍耳又被划分为3个不同级别的分支,但其Bootstrap检验支持率较低,说明这3种苍耳属杂草独立性相对较弱。北美苍耳、球果苍耳、宾州苍耳、沃式苍耳、西方苍耳也被分为各自独立的分支,但其Bootstrap检验支持率较低,说明这5种苍耳属植物独立性相对较弱。
3结论与讨论
苍耳属杂草种间的传统鉴定方法主要依赖于形态鉴定,依靠总苞的大小、颜色,苞刺的长短、疏密、碾去时的难易程度及果柄横切面和总苞表皮毛茸的显微特征等方面[12]。近些年发展为化学分析层面,从果实粉末的薄层色谱、紫外吸收光谱的差异分析来区分苍耳属种间的亲缘关系[13]。然而在苍耳属杂草检疫鉴定过程中存在鉴定材料不完整、化学分析耗时长等问题,难以满足过关检疫快速准确的要求,因此笔者依据当代分子生物学技术,利用DNA条形码技术,开发了可适用于苍耳属杂草的鉴定条形码trnH-psbA序列。从试验结果可以看出,trnH-psbA序列可作为DNA条形码对10种苍耳属杂草进行区分,9种检疫性苍耳可与本地苍耳进行鉴定区分;将10种苍耳属杂草分为3个大类群,并在基因位点上进一步区分,各不同检疫性苍耳处于独立分支,可通过构建进化树进行区别。但不足之处是,柱果苍耳、意大利苍耳、河岸苍耳仅在序列397位存在点突变,在进化树中分支的Bootstrap支持率较低;西方苍耳、宾州苍耳在序列上完全相同,不存在变异变点,在进化树中分支的Bootstrap支持率较低。为了更好地区分其他苍耳物种之间的区别,建议开发其他条形码序列进行补充。
参考文献:
[1]伏建国,安榆林,杨晓军.杂草风险分析概述[J].植物检疫,2009,23(增刊1):39-44.
[2]谌运清,刘翔,杨万风,等.进境大豆植物检疫截获疫情分析及工作建议[J].植物检疫,2009,23(增刊1):61-64.
[3]熊颖,刘启德,宓穗卿.苍耳子化学研究进展[J].广东药学,2005,15(6):65-68.
[4]高锐红,龚大坤,张星明.苍耳子及其伪品单喙苍耳子的比较鉴别[J].中药材,2001,24(6):410-411.
[5]CBOLPlantWorkingGroup.ADNAbarcodeforlandplants[J].ProceedingsoftheNationalAcademyofSciences,2009,106(31):12794-12797.
[6]丁潜,邢光东,胡肄农,等.识别杜洛克猪个体身份的DNA条形码[J].江苏农业学报,2014,30(5):1058-1063.
[7]HollingsworthPM,GradamSW,LittleDP.ChoosingandusingaplantDNAbarcode[J].PlosOne,2011,6(5):e19254.
[8]张伟,范晓虹,邵秀玲,等.DNA条形码在检疫性杂草银毛龙葵鉴定中的应用研究[J].植物检疫,2013,27(3):60-65.
[9]陈冬美.毒麦属6个种的分子检测检疫应用研究[D].长沙:湖南农业大学.2007.[ZK)]
[10]GaoT,YaoH,SongJ,etal.EvaluatingthefeasibilityofusingcandidateDNAbarcodesindiscriminatingspeciesofthelargeAsteraceaefamily[J].BMCEvolutionaryBiology,2010,10(1):324.
[11]程佳月,王麗华,彭克美,等.国际生命条形码计划-DNABarcoding[J].中国畜牧兽医,2009(8):49-53.
[12]吕益涛,侯海宫,苏耀海,等.苍耳属植物的鉴别研究[J].中国中药杂志,2001,26(1):17-20.
[13]阮贵华,李攻科.苍耳子的化学成分及其分离分析研究进展[J].中成药,2008,30(3):421-426.
