常规和倒置A2/O工艺活性污泥微生物群落结构的比较
2017-04-11毕学军汝少国山东省环境保护科学研究设计院山东济南5001中国海洋大学海洋生命学院山东青岛6600青岛理工大学环境与市政工程学院山东青岛660
李 鹏,毕学军,王 军,汝少国*(1.山东省环境保护科学研究设计院,山东 济南 5001;.中国海洋大学海洋生命学院,山东 青岛 6600;.青岛理工大学环境与市政工程学院,山东 青岛 660)
常规和倒置A2/O工艺活性污泥微生物群落结构的比较
李 鹏1,2,毕学军3,王 军2,汝少国2*(1.山东省环境保护科学研究设计院,山东 济南 250013;2.中国海洋大学海洋生命学院,山东 青岛 266003;3.青岛理工大学环境与市政工程学院,山东 青岛 266033)
为了探究倒置A2/O工艺脱氮除磷效果优于常规A2/O工艺的机理,利用聚合酶链式反应-变形梯度凝胶电泳(PCR-DGGE)技术与纯培养方法分析了2种工艺活性污泥中微生物群落结构.结果显示,2种工艺活性污泥的优势菌群均含有β-变形菌与γ-变形菌;倒置A2/O工艺活性污泥中检测到大量的亚硝化螺旋菌、红环菌和4种未培养的拟杆菌,而这些菌类在常规A2/O工艺中含量较少;利用纯培养方法在倒置A2/O工艺活性污泥检测到的γ-变形菌纲希瓦氏菌属、克雷伯氏菌属和类球红细菌在常规A2/O工艺样品中未检测到.推测以上菌种的存在可能是倒置A2/O工艺具有较高脱氮除磷效果的主要原因.此外,扫描电镜结果显示倒置A2/O工艺活性污泥较为疏松,丝状细菌含量较少,且不存在念珠状细菌.
活性污泥;微生物群落结构;PCR-DGGE;倒置A2/O
目前,我国 80%以上的城市污水处理厂采用活性污泥法降解有机污染物,降解效率主要取决于活性污泥中微生物的结构与功能[1–2].传统污水处理厂多采用 A2/O(厌氧-缺氧-好氧)工艺,该工艺兼顾脱氮和除磷双重功能,由于2种功能对动力条件的要求不一致,导致除磷效果不尽理想
[3].为进一步强化除磷效果,研究者设计了倒置A2/O(缺氧-厌氧-好氧)工艺,以期解决常规 A2/O工艺存在的问题[4].研究表明,倒置 A2/O工艺对有机污染物、氨氮与磷酸盐的去除效率明显优于常规A2/O工艺[5],但是2种工艺的微生物结构和功能存在哪些差异,从而导致脱氮、除磷效果不一致尚不清楚.因此,本研究从常规 A2/O与倒置A2/O工艺的好氧区末端采集活性污泥样品,利用聚合酶链式反应-变性梯度凝胶电泳(PCRDGGE)技术鉴定了 2种工艺活性污泥中微生物群落结构,同时采用显微镜观察和分离培养技术得到单一菌株,并对可培养菌株进行鉴定,以探究倒置A2/O工艺脱氮除磷效果优于常规A2/O工艺的机理.
1 材料与方法
1.1 材料
将青岛市团岛污水处理厂(以处理城市生活污水为主)曝气沉砂池出水引入 A2/O与倒置A2/O工艺的小试试验装置(图1).2种工艺的有效容积均为 289L,进水量 400mL/min,水力停留时间为12h,溶解氧为2.0~3.0mg/L,泥龄为16d.每天下午采集进水与出水水样,参照《污水综合排放标准》(GB18918-2002)测定水样中的氮、磷含量.运行30d后,于好氧池末端采集活性污泥样品,取样时间为下午第2次排泥之前.
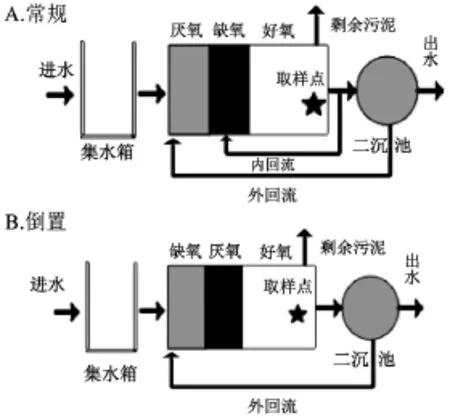
图1 常规A2/O和倒置A2/O工艺试验装置Fig.1 Lab-scale testing units of conventional A2/O and reversed A2/O processes
1.2 16S rDNA V3区的PCR-DGGE分析
1.2.1 DNA的提取与纯化 活性污泥中微生物基因组DNA提取采用SDS细胞裂解法[6].首先,向活性污泥中加入270µL含有50µg蛋白酶K的DNA提取液,于37℃、225r/min震荡30min;随后,加入30µL 20%的SDS,65℃水浴2h,每隔20min轻轻摇动1次;6000r/min离心10min,取上清液;向沉淀中加入 90µL DNA提取液,20µL 10% SDS,65℃水浴10min,6000r/min离心10min.将2次获得的上清合并后,利用 DNA胶回收试剂盒对粗提液DNA进行纯化.
1.2.2 16S rDNA的扩增 参照 Bosshard等[7]的方法,以纯化的总 DNA为模板,利用细菌 16S rDNA的通用引物27F (5’-AGAGTTTGATCCTGGCTCAG-3’)和1492R (5’-GGTTACCTTGTTACGACTT-3’),采用 PCR方法扩增出微生物的16S rDNA.PCR反应条件参照杨倩等[8]报道的方法,扩增产物用1%琼脂糖凝胶电泳检测.
1.2.3 变性梯度凝胶电泳(DGGE) 采用Dcode通用突变检测系统对PCR反应产物进行分离,聚丙烯酰胺凝胶浓度为 8%,变性胶梯度从 30%到60% (100%变性剂含7mol/L尿素和40%去离子甲酰胺).PCR样品上样量为20μL,60℃、150V电泳4h,经SYBR-Green I染色后,利用DOC1000凝胶成像系统拍照.
1.2.4 DGGE条带的切割和 DNA回收 从DGGE胶上切下目标条带,经无菌双蒸水浸洗 3次后,破碎凝胶,加入30µL TE缓冲液(pH 7.6),于50℃水浴中融化,6000r/min离心5min,取上清.以回收的DNA为模板,利用大多数细菌16S rDNA基因V3区具有的特异性引物341f和518r进行PCR扩增.
1.2.5 16S rDNA基因V3区的克隆、测序与分析 PCR产物切胶后,利用DNA胶回收试剂盒对16S rDNA进行回收纯化.将纯化的PCR产物与pGM-T载体连接后,转化到感受态细胞,涂布于加入IPTG和X-gal的LB固体培养基.随机挑选白色菌落,置于5mL LB液体培养基(含100μL相应抗生素)中,37℃培养过夜,提取质粒,将含有目的基因的菌株送生工生物工程(上海)有限公司测序.利用BLAST软件分析16S rDNA基因序列与GenBank中收录的16S rDNA序列的同源性,用Mega5.0软件对扩增的细菌进行聚类,采用邻位相连法(Neighbor-joining)构建系统发生树,并通过自举分析(bootstrap)进行置信度检测,自举数据集为1000次[9].
1.3 微生物的形态观察
将活性污泥菌胶团悬液涂布于干净的载玻片上,自然干燥后在光学显微镜下镜检,选出菌体较密且分散度较好的样品.将样品置于 2%戊二醛磷酸缓冲液(pH 7.2)中,4℃固定过夜;次日,用40%、70%、90%和 100%的乙醇依次脱水,并用醋酸戊脂置换乙醇;将上述制备好的样品置于临界点干燥器中,浸泡于液态二氧化碳中,加热至临界点温度(31.4℃,7.28MPa)以上,使之气化干燥;在真空镀膜机内把金喷镀到样品表面后,利用扫描电镜观察.
1.4 活性污泥微生物的纯化与鉴定
1.4.1 微生物的纯化 将10mL活性污泥放入含有3g玻璃珠(直径0.11~0.12mm)的50mL离心管中,旋涡混合器击打15min去絮凝.自然沉降1h,收集上清液,用灭菌蒸馏水将上清液稀释102~ 107倍后,吸取0.1mL均匀涂布于普通肉汤固体培养基,倒置于 37℃恒温培养箱中培养至形成单菌落.挑取单菌落进行划线分离纯化,将分离纯化的单菌落接种到斜面培养基上,37℃培养至菌落形成.
1.4.2 微生物的鉴定 16S rDNA基因序列测定和分析:利用酚氯仿抽提法提取菌株的基因组DNA,利用1.3的方法扩增16S rDNA基因.利用DNA胶回收试剂盒回收PCR产物,为了剔除可能相同的菌株,选用内切酶Rsa I和Hha I对所有菌株的PCR纯化产物进行酶切,并利用2%琼脂糖凝胶对酶切结果进行检测.将酶切带谱差异较大菌株的PCR产物进行纯化回收后,送生工生物工程(上海)有限公司进行测序.所得的16S rDNA序列用BLAST软件与GenBank中已知的序列进行同源性比较,确定种属.
细菌的BIOLOG鉴定:选取酶切带谱差异较大的菌株在 BIOLOG专用培养基上连续转接2~3次,划线接种到培养基上,37℃培养24h.用无菌棉签蘸生理盐水湿润后轻轻擦下培养基上的菌落,转入无菌稀释缓冲液中,混匀得到菌悬液,测定其浊度,控制在浊度标准的±3%以内.将 150 µL菌悬液加入到鉴定板的各孔中,盖上鉴定板盖,置于37℃培养箱内,4~6h时读1次,之后16~24h再读1次.
2 结果与讨论
2.1 2种工艺氮磷去除效率的比较
NH3-N含量测定结果显示,运行前期2种工艺的氨氮去除率都不高,16d后倒置A2/O工艺的氨氮去除率(50%)要明显高于常规 A2/O工艺(13%)(图 2A).PO4-P含量测定结果显示,常规A2/O工艺对磷的去除效率波动较大,在处理前期磷的去除率仅为50%,2周后能达到80%以上,倒置A2/O工艺对磷的去除率则一直维持在90%以上,表现更加稳定(图2B).
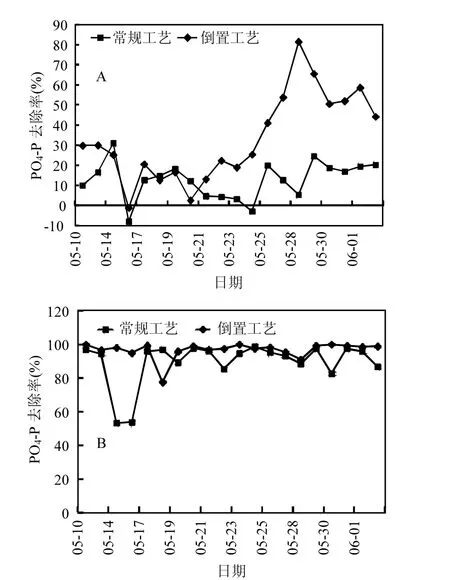
图2 常规A2/O工艺与倒置A2/O工艺对NH3-N与PO4-P的去除率Fig.2 Removal efficiency of NH3-N and PO4-P of conventional A2/O and reversed A2/O processes
2.2 细菌 16S rDNA V3区的纯化与特征片段DGGE指纹图谱
从常规和倒置A2/Oc23kb的DNA片段,这与Bourrain等[11]从活性污泥中提取的DNA大小相同,表明获得了完整的基因组 DNA.随后利用胶回收试剂盒成功纯化了基因组 DNA,以纯化的DNA为模板,对细菌16S rDNA V3区序列进行PCR扩增,2个样品均得到了约为200bp的单一条带,条带的亮度和纯度较好,且无明显非特异性扩增.PCR-DGGE技术是目前分析活性污泥中微生物群落结构常用的方法[11–12],本研究利用该技术比较了常规和倒置A2/O工艺活性污泥中的微生物组成,发现条带1、2、3、4、6、9、10、11、12、13、14、16、18、19在2种样品的DGGE图像中均存在,且比较亮,含量较高,而条带5、7、8、15、17、20在常规工艺活性污泥DGGE图像相对较暗,含量较低,但是在倒置 A2/O工艺样品中亮度较大,含量较高(图3).
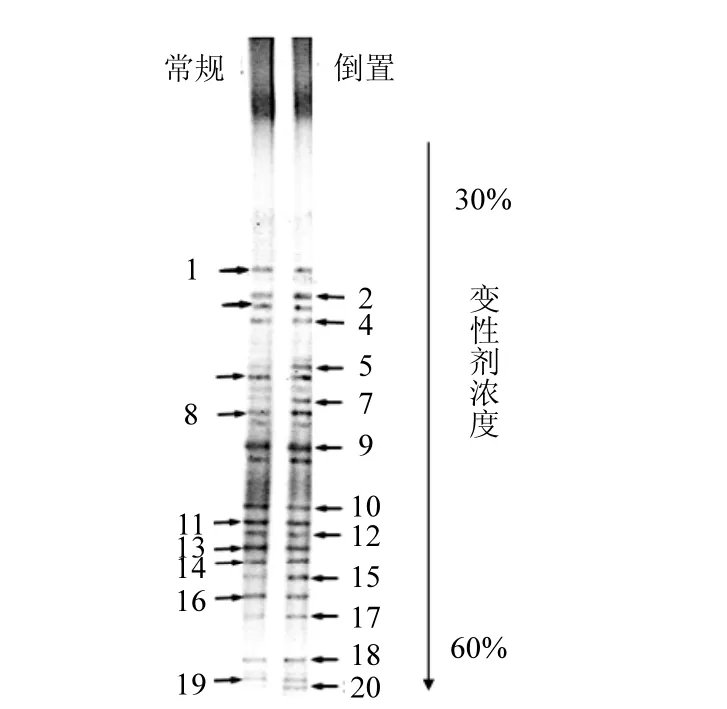
图3 活性污泥样品中细菌16S rDNA V3区片段的DGGE图谱Fig.3 DGGE profiles of PCR amplified 16S rDNA V3region fragments from different samples of activated sludge
2.3 细菌16S rDNA序列同源性与聚类分析
将DGGE分离后可分辨的电泳条带进行回收、扩增及测序,共获得19条170~195bp的序列(条带7未测出),其中有11条序列是环境中未培养的菌株.16S rDNA序列的相似性比较和聚类分析结果表明,这些菌株主要属于拟杆菌属(Bacteroides,55%)、变形菌门(Proteobacteria, 25%)、Candidate division TM7类群(10%)和厚壁菌门(Firmicutes,5%),其中拟杆菌属和变形菌(主要是 β、γ-变形菌)为优势菌类,这与此前研究者对活性污泥中微生物多样性的研究结果相一致
[13–14].Buchan[15]则发现,生物除磷曝气池活性污泥中的优势菌为不动杆菌(Acinetobacter spp.),而本研究采用 PCR-DGGE技术未检测到不动杆菌.
进一步分析DGGE图谱可知,2工艺样品中共有的菌类为盐单胞菌属(Halomonas sp.)、红环菌属(Rhodoferax sp.)、普氏菌属(Prevotella sp.)、黄杆菌科(Niablella koreensis strain)、不可培养的拟杆菌(Bacteroidetes bacterium)、产丁酸菌(butyrate-producing bacterium)、屈挠杆菌属(Flexibacteraceae sp.)、 鞘 氨 醇 杆 菌 属(Sphingobacterium sp.)、不可培养的 candidate division TM7、不可培养的PHOS-HE28菌与γ-变形菌纲(Gammaproteobacteria) (表1).相比之下,可培养的亚硝化螺菌(Nitrosospira sp.,条带18)、红环菌属(Rhodocyclus sp.,条带15)与4种不可培养的拟杆菌(条带5、17、19、20)在常规工艺中含量较少,但在倒置工艺中含量较高.亚硝化螺菌对亚硝酸盐具有很低的米氏常数(Km值),并且对有限氧表现出很强的竞争力[16],刘惠军等[17]发现,亚硝化螺菌是曝气生物反应器中起主要氨氧化作用的细菌,占细菌总数的 21%;红环菌通常是磷含量高的生物除磷体系中的优势菌[18],例如 Bond等[19]发现,红环菌在高效除磷系统中含量较高,而在低效除磷系统中含量较少,推测红环菌可能是造成两系统除磷功能差异的主要原因.Zilles等[20]也报道,红环菌是生物强化除磷系统中的优势聚磷菌,发挥着主要除磷作用.因此,推测亚硝化螺菌与红环菌属可能是造成倒置 A2/O工艺具有较高脱氮除磷效果的主要原因之一.
为了分析鉴定菌种与已知菌种的亲缘关系,进一步构建了系统发生树(图4),常规A2/O工艺活性污泥中获得的18条序列中,有10条属于拟杆菌门(Bacteroidetes),包括2条鞘脂杆菌纲的鞘脂杆菌属(Sphingobacterium sp.)、1条拟杆菌纲普雷沃氏菌属(Prevotella sp.)、1条黄杆菌纲黄杆菌科(Flavobacteriaceae)和1条噬纤维菌-屈挠杆菌-拟杆菌(Cytophaga—Flexibacter- Bacteroides,CFB);5条序列属于变形菌门(Proteobacteria),包括β-变形细菌(3条)和γ-变形细菌(2条);1条序列属于厚壁菌门梭菌目(Clostridiales).倒置 A2/O工艺活性污泥中鉴定的菌属与常规A2/O工艺活性污泥相似,唯一多出的条带 20属于拟杆菌门(Bacteroidetes).

表1 DGGE回收DNA片段序列分析结果Table 1 Sequence identification of 16S rDNA DGGE fragments
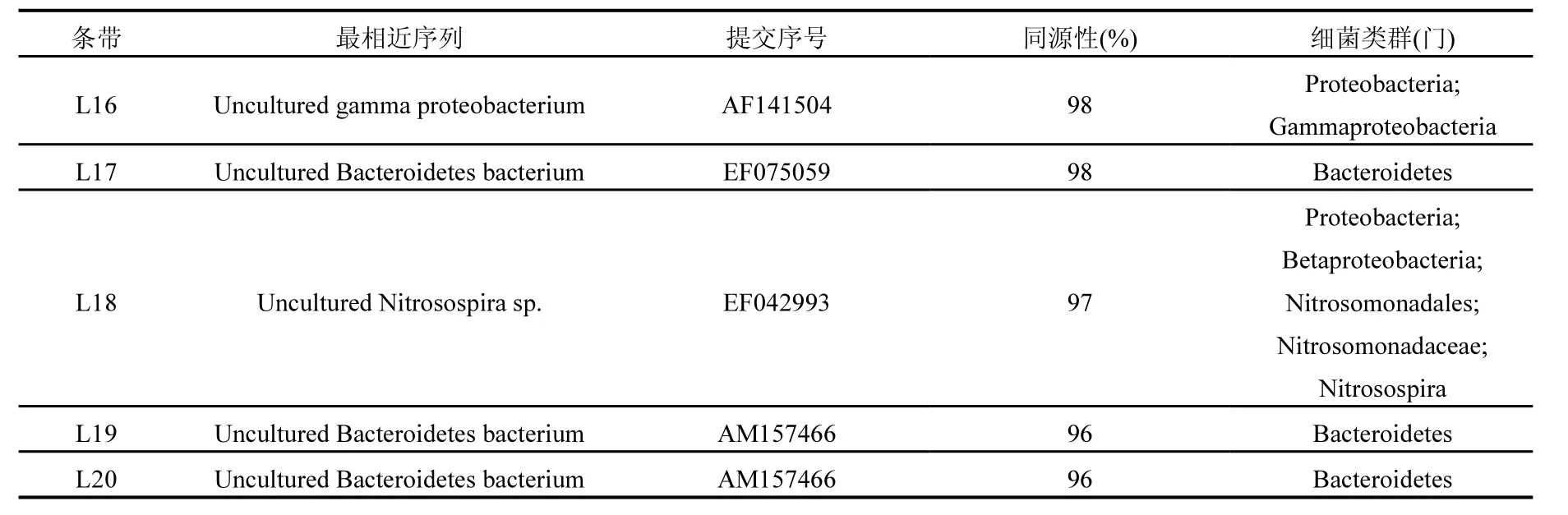
续表1

图4 DGGE条带16S rDNA序列聚类分析Fig.4 Cluster analysis of DGGE patterns from 20samples
2.4 活性污泥中微生物的形态观察
扫描电镜结果显示,常规A2/O与倒置A2/O工艺活性污泥中均含有大量的球状菌和杆状菌,这与SBR工艺活性污泥的研究结果相近[21].常规A2/O工艺活性污泥菌胶团比较致密,含有大量的丝状细菌和部分念珠状细菌(图 5),而倒置 A2/O工艺活性污泥则较为疏松,丝状细菌含量较少,且未发现念珠状细菌(图6).丝状细菌能显著影响絮状活性污泥的沉降性(污泥膨胀)或引起生物量变化和泡沫形成(污泥发泡),严重时可能导致活性污泥运行的瘫痪[22].可见,与常规 A2/O工艺相比,倒置A2/O工艺能够避免污泥膨胀等异常问题,更有助于维持活性污泥的处理效率.

图5 常规A2/O工艺曝气池末端活性污泥扫描电镜图Fig.5 Scanning electron micrograph of activated sludge in A2/O procedureA:丝状细菌; B:球状菌; C:杆状菌; D:念珠状菌; 4:菌胶团

图6 倒置A2/O工艺曝气池末端活性污泥扫描电镜图Fig.6 Scanning electron micrograph of activated sludge in reversed A2/O procedureA:丝状细菌; B:球状菌; C:杆状菌; 4:菌胶团
2.5 微生物的分离与鉴定

图7 34株纯化菌的ARDRA结果分析Fig.7 ARDRA analysis of 34bacterial isolatesC1~C19:常规工艺中分离的菌株;D1-D15:倒置工艺中分离的菌株
采用培养方法从活性污泥样品中共分离得到34株菌,聚类进化树结果显示它们属于11种菌株(图7).BIOLOG分析将其中3株鉴定到了属,另外8株则准确鉴定到了种.16S rDNA同源性比对结果显示这些菌种可分为 5大类:α-变形杆菌纲(Alphaproteobacteria)、β-变形杆菌纲(Betaproteobacteria)、γ-变形杆菌纲(Gammaproteobacteria)、放线菌纲(Actinobacteria)、芽孢杆菌纲(Bacillibacteria),其中优势菌为 β-变形菌纲、γ-变形菌纲和放线菌纲(表2),这与PCR-DGGE技术鉴定得到的优势菌种相类似.然而,PCR-DGGE检测到的红环菌在纯培养研究中并未分离到,而 PCRDGGE技术未检测到的不动杆菌却获得了纯培养.对比2种工艺发现,γ-变形菌纲中的希瓦氏菌属(Shewanella algae sp.)、克雷伯氏菌属(Klebsiella sp.)和类球红细菌(Rhodobacter sphaeroides)只在倒置工艺中分离得到,而放线菌纲的滕黄微球菌(Micrococcus luteus)只在常规工艺中分离到.Fuhs等[23]发现,高效除磷的活性污泥中可培养的细菌主要是 γ-亚纲变形菌的不动杆菌;Suresh等[24]发现,假单胞菌和克雷伯氏菌具有很强的除磷能力.此外, Liu等[25]与 Gieseke等[26]在具有较高除磷能力的活性污泥中都检测到了倒置 A2/O工艺特有的这 3种微生物大量存在.因此,推测以上菌株的差异也可能是造成两工艺脱氮除磷差异的原因.
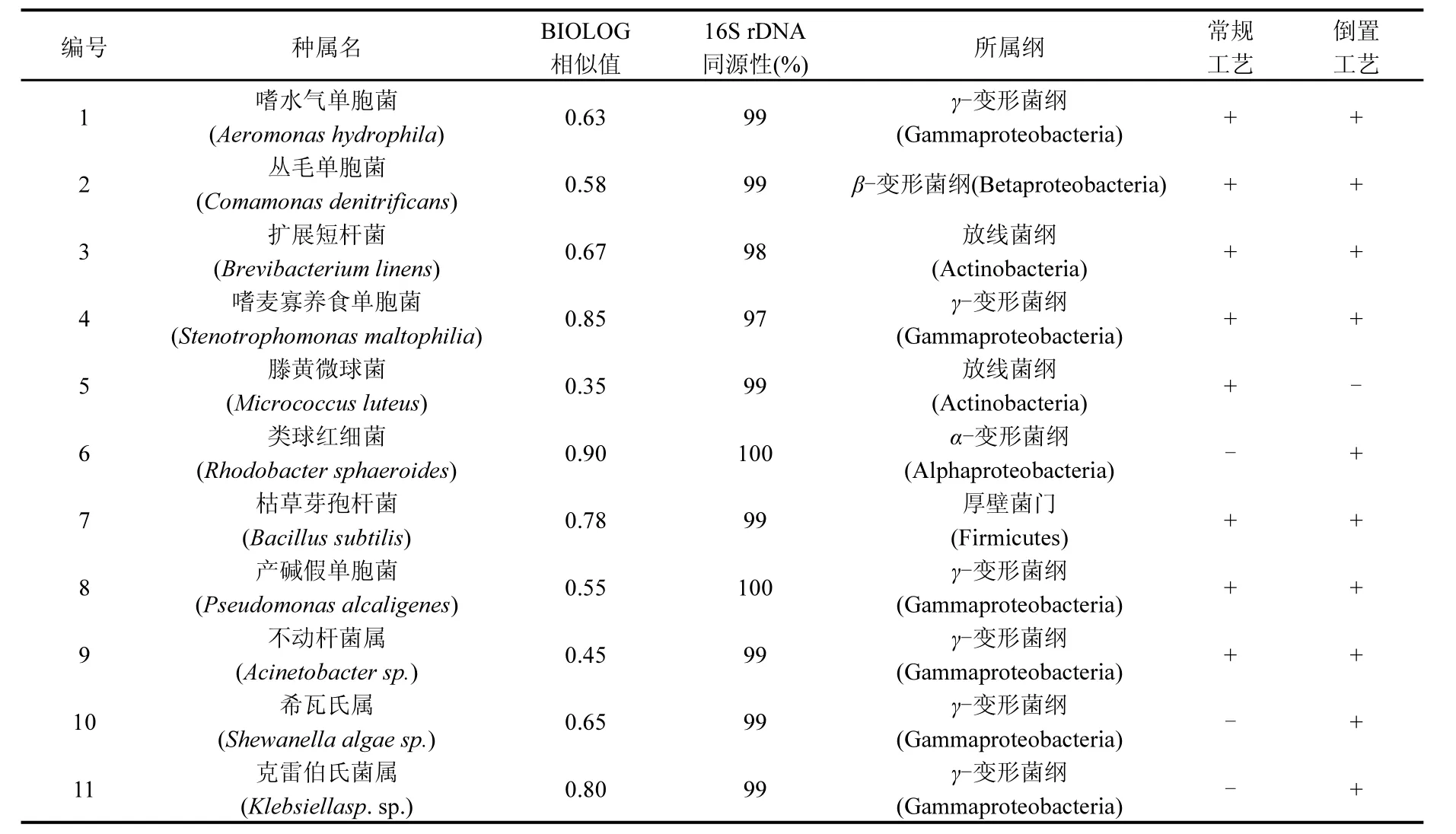
表2 BIOLOG与16S rDNA同源性鉴定结果Table 2 Bacterial strains identification by BIOLOG technique and 16S rDNA gene analysis
3 结论
3.1 利用PCR-DGGE技术检测了常规A2/O和倒置 A2/O工艺活性污泥中的微生物群落结构,发现亚硝化螺旋菌、红环菌和4种不可培养的拟杆菌在倒置A2/O工艺活性污泥中大量存在,但在常规工艺中含量较少.推测这些特殊菌种的存在可能是造成倒置A2/O工艺具有较高脱氮除磷效果的主要原因.
3.2 常规 A2/O工艺活性污泥菌胶团比较致密,含有大量的丝状细菌、杆状菌和球状菌,同时还有部分念珠状细菌,而倒置A2/O工艺活性污泥菌胶团比较疏松,丝状细菌相对较少,且不存在念珠状细菌.γ-变形菌纲中的希瓦氏菌属、克雷伯氏菌属和类球红细菌只在倒置工艺中分离到,这也可能是造成两种工艺脱氮除磷效果差异的原因.
3.3 PCR-DGGE技术与纯培养方法均发现两种工艺活性污泥中的优势菌群均包括 β-变形菌与 γ-变形菌.此外,PCR-DGGE技术检测到的红环菌在纯培养研究中未分离到,而 PCR-DGGE技术未检测到的不动杆菌却获得了纯培养,表明两种方法相结合可以取得更加高效准确的鉴定结果.
[1] Liu C X, Zhang Y, Yang M, et al. Analysis of bacterial community structures in two sewage treatment plants with different sludge properties and treatment performance by nested PCR-DGGE method [J]. Journal of Environmental Sciences, 2007,19(1):60—66.
[2] 朱海霞,陈林海,张大伟,等.活性污泥微生物菌群研究方法进展[J]. 生态学报, 2007,27(1):314—322.
[3] 王振江,武鹏崐,毕学军.倒置A2/O生物脱氮除磷工艺的生产性试验 [J]. 中国给水排水, 2006,22(19):36—38.
[4] 张 波,高迁耀.倒置A2/O工艺的原理与特点研究 [J]. 中国给水排水, 2000,16(7):11—15.
[5] 李亚静,孙力平.常规A2/O工艺和倒置A2/O工艺处理城市污水比较研究 [J]. 环境工程, 2015,33:967—970.
[6] Zhou J, Bruns M A, Tiedje J M. DNA recovery from soils of diverse composition [J]. Applied and environmental microbiology, 1996,62(2):316—322.
[7] Bosshard P P, Santini Y, Grüter D, et al. Bacterial diversity and
community composition in the chemocline of the meromicticalpine Lake Cadagno as revealed by 16S rDNA analysis [J]. FEMS Microbiology Ecology, 2000,31(2):173—182.
[8] 杨 倩,蒋阳月,王小军,等.不同区域污水处理厂活性污泥中微生物菌落结构分析 [J]. 化工进展, 2014,33(12):3329—3336.
[9] 葛 峰,郭 坤,周广灿,等.南京市 4个污水处理厂的活性污泥中细菌的分离鉴定和抗生素耐药性分析 [J]. 环境科学, 2012,33(5):1646—1651.
[10] Bourrain M, Achouak W, Urbain V, et al. DNA extraction from activated sludges [J]. Current microbiology, 1999,38(6):315—319.
[11] Dar S A, Kuenen J G, Muyzer G. Nested PCR-denaturing gradient gel electrophoresis approach to determine the diversity of sulfate-reducing bacteria in complex microbial communities [J]. Applied and environmental microbiology, 2005,71(5):2325—2330.
[12] Chen Y, Ren C G, Yang B, et al. Priming effects of the endophytic fungus Phomopsis liquidambari on soil mineral N transformations [J]. Microbial ecology, 2013,65(1):161-170.
[13] 刘新春,吴成强,张 昱,等. PCR-DGGE法用于活性污泥系统中微生物群落结构变化的解析 [J]. 生态学报, 2005,25(4): 842—847.
[14] 王海燕,周岳溪,蒋进元.强化生物除磷系统的微生物种群及其表征技术 [J]. 微生物学通报, 2005,32(1):118—122.
[15] Buchan L. Possible biological mechanism of phosphorus removal [J]. Water Science and Technology, 1983,15(3/4):87—103.
[16] Laanbroek H J, Gerards S. Competition for limiting amounts of oxygen between Nitrosomonas europaea and Nitrobacter winogradskyi grown in mixed continuous cultures [J]. Archives of Microbiology, 1993,159(5):453—459.
[17] 刘惠军,杨凤林,张捍民,等.炭膜曝气生物膜反应器硝化作用及其微生物群落结构分析 [J]. 环境科学, 2007,28(9):2123—2128.
[18] Hesselmann R P X, Werlen C, Hahn D, et al. Enrichment, phylogenetic analysis and detection of a bacterium that performs enhanced biological phosphate removal in activated sludge [J]. Systematic and Applied Microbiology, 1999,22(3):454—465.
[19] Bond P L, Keller J, Blackall L L. Characterisation of enhanced biological phosphorus removal activated sludges with dissimilar phosphorus removal performances [J]. Water Science and Technology, 1998,37(4):567—571.
[20] Zilles J L, Hung C H, Noguera D R. Presence of Rhodocyclus in a full-scale wastewater treatment plant and their participation in enhanced biological phosphorus removal [J]. Water Science and Technology, 2002,46(1/2):123—128.
[21] Van Houten R, Evenblij H, Keijmel M. Membrane bioreactors hit the big time: ten years of research in The Netherlands [J]. H2O MBR Special, 2001:26—31.
[22] Guo F, Zhang T. Profiling bulking and foaming bacteria in activated sludge by high throughput sequencing [J]. Water research, 2012,46(8):2772—2782.
[23] Fuhs G W, Chen M. Microbiological basis of phosphate removal in the activated sludge process for the treatment of wastewater [J]. Microbial Ecology, 1975,2(2):119—138.
[24] Suresh N, Warburg R, Timmerman M, et al. New strategies for the isolation of microorganisms responsible for phosphate accumulation [J]. Water science and technology, 1985,17(11/12): 99—111.
[25] Liu P, Liu K, He J. A novel analysis to the ridged cylinder TEM transmission line [J]. International journal of infrared and millimeter waves, 1995,16(6):1083—1090.
[26] Gieseke A, Arnz P, Amann R, et al. Simultaneous P and N removal in a sequencing batch biofilm reactor: insights from reactor-and microscale investigations [J]. Water Research, 2002, 36(2):501—509.
Microbial diversity in activated sludges of conventional and reversed A2/O processes.
LI Peng1,2, BI Xue-jun3, WANG Jun2, RU Shao-guo2*(1.Shandong Academy of Environmental Science, Jinan 250013, China;2.Marine Life Science College, Ocean University of China, Qingdao 266003, China;3.School of Environmental and Municipal Engineering, Qingdao Technological University, Qingdao 266033, China). China Environmental Science, 2017,37(3):1137~1145
To explore the reason why removal efficiency of nitrogen and phosphorus in reversed anoxic-anaerobic-oxic (A2/O) process is higher than that in conventional A2/O process, bacterial community compositions of activated sludges from the two procedures were investigated by PCR-DGGE (polymerase chain reaction-denaturing gradient gel electrophoresis) technique and cultivation method. Betaproteobacteria and Gammaproteobacteria were detected as dominant bacteria in activated sludge from both processes. In the activated sludge of the reversed A2/O process, a large amount of Nitrosospira sp., Rhodocyclus sp., and four uncultured bacteroidetes bacterium were detected by PCR-DGGE technique, while less of these bacteria were found in the conventional A2/O process. Shewanella algae sp., Klebsiella sp., and Rhodobacter sphaeroides of Gammaproteobacteria were separated from the activated sludge of reversed A2/O process by cultivation method, while they were absent from the conventional A2/O process. The diversity of these above-mentioned bacteria species could account for the higher removal efficiency of nitrogen and phosphorus in the reversed A2/O process. Additionally, scanning electron microscopy showed that the activated sludge of reversed A2/O process was relatively loose, with less Filamentous bacteria and no Nostoc.
activated sludge;microbial community structure;PCR-DGGE;reversed A2/O process
X172
A
1000-6923(2017)03-1137-09
李 鹏(1981-),男,山东兖州人,高级工程师,硕士,主要从事污水处理活性污泥微生物等方面研究.发表论文10余篇.
2016–07–22
山东省自然科学基金重点项目(ZR2014DZ001); 高等学校博士学科点专项科研基金资助课题(20120132110011)
* 责任作者, 教授, rusg@ouc.edu.cn
