乌鲁木齐牛羊肉源沙门氏菌对喹诺酮类药物的耐药状况及相关基因分析
2017-03-27杨保伟吴浩天张亚南马文瑞
葛 琨,武 运,*,杨保伟,吴浩天,王 威,张亚南,田 歌,马文瑞
(1.新疆农业大学食品科学与药学学院,新疆 乌鲁木齐 830052;2.西北农林科技大学食品科学与工程学院,陕西 杨凌 712100)
乌鲁木齐牛羊肉源沙门氏菌对喹诺酮类药物的耐药状况及相关基因分析
葛 琨1,武 运1,*,杨保伟2,吴浩天1,王 威1,张亚南1,田 歌1,马文瑞1
(1.新疆农业大学食品科学与药学学院,新疆 乌鲁木齐 830052;2.西北农林科技大学食品科学与工程学院,陕西 杨凌 712100)
目的:研究牛羊肉源沙门氏菌对15 种抗生素的药敏性以及对喹诺酮类药物的相关耐药基因,更好地了解沙门氏菌耐药性的产生和传播途径,为预防与控制沙门氏菌疾病提供基础信息。方法:用琼脂稀释法测定沙门氏菌的药敏性,用聚合酶链式反应和基因序列测定法确定耐药沙门氏菌中与喹诺酮类抗生素耐药相关的喹诺酮类抗性决定区基因突变及质粒携带的耐药基因。结果:30 株沙门氏菌中对甲氧苄啶、氯霉素、萘啶酮酸、四环素、磺胺异甲二唑、链霉素、甲氧嘧啶/磺胺异恶唑、阿莫西林、氨苄西林的耐药率分别为100%、86.7%、66.7%、60.0%、50.0%、33.3%、26.7%、6.7%、6.7%,对环丙沙星、头孢曲松、庆大霉素、卡那霉素、头孢西丁、阿米卡星均表现为敏感;qnrB、qnrA、qnrS、aac(6′)-Ib-cr基因的检出率分别为5.0%、45.0%、0%、5.0%;30 株沙门氏菌发生gyrA基因突变的菌株数为14 株,主要突变类型是Ser83Phe,发生parC基因突变的菌株数为25 株,主要突变类型是Thr57Ser。结论:乌鲁木齐牛羊肉源沙门氏菌的耐药情况较为严重,对喹诺酮类药物的耐药状况应当予以关注,其耐药突变基因及质粒携带的耐药基因在一定程度上是导致沙门氏菌耐药的重要原因。
沙门氏菌;药敏性;喹诺酮;耐药基因
沙门氏菌病是公共卫生学上有重要意义的人畜共患病之一,对人类和动物健康都具有极大威胁[1]。目前,由沙门氏菌引起的食源性疾病不仅成为全世界非常重视的公共卫生问题,对全球食品安全和人类健康也造成了巨大威胁[2]。近年来,由于抗生素在农业、畜牧业和临床治疗中广泛使用甚至滥用,导致病原菌的耐药性逐步增强,耐药谱逐渐加宽,因此,加强该菌耐药性检测具有重要的公共卫生意义[3-6]。
喹诺酮类药物是人和动物的沙门氏菌性胃肠炎疾病的常规用药,有时为让动物快速生长,会将该类抗生素药物伴随饲料添加到动物的食物中,长时间后多种血清型沙门氏菌对喹诺酮类药物的敏感性下降,导致多重耐药性沙门氏菌越来越常见,因此了解沙门氏菌的耐药机制对预防与控制沙门氏菌疾病有着重要意义[7-10]。
目前,国内外研究者认为,编码DNA旋转酶A、B、C、D亚单位中任一亚基的基因发生突变均可引起喹诺酮类的耐药性,其中DNA旋转酶亚基GyrA和拓扑异构酶Ⅳ亚基ParC中的部分基因又是影响耐药性的主要亚基基因。另外,有研究表明,导致沙门氏菌对喹诺酮类药物产生抗性的另一原因是质粒携带耐药基因的转移。许多耐药质粒含有多种耐药基因,如qnrA、qnrB、qnrS、aac(6′)-Ib-cr质粒基因,其广泛存在于自然界中的许多细菌中,并且能在细菌之间相互传播,导致沙门氏菌对喹诺酮类抗生素药物的抗性不断提高[11-12]。
本研究主要分析了30 株牛羊肉源沙门氏菌对15 种抗生素药物的药敏性情况,及喹诺酮相关耐药基因,以更好地了解沙门氏菌耐药性的产生和传播途径,为预防与控制沙门氏菌疾病提供理论依据。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株
沙门氏菌(Salmonella typhimurium),分离自2013—2014年采集于乌鲁木齐地区农贸市场的325 份零售羊肉样品(21 株)及210 份零售牛肉样品(9 株)。沙门氏菌标准菌株LT2、药敏实验质控菌株大肠埃希菌ATCC25922和粪肠球菌ATCC29212均为西北农林科技大学食品科学与工程学院微生物实验室保存。
1.1.2 培养基与试剂
LB培养基 北京陆桥技术有限责任公司。
沙门氏菌属O多价抗血清A-F 宁波天润生物药业有限公司;Taq DNA聚合酶、dNTPmix、10×Buffer、DL2000 DNA Marker、DL100 DNA Marker 宝生物工程(大连)有限公司;氨苄西林(ampicillin,AMP)、阿莫西林-克拉维酸(amoxicillin-clavulanic acid,AMC)、头孢曲松(ceftriaxone,CRO)、头孢西丁(cefoxitin,CEF)、氯霉素(chloramphenicol,CHL)、四环素(tetracycline,TET)、链霉素(streptomycin,STR)、阿米卡星(amikacin,AMK)、庆大霉素(gentamicin,GEN)、卡那霉素(kanamycin,KAN)、磺胺甲基异恶唑-甲氧苄啶(sulfamethoxazole-trimethoprim,SXT)、磺胺二甲异唑(sulfadimethisoxazol,SMX)、甲氧苄啶(trimethoprim,TIO)、萘啶酮酸(nalidixic acid,NAL)、环丙沙星(ciprofloxacin,CIP) 美国Sigma公司。
1.1.3 PCR扩增引物
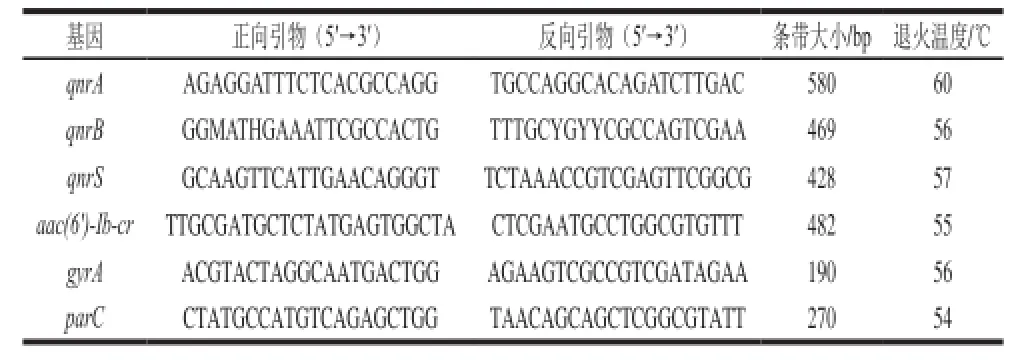
表1 PCR扩增基因与引物Table1 PCR primers for the target genes
qnrA、qnrB、qnrS、aac(6′)-Ib-cr、gyrA和parC基因扩增用引物均使用Primer Premier 5软件设计,由上海捷锐生物工程有限公司合成(表1)。
1.2 仪器与设备
超净工作台 苏州净化设备有限公司;微波炉 广州美的微波炉制造有限公司;隔水式培养箱 江苏东鹏仪器制造有限公司;三用恒温水箱 常州中捷实验仪器制造有限公司;鼓风干燥箱 宝康电器设备有限公司;超纯水器 德国Eppendoff公司;4 ℃冰箱 海尔电器股份有限责任公司;-40 ℃低温冰箱、-80 ℃超低温冰箱日本三洋公司;高压灭菌锅 上海申安医疗器械厂;旋涡振荡器 海门其林贝尔仪器制造有限公司;分析天平 上海民桥精密科学仪器有限公司;磁力加热搅拌器 美国Thermo Fisher公司;高速台式离心机 上海安亭科学仪器厂;聚合酶链式反应(polymerase chain reaction,PCR)仪 美国Bio-Rad公司。
1.3 方法
1.3.1 药敏性测定
对经分离鉴定的沙门氏菌按照美国临床实验室标准化委员会(clinical and laboratory standards institute,CLSI)推荐的琼脂稀释法测定抗生素的最小抑菌浓度,按照CLSI标准判读结果并确定耐药表型进行耐药性检测[13]。药敏测定中使用大肠埃希菌ATCC25922和粪肠球菌ATCC29212质控菌株。
1.3.2 喹诺酮类抗生素相关耐药基因检测
1.3.2.1 沙门氏菌DNA提取
用无菌棉签蘸取适量在LB培养基上过夜培养的沙门氏菌,将其均匀洗涤于无菌生理盐水中,制成麦氏浊度为0.5的菌悬液,取800 μL菌悬液于1.5 mL无菌离心管中,100 ℃左右加热煮沸10 min,13 200 r/min离心5~8 min后,吸取上清液,备用。
1.3.2.2 PCR扩增
PCR体系(25 μL)为:13.25 μL ddH2O、2.5 μL10×PCR Buffer、2.0 μL dNTP、1.5 μL MgCl2、0.25 μL Taq酶、上游引物、下游引物和5 μL DNA模板。PCR条件为:94 ℃预变性10 min;94 ℃变性1 min,60 ℃退火1 min,72 ℃延伸1 min,35 个循环;72 ℃延伸10 min。不同基因扩增过程的退火温度应由相应的引物序列来确定。PCR产物经琼脂糖凝胶电泳分离后,使用凝胶成像系统照相。
1.3.2.3 DNA序列测定
gyrA和parC基因扩增产物由上海桑尼生物科技有限公司纯化后测序。
1.3.2.4 gyrA、parC基因突变位点的确定
将测定得到的DNA序列输入基因库,采用软件BLAST程序在线比对,在确定标准菌株Salmonella typhimurium LT2的DNA序列和基因库序列完全吻合以后,比对分析供试菌株相应的DNA序列,来确定突变点和突变的种类。
2 结果与分析
2.1 药敏性结果
2.1.1 沙门氏菌对供试抗生素的药敏性
由图1可知,30 株沙门氏菌均对TIO有抗性,但对KAN、CRO、GEN、CEF、AMK和CIP均表现敏感,对其他供试抗生素的总耐药率依次为CHL 86.7%、NAL 66.7%、TET 60.0%、SMX 50%、STR 33.3%、SXT 26.7%、AMP 6.7%、AMC 6.7%。在每一特定数量的抗生素谱系中,以4 种和5 种抗生素组成的耐药谱种类最多(4 类),其次为2 种抗生素组成的耐药谱(3 类)和3 种(2 类)、6 种(2 类)、7 种(2 类)及8 种(1 类)抗生素组成的耐药谱。

图1 牛羊肉源沙门氏菌对抗生素的耐药率Fig.1 Antibiotic resistance of beef and mutton-borne Salmonella
2.1.2 牛羊肉源沙门氏菌的耐药谱

表2 牛羊肉源沙门氏菌的耐药谱和相应菌株数Table2 Antimicrobial resistance prof i les of beef and mutton-borne Salmonella and corresponding numbers
由表3可知,30 株乌鲁木齐地区牛羊肉源沙门氏菌中,同时对2~3 种抗生素产生抗性菌株数为8 株(占比26.6%),同时对4~5 种抗生素产生抗性菌株数15 株(50.0%),同时对6~7 种抗生素产生抗性菌株数为6 株(20.0%),同时对8 种抗生素产生抗性菌株数为1 株(3.3%)。
2.2 喹诺酮类抗生素耐药相关gyrA、parC基因扩增及突变结果
由图2、3可知,gyrA、parC基因的DNA大小分别为190 bp和270 bp。由表3可知,30 株沙门氏菌的gyrA、parC基因中共检出39 个点突变,25 株(83.33%)沙门氏菌parC基因中发生的Thr57Ser变异为常见突变,在该位点,由于基因突变而使苏氨酸突变为丝氨酸。14 株沙门氏菌gyrA基因突变株中,有11 株在83位氨基酸处发生突变,突变类型为Ser83Phe或Ser83Tyr。另外,有2 株在87位氨基酸处发生突变,突变类型为Asp87Tyr或Asp87Asn,只有1 株沙门氏菌突变类型为Gly75Phe。

图 2gyrA基因PCR扩增结果Fig.2 PCR amplifi cation results of gyrA gene

图 3parC基因PCR扩增结果Fig.3 PCR amplifi cation results of parC gene

表3 30 株沙门氏菌gyrA、parC 基因突变结果Table3 gyrAandparC mutations in 30Salmonella isolates
在20 株耐萘啶酮酸的菌株中,13 株检测到gyrA与parC同时突变,2 株仅parC突变,3 株仅gyrA突变,gyrA和/或parC基因突变导致菌株耐萘啶酮酸的比例达90.0%。在25 株沙门氏菌parC基因突变株中,耐萘啶酮酸菌株为15 株(占比60.0%),在18 株沙门氏菌gyrA突变株中,耐萘啶酮酸菌株为16 株(占比88.89%)。
2.3 喹诺酮类抗生素耐药相关质粒基因检出结果

图4 qnrA基因PCR扩增结果Fig.4 PCR amplifi cation results of qnrA gene

图5 qnrB基因PCR扩增结果Fig.5 PCR amplifi cation results of qnrB gene

图6 aac(6ʹ)-Ib-cr基因PCR扩增结果Fig.6 PCR amplifi cation results of aac(6ʹ)-Ib-cr gene
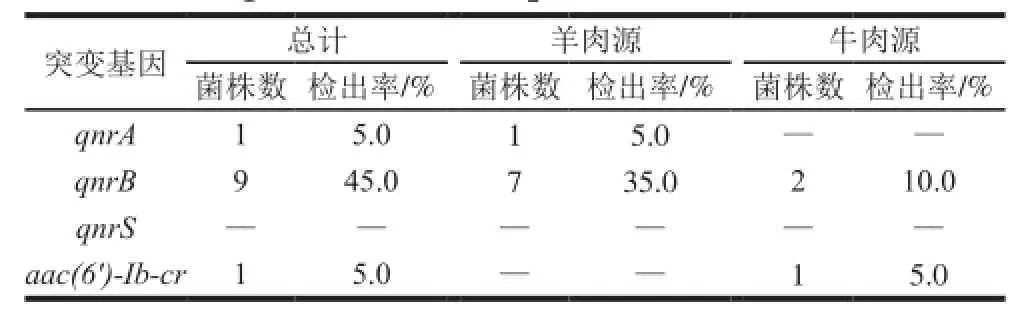
表4 牛羊肉源沙门氏菌相关质粒基因检出结果Table4 Results of detection of plasmid-mediated quinolone resistance genes in beef and lamp-borne Salmonella
由图4~6可知,通过PCR扩增基因产物进行电泳后得qnrA、qnrB、aac(6′)-Ib-cr基因的DNA大小分别为580、469、428 bp。由表4可知,20 株耐一代喹诺酮NAL的沙门氏菌菌株中,qnrB基因的检出率最高,为45.0%,qnrA和aac(6′)-Ib-cr基因的检出率均为5.0%,qnrS基因未检出,qnrB质粒基因的检出率要明显高于其他3 种质粒基因。其中羊肉源中沙门氏菌qnrA、qnrB基因的检出率分别为5.0%、35.0%,qnrS、aac(6′)-Ib-cr基因均未检出,而牛肉源中沙门氏菌qnrB、aac(6′)-Ib-cr基因的检出率分别为10.0%、5.0%,qnrA、qnrS基因均未检出。
3 讨 论
沙门氏菌是目前最常见的食源性致病菌,我国每年大约有3 亿人因沙门氏菌感染而患病,占我国病原菌型食源性疾病总数的70%~80%[13]。因此,为治疗沙门氏菌性疾病,大量的抗生素药物开始被使用,使沙门氏菌对许多抗生素药物产生耐药性。从检测结果来看,乌鲁木齐地区牛羊肉源沙门氏菌对TIO(100%)、CHL(86.7%)、NAL(66.7%)、TET(60.0%)、SMX(50.0%)5 种抗生素的耐药率均在50.0%以上,这可能源于这些抗生素在该地区牛羊肉生产过程中的广泛使用致使细菌的耐药性普遍偏高。NAL是应用于治疗沙门氏菌疾病的第1代喹诺酮类药物[14],本研究中牛羊肉源沙门氏菌分离株对NAL的耐药程度比较严重,耐药率高达66.7%,但对3代喹诺酮CIP完全敏感,这一结果与目前许多的研究结果是相一致的,这对掌握新疆乌鲁木齐沙门氏菌的耐药表型从而选择相应的应对措施有着重要意义[15]。
目前,病原沙门氏菌染色体DNA中编码解旋酶和拓扑异构酶Ⅳ亚基上的氟喹诺酮类抗生素抗性决定区基因突变是国内外研究认为沙门氏菌对喹诺酮的耐药的主要机制之一[16],而编码基因gyrA和parC又分别是解旋酶和拓扑异构酶Ⅳ上发生突变的主要编码基因。检测结果表明,在20 株耐萘啶酮酸的菌株中,gyrA与parC编码基因中有一种发生突变导致菌株耐萘啶酮酸的比例达到了90.0%,从突变结果上来看,gyrA编码基因的突变类型主要是Ser83Phe,即83位的丝氨酸被苯丙氨酸替代,另外也有少部分其他的突变类型,如Ser83Tyr、Asp87Tyr、Asp87Asn、Gly75Phe;parC基因突变类型主要是是Thr57Ser,即57位的苏氨酸突变为丝氨酸,这些突变类型也已经被许多研究者证实[17]。
另外,编码基因gyrA与parC的突变在导致菌株的耐药程度上又存在差异[18],在25 株检测到parC发生突变的沙门氏菌菌株中,对NAL的产生抗性的菌株数为15 株,所占比例为60.0%,在18 株检测到gyrA发生突变的沙门氏菌菌株中,对NAL产生抗性的菌株数为16 株,所占比例为88.9%,由此可以得出的结论是,编码基因gyrA基因突变在一定程度上比parC基因突变更容易导致沙门氏菌对NAL产生抗性,这与现有的结论是一致的。对沙门氏菌进行耐喹诺酮类药物相关基因的持续监控是减少耐药性产生的重要措施之一[19]。
导致沙门氏菌对喹诺酮类药物产生抗性的另外一个机制是质粒携带耐药基因的转移[20]。许多耐药质粒含有多种耐药基因,它们广泛存在于自然界中的许多细菌中,并且能在许多细菌之间相互传播。目前,许多研究结果表明,qnrA与qnrB两种质粒基因在大肠杆菌等肠杆菌属中均有检出[21],qnrS和aac(6′)-Ib-cr基因的相关研究报道还很少。本实验检测结果表明,在20株耐萘啶酮酸的沙门氏菌菌株中,qnrB基因的检出率要明显高于其他3 种质粒基因,共检测出9 株(45.0%),qnrA和aac(6′)-Ib-cr基因的检出数均为1 株(5.0%),qnrS基因未检出,但由于样本数量较小的原因,由此得出的结论与现有的研究结果并不完全一致,需要做进一步的调查研究。
沙门氏菌对一代喹诺酮的耐药情况与携带耐药基因的相关质粒基因的检出与否存在一定关系[22-24],在检出qnrA、qnrB、aac(6′)-Ib-cr质粒基因的12 株沙门氏菌菌株中,对一代喹诺酮药物NAL产生抗性的菌株数为10株,耐药比例达到了83.33%,这说明,携带耐药基因的相关质粒在一定程度上可以导致沙门氏菌对一代喹诺酮药物产生抗性。
本研究结果表明,新疆乌鲁木齐地区牛羊肉源沙门氏菌对常用抗生素药物的耐药情况较为严重,并且进一步分析了喹诺酮相关耐药基因的情况。另外,耐药基因的检测有助于了解乌鲁木齐地区流行株的耐药机制,从而丰富和完善牛羊肉源致病性沙门氏菌耐药性的分子流行病学资料,也为临床治疗沙门氏菌疾病的合理用药及沙门氏菌多重耐药机制的进一步研究提供实验依据。
[1] 杨保伟, 申进玲, 席美丽, 等. 2007—2008年西安地区鸡肉源沙门氏菌相关特性分析[J]. 食品科学, 2011, 32(19): 130-136.
[2] 张凌云. 鸡场沙门氏菌的分离鉴定及耐药硏究[D]. 长春: 吉林农业大学, 2011: 11-14.
[3] 方翟. 湖北省鸡源沙门氏菌的分离鉴定及耐药性分析[D]. 武汉:华中农业大学, 2014: 2-12.
[4] BAUCHERON S, TYLER S, BOYD D, et al. AcrAB-TolC directs efflux-mediated multidrug resistance in Salmonella enterica serovar typhimurium DT104[J]. Antimicrobial Agents and Chemotherapy, 2004, 48: 3729-3735. DOI:10.1128/AAC.48.10.3729-3735.2004.
[5] 郝宏珊. 八省市鸡肉源沙门氏菌污染状况调查及其特性研究[D]. 杨凌: 西北农林科技大学, 2012: 3-5.
[6] 吴云凤, 袁宝君, 乔昕, 等. 肉鸡胴体中沙门氏菌的分离鉴定及多重耐药谱研究[J]. 南京医科大学学报, 2012, 32(1): 125-128.
[7] ALCAINE S D, WARNICK D L, WIEDMANN M. Antimicrobial resistance in nontyphoidal Salmonella[J]. Journal of Food Protection, 2007, 70(3): 780-790 .
[8] 刘渠, 刘衡川, 李灶平, 等. 食品中沙门氏菌的耐药性研究[J]. 现代预防医学, 2004, 31(3): 330-332. DOI:10.3969/ j.issn.1003-8507.2004.03.007.
[9] ROBICSEK A, STRAHILEVITZ J, SAHM D F, et al. qnr prevalence in ceftazidime-resistant Enterobacteriaceae isolates from the United States[J]. Antimicrobial Agents and Chemotherapy, 2006, 50(8): 2872-2874. DOI:10.1128/AAC.01647-05.
[10] 邹明, 魏蕊蕊, 张纯萍, 等. 鸡源沙门氏菌的血清型、耐药性和耐药机制调查[J]. 农业生物技术学报, 2013, 21(7): 855-862. DOI:10.3969/j.issn.1674-7968.2013.07.012.
[11] 林居纯, 覃春红, 赖婧, 等. 食品动物源沙门氏菌质粒介导喹诺酮类耐药基因的检测与分析[J]. 畜牧兽医学报, 2012, 43(5): 803-809.
[12] 杜雄伟, 李叶, 王晓辉. 沙门氏菌耐药机制的研究进展[J]. 江苏农业科学, 2010(6): 487-490. DOI:10.3969/j.issn.1002-1302.2010.06.194.
[13] Clinical and Laboratory Standards Institute. M100-S20: performance standards for antimicrobial susceptibility testing[S]. Wayne: CLSI, 2010.
[14] 郝宏珊, 杨保伟, 师俊玲, 等. 鸡肉源沙门氏菌对喹诺酮和氟喹诺酮类抗生素耐药状况及相关基因[J]. 微生物学报, 2011, 51(10): 1413-1420. [15] 王晓泉, 陈祥, 吴双, 等. 沙门氏菌gyrA基因的变异对氣瞎诺砸类药物敏感性的影响[J]. 中国人兽共患病学报, 2007, 23(9): 891-894; 898. DOI:10.3969/j.issn.1002-2694.2007.09.011.
[16] 马婧嘉, 施春雷, 李可, 等. 沙门氏菌耐药谱及质粒耐药基因的筛查[J].中国食品学报, 2014, 14(4): 184-190.
[17] KEHRENBERG C, FRIEDERICHS S, de JONG A, et al. Identif i cation of the plasmid-borne quinolone resistance gene qnrS in Salmonella enterica serovar Infantis[J]. Journal of Antimicrobial Chemotherapy, 2006, 58(l): 18-22. DOI:10.1093/jac/dkl213.
[18] WANG Minghua, GUO Qinglan, XU Xiaogang, et al. New plasmidmediated quinolone resistance gene, qnrC, found in a clinical isolate of Proteus mirabilis[J]. Antimicrobial Agents and Chemotherapy, 2009, 53(5): 1892-1897. DOI:10.1128/AAC.01400-08.
[19] 符浩, 夏兴, 陈代杰. 肠杆菌科细菌质粒介导的喹诺酮耐药基因研究进展[J]. 世界临床药物, 2011, 32(11): 680-685.
[20] 魏秀丽, 陈杖榴. 沙门氏菌耐药株gyrA基因和parC基因突变特征分析[J]. 中国兽医杂志, 2006, 42(9): 6-8. DOI:10.3969/ j.issn.0529-6005.2006.09.002.
[21] 刘贵深, 于涛. 食源性沙门氏菌耐药性及质粒介导喹诺酮耐药基因检测[J]. 生物技术通报, 2014, 46(8): 201-207.
[22] 刘芳萍, 赵玉林, 李昌文, 等. 鸡源性沙门氏菌耐药基因检测与耐药相关性分析[J]. 中国预防兽医学报, 2013, 35(8): 627-630. DOI:10.3969/j.issn.1008-0589.2013.08.06.
[23] THONG K L, MODARRESSI S. Antimicrobial resistant genes associated with Salmonella from retail meats and street foods[J]. Food Research International, 2011, 44(9): 2641-2646. DOI:10.1016/ j.foodres.2011.05.013.
[24] 吴浩天, 武运, 尹明远, 等. Salmonella hadar对喹诺酮类药物耐药性及其耐药基因分析[J]. 食品科学, 2016, 37(17): 150-155. DOI:10.7506/spkx1002-6630-201617025.
Quinolone Resistance Characteristics and Related Gene Analysis of Salmonella in Beef and Mutton Retailed in Ürümqi
GE Kun1, WU Yun1,*, YANG Baowei2, WU Haotian1, WANG Wei1, ZHANG Yanan1, TIAN Ge1, MA Wenrui1
(1. College of Food Science and Pharmaceutical Science, Xinjiang Agricultural University, Ürümqi 830052, China; 2. College of Food Science and Engineering, Northwest A&F University, Yangling 712100, China)
Objective: To determine the antibacterial susceptibility to 15 antibiotics and quinolone resistance genes of 30 Salmonella isolates from beef and mutton Ürümqi, Xinjiang for the purpose of better understanding the development and transmission pathways of antibiotic resistance in Salmonella and hence providing basic information for preventing and controlling Salmonella-related diseases. Methods: The drug sensitivity of Salmonella isolates was evaluated by the agar dilution method. In addition, polymerase chain reaction and gene sequencing were used to detect the quinolone resistancedetermining regions mutations and the plasmid-mediated quinolone resistance genes. Results: The drug resistance rates of 30 Salmonella isolates to trimethoprim, chloramphenicol nalidixic acid, tetracycline, sulf i soxazole, streptomycin, trimethoprim/ sulfamethoxazole, amoxicillin/clavulanic, ampicillin were 100%, 86.7%, 66.7%, 60.0%, 50.0%, 33.3%, 26.7%, 6.7% and 6.7%, respectively. All the Salmonella isolates were sensitive to ciprof l oxacin, ceftriaxone, gentamicin, kanamycin, cefoxitin and amikacin. The number of Salmonella carrying gyrA mutations was 14, and the main type of mutation was Ser83Phe; the number of Salmonella having parC mutations was 25, and the main type of mutation was Thr57Ser. Conclusions: The Salmonella isolates from beef and mutton retailed in Ürümqi are resistant to multiple antibiotics. Therefore, we should be seriously concerned about this phenomenon. The antibiotic resistance mutations and the plasmid-mediated quinolone resistance genes may be an important cause of the antimicrobial resistance of Salmonella isolates from beef and mutton in some extend.
Salmonella; antibacterial susceptibility; quinolone; antibiotic resistance gene
10.7506/spkx1002-6630-201704018
TS201.3
A
1002-6630(2017)04-0107-06
2016-05-26
新疆维吾尔自治区“十三五”重大专项(2016A01001-5)
葛琨(1990—),男,硕士研究生,研究方向为食品生物技术。E-mail:gekunwlmq@163.com
*通信作者:武运(1965—),女,教授,硕士,研究方向为食品生物技术。E-mail:wuyunster@sina.com
葛琨, 武运, 杨保伟, 等. 乌鲁木齐牛羊肉源沙门氏菌对喹诺酮类药物的耐药状况及相关基因分析[J]. 食品科学, 2017, 38(4): 107-112. DOI:10.7506/spkx1002-6630-201704018. http://www.spkx.net.cn
GE Kun, WU Yun, YANG Baowei, et al. Quinolone resistance characteristics and related gene analysis of Salmonella in beef and mutton retailed in Ürümqi[J]. Food Science, 2017, 38(4): 107-112. (in Chinese with English abstract)
10.7506/ spkx1002-6630-201704018. http://www.spkx.net.cn
