一种新的氨基酸描述符及其在肽QSAR中的应用
2017-03-09仝建波李康楠吴英纪李玲霄
仝建波,李康楠,吴英纪,占 培,李玲霄
(陕西科技大学 化学与化工学院,陕西 西安 710021)
一种新的氨基酸描述符及其在肽QSAR中的应用
仝建波*,李康楠,吴英纪,占 培,李玲霄
(陕西科技大学 化学与化工学院,陕西 西安 710021)
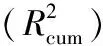
定量构效关系;氨基酸描述符;多元线性回归;肽
多肽类药物在治疗癌症、代谢类疾病、心血管疾病、传染性疾病、内分泌类疾病、血液病和疼痛缓解等众多方向均有显著的疗效和广泛的应用前途。与小分子化学药物相比,多肽类药物更为安全、副作用更小、特异性更强,很少引起严重的免疫反应[1]。随着化学和生命科学的进步,近年来多肽类药物的研发和上市出现了逐步加速的趋势。
肽类药物定量构效关系(Quantitative structure activity relationship,QSAR)是应用数学统计学方法来定量描述肽类药物或其类似物的化学结构与特定的肽药物活性之间的关系[2]。采取实验测量评估肽类药物活性的方法极大地耗费人力和时间[3],因此肽类药物定量构效关系在肽类药物的研发中具有重要意义。在肽类药物定量构效关系中,最为重要的是肽药物结构的表征[4]。Sneath等[5]最先使用几个半定量实验参数对20种天然氨基酸分子结构进行表征,并成功用于后叶催产素类似物活性的预测。Kidera等[6]用因子分析法分析了20种天然氨基酸的188种结构参数,得到10个正交因子,并将其运用到肽类药物活性预测,取得较好结果。其后,Hellberge等[7-11]用主成分分析法分析了20种天然氨基酸的29个物理化学性质,得到3个显著主成分,将其作为氨基酸描述符成功建立了一些肽的定量构效关系模型。此后不断有新的氨基酸描述符提出,包括ISA-ECI[12],MS-WHIM[13],Z-scale[14],SVHEHS[15],SZOTT[16]等均在肽类药物定量关系研究中取得满意成果,为肽药物的研究和开发提供了很大的指导和帮助。
1 方法与原理
1.1 氨基酸描述符的选取
首先通过ChemOffice 2010绘制出20种基本氨基酸分子的初始结构,而后通过Gaussian 9.8在DFT水平上采用Berrny能量梯度法和广义梯度近似法(GGA)的B3LYP/6-31G**理论优化氨基酸分子的空间立体结构及单点计算[17-19]。然后将氨基酸分子的最佳构象分别导入软件dragon 6[20]计算出20种天然氨基酸的41个randic molecular profiles非零描述符、44个eigenvalue based indices非零描述符和47个walk and path counts非零描述符。然后对3类原始变量分别进行主成分分析以剔除原始数据中的噪音信息和无关变量,经主成分分析每个非零描述符的前3,4,3个主成分分别可解释原始变量98.90%,98.33%,95.89%。可见这10个主成分足以表达原始变量所含的信息。用这10个主成分代替原始变量,作为新的氨基酸描述符-SVREW用于肽类药物定量构效的研究。
1.2 模型的建立与检测
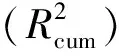
(1)

(2)
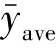
(3)
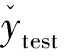
(4)
式中:n为样本总数,yobs为肽活性实验值,ypre为肽活性预测值。
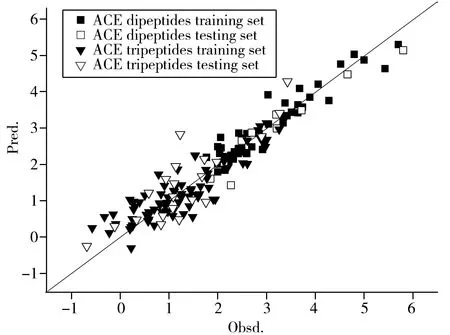
图1 ACE抑制二肽和三肽样本的实验值与预测值的关系图Fig.1 Plot of the predicted activity against the observed activity of ACE inhibitor dipeptides and tripeptides
2 结果与讨论
2.1 ACE抑制肽的QSAR研究
从图1可以发现,所有样本集中分布在过原点45°对角线附近,说明SMR-MLR所建模型的拟合能力与预测能力较强。SMR-MLR所建多元线性回归方程如下:Y=2.785-0.230×υ18+0.095×υ9+0.063×υ15+0.110×υ5-0.208×υ6+0.591×υ12-0.565×υ11+0.777×υ17+0.026×υ19+1.315×υ13+0.858×υ14-0.158×υ16。
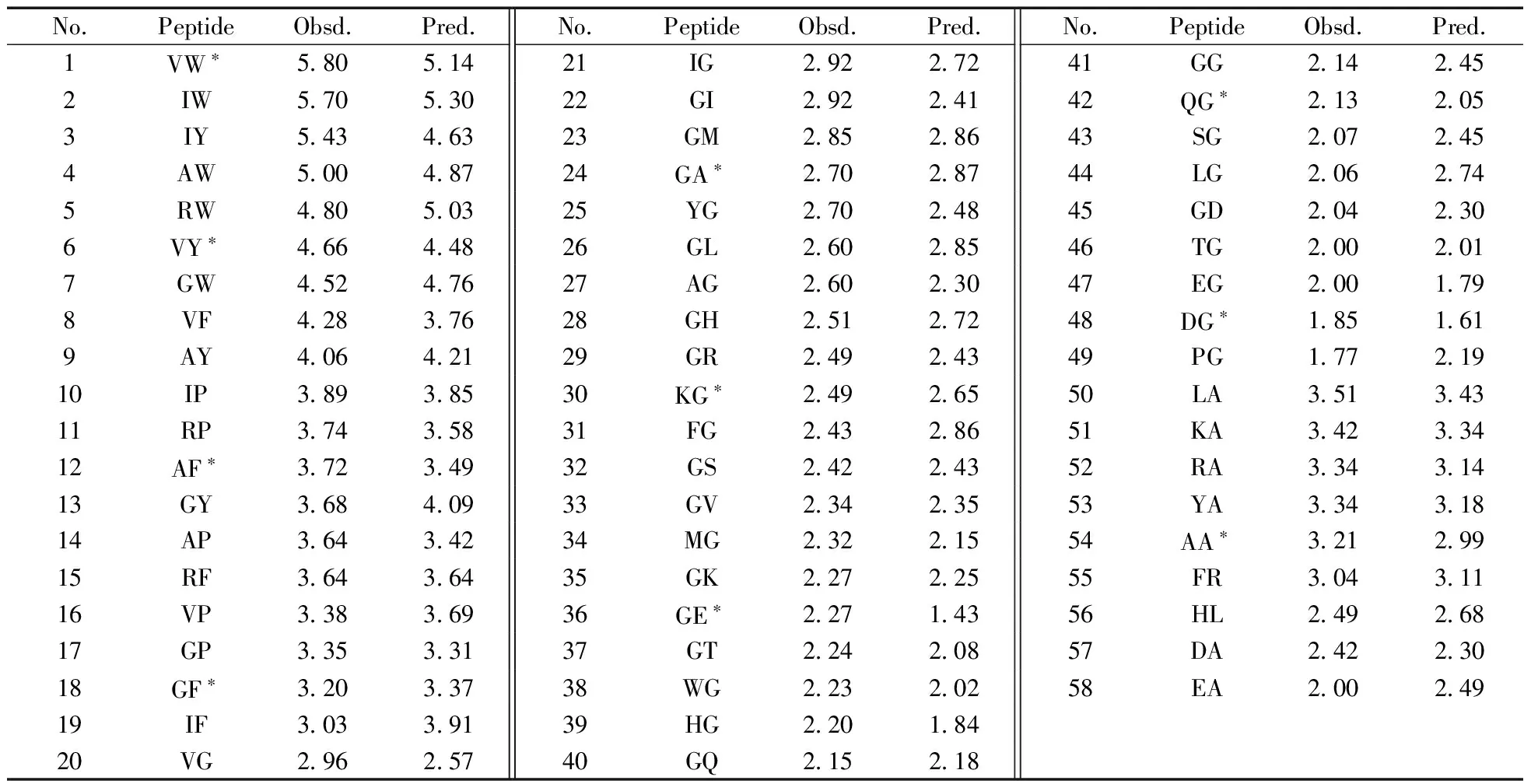
表1 58个ACE抑制二肽序列及其生物活性的实验值与预测值Table 1 Sequences of 58 ACE inhibitor dipeptides with observed activity and predicted activity
*test set

图2 苦味二肽和四肽样本的实验值与预测值的关系图Fig.2 Plot of the predicted activity against the observed activity of BTT dipeptides and tetrapeptides
2.2 苦味肽QSAR研究
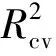
从图2可以发现所有的样本均集中分布在过原点45°对角线附近,说明SMR-MLR所建模型的拟合能力与预测能力较强。SMR-MLR所建的多元线性回归方程如下:Y=2.019-0.017×υ8+0.156×υ15+0.022×υ14-0.233×υ2+0.445×υ17+0.188×υ3+0.041×υ4。
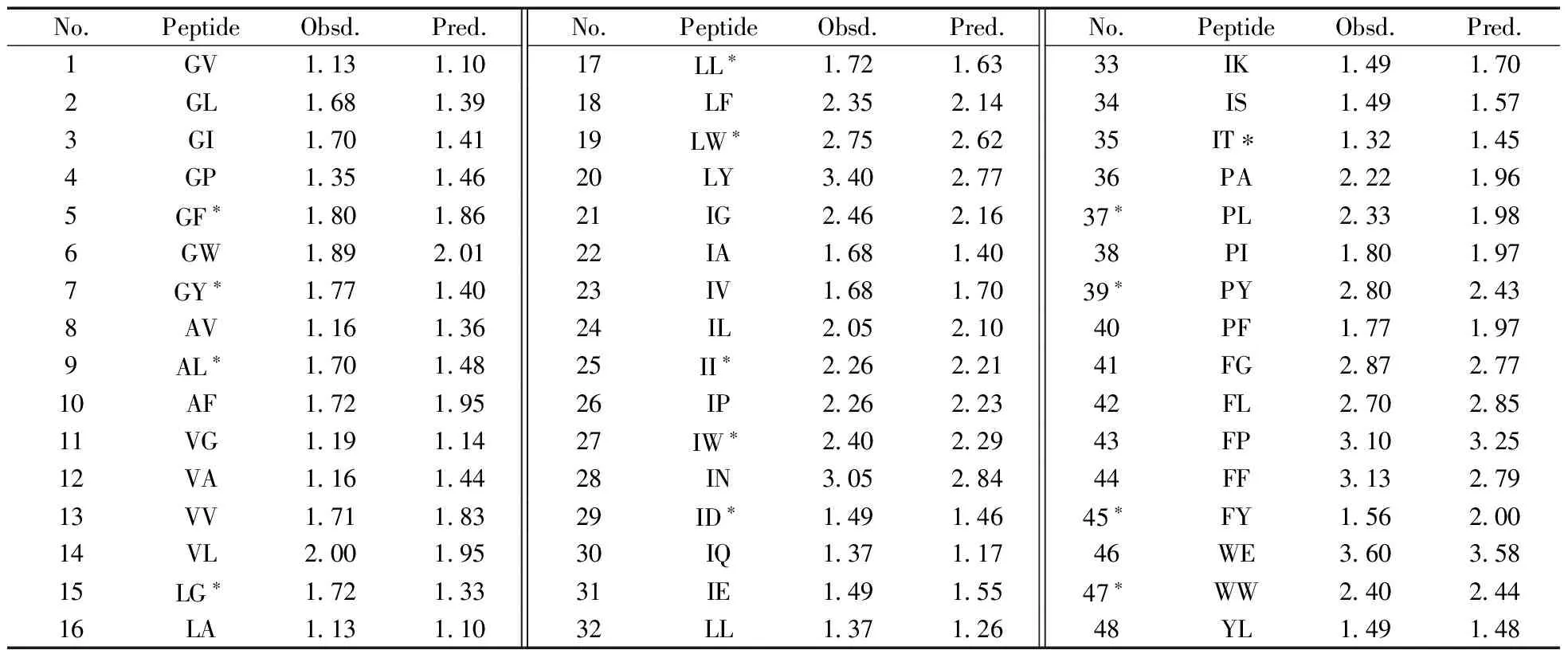
表2 48个苦味活性二肽氨基酸序列及其活性的观察值与模型预测值Table 2 Sequences of 48 BTT dipeptides with observed activity and predicted activity
*test set

表3 23个苦味四肽氨基酸序列及其活性的观察值与模型预测值Table 3 Sequences of 23 BTT tetrapeptides with observed activity and predicted activity
*test set
2.3 后叶催产素的QSAR研究
后叶催产素是由大脑垂体后叶产生的一种激素,具有促进子宫收缩从而催产以及促进泌乳的作用[26]。此外研究证明[27],当人体催产素含量上升时,会随之释放出大量能够缓解压力、延缓衰老的激素,并能促进细胞重生。人体在分娩和哺乳等情况下会大量分泌后叶催产素,它能够产生止痛或镇定的作用。
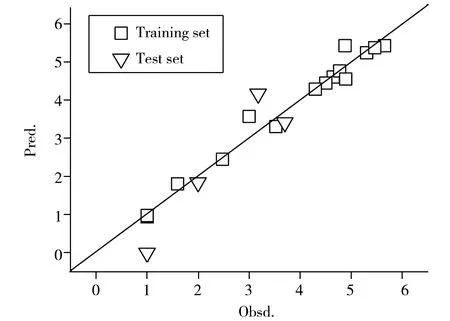
图3 后叶催产素样本实验值和预测值的关系图Fig.3 Plot of calculated versus observed activities for 21 oxytocin analogues
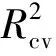
从图3可以发现,所有样本都集中分布在过原点45°对角线附近,说明SMR-MLR所建模型的拟合能力与预测能力较强。SMR-MLR所建多元线性回归方程如下:Y=3.160+3.568×υ16+0.544×υ8-0.435×υ23-0.479×υ18+14.761×υ17。

表4 后叶催产素序列及其生物活性的实验值与预测值Table 4 Sequences of oxytocin with observed and calculated activities
*test set
2.4 HLA-A*0201限制性CTL表位肽的QSAR研究

图4 HLA-A*0201限制性CTL表位肽实验值和预测值的关系图Fig.4 Plot of calculated versus observed activities for HLA-A* 0201 restricted CTL epitopes
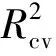
3 结 论
本文提出新的氨基酸描述符-SVREW,此描述符完全是由理论计算获得的参数,具有操作简单、形式统一、数据易获取等特点,将其应用于 ACE抑制肽、苦味肽、后叶催产素类似物、HLA-A*0201限制性CTL表位肽的结构表征,应用逐步回归结合多元线性回归建立QSAR模型。所得模型的拟合能力和预测能力较强,有望在其他肽类药物定量构效关系中得到应用和推广。
[1] Chen G H,Chi J G,Qiu W Z,Wang J N,Sun Y J.ShandongSci.(陈贯虹,迟建国,邱维忠,王加宁,孙元军.山东科学),2008,21(3):42-48.
[2] Zhang Y,Ding Y,Wang Y Q,Wang B C.ChongqingUniv.Technol.:Nat.Sci.(张娅,丁元,王远强,王伯初.重庆理工大学学报:自然科学版),2011,25(5):35-41.
[3] Schultz T W,Cronin M T D,Netzeva T I.Mol.Struct.Theochem.,2003,622(2):23-38.
[4] Tong J B,Chang J,Zhao X,Bai M.Mol.Sci.(仝建波,常佳,赵翔,白敏.分子科学学报),2014,30(4):280-286.
[5] Sneath P H A.J.Theor.Biol.,1966,12(2):157-195.
[6] Kidera A,Konishi Y,Oka M,Ooi T.ProteinJ.,1985,4(1):23-55.
[7] Hellberg S,Sjöström M,Sjostrom M,Wold S.ActaChem.Scandinavica,1986,40(2):135-140.
[8] Hellberg S,Sjöström M,Skagerberg B,Wold S.Med.Chem.1987,30(7):1126-1135.
[9] Hellberg S,Eriksson L,Jonsson J,Lindgren F,Sjöström M.Int.PeptideProteinRes.1991,37(5):414-424.
[10] Wold S,Eriksson L,Hellberg S,Jonsson J,Sjöström M.CanadianJ.Chem.,2011,65(65):1814-1820.
[11] Jonsson J,Eriksson L,Hellberg S,Sjöström M,Wold S.Mol.Inf.,1989,8(3):204-209.
[12] Collantes E R,Rd D W.Med.Chem.,1995,38(14):2705-2713.
[13] And A Z,Gancia E.Chem.Inf.Model.,1999,39(3):525-533.
[14] Tian F,Zhou P,Li Z.J.Mol.Struct.,2007,830(S 1/3):106-115.
[15] Liu J,Peng J Q,Guan X.Anal.Sci.(刘静,彭剑秋,管骁.分析科学学报),2012,28(1):16-22.
[16] Liang G Z ,Zhou P,Zhou Y.ActaChim.Sin.,2006,64(5):393-396.
[17] Kohn W,Sham L J.Phys.Rev.,1965,140(4A):1133-1138.
[18] Hohenberg P,Kohn W.Phys.Rev.,1964,136(3):864-871.
[19] Maczyński M,Zimecki M,Taraszkiewicz M.ActaPoloniaePharm.,2008,65(5):543-549.
[20] Todeschini R,Consonni V,Mauri A,Pavan M.Anal.Chim.Acta,2004,515(1):199-208.
[21] Tropsha A.Gramatica P.QsarComb.Sci.,2003,22(1):69-77.
[22] Tong J B,Chang J,Liu S L,Bai M.Serb.Chem.Soc.,2015,80(3):343-353.
[23] Tong J B,Chang J,Liu S L,Bai M.Serb.Chem.Soc.,2014,65(5):550-555.
[24] Chen J W,Liu S S,Cai G X.FoodSci.(陈季旺,刘珊珊,蔡广霞.食品科学),2013,34(9):19-23.
[25] Kim H O,Lichan E C.Agric.FoodChem.,2006,54(26):10102-10111.
[26] Bull P M,Douglas A J,Russell J A.Neuroendocrinol.,1994,6(3):267-274.
[27] Shang S Y,Su Y J.Psychology:TechniquesandApplications(尚思源,苏彦捷.心理技术与应用),2016,(4):224-235.
[28] Dnyaneshwar W,Preeti C,Kalpana J,Bhushan P.Biol.Pharm.Bull.,2006,29(11):2313-2316.
[29] Chen T,Li M P,Zhang S W.Comput.Appl.Chem.(陈婷,李美萍,张生万.计算机与应用化学),2011,28(1):78-82.
[30] Lin Z H,Hu Y,Wu Y Z.ActaChim.Sin.(林治华,胡勇,吴玉章.化学学报),2004,62(18):1835-1840.
A New Descriptor for Amino Acids and Its Applications in Peptide QSAR
TONG Jian-bo*,LI Kang-nan,WU Ying-ji,ZHAN Pei,LI Ling-xiao
(College of Chemistry & Chemical Engineering,Shaanxi University of Science & Technology,Xi'an 710021,China)
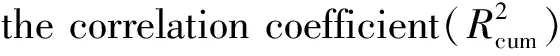
quantitative structure-activity relationship;amino acid descriptor;multiple linear regression;peptides
2016-07-05;
2016-08-30
国家自然科学基金(21475081);陕西省自然科学基础研究计划(2015JM2057);陕西科技大学研究生创新基金
10.3969/j.issn.1004-4957.2017.02.012
O641.121;O629.76
A
1004-4957(2017)02-0224-07
*通讯作者:仝建波,博士,教授,研究方向:计算机辅助药物设计与相关化学信息及计量学、能源化工、食品化学的研究,E-mail:jianbotong@aliyun.com
