DNA条形码在主要滇产石斛鉴别中的应用
2017-03-07张爱丽高亚芳黎太米谭文红钱
张爱丽++高亚芳++黎太米++谭文红 钱子刚

【摘要】目的:选择云南境内应用面较广、市场流通量较大的14个石斛品种,建立其 DNA条形码鉴定方法。方法:结合Genbank数据库序列,选择ITS、psbA-trnH、matK、rbcL等4条常用序列对石斛DNA样品进行扩增和测序,利用MEGA50计算种间种内 K2P遗传距离并构建NJ树,同时采用Taxon DNA软件计算Barcoding gap(条形码种内種间遗传差异)。结果:ITS序列的K2P种内遗传距离小于种间遗传距离,NJ树可以很好地将不同品种的石斛分开,且有较明显的Barcoding gap。结论:ITS序列可以作为鉴别这14种滇产主要石斛的优选序列。
【关键词】滇产石斛;DNA条形码;分子鉴别
【中图分类号】R197【文献标志码】 A【文章编号】1007-8517(2017)02-0011-06
Application of DNA Barcoding in species identification of Dendrobium widely used in Yunnan
ZHANG AiliGAO YafangLI TaimiTAN Wenhong*QING Zigang*
Yunnan University of Chinese Traditional Medicine,Kunming 650500,China
Abstract:Objective To identify 14 species of Dendrobium which widely used in Yunnan province by DNA barcoding. Methods Three chloroplast sequences including psbA-trnH, matK, rbcL and an ribosomal DNA ITS sequence of the 14 species were comparatively analyzed. K2P distances were calculated and NJ Tree was performed applying MEGA 50Meanwhile, Barcoding gap were calculated by Taxon DNA. Results The interspecific K2P distance of ITS were higher than the intraspecific one. And NJ tree could also divide species into different clades. The barcoding-gap were obvious between intra- and inter-species. Conclusions ITS sequence can be the optimal barcode to identify the 14 species of Dendrobium.
Keywords:Genus of Dendrobium in Yunnan; DNA Barcoding; Molecular Identification
石斛为我国著名中药材,具有生津益胃、滋阴清热、润肺止咳等功效[1]。是历代本草和历版《中国药典》收载的品种。《中国药典》(2015年版)收载的石斛药材为兰科植物金钗石斛(Dendrobium nobile)、鼓槌石斛(Dendrobium chrysotoxum)、流苏石斛(Dendrobium fimbriatum)的栽培品及同属植物的近似种[2]。
石斛作为云南特有药材之一,产量占全国的60%。在云南境内,除药典品种铁皮石斛以外,市场流通的石斛品种多达20几种[3]。但这些品种没有专属的质量标准,使得这类石斛药材质量得不到控制。因此,控制其质量、保证临床用药的安全和疗效,必先对石斛药材的来源进行严格的鉴定。但是,石斛属 (Dendrobium SW)作为兰科 (Orchidaceae) 最大的属之一,其种类繁多,且许多种类形态非常相似,从外观形态上对石斛属植物进行鉴别有一定的困难[4]。
DNA条形码鉴定是现阶段一个精准、稳定并且易于操作的鉴定方法。本实验收集了云南省境内应用面较广、市场流通量较大的14个石斛品种,选择ITS、psbA-trnH、matK、rbcL等4条常用条形码片段对其进行了序列的分析比较,以期为鉴别这些主要滇产石斛品种提供分子依据,使得其基源鉴定更为准确可靠,为今后石斛的质量控制研究奠定基础。
1仪器和材料
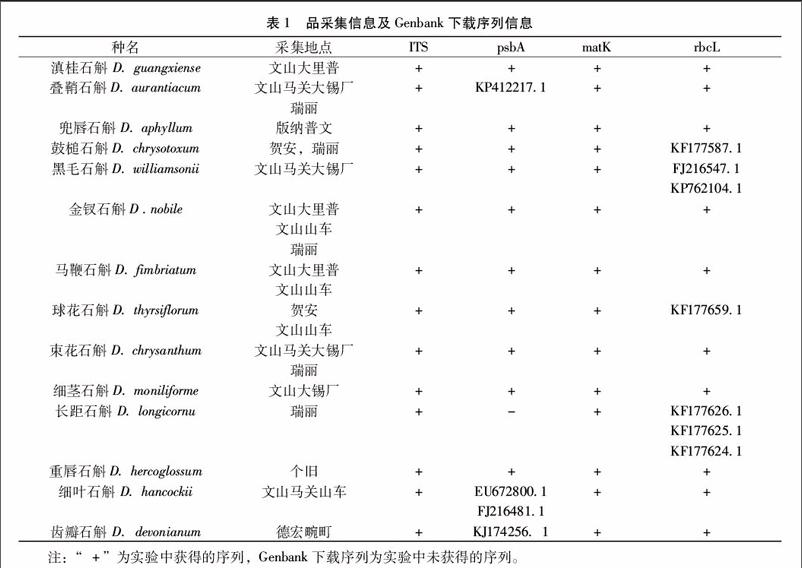
11样品来源实验通过走访调查,文献查阅,收集了云南省境内应用面广、市场流通大的14个石斛品种,每个品种保证至少3个或以上重复样品,实验中不能获得的序列信息,均从Genbank下载。具体信息见表1。
12仪器SW-CJ-1D洁净工作台(江苏苏洁净化设备厂);高速冷冻离心机(HITACHI);电泳仪(Bio-Rad);微型电泳槽(Bio-Rad);凝胶成像系统(Bio- Rad);PCR反应扩增仪(Bio-Rad);移液器(范围100~1000μL,20~200μL,05~10μL(Eppordorf)。
[JP3]13试剂4S Red Plus核酸染色剂[BF](上海生工,生产批号:A606695);琼脂糖(Biowest,生产批号:111860);Mix Tag酶(Takara公司,生产批号:A3201A)。[BFQ][JP]
2方法
21DNA提取采集的新鲜石斛叶片用变色硅胶干燥, 采用植物基因组DNA快速提取试剂盒 (Bioteke, 北京),提取石斛样品的总DNA。
22PCR扩增PCR 体系总体积为 25 μL, 其中包含: 2×Taq PCR Mix 125 μL、DNA 模板 2 μL、ddH2O 85 μL、引物各 1 μL。PCR 反应条件及引物序列见表2。扩增结束后,用1% 琼脂糖凝胶电泳检测,凝胶成像分析系统分析。
23纯化测序PCR产物送上海生工进行纯化后,测序。测序工作由上海生物的ABI-PRISM3730DNA自动测序仪完成。每个产物样品采用双向测序。
24数据处理测序峰图采用Contigexpress序列拼接软件校对拼接,去除低质量序列及引物区,利用MEGA 50对拼接后序列进行手工排序矫正后,比对分析;同时从 GenBank 数据库中搜集并下载未获得的ITS、 psbA-trnH、rbcL、matK序基因序列;共计221条(实验获得和GenBank下载);运用 MEGA 50 软件,以密花石豆兰 (Bulbophyllum odoratissimum) 为外类群[5],并基于K2P 距离模型构建NJ系统发育树[6],计算种间种内的K2P遗传距离;采用Taxon DNA软件做出各候选序列的barcoding gap图。
3结果与分析
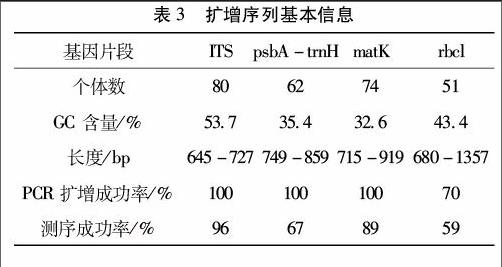
31序列信息、PCR 扩增效率和测序成功率PCR 扩增效率和测序成功率是评价DNA 条形码的一个重要指标,本实验对4个候选序列的 PCR扩增效率(出现明显 PCR 条带即判定成功)和测序成功率(测序重复2次后获得高质量序列即为成功)进行了统计。其中rbcL序列由于长度较长,其扩增和测序成功率都非常底,不到70%和60%。psbA-trnH序列的扩增成功率为100%,但其测序成功率较低,仅为67%。matK序列的扩增成功率为100%,其测序成功率为89%。ITS序列无论扩增还是测序其成功率都最高,都接近100%。另外,各序列 GC 含量差异较大,ITS序列最高,平均含量达到 537%,matK序列GC 含量较低,平均仅为326%。详细信息见表3。
32不同DNA条形码序列种类种间差距种内种间差异也是评价条形码序列的重要指标。合适的条形码序列其种间变异要大于种内变异,以便准确鉴别同属不同种的物种[7]。研究基于K2P模型,计算了14个石斛品种的种间种内遗传距离 (表4)。结果显示,4条序列中,ITS序列的种间遗传距离为001~020,种内遗传距离为000~008,除叠鞘石斛(Daurantiacum)以外,其余品种的种间遗传距离都大于种内遗传距离。而psbA-trnH、matK及rbcL序列的种间遗传距离基本与种内遗传距离相一致。见表4。
33不同序列 barcoding gap检验 Barcoding gap是指序列种内与种间遗传变异由于显著差异而形成的间隔区[8-9],如图1为4条序列的barcoding gap 图。matK、psbA-trnH及rbcL序列均无明显的间隔区。相对其他序列,ITS序列有较为明显的间隔区,种内与种间变异明显分为两大部分,且其分布较高的遗传变异也可明显区分,因此在barcoding gap 分析方面,ITS序列为鉴别不同石斛较理想条形码序列。
34NJ系统树的构建与分析NJ 树是基于 K2P 遗传距离建立,通过分析不同物种间的亲缘关系远近以鉴别物种的系统发育树,该方法可直观展示不同物种的系统关系。如图(2-5)所示,以密花石豆兰 (Bulbophyllum odoratissimum) 为外类群[5],基于K2P 距离模型构建NJ系统发育树[6],结果显示ITS序列可以将不同物种分到不同的分支上,将不同品种的石斛区分开来,而matK、 psbA-trnH及rbcL序列均不能将不同品种的石斛完全具为1个分支。
4讨论
DNA 条形码是指利用有足够变异、易扩增且相对较短的标准DNA片段,在种内特异性与种间多样性中建立的一种新的生物身份识别系统,从而实现对物种进行快速、准确地识别和鉴定[10]。理想的条形码序列应具有足够的种间差异(种间差异应小于种内差异),易于扩增,且引物通用性好,能够通过双向测序得到高质量的序列[11]。本实验选取了当前在植物学界推荐的4个热点候选序列(ITS、matK、rbcL、psbA-trnH序列)对主要的14个滇產石斛品种进行鉴别[12],四个片段的鉴别能力各有差别。
41matK序列对不同品种石斛的鉴定能力评价matK及片段有较高的的扩增和测序成功率,分别为100%和89%。计算K2P遗传距离表明,其种内和种间差异保守。barcoding gap分析显示,并没有明显的barcoding gap。NJ树也不能将不同品种的石斛很好的聚为一支(图3)。同时,其作为条形码的最主要争议是引物通用性差[13-14],在不同物种间的通用性较小。鉴于此,本实验中不建议选用该片段作为鉴定片段。
42psbA-trnH序列对不同品种石斛的鉴定能力评价psbA-trnH片段的PCR扩增和测序成功率,分别为100%和67%。根据barcoding gap分析显示,其分布较高的遗传变异与分布较低的遗传变异可以明显区分。但K2P遗传距离表明,其种间遗传距离没有明显大于种内遗传距离,不能将不同种间的差异表现出来。同时,NJ树也不能将不同品种的石斛很好的聚为一支。鉴于此,本实施不推荐psbA-trnH序列作为区分这14个石斛品种的理想条形码片段。
43rbcL序列对不同品种石斛的鉴定能力评价与其它三个片段相比较,rbcL的序列长度最长,在本实验中,其最长片段长度接近1400bp。因此,其扩增和测序的成功率都不高,仅为70%和59%。计算其K2P遗传距离表明,其种间种内遗传距离无明显区别(000~001)。基于K2P遗传距离构建的NJ树也不能将不同品种的石斛很好的聚为一支。文献研究报道[15],rbcL的变异主要存在于种以上的水平,其鉴别主要应用在科属及以上水平。鉴于此,rbcL也并不是鉴别该14个品种石斛的最佳条形码。
44ITS序列对不同品种石斛的鉴定能力评价对于ITS片段,由于具有序列长度较短,易于扩增,对样本要求较低。在本实验中,其扩增和测序成功率均接近100%。根据实验数据分析,除了叠鞘石斛(D aurantiacum)的K2P种内遗传距离为006,大于种间遗传距离,其余各品种的种间遗传距离均大于种内遗传距离。使用Mega 50软件进行1000次自展计算,对14个石斛品种进行NJ树的构建,由图2可以看出,以密花石豆兰 (Bulbophyllum odoratissimum)为外类群[6],基于ITS序列构建的NJ系统聚类树,不同的石斛品种基本上都聚在一起。同时barcoding gap分析显示,其种内与种间变异区分为两大部分,有明显的barcoding gap,其分布较高的遗传变异也可以明显区分。综合以上分析,ITS序列可以作为鉴别不同石斛物种的优选序列。
综上,本实验筛选了4个热点DNA条形码片段,用以区分云南市场上流通量较大的14个石斛品种。实验结果表明,ITS序列是鉴别这14个石斛品种较为理想的DNA条形码片段。ITS序列作为国际上承认的通用核心条形码,在植物类的鉴别中有着广泛的应用前景。
彭禄[16]等通过ITS序列鉴定了独活的17个种,得到四带芹类可作为独活的替补品的结论。刘静[17]等研究也发现,ITS序列可将17种药用石斛完全鉴别开来。
ITS条形码在中药鉴定领域的广泛应用将实现对中药原植物及其药材和饮片的准确快速鉴。
参考文献
[1]郁美娟,孟庆华,黄德音,等. 石斛属植物有效成分及药理作用研究 [J]. 中成药,2003,25(11):918-921.
[2]国家药典委员会.中华人民共和国药典(第一部)[S]. 北京:中国医药科技出版社,2015:93-94.
[3]云南省药材公司.云南中药资源名录 [M]. 北京:科学出版社,1993:669-672.
[4]Adams P.B. Systematics of Dendrobiinae (Orchidaceae), with special reference to Australian taxa [J]. Bot J Linn Soc, 2011, 166: 105-126.
[5]Xiang X. G., Schuiteman A., Li D.Z., et al. Molecular systematic of Dendrobium (Orchidaceae, Dendrobieae) from mainland Asia based on plastid and nuclear sequences [J]. Mol Phylogenet Evol, 2013, 69: 950-960.
[6]Chen S. L., Yao H., Han J. P., et al. Validation of the ITS2 regionas a novel DNA barcode for identifying medicinal plant species [J]. PLoS One, 2010, 5: e8613.
[7]郑司浩,李亚康,任伟光,等.基于DNA条形码的百合属分子鉴定[J].药学学报,2014,49(12):1730-1738.
[8]Meyer, C. P., Paulay, G. DNA barcoding: error rates based on comprehensive sampling [J]. PLoS Biol, 2005, 3: e422.
[9]Lahaye, R., Bank, M., Bogarin, D., et al. DNA barcoding the floras of biodiversity hotspots [J]. Proc Natl Acad Sci USA, 2008, 105: 2923-2928.[ZK)]
[10]Yuan, Y. W. Olmstead, R. G. Evolution and phylogenetic utility of the PHOT gene duplicates in Verbena complex (Verbenaceae): dramtic intron size variation and footprint of abcestral recombination [J]. American jouranal of bantany, 2008, 95(9), 1166-1176.
[11]陈士林,宋经元,姚辉. 药用植物DNA 条形码鉴定策略及關键技术分析 [J]. 中国天然药物,2009,7 (5):322-327.
[12]陈士林,姚辉,宋经元,等. 基于DNA barcoding (条形码) 技术的中药材鉴定 [J]. 世界科学技术-中医药现代化,2007,9(3):7-12.
[13]Chase, M. W., Cowan, R. S., Hollingsworth, P.M., et al. Hollingsworth ML. A proposal for a standardized protocol to barcode all land plants [J]. Taxon. 2007, 56: 295-299.
[14]Hollingsworth, P. M. DNA barcoding plants in biodiversity hot spots: Progress and outstanding questions [J].Heredity, 2008, 101: 1-2.
[15]Newmaster, S. G., Fazekas, A. J., Ragupathy, S. DNA barcoding in land plants: evaluation of rbcL in a multigenetiered approach [J]. Can J Bot,2008, 84: 335-341.
[16] 彭禄,余言,王志新,等. 基于ITS序列对独活17个种的分子鉴定 [J]. 中草药,2013,43(12):1648-1653.
[17] 刘静,何涛,淳泽. 基于ITS序列的中国药用石斛及其混伪品的分子鉴定 [J]. 中国中药杂志,2009,34(22):2853-2856.
(编辑:梁志庆)
