基于文献计量的基因组编辑技术发展态势分析
2017-02-24王慧媛袁天蔚阮亮亮熊燕
王慧媛,袁天蔚,阮亮亮,熊燕
1.中国科学院上海生命科学信息中心,上海 200031
2.上海医药临床研究中心,上海 200032
3.上海生物样本库工程技术研究中心,上海 200031
基于文献计量的基因组编辑技术发展态势分析
王慧媛1,袁天蔚1,阮亮亮2,3,熊燕1
1.中国科学院上海生命科学信息中心,上海 200031
2.上海医药临床研究中心,上海 200032
3.上海生物样本库工程技术研究中心,上海 200031
近年来,以CRISPR为代表的基因组编辑技术发展迅速,围绕该技术的一系列基础和应用研究成为全球研究热点。利用Web of Science信息检索平台中的SCI-Expanded 来源数据库和CiteSpace分析软件,采用文献统计、共被引分析和关键词共现分析等文献计量方法,对基因组编辑技术研究态势进行分析,为相关研究提供参考。
基因组编辑;CRISPR;文献计量;CiteSpace
自2013年首次报道CRISPRCas9系统成功在哺乳动物基因组编辑中应用以来,以CRISPR为代表的基因组编辑技术受到广泛关注。基因编辑技术是对目标基因序列进行插入、删除或者改变的操作方法。从锌指核糖核酸酶(ZFN)技术、转录激活因子样效应物核酸酶(TALEN)技术到现在的RNA介导的成簇规律间隔短回文重复序列系统(CRISPR)技术,基因组编辑技术成为生命科学研究的一项重要技术。尤其是CRISPR技术,自2012年问世以来,在短短几年内迅速席卷全球各实验室,并展现出广阔的应用前景,被认为是遗传研究领域的革命性技术,已应用于作物育种、疾病模型构建、靶向药物研发等各个领域。采用文献统计、共被引分析和关键词共现分析等文献计量方法,对基因组编辑技术近十年的相关文献进行分析,总结技术发展路径和研究现状,并对未来的研究热点趋势进行展望。
1 基因组编辑技术发展迅速
基因组编辑依赖于位点特异的核酸内切酶在剪切位点通过DNA修复系统触发序列修改。目前,在该领域主要有三种技术,即ZFN技术、TALEN技术和CRISPR技术。
TALEN技术与ZFN技术组成了一大类强有力的基因组编辑工具,由一个可编码的序列特异性DNA结合模块与一个非特异性的DNA切割结构域所组成,通过诱导DNA双链断裂来刺激容易出错的非同源末端连接或在特定基因所在的位置进行的同源定向修复,完成一系列基因编辑操作。CRISPR技术是最新出现的一种基因组编辑工具,与其他基因组编辑工具相比,更易于操作,有更强的可扩展性。
利用Web of Science数据库,检索2006~2016年的基因组编辑技术相关文献(检索时间为2016年11月8日,数据更新时间为2016年11月4日)。2006年以来,共获得基因组编辑技术研究相关文献8176篇。2015年发文量为1964篇,是2006年发文量(62篇)的31.68倍。2016年(数据截至2016年11月8日)为2145篇,已经比2015年增长了9.22%。2006~2015年,基因组编辑技术研究发文量的平均年增长率为47.94%,发文量呈快速增长的趋势(图1)。
2 美国引领基因组编辑技术研究
2006~2016年全球发表的基因组编辑技术论文中,美国以4090篇的发文量位居世界第一,占全球该领域发文总量的50.02%。中国以992篇的发文量排名第二,占全球该领域发文总量的12.13%,发文量排名前五的国家还有德国、英国、日本(图2)。
2006~2015年,基因组编辑技术论文发文量排名前五的国家中,美国的发文量增长速度最快,从2006年的36篇增长到2015年的993篇(2016年数据不全,此处只做趋势观察,不做年度统计),发文量增长了近26.58倍,平均年增长率达44.90%。中国从2007年才开始有基因组编辑技术的相关文献报道,发文量以缓慢的速度平稳增长。2012年开始,中国的发文量快速增长,2014年达到189篇,超越德国,位居全球第二(图3)。
2006~2016年,在基因组编辑技术领域,美国发表的论文的篇均被引频次达到26.93次,h指数高达135,拥有较多的高质量论文。中国尽管在发文量上排在全球第二,但其论文的总被引频次只有15 416次,篇均被引频次仅为15.54次,在发文量排名前十位的国家中排名靠后,而法国、加拿大、荷兰、意大利、西班牙的篇均被引频次都达到20次以上(表1)。

图1 2006~2016年基因组编辑技术相关文献年度分布
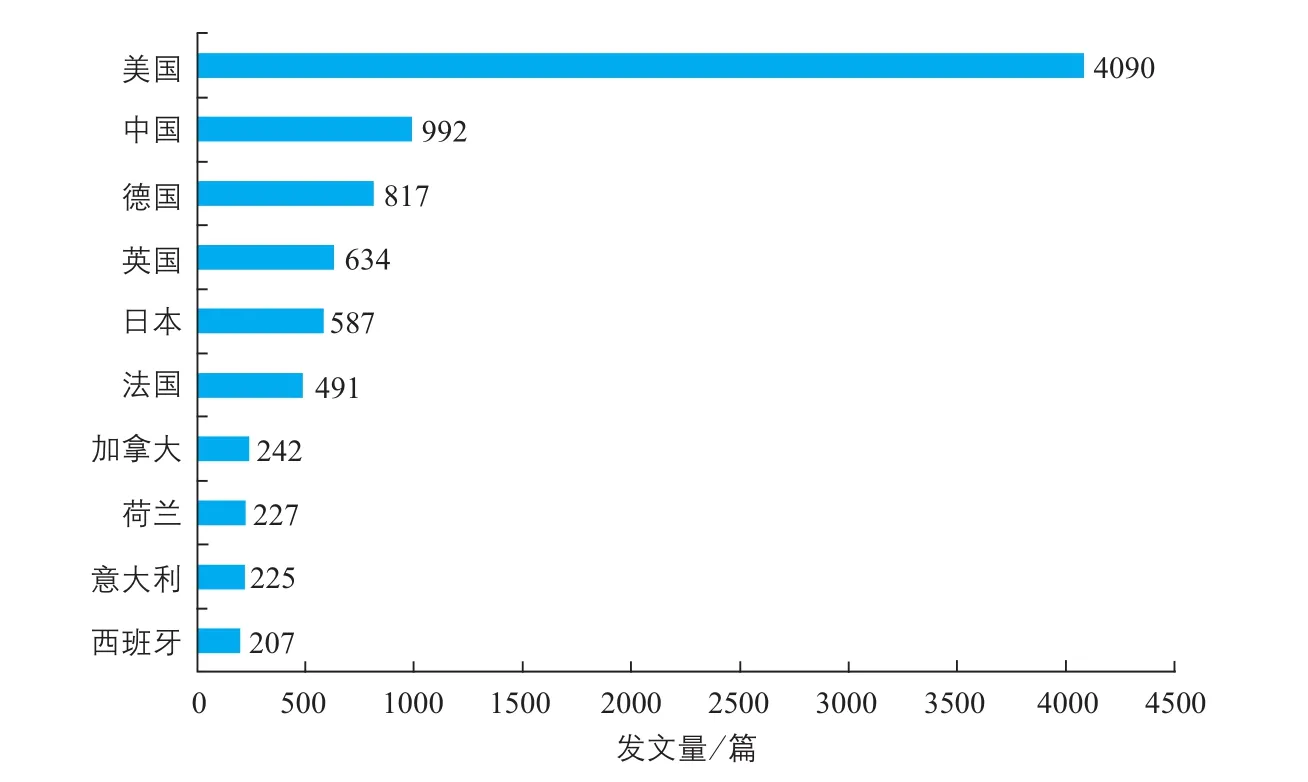
图2 2006~2016年基因组编辑技术论文发文量前十国家分布
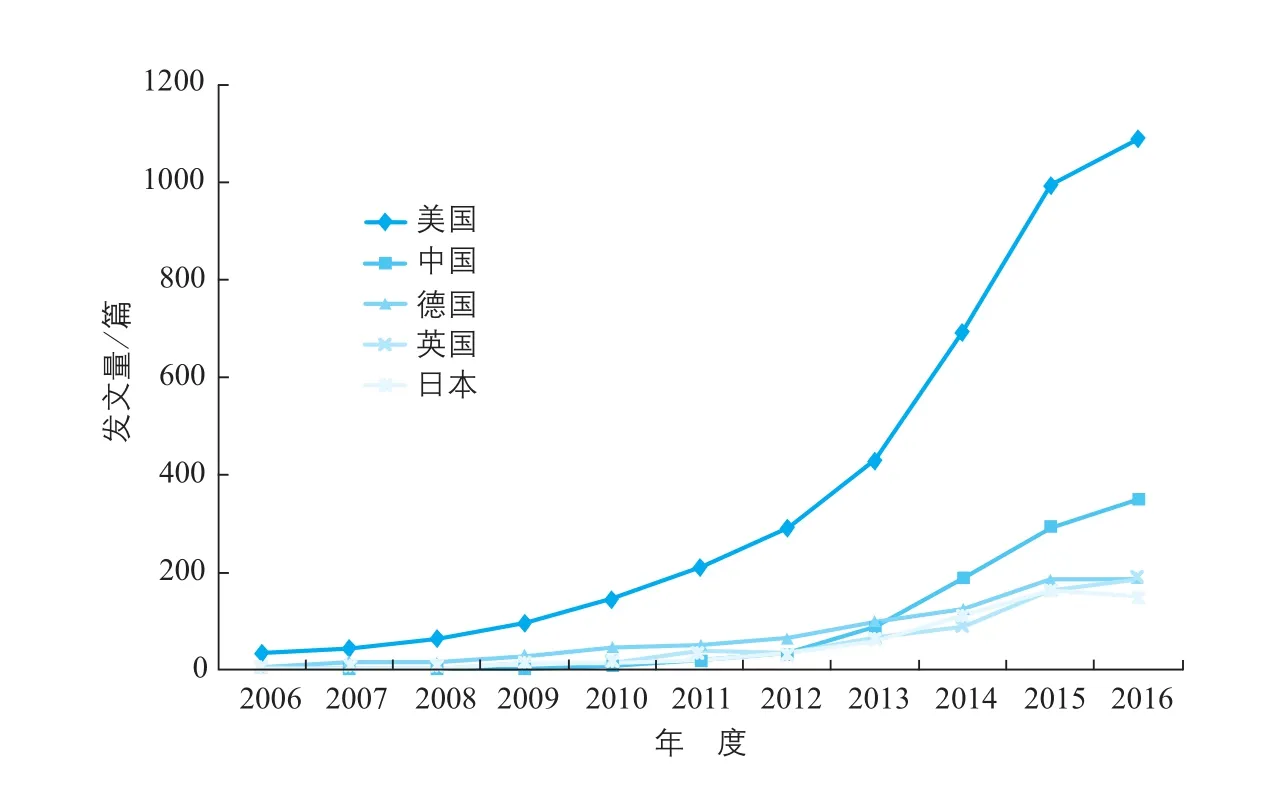
图3 2006~2016年基因组编辑技术论文发文量排名前五国家的发文量年度分布
2006~2016年,全球基因组编辑技术相关论文8176篇,总被引频次为155 810次,篇均被引频次为19.06次。将某一时间段各国论文的篇均被引频次与该时间段全球篇均被引频次相比,得到各国篇均被引频次的相对数值,将这一指标称为标准引文影响指数(normalized citation impact,NCI),国际篇均被引频次作为基线1,NCI可以从一个角度反映一个国家在该领域的研究水平。
发文量排名前十位的国家中,美国、法国、加拿大、荷兰和西班牙在基因组编辑领域的研究高于全球平均水平,德国和意大利与全球平均水平相当,英国略低于全球平均水平,而中国和日本的NCI分别为0.82和0.66,低于全球平均水平(图4)。
3 中国研究机构在基因编辑技术研究领域论文发文量表现突出
2006~2016年全球基因组编辑技术论文发文量排名前十位的研究机构如图5所示。哈佛大学以390篇的发文量排名首位;中国科学院排名第二(260篇),是唯一进入全球前十的中国研究机构。
2006~2016年,基因组编辑技术论文发文量排名前十的机构中,美国麻省理工大学的篇均被引频次最高,达到94.38次,其次是哈佛大学,篇均被引频次为69.80次。中国科学院尽管发文量排名第二位,但篇均被引频次仅为17.41次,与其他机构相比,整体发文质量仍有一定差距。从h指数来看,哈佛大学的h指数较高(74),拥有较多的高质量论文(表2)。

表1 基因组编辑技术论文被引频次前十国家分布

图4 2006~2016 年基因组编辑技术发文量排名前十国家的NCI分布

图5 2006~2016年全球基因组编辑技术论文发文量排名前十机构分布
2006~2016年,基因组编辑技术论文发文量排名前十的中国机构中,中国科学院以260篇的发文量排名第一,占中国基因组编辑技术研究论文总量的26.21%。从h指数来看,中国科学院的h指数为31,遥遥领先于其他国内机构。清华大学的篇均被引频次最高,达到64.10次,其次是北京大学,篇均被引频次为38.01次。这两所高校在基因组编辑技术领域的论文总体质量较高(表3)。
4 基因组编辑技术领域的革命性技术——CRISPR技术
为了研究基因组编辑的技术发展路径,运用信息可视化软件CiteSpace 绘制知识图谱并计量分析数据,对2006~2016年基因组编辑相关文献的共被引和关键词共现情况进行分析(图6),梳理基因组编辑的技术路径,可以看出,CRISPR技术无疑是基因组编辑技术领域的革命性突破。CiteSpace是应用于Java 环境下的可视化软件,基于对特定领域文献集合的计量,通过知识图谱的绘制,能够对CRISPR技术的知识结构、研究演进以及热点和前沿主题进行定量与定性分析。
CRISPR是一类广泛分布于细菌和古菌基因组中的重复结构。研究表明,CRISPR与一系列相关蛋白、前导序列一起,能为原核生物提供对抗噬菌体等外源基因的获得性免疫能力。这种结构的作用机理可能与真核生物的RNA干扰过程类似,最早于1987年在大肠杆菌(Escherichia coli)K12的凋亡蛋白抑制因子(inhibitor of apoptosis protein)基因侧翼序列中被发现 。2005年左右,CRISPR位点附近高度保守的Cas基因(CRISPR-associated genes)引起了研究者关注。Cas基因是一类基因家族,编码具有核酸酶和解旋酶结构域的蛋白质。2005年,Daniel等[2]在原核生物中发现了基因序列中包含45个CRISPR-Cas系统的蛋白家族和多个CRISPRCas亚型的集合,这些集合显示了高度的灵敏性和特异性。2007年3月,Barrangou等[3]在《Science》上发表文章称,CRISPR与Cas构成的CRISPR-Cas系统能够使细菌抵抗噬菌体,从而免受病毒和质粒侵害,在此基础上,研究人员逐步探明了CRISPR-Cas系统详细的免疫机制,为CRISPR-Cas系统发展成为通用的RNA介导的可编程DNA核酸内切酶辅平了道路,这篇文章成为了CRISPRCas系统作为基因组编辑工具的奠基性研究。随后,科学家们陆续对CRISPR-Cas的作用机制进行了深入研究,例如2008年荷兰瓦赫宁根大学的Brouns等[4]发表在《Science》上的文章,报道了原核生物中小CRISPR RNA(small CRISPR RNAs)介导的抗病毒防御,阐述了包含在CRISPR-Cas中的病毒衍生序列如何使用来自宿主的CRISPR相关蛋白Cas介导抵抗感染的抗病毒反应。直到2012年8月,加州大学伯克利分校的Jinek等[5]发表在《Science》杂志上的文章,正式提出利用RNA介导CRISPR-Cas系统可实现基因组编辑,证明CRISPRCas系统可作为基因组编辑工具,CRISPR-Cas基因组编辑技术瞬间吸引了全球研究者的目光。该文章被引用1290次,成为近几年来CRISPR技术大热的起点。

表2 2006~2016年基因组编辑技术论文发文量排名前十机构的被引频次及h指数

表3 2006~2016年基因组编辑技术论文发文量排名前十的中国机构的被引频次及h指数
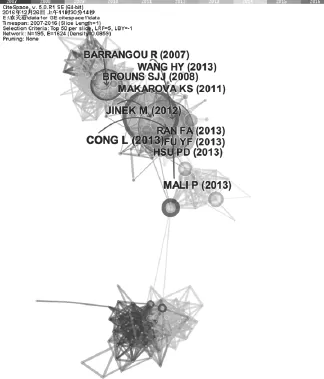
图6 基因组编辑技术文献的共被引知识图谱示意
5 有效性验证和应用研究是基因组编辑技术研究的热点
利用作者从论文中抽取用于表征论文的主题特征的关键词,可以分析出一定研究领域的热点和前沿主题。在CiteSpace软件中,选择网络节点类型为“keywords”(关键词),参数阈值设置为“top 50”(每年出现频次最高的前50 位关键词),绘制关键词共现知识图谱(图7)。分析2006~2016年基因组编辑技术相关的8176篇文献,共出现14 142个关键词。综合来看,2006~2013年,频次和中介中心性较高的关键词分别是zinc f nger nuclease(锌指核酸酶 )、DNA express system(DNA表达系统)、Escherichia coli(大肠杆菌)、DNA binding specif city(DNA结合特异性)、homologous recombination(同源重组)、双链断裂fragment-length-polymorphism(片段多态性)、endonuclease(核酸内切酶)等。这期间的研究热点在于对上一代基因组编辑技术ZFNs、TALANs等的深入研究和对CRISPR技术的分子机制研究。2013年以后的高频词和高中介中心性关键词为prokaryote(原核细胞)、mice( 小 鼠)、mammalian cell(哺乳细胞)、CRISPR/Cas9、pluripotent stem cell(多能干细胞)等,研究热点集中在以CRISPR技术为代表的基因组编辑技术在不同系统中的有效性验证及应用。
2013年以来,基因组编辑技术领域不断取得突破性进展,大部分都是围绕CRISPR技术展开。科学家们纷纷在细菌、斑马鱼、小鼠、小麦、水稻、哺乳动物甚至人类等不同生物系统上验证CRISPR的有效性,例如,Hsu等[6]利用产脓链球菌(Streptococcus pyogenes)中的Cas酶(SpCas)和RNA,在小鼠和人类细胞的DNA中进行了插入、修改、敲除基因片段,首次证明了Cas9核酸酶能用于哺乳动物细胞基因组的编辑;Cong等[7]将多个引导序列编码到单个CRISPR阵列中,使得能够同时编辑小鼠基因组内的多个位点,展示CRISPR技术在小鼠体内进行多位点编辑的有效性;Jiang等[8]利用CRISPR-Cas系统进行RNA指导的细菌基因组编辑,利用Cas9核酸内切酶结合双股RNA在肺炎链球菌和大肠埃希氏菌基因组中实现精确的基因突变;Hwang等[9]发现细菌II型CRISPR-Cas能够在斑马鱼胚胎中诱导产生靶向遗传修饰,其效果与ZFN和TALEN的效果相类似;Ding等[10]利用CRISPR技术进行人多能干细胞基因组编辑,并指出CRISPR较ZFN和TALEN具有极大的优势,CRISPR更易于操作,也具有更强的扩展性;Shan等[11]利用CRISPR-Cas系统定点突变了水稻和小麦两个作物的OsPDS和TaMLO等5个基因,首次证实CRISPR-Cas系统能够用于植物的基因组编辑;Niu等[12]利用CRISPR技术实现猴的基因编辑,提示该技术进行人基因组编辑的可能性。

图7 基因组编辑技术的关键词共现知识图谱示意
同时,以CRISPR为代表的基因组编辑技术在疾病治疗、动植物品种改造等应用方面的研究也取得较大进展。研究人员已经利用CRISPR技术在疾病治疗领域取得诸多突破,包括构建衰老模型[13]、改造艾滋病毒[14]等,尤其是成功编辑T细胞[15],为与自身免疫相关的1型糖尿病、艾滋病、癌症等多发性疾病提供了新的治疗途径。在癌症治疗领域,研究人员利用CRISPR技术鉴别和筛选癌症相关基因[16]、精准控制基因表达[17]、构建癌症类器官[18]等。此外,CRISPR也可用于设计干细胞,开发疾病研究和药物测试模型[19],并有望应用到诱导多能干细胞中[20],实现个性化的干细胞治疗,造福多种遗传学疾病的患者。在动植物分子育种方面,在利用CRISPR-Cas系统定点突变了水稻和小麦基因的基础上,Ji等[21]利用CRISPR-Cas特异识别病毒和外源DNA的特性,将CRISPR切割系统引入植物,在植物中建立了DNA病毒防御体系;CRISPR技术也被广泛用于山羊、猪、狗等动物品种的改良上。除了应用于疾病治疗和物种改良,CRISPR在其他应用领域也发挥着重要的作用,如销除“转基因”生物中特定序列[22]、光控CRISPR-Cas9系统[23]、制备新型gRNA文库[24]等。
在多种系统中成功应用CRISPR技术之后,科学家们更加关注CRISPR技术应用于人类基因组编辑。2015年,我国中山大学生命科学学院黄军就在《Protein & Cell》杂志发表文章称,其团队成功修改了人类胚胎的DNA,为治疗地中海贫血症提供了可能[25]。这是CRISPR技术首次应用于人类胚胎编辑,尽管引发了基因组编辑技术伦理和监管问题的巨大争议,但仍是该基因组编辑技术应用研究的重要突破,黄军就也因此入选了《Nature》2015年度十大人物。
6 总结与展望
随着基因组编辑技术飞速发展,尤其是CRISPR技术的不断应用与探索,相关研究发文量逐年迅速增长。美国引领了基因组编辑技术的研究,其发文量占全球发文量的近一半,且发文质量整体水平较高。中国后来居上,在基因组编辑领域的研究实力强劲,尤其是中国科学院,在发文量上仅次于哈佛大学,其发文整体实力与高质量论文量在国内也处于领先地位。基因组编辑技术发展过程中,CRISPR技术无疑是该领域的革命性技术。
近年来,基因组编辑的研究热点都围绕着CRISPR技术展开,从作用机制到不同系统的有效性验证,再到技术应用,基因组编辑的应用潜力不断被挖掘,在药物开发、疾病治疗、动植物改造等领域发挥着越来越重要的作用。利用基因组编辑技术寻找、确定和制备药物筛选靶(分子药靶)已成为新药研发的新思路;基因组编辑技术越来越普遍地用于作物育种。此外,研究人员也在不断探索如何减少基因组编辑技术的脱靶效应,提高系统效率和特异性,寻找更高效的编辑系统或工具等,对脱靶效应的研究使得该技术更为精准,“精准基因组编辑”正逐步实现。而具有多重功能或更大识别序列的新系统也在不断被发现。此外,随着基因组编辑技术成功应用于人类胚胎,该技术的快速发展所带来的伦理和监管问题也得到广泛关注和探讨。
[1] ISHINO Y,SHINAGAWA H,MAKINO K,et al.Nucleotide sequence of the iap gene,responsible for alkaline phosphatase isozyme conversion in Escherichia coli,and identif cation of the gene product[J]. Journal of Bacteriology,1987,169(12):5429-5433.
[2] DANIEL H H,JEREMY D S,EMMANUEL F M,et al. A guild of 45 CRISPR-associated(Cas)protein families and multiple CRISPR/Cas subtypes exist in prokaryotic genomes[J]. PLoS Computational Biology,2005,1(6):0474-0483.
[3] BARRANGOU R,FREMAUX C,DEVEAU H,et al. CRISPR provides acquired resistance against viruses in prokaryotes[J]. Science,2007,315:1709-1712.
[4] BROUNS S,JORE M,LUNDGREN M,et al.Small CRISPR RNAs guide antiviral defense in prokaryotes[J]. Science,2008,321(5891):960-964.
[5] JINEK M,CHYLINSKI K,FONFARA L,et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science,2012,337(6096):816-821.
[6] HSU P D,SCOTT D A,WEINSTEIN J A,et al. DNA targeting specif city of RNA-guided Cas9 nucleases[J]. Nature Biotechnology,2013,31(9):827-832.
[7] CONG L,RAN F A,COX D,et al. Multiplex genome engineering using CRISPR/Cas systems[J]. Science,2013,339(6121):819-823.
[8] JIANG W,BIKARD D,COX D,et al. RNA-guided editing of bacterial genomes using CRISPR-Cas systems[J]. Nature Biotechnology,2013,31(3):233-239.
[9] HWANG W Y,FU Y,REYON D,et al. Eff cient genome editing in zebraf sh using a CRISPR-Cas system[J]. Nature Biotechnology,2013,31(3):227-229.
[10] DING Q,REGAN S N,XIA Y,et al. Enhanced eff ciency of human pluripotent stem cell genome editing through replacing TALENs with CRISPRs[J]. Cell Stem Cell,2013,12(4):393-394.
[11] SHAN Q W,WANG Y P,LI J,et al. Targeted genome modif cation of crop plants using a CRISPR-Cas system[J]. Nature Biotechnology,2013,31(8):686-688.
[12] NIU Y,SHEN B,CUI Y,et al. Generation of gene-modif ed cynomolgus monkey via Cas9/RNA-mediated gene targeting in one-cell embryos[J]. Cell,2014,156(4):836-843.
[13] ITAMAR H,BERENICE A B,BEN M,et al. A platform for rapid exploration of aging and diseases in a naturally short-lived vertebrate[J]. Cell,2015,160(5):1013-1026.
[14] LIAO H K,GU Y,DIAZ A,et al. Use of the CRISPR/Cas9 system as an intracellular defense against HIV-1 infection in human cells[J]. Nature Communication,2015,6.
[15] KATHRIN S,STEVEN L,ERIC B,et al. Generation of knock-in primary human T cells using Cas9 ribonucleoproteins[J]. PNAS,2015,112(33):10437-10442.
[16] TAGLIABRACCI V S,WILEY S E,GUO X,et al. A single kinase generates the majority of the secreted phosphoproteome[J]. Cell,2015,161(7):1619-1632.
[17] HART T,CHANDRASHEKHAR M,AREGGER M,et al. High-Resolution CRISPR screens reveal fitness genes and genotype-specific cancer liabilities[J]. Cell,2015,163(6):1515-1526.
[18] DROST J,VAN JAARSVELD R H,PONSIOEN B,et al. Sequential cancer mutations in cultured human intestinal stem cells[J]. Nature,2015,521(7550):43-47.
[19] SMITH C,ABALDE-ATRISTAIN L,HE C,et al. Eff cient and allele-specif c genome editing of disease loci in human iPSCs[J]. Molecular Therapy,2015,23(3):570-577.
[20] HOWDEN S E,MAUFORT J P,DUFFIN B M,et al. Simultaneous reprogramming and gene correction of patient fibroblast[J]. Stem Cell Reports,2015,5(6):1109-1118.
[21] JI X,ZHANG H W,ZHANG Y,et al. Establishing a CRISPR-Cas-like immune system conferring DNA virus resistance in plants[J]. Nature plants,2015,1(10).
[22] CALIANDO B J,VOIGT C A. Targeted DNA degradation using a CRISPR device stably carried in the host genome[J]. Nature Communication,2015,6.
[23] NIHONGAKI Y,KAWANO F,NAKAJIMA T,et al. Photoactivatable CRISPR-Cas9 for optogenetic genome editing[J]. Nataure Biotechnology,2015,33(7):755-760.
[24] VIDIGAL J A,VENTURA A. Rapid and eff cient one-step generation of paired gRNA CRISPR-Cas9 libraries[J]. Nature Communication,2015,6.
[25] LIANG P,XU Y,ZHANG X,et al. CRISPR/Cas9-mediated gene editing in human tripronuclear zygotes[J]. Protein Cell,2015,6(5):363-272.
Development trend analysis of genome editing technologies based on bibliometric
WANG Huiyuan1,YUAN Tianwei1,RUAN Liangliang2,3,XIONG Yan1
1. Shanghai Information Center for Life Sciences, CAS, Shanghai 200031, China
2. Shanghai Clinical Research Center, Shanghai 200032, China
3. Shanghai Engineering Research Center of Biobank, Shanghai 200031, China
In recent years, new genome editing technologies represented by CRISPR based on novel and eff cient DNA targets have been widely applied in biology and life science research area. Based on SCI-Expanded database in Web of Science and Citespace analysis software, development trend of genome editing is analyzed through bibliometric analysis methods.
genome editing; CRISPR; bibliometric; CiteSpace
10.3969/j.issn.1674-0319.2017.01.002
王慧媛,硕士,馆员。主要从事生命科学及相关领域学科情报和战略情报研究。
E-mail:hywang1213@sibs.ac.cn
国家自然科学基金委员会-中国科学院学科发展战略研究合作项目(L1222037),上海市科学技术委员会科研计划项目(15DZ2292100,16DZ0500600)