陆地棉转录因子GhNAC7的克隆及功能分析
2017-02-16郑学伟SHAHSyedTariq范术丽魏恒玲庞朝友李鸿彬喻树迅
郑学伟,SHAH Syed Tariq,范术丽,魏恒玲,庞朝友,李鸿彬,喻树迅
(1石河子大学生命科学学院,新疆石河子 832000;2中国农业科学院棉花研究所/棉花生物学国家重点实验室,河南安阳 455000)
陆地棉转录因子GhNAC7的克隆及功能分析
郑学伟1,2,SHAH Syed Tariq2,范术丽2,魏恒玲2,庞朝友2,李鸿彬1,喻树迅2
(1石河子大学生命科学学院,新疆石河子 832000;2中国农业科学院棉花研究所/棉花生物学国家重点实验室,河南安阳 455000)
【目的】从陆地棉中克隆GhNAC7,分析其结构和功能,研究其在棉花不同组织中以及叶片不同发育时期的表达量。并转入拟南芥进一步探究其在棉花叶片衰老过程中的作用。【方法】利用中国农业科学院棉花研究所棉花生物学国家重点实验室建立的棉花衰老叶片cDNA文库中的序列,获得1个含有NAM结构域的EST,使用Oligo 6.71设计引物,重新在陆地棉叶片cDNA中进行克隆。使用Gene Structure Display Server软件分析GhNAC7结构,使用在线工具PlantCARE分析启动子序列,利用在线工具GenScan进行氨基酸序列翻译。同时,利用拟南芥基因组数据库(TAIR)进行序列比对,选取得分较高的NAC家族基因,使用MEGA 6.06软件和GeneDoc软件进行进化树分析和氨基酸比对。以XbaⅠ和SacⅠ为酶切位点构建35S::GhNAC7-GFP融合表达载体,分析其在洋葱表皮细胞中的瞬时表达,进行亚细胞定位。利用实时荧光定量PCR技术分析GhNAC7在棉花不同组织、不同叶片发育时期以及在200 μmol·L-1ABA调控下的表达量。通过构建pGhNAC7-GUS融合表达载体并转拟南芥,分析其启动子特异性。以EcoRⅠ和SalⅠ为酶切位点,利用pBI101和pBI121载体,分别构建融合表达载体并转拟南芥进行过表达分析。【结果】从陆地棉中成功克隆GhNAC7,其全长为1 064 bp,包含3个外显子,2个内含子。生物信息学分析结果表明,GhNAC7开放阅读框为834 bp,可编码277个氨基酸,其蛋白质分子量为31.35 kD,等电点为9.22。结构域分析表明其属于NAC转录因子的NAM亚家族,进化树分析显示GhNAC7与ANAC041、ANAC083同源性最高,其中,GhNAC7与ANAC083结构域位置均为17—58 aa。其启动子核心元件包含一系列与衰老、激素、胁迫相关的顺式作用元件。亚细胞定位表明其蛋白为核蛋白。组织特异性表明GhNAC7在真叶、子叶、花、花药和衰老真叶中均明显表达,其中在衰老的真叶中表达量最高。启动子特异性分析表明,其GUS活性在衰老的叶片中最强。在拟南芥中过表达该基因,转基因植株比野生型表现出明显的衰老症状。荧光定量PCR分析表明,ABA处理后6 h GhNAC7明显上调表达,并在48 h表达量达到最高,这表明ABA可调控GhNAC7表达从而调节棉花叶片衰老。【结论】GhNAC7可以促进棉花叶片衰老并受ABA的调控。
陆地棉;NAM结构域;叶片衰老;GhNAC7;过表达
0 引言
【研究意义】棉花作为世界上种植最为广泛的农作物之一,其纤维是纺织工业中应用最普遍的天然纤维。但是,棉花在实际种植过程中受各种环境因素的影响(例如干旱、盐胁迫、黄萎病等),严重影响了棉花纤维的质量和产量。越来越多的研究表明 NAC家族转录因子广泛参与各种胁迫过程,并发挥着显著作用[1-4],NAC家族转录因子也参与一些高度复杂的网络调控,并对环境信号和多种生长发育过程进行有效整合,进而调节叶片衰老[5]。因此,开展棉花特别是陆地棉 NAC基因的功能研究显得格外重要。【前人研究进展】NAC转录因子广泛分布于陆生植物中,它是植物中所特有的具有多种生物功能的转录因子。随着SOUER等[6]从矮牵牛中成功克隆到第一个NAC转录因子NAM,越来越多的NAC家族转录因子在不同植物中被报道。拟南芥中包含138个NAC转录因子,水稻中有158个,狗尾草中有149个,三叶杨中有 289个[7],其他植物(如大麦、玉米、柑橘南瓜、甘蔗、陆地棉、小麦等)中也发现了 NAC基因家族相关基因。因此,NAC家族转录因子被认为是植物中最大转录因子家族之一[8]。NAC转录因子具有多种功能,在植物生长发育、逆境胁迫应答和激素信号转导等过程中具有重要作用[9]。GhNAP可以缓解atnap突变体的延迟衰老,引起拟南芥早衰。在棉花中,GhNAP的下调延迟了叶片衰老。同时,GhNAP受ABA的诱导表达并通过 ABA调控途径调节叶片衰老[10]。EVANS等[11]研究还发现GhNAC18受水杨酸、茉莉酸甲酯和乙烯诱导上调表达,但是在脱落酸的诱导下却下调表达。胁迫处理中,在干旱,过氧化氢,低温(4℃)下其表达上调,但是在盐胁迫中表达受到抑制。有趣的是,GhNAC18的转录激活功能在衰老叶片中受到抑制,其表达量下降,进一步研究发现其在衰老的起始阶段发挥着重要作用。研究证实,GhATAF1在非
生物胁迫中发挥着重要作用,其在ABA、冷胁迫和盐胁迫的调控下上调表达。GhATAF1也受茉莉酸,水杨酸和黄萎病菌的诱导表达,这暗示了其不仅参与非生物胁迫,而且参与了生物胁迫过程。此外,过表达GhATAF1增加了棉花对黄萎病和灰霉病的易感性,并伴随着茉莉酸介导的信号传导的抑制和水杨酸介导的信号传导的激活[12]。同样,SlNAC35表达受干旱胁迫、盐胁迫、细菌病原体和信号分子诱导,暗示其与生物和非生物刺激相关。研究发现SlNAC35可以通过调节NtARF的表达,从而促进根的生长发育[13]。NIU等[14]发现BnaNAC55能够激活一些与活性氧和防御有关基因的表达。无论是在油菜和本氏烟草中表达BnaNAC55,都会导致ROS积累,并最终导致植物过敏性细胞死亡。值得关注的是,大麦中HvNAC005与发育衰老相关,是大麦衰老的正调节因子,且在ABA处理下,受其启动子中 ABA介导的元件的响应,其表达明显上调[15]。此外,水稻OsNAC6[16]和SNAC2[17]、辣椒 CaNAC1[18]、拟南芥 NAC1[19]等基因在相关植物胁迫应答和生长发育中发挥着重要作用。【本研究切入点】NAC家族转录因子功能在拟南芥、水稻等植物中被广泛的研究,但是对于棉花尤其是陆地棉相关的NAC家族转录因子研究却很少,而关于调节衰老相关的 NAC基因更是鲜有报道。【拟解决的关键问题】本研究从陆地棉中克隆获得一个全新的 NAC基因——GhNAC7,进一步分析其在棉花叶片衰老中的作用,为进一步研究其参与叶片衰老的机制奠定基础,为其他棉花 NAC转录因子基因的后续深入研究提供重要的借鉴。
1 材料与方法
1.1 试验材料
试验材料为中棉所10号(CCRI 10),于2013年4月种植于中国农业科学院棉花研究所白壁镇试验田,田间采用常规大田管理。
温室试验处理所用材料中棉所 10号和拟南芥于2013年6月均种植于中国农业科学院棉花研究所棉花生物学国家重点实验室温室,试验条件分别设置为25℃,光照16 h/黑暗8 h;22℃,光照16 h/黑暗8 h。
1.2 试验处理及取样
大田棉花分别取花、花药、纤维(0、5、10、15、20和25DPA)、子叶、真叶、衰老叶片、根和茎为试验材料,每种样品3个重复,所取样品均用液氮迅速冷冻,-80℃保存备用。
温室样品选取长势一致的中棉所 10号棉花幼苗进行处理。其中,用200 μmol·L-1脱落酸(ABA)进行喷洒叶片处理。温室样品处理时均以无菌水处理的棉花幼苗为空白对照。分别在处理后 6、12、24、36和48 h取子叶。所取样品迅速用液氮冷冻,-80℃保存用于提取DNA和RNA。
为研究GhNAC7在自然衰老中的表达情况,从棉花材料中棉所10号的七叶期开始进行统一挂牌,选取叶片生长发育过程的5个时期作为研究对象,为了便于研究,将衰老子叶和叶片的5个发育时期分为以下5个阶段,不衰老期(non-senesent,NS)、衰老初始期(initial-senescent,IS,叶片衰老面积大于10%)、衰老早期(early-senescent,ES,叶片衰老面积小于30%)、衰老晚期(late-senescent,LS,叶片衰老面积大于 50%)和完全衰老期(completely-senescent,CS,叶片衰老面积大于90%),在叶绿素测定和基因表达分析中,以NS作为对照组。
1.3 DNA和RNA的提取
采用改良的CTAB方法[20]提取样品DNA,-20℃保存备用,采用试剂盒(天根生化科技有限公司(北京)的RNAprep Pure Plant Kit(DP441))提取全RNA,方法完全参照说明书进行,-80℃保存备用。cDNA反转录选用 PrimeScript First-Strand cDNA Synthesis Super Mix(TaKaRa,Japan)试剂盒进行。
1.4 GhNAC7的克隆及生物信息学分析
通过对实验室自建的棉花衰老叶片cDNA文库测序,获得1个含有NAM结构域的NAC转录因子的EST(未公布),与雷蒙德氏棉(Gossypium raimondii)D 基因组(http://cgp.genomics.org.cn/page/species/ download.jsp)比对,并设计引物(表1),以陆地棉叶片cDNA为模板,利用基于PCR的改良染色体步移法[21]扩增启动子,扩增程序为94℃ 5 min;94℃ 30 s,58℃ 30 s,72℃ 1 min,30个循环;72℃ 5 min。PCR产物经检测、纯化后,与克隆载体pMD18-T(TaKaRa,Japan)连接,并转化大肠杆菌DH5α,然后挑取单克隆进行测序。DNA测序和引物合成均由苏州金唯智生物科技有限公司完成。
使用 Oligo 6.71设计引物(表 1)。使用 Gene Structure Display Server(http://gsds.cbi.pku.edu.cn/)分析GhNAC7结构[22],利用NCBI在线工具ORF Finder(http://www.ncbi.nlm.nih.gov/gorf/gorf.html)查找GhNAC7的ORF。利用在线工具GenScan(http://genes. mit.edu/GENSCAN.html)进行氨基酸序列翻译。通过在线工具 Compute pI/Mw(http://web.expasy.org/ compute_pi/)预测其蛋白分子量和等电点。使用在线工具 PlantCARE(http://bioinformatics.psb.ugent.be/ webtools/plantcare/html/)对其启动子进行分析。利用美国拟南芥基因组数据库(TAIR,http://www. arabidopsis.org/)进行BLAST分析。氨基酸序列比对利用GeneDoc软件,进化树的构建利用MEGA 6.06软件,采用ClustalW法进行比对,并使用Neighborjoining方法构建进化树。

表1 本研究中的引物Table 1 The primer sequences in this study
1.5 荧光定量PCR
以Ghactin为内参基因,并设计引物(表1)。以反转录 cDNA为模板,利用 ABI 7500系统(Applied Biosystems,美国),选用 UltraSYBR Mixture(With ROX)(康为,北京)试剂盒进行qRT-PCR试验,反应体系和步骤按照SYBR GreenⅠ的说明书进行,GhNAC7相对表达量利用 2-ΔΔCt方法计算。
1.6 叶绿素浓度测定
叶绿素的测定参照LICHTENTHALER[23]的方法,将样品浸泡于80%丙酮中,置于4℃,黑暗处理48 h。取出后轻摇3 min,分别在波长为663和645 nm测定吸光度。根据以下公式计算浓度:
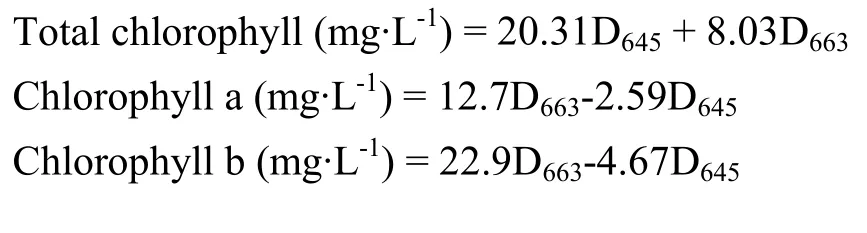
1.7 亚细胞定位
参照SHAH等[4]方法进行GhNAC7亚细胞定位,用 XbaⅠ和 SacⅠ分别对测序正确的 GhNAC7和pBI121载体进行双酶切,并使用T4DNA连接酶连接,构建 35S::GhNAC7-GFP融合表达载体,同时以35S::GFP载体为空白对照。通过冻融法将载体转入农杆菌LBA4404感受态细胞,利用卡那霉素和PCR进行阳性菌液筛选,筛选出的阳性菌在LB液体培养基中扩摇至OD600值为0.6,然后侵染洋葱表皮30 min,无菌滤纸吸净表面菌液于暗处在 1/2MS培养基培养12 h,培养温度设置为25℃。
1.8 GhNAC7启动子的特异性分析
按照1.7方法同时构建pGhNAC7-GUS融合表达载体。将构建好的融合表达载体转入拟南芥,鉴定转基因拟南芥,取叶片、茎、花和根,分别置于 GUS染色液中,37℃培养箱中温育过夜。将浸染过的样品转入70%酒精中脱色2—3次,至阴性对照材料呈白色,然后置于体式镜下观察拍照。
1.9 拟南芥的遗传转化
使用EcoRⅠ和SalⅠ分别对测序正确的GhNAC7和pBI101双元载体进行酶切,用T4连接酶连接过夜,并转化大肠杆菌 DH5α,挑取单克隆,测序后提取质粒转化农杆菌LBA4404感受态细胞,筛选阳性菌落。将以上2种转化后的含有正确质粒的农杆菌转化拟南芥,步骤如下:(1)将农杆菌接种到LB培养基(50 μg·mL-1卡那霉素、50 μg·mL-1链霉素和25 μg·mL-1利福平)中,25℃,175 r/min震荡过夜。(2)扩大培养至OD600=1.3,500 r/min离心15 min,并收集菌体。(3)将收集的菌体重悬于渗透液(5%蔗糖、121℃灭菌15 min,加重浓度0.02%的SilwettL-77),以渗透液为参照,调节OD600至0.8—1.0。(4)将拟南芥的花在重悬液中浸染30—50 s,浸染后的拟南芥置于暗处培养24 h。然后在长日照下培养,直至收种子。
转基因拟南芥的筛选参照张文香等[24]方法。
2 结果
2.1 GhNAC7及其启动子的生物信息学分析
根据中国农业科学院棉花研究所棉花生物学国家重点实验室建立的棉花衰老叶片 cDNA文库进行扩增,经测序获得1 064 bp序列(GenBank:JX155857.1),并依序将其命名为 GhNAC7。使用 Gene Structure Display Server对其结构进行分析,结果显示,其包含3个外显子,2个内含子,其ORF为834 bp(图1)。其启动子包含一系列的顺式作用元件(图 2)。其中,激素响应元件包括ABA响应元件ABRE、茉莉酸甲酯响应元件CGTCA和TGACG。胁迫响应元件包括热胁迫元件HSE、低温胁迫响应元件LTR和干旱胁迫诱导的MYB结合位点MBS。与衰老相关的G-box元件,涉及昼夜节律控制的调控元件circadian以及响应光照的顺式作用元件Gap-box、I-box和Sp1等。这些激素、胁迫和衰老相关的顺式作用元件预示着GhNAC7参与棉花逆境响应和衰老过程,并发挥重要作用。
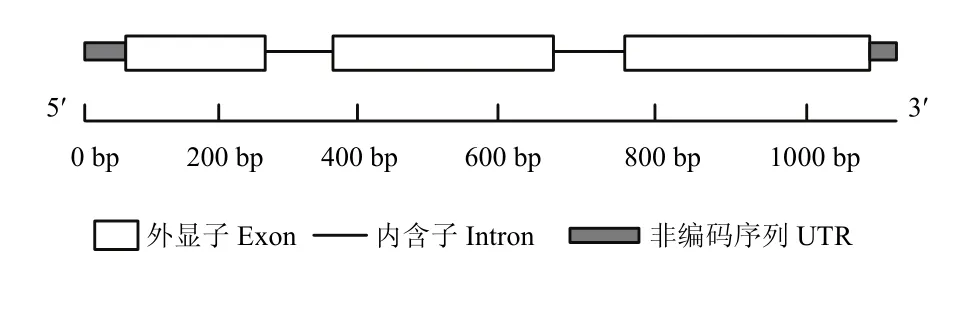
图1 GhNAC7结构示意图Fig. 1 Schematic representation of the genomic DNA structure of GhNAC7
2.2 GhNAC7蛋白结构及进化树分析
由 GenScan在线预测编码氨基酸序列,发现GhNAC7共编码277个氨基酸,进一步分析,可知其蛋白分子质量约为31.35 kD,等电点为9.22,结构域分析表明GhNAC7含有一个NAM结构域,属于NAM亚家族。使用GhNAC7全长CDS在TAIR中进行比对,选取得分较高的NAC家族基因(图3-A),利用MEGA 6.06软件并结合GeneDoc软件,对GhNAC7构建进化树,结果显示,GhNAC7与ANAC041、ANAC083同源性最高(图3-A)。对三者氨基酸序列进行比对,其保守结构域A、B、C、D、E 5个部分如图所示(图3-B)。结果表明,3种蛋白N末端表现出高度的相似性,然而其 C末端表现出明显的差异。其中,GhNAC7与ANAC083同源性最高,其结构域位置均为17—58 aa(图3-B),预示GhNAC7蛋白在植物生长过程中与ANAC083发挥着相似或相近的生物学功能。
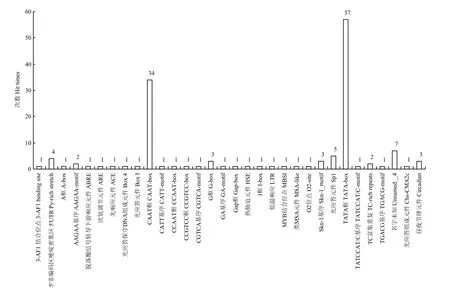
图2 GhNAC7启动子分析示意图Fig. 2 Schematic representation of GhNAC7 promoter

图3 GhNAC7的聚类分析和氨基酸序列比对Fig. 3 Cluster analysis of GhNAC7 and Sequence alignment of amino acids
2.3 GhNAC7蛋白的亚细胞定位
将构建的 35S::GhNAC7-GFP融合表达载体转化到洋葱表皮细胞,暗处培养12 h后,利用共聚焦激光扫描显微镜观察绿色荧光在细胞中的分布(图 4),只在细胞核中观察到绿色荧光,说明GhNAC7被定位在细胞核中;然而,作为对照组,绿色荧光则分布在细胞膜、细胞质和细胞核中。这也说明了GhNAC7启动子具有转录激活活性,进一步说明GhNAC7只作用于细胞核,属于核蛋白。
2.4 GhNAC7在陆地棉中的表达模式分析
通过研究 GhNAC7在不同组织中的表达情况(图5-A),发现GhNAC7在不同组织中表达情况与先前报道的 GhNAC家族基因表达相似[25]。GhNAC7在纤维中几乎不表达,在根、茎、花药中少量表达,在真叶、子叶、衰老叶片和花中表达量较高,尤其在子叶和衰老叶片中表达较明显。为了进一步阐明 GhNAC7在子叶和衰老叶片中的表达情况,分别取正常衰老的真叶和子叶对 GhNAC7的表达量进行分析(图 5-B、5-C),结果表明,在2种组织中GhNAC7随着衰老程度的加深,其表达量增加。在子叶和真叶完全衰老时,其表达量达到最大,尤其是在完全衰老的真叶中,其表达量约是不衰老的 50倍之多,说明该基因在叶片衰老过程中有重要作用。
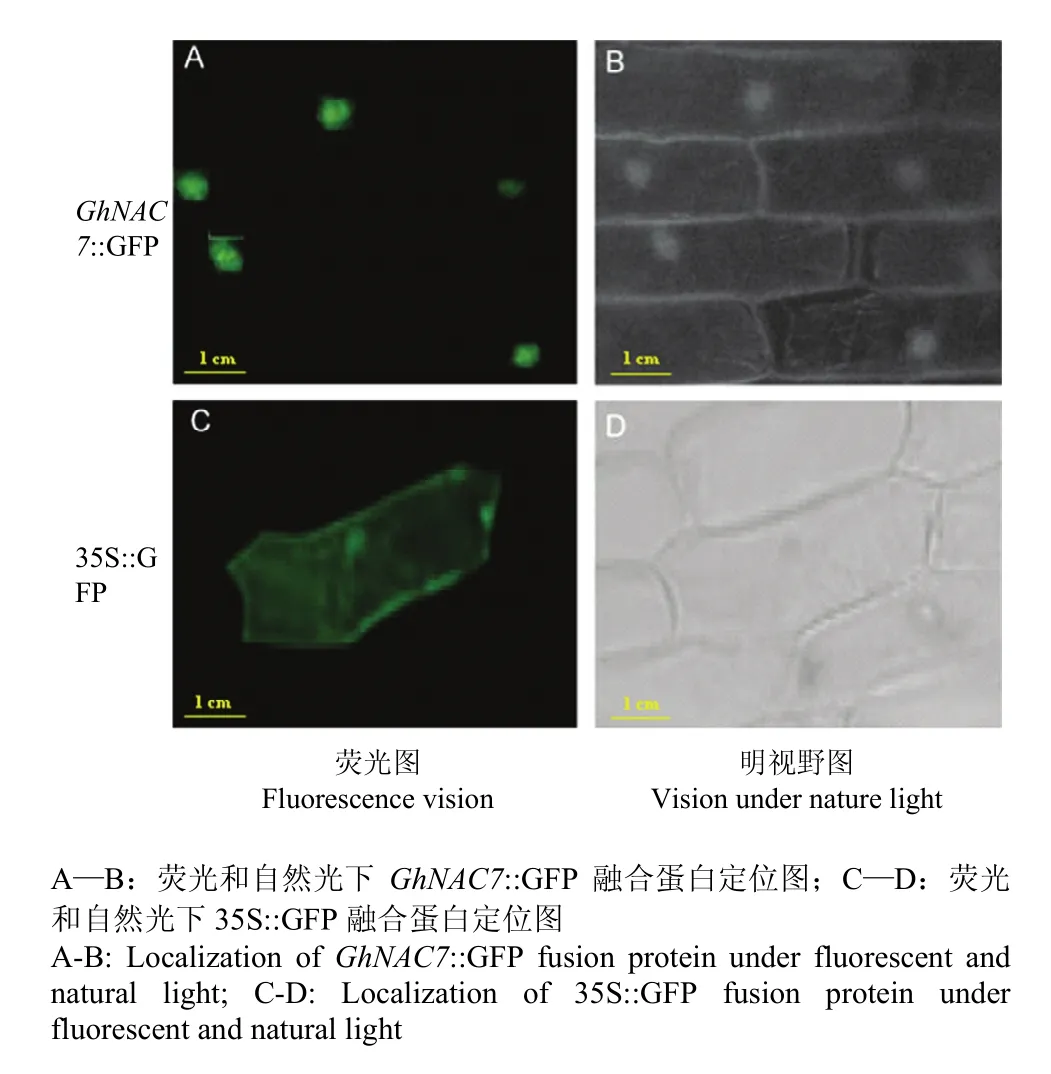
图4 GhNAC7蛋白亚细胞中定位分析Fig. 4 Subcellular localizations of GhNAC7 protein in onion epidermis cells
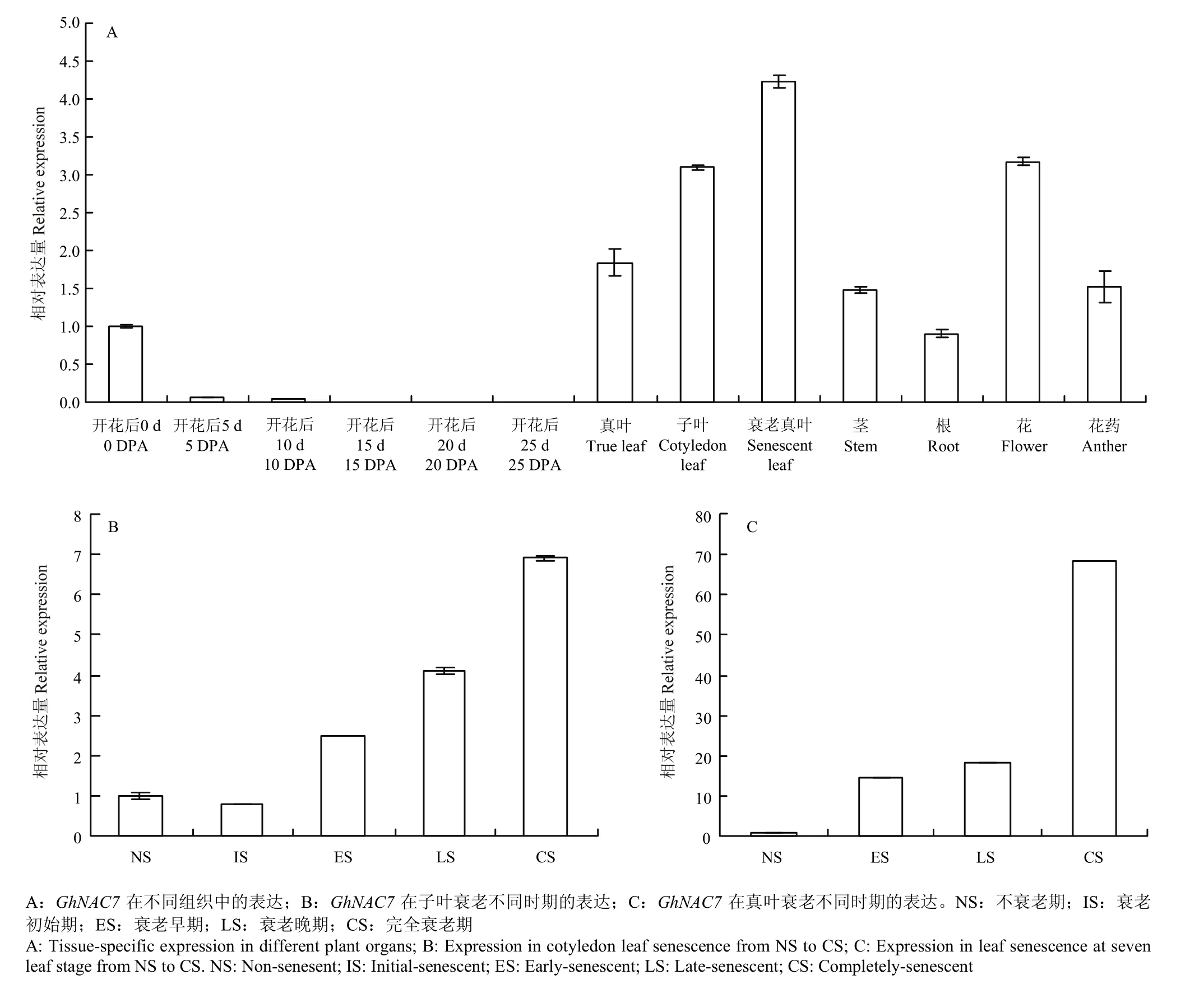
图5 GhNAC7的表达分析Fig. 5 Expression analysis of GhNAC7 gene in cotton
2.5 GhNAC7启动子特异性分析
为进一步验证GhNAC7的组织特异性,通过构建pGhNAC7::GUS融合表达载体,转化拟南芥,并对转基因株系各部分组织进行GUS染色。结果显示,在转基因拟南芥的叶片和根中均检测到GUS活性(图6-B和图6-C),其中,茎生绿叶和根中GUS活性比较微弱,然而在衰老叶片中,其GUS活性最强。而在茎和幼叶、花和种皮中没有检测到GUS活性(图6-A)。
2.6 转基因拟南芥过表达分析
为研究GhNAC7在植物生长过程中可能的生物学功能,构建GhNAC7植物过表达载体,并转化拟南芥进行表型分析。2个载体各筛选出5株转基因株系,进行鉴定(图7)。与野生型植株相比,pBI101载体的过表达株系叶片出现衰老现象(图8-A—图8-C),且观察发现,衰老首先在莲座叶出现,随后出现在茎生叶中。而pBI121载体的过表达株系也同样出现了叶片衰老(图8-D—图8-F)。说明GhNAC7可以促进拟南芥叶片衰老。
2.7 ABA对GhNAC7的调控分析
通过对喷施200 μmol·L-1ABA 6 h后的棉花幼苗子叶进行荧光定量PCR分析(图9),在ABA处理下GhNAC7表达量显著高调,且在处理后48 h达到最大值。结果表明,ABA调控GhNAC7,从而调节棉花叶片的衰老。

图6 转基因拟南芥GUS组织染色Fig. 6 GUS analysis of pGhNAC7::GUS in transgenic Arabidopsis
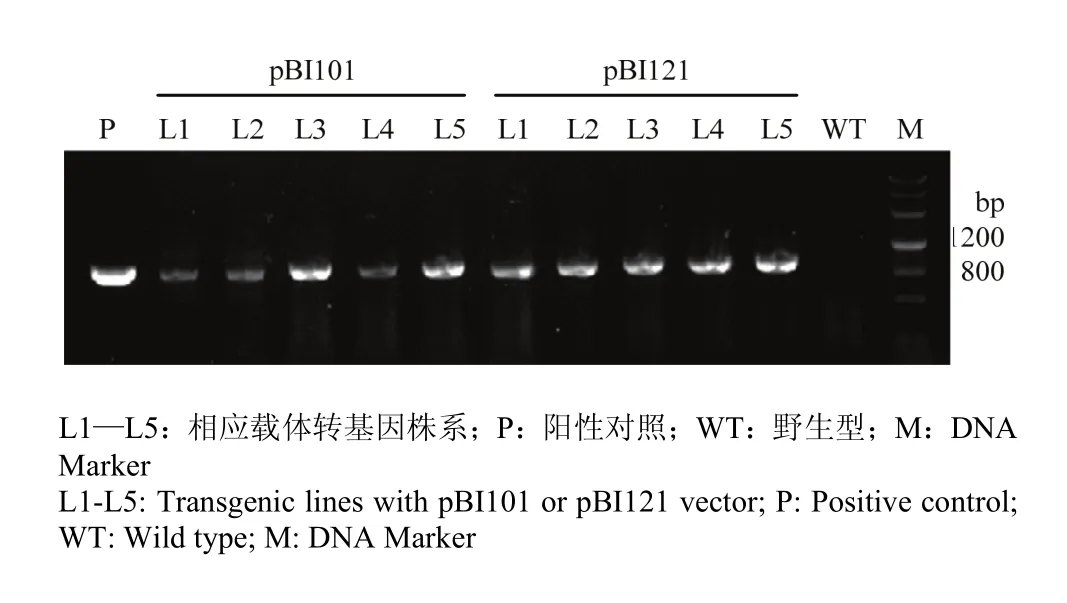
图7 转基因拟南芥DNA检测Fig. 7 DNA identification of transgenic Arabidopsis
3 讨论
NAC家族转录因子是植物中主要的调节因子,其功能在拟南芥、水稻等植物中被广泛的研究,它在调节胁迫应答和植物生长过程中发挥着重要的作用[26]。但是在棉花中,尤其是陆地棉相关的 NAC家族转录因子研究却很少,而调节衰老相关的 NAC基因更是鲜有报道。
本文从陆地棉中成功克隆获得一个全新的 NAC家族转录因子GhNAC7,生物信息学研究发现其拥有一个NAM结构域,这也是NAM亚家族转录因子的特有典型结构。序列比对显示其N端高度保守,C端保守性较低,拥有多个重复出现的氨基酸序列。
荧光定量结果显示,GhNAC7组织特异性表达其与棉花生长发育有着密切的关系,其表达量在衰老的真叶中最高。且进一步研究发现,GhNAC7表达量随着棉花真叶和子叶衰老程度的加深而逐渐增加,这充分表明在棉花叶片衰老过程中GhNAC7扮演着某种重要的作用。ZHAO等[27]研究表明,GhNAC12在早衰和衰老的子叶中,其表达量逐渐增加,最终导致棉花早衰。本研究中,GhNAC7主要在衰老的真叶中表达,说明GhNAC7主要参与调控了棉花真叶衰老的过程。ODA-YAMAMIZO等[28]证实,与野生型拟南芥相比,ANAC046转基因拟南芥明显出现了早衰的表型,说明ANAC046是拟南芥叶片衰老的正向调节因子。为了进一步探究GhNAC7相关的生物学功能,在拟南芥模式植物中对其进行了过表达研究,结果发现,相对于野生型拟南芥,转基因拟南芥表型发生了较大的改变,其中转基因拟南芥表现出明显的衰老现象,推测可能是GhNAC7的过表达导致了转基因拟南芥表型的变化。这也说明GhNAC7在调节叶片衰老的过程中可能扮演着正向调节因子的作用,因此,认为GhNAC7是调控叶片衰老的基因之一。但是,对于调节叶片衰老的具体机制还不得而知,需要进一步加以研究。

图8 转基因拟南芥过表达分析Fig.8 Overexpression of GhNAC7 gene in Arabidopsis
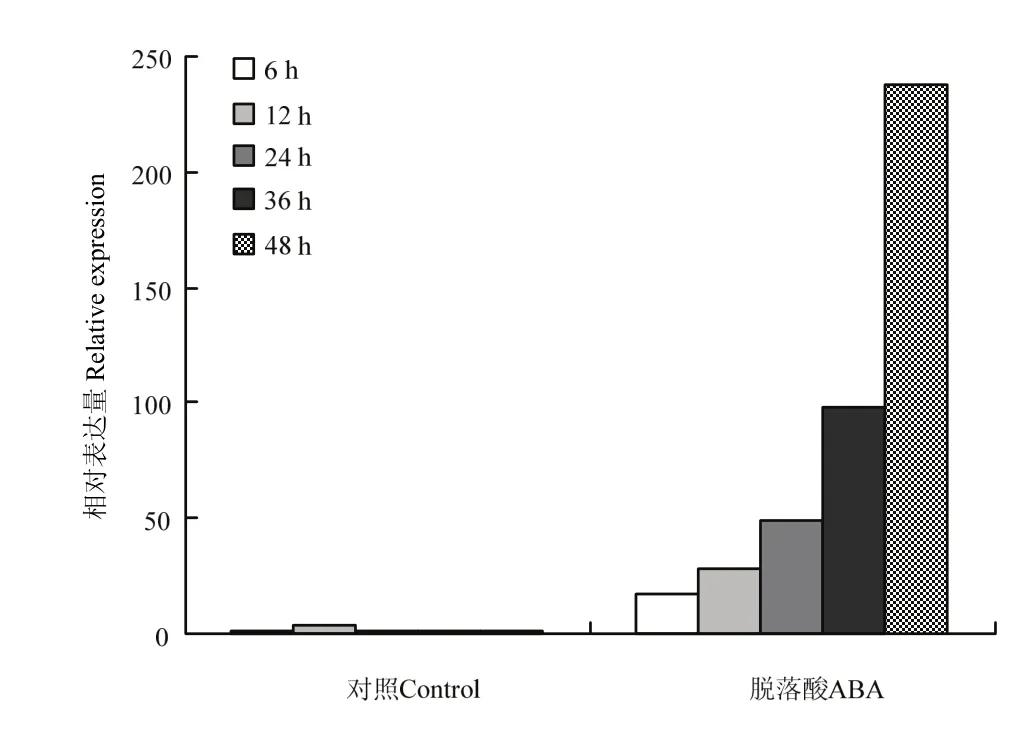
图9 ABA处理下GhNAC7在棉花幼苗中的表达Fig. 9 Expression profile of GhNAC7 gene in ABA treatment in cotton seedlings
进化树分析表明,GhNAC7与ANAC083具有很高的一致性。这也暗示着GhNAC7在植物生长过程中与其发挥着相似的生物学作用。研究表明,ABA是叶片衰老中的正调节因子,可以促进叶片衰老[29]。SHAH等[30]研究也表明,对棉花幼苗施加外源脱落酸和乙烯,NAC家族基因对ABA响应最为明显。此外,ABA应答基因 VND-INTERACTING2(VNI2,ANAC083)可以通过整合 ABA介导的非生物胁迫信号来调节相关基因的表达,如COR(受冷胁迫调节)和RD(受失水调节),进而调节叶片衰老[31]。本文启动子分析显示其包含了ABA响应元件ABRE,荧光定量试验也进一步证实对棉花幼苗施加ABA 6 h后其表达量明显上调,说明在棉花中ABA可以通过调控GhNAC7的表达,调节棉花叶片衰老。研究表明,NAC家族基因在胁迫诱导衰老中发挥着正调节因子的作用[32-33]。综上所述,本文得到了一个棉花叶片的正调控因子,为进一步分析该基因在叶片衰老过程中的作用机制打下基础;同时,利用棉花叶片衰老特异启动子和 RNAi载体,可以降低该基因在叶片衰老过程中的表达量,有望获得抑制棉花叶片衰老的新种质,为该基因的生产应用打下基础。
4 结论
获得GhNAC7全长cDNA,该基因属于NAM转录因子亚家族,其全长为1 064 bp,ORF为834 bp,编码277个氨基酸,其蛋白为核蛋白。GhNAC7启动子核心元件包含一系列与衰老、激素、胁迫相关的顺式作用元件。GhNAC7的表达具有较高的组织特异性,在衰老的真叶中表达量最高,其启动子特异性在衰老的真叶中也最强。在拟南芥中过表达该基因,转基因植株比野生型表现出明显的衰老症状。GhNAC7可促进棉花叶片衰老,并受ABA的调控。
[1] GUNAPATI S, NARESH R, RANJAN S, RANJAN S, NIGAM D, HANS A, VERMA P C, GADRE R, PATHRE U V, SANE A P, SANE V A. Expression of GhNAC2 from G. herbaceum, improves root growth and imparts tolerance to drought in transgenic cotton and Arabidopsis. Scientific Reports, 2016, 6: 24978.
[2] WANG J Y, WANG J P, YANG H F. Identification and functional characterization of the NAC gene promoter from Populus euphratica. Planta, 2016, 244(2): 417-427.
[3] RAHMAN H, RAMANATHAN V, NALLATHAMBI J, DURAIALAGARAJA S, MUTHURAJAN R. Over-expression of a NAC67 transcription factor from finger millet (Eleusine coracana L.) confers tolerance against salinity and drought stress in rice. BMC Biotechnology, 2016, 16(1): 7.
[4] SHAH S T, PANG C, HUSSAIN A, FAN S, SONG M, ZAMIR R, YU S. Molecular cloning and functional analysis of NAC family genes associated with leaf senescence and stresses in Gossypium hirsutum L.. Plant Cell, Tissue and Organ Culture, 2014, 117(2): 167-186.
[5] KIM H J, NAM H G, LIM P O. Regulatory network of NAC transcription factors in leaf senescence. Current Opinion in Plant Biology, 2016, 33: 48-56.
[6] SOUER E, VAN HOUWELINGEN A, KLOOS D, MOL J, KOES R. The no apical meristem gene of Petunia is required for pattern formation in embryos and flowers and is expressed at meristem and primordia boundaries. Cell, 1996, 85(2): 159-170.
[7] JIN J, ZHANG H, KONG L, GAO G, LUO J. PlantTFDB 3.0: A portal for the functional and evolutionary study of plant transcription factors. Nucleic Acids Research, 2014, 42(Database issue): D1182.
[8] 康桂娟, 曾日中, 聂智毅, 黎瑜, 代龙军, 段翠芳. 植物NAC转录因子的研究进展. 生物技术通报, 2012, 28(11): 21-26.
KANG G J, ZENG R Z, NIE Z Y, LI Y, DAI L J, DUAN C F. Research progress of plant NAC transcription factors. Biotechnology Bulletin, 2012, 28(11): 21-26. (in Chinese)
[9] 陈娜, 蒋晶, 曹必好, 雷建军,陈长明. 植物NAC转录因子功能研究新进展. 分子植物育种, 2015, 13(6): 1407-1414.
CHEN N, JIANG J, CAO B H, LEI J J, CHEN C M. The latest progresses on plant NAC transcription factors function. Molecular Plant Breeding, 2015, 13(6): 1407-1414. (in Chinese)
[10] FAN K, BIBI N, GAN S,LI F, YUAN S, NI M, WANG M, SHEN H, WANG X. A novel NAP member GhNAP is involved in leaf senescence in Gossypium hirsutum. Journal of Experimental Botany, 2015, 66(15): 4669-4682.
[11] EVANS O, DOU L, GUO Y, PANG C, WEI H, SONG M, FAN S, YU S. GhNAC18, a novel cotton (Gossypium hirsutum L.) NAC gene, is involved in leaf senescence and diverse stress responses. African Journal of Biotechnology, 2016, 15(24): 1233-1245.
[12] HE X, ZHU L, XU L, GUO W, ZHANG X. GhATAF1, a NAC transcription factor, confers abiotic and biotic stress responses by regulating phytohormonal signaling networks. Plant Cell Reports, 2016, 35(10): 2167-2179.
[13] WANG G, ZHANG S, MA X, WANG Y, KONG F, MENG Q. A stress-associated NAC transcription factor (SlNAC35) from tomato plays a positive role in biotic and abiotic stresses. Physiologia Plantarum, 2016, 158(1): 45-64.
[14] NIU F, WANG C, YAN J, GUO X, WU F, YANG B, DEYHOLOS M K, JIANG Y. Functional characterization of NAC55 transcription factor from oilseed rape (Brassica napus L.) as a novel transcriptional activator modulating reactive oxygen species accumulation and cell death. Plant Molecular Biology, 2016, 92(1): 89-104.
[15] CHRISTIANSEN M W, MATTHEWMAN C, PODZIMSKA-SROKA D, O’SHEA C, LINDEMOSE S, MØLLEGAARD N E, HOLME L B, HEBELSTRUP K, SKRIVER K, GREGERSEN P L. Barley plants over-expressing the NAC transcription factor gene HvNAC005 show stunting and delay in development combined with early senescence. Journal of Experimental Botany, 2016, 67(17): 5259-5273.
[16] NAKASHIMA K, TRAN L S P, VAN NGUYEN D, FUJITA M, MARUYAMA K, TODAKA D, LTO Y, HAYASHI N, SHINOZAKI K, YAMAGUCHI-SHINOZAKI K. Functional analysis of a NAC-typetranscription factor OsNAC6 involved in abiotic and biotic stress-responsive gene expression in rice. The Plant Journal, 2007, 51(4): 617-630.
[17] HU H, YOU J, FANG Y, ZHU X, QI Z, XIONG L. Characterization of transcription factor gene SNAC2 conferring cold and salt tolerance in rice. Plant Molecular Biology, 2008, 67(1/2): 169-181.
[18] OH S K, LEE S, YU S H, CHOI D. Expression of a novel NAC domain-containing transcription factor (CaNAC1) is preferentially associated with incompatible interactions between chili pepper and pathogens. Planta, 2005, 222(5): 876-887.
[19] XIE Q, FRUGIS G, COLGAN D, CHUA N H. Arabidopsis NAC1 transduces auxin signal downstream of TIR1 to promote lateral root development. Genes & Development, 2000, 14(23): 3024-3036.
[20] 宋国立, 崔荣霞, 王坤波, 郭立平, 黎绍惠, 王春英, 张香娣. 改良CTAB法快速提取棉花DNA. 棉花学报, 1998, 10(5): 273-275.
SONG G L, CUI R X, WANG K B, GUO L P, LI S H, WANG C Y, ZHANG X D. A rapid improved CTAB method for extraction of cotton genomic DNA. Cotton Science, 1998, 10(5): 273-275. (in Chinese)
[21] WU A, LIU J. An improved method of chromosome walking for promoter sequences cloning. Chinese Journal of Biochemistry and Molecular Biology, 2005, 22(3): 243-246.
[22] GUO A Y, ZHU Q H, CHEN X, LUO J C. GSDS: A gene structure display server. Yi Chuan, 2007, 29(8): 1023-1026.
[23] LICHTENTHALER H K. [34] Chlorophylls and carotenoids: Pigments of photosynthetic biomembranes. Methods in Enzymology, 1987, 148: 350-382.
[24] 张文香, 范术丽, 宋美珍, 庞朝友, 魏恒玲, 喻树迅. 棉花GhMADS29启动子克隆及表达分析. 棉花学报, 2015, 25(4): 309-315.
ZHANG W X, FAN S L, SONG M Z, PANG C Y, WEI H L, YU S X. Cloning and expression analysis of the promoter of GhMADS29 from cotton. Cotton Science, 2015, 25(4): 309-315. (in Chinese)
[25] MENG C, CAI C, ZHANG T, GUO W. Characterization of six novel NAC genes and their responses to abiotic stresses in Gossypium hirsutum L.. Plant Science, 2009, 176(3): 352-359.
[26] HE X J, MU R L, CAO W H, ZHANG Z G, ZHANG J S, CHEN S Y. AtNAC2, a transcription factor downstream of ethylene and auxin signaling pathways, is involved in salt stress response and lateral root development. The Plant Journal, 2005, 44(6): 903-916.
[27] ZHAO F, MA J, LI L, FAN S, GUO Y, SONG M, WEI H, PANG C. GhNAC12, a neutral candidate gene, leads to early aging in cotton (Gossypium hirsutum L.). Gene, 2016, 576(1): 268-274.
[28] ODA-YAMAMIZO C, MITSUDA N, SAKAMOTO S, OGAWA D, OHME-TAKAGI M, OHMIYA A. The NAC transcription factor ANAC046 is a positive regulator of chlorophyll degradation and senescence in Arabidopsis leaves. Scientific Reports, 2016, 6: 23609.
[29] GAO S, GAO J, ZHU X, SONG Y, LI Z, REN G, ZHOU X, KUAI B. ABF2, ABF3 and ABF4 promote ABA-mediated chlorophyll degradation and leaf senescence by transcriptional activation of chlorophyll catabolic genes and senescence-associated genes in Arabidopsis. Molecular Plant, 2016, 9(9): 1272-1285.
[30] SHAH S T, PANG C, FAN S, SONG M, ARAIN S, YU S. Isolation and expression profiling of GhNAC transcription factor genes in cotton (Gossypium hirsutum L.) during leaf senescence and in response to stresses. Gene, 2013, 531(2): 220-234.
[31] YANG S D, SEO P J, YOON H K, PARK C M. The Arabidopsis NAC transcription factor VNI2 integrates abscisic acid signals into leaf senescence via the COR/RD genes[W]. The Plant Cell, 2011, 23(6): 2155-2168.
[32] PIMENTA M R, SILVA P A, MENDES G C, ALVES J R, CAETANO H D N, MACHADO J P B, BRUSTOLINI O J B, CARPINETTI P A, MELO B P, SILVA J C F, ROSADO G L, FERREIRA M F S, DAL-BIANCO M, PICOLI E A d T, ARAGAO F J L, RAMOS H J O, FONTES E P B. The stress-induced soybean NAC transcription factor GmNAC81 plays a positive role in developmentally programmed leaf senescence. Plant and Cell Physiology, 2016, 57(5): 1098-1114.
[33] MAHMOOD K, EL-KEREAMY A, KIM S H, NAMBARA E, ROTHSTEIN S J. ANAC032 positively regulates age-dependent and stress-induced senescence in Arabidopsis thaliana. Plant and Cell Physiology, 2016: pcw120.
(责任编辑 李莉)
Molecular Cloning and Functional Analysis of GhNAC7 in Upland Cotton (Gossypium hirsutum L.)
ZHENG XueWei1,2, SHAH Syed Tariq2, FAN ShuLi2, WEI HengLing2, PANG ChaoYou2, LI HongBin1, YU ShuXun2
(1College of Life Science, Shihezi University, Shihezi 832000, Xinjiang;2Institute of Cotton Research, Chinese Academy of Agricultural Sciences/State Key Laboratory of Cotton Biology, Anyang 455000, Henan)
【Objective】The primary objectives of this experiment are to clone GhNAC7 gene, analyze its structure, detect its expression in different tissues of cotton and at different developmental leaf senescence stages. Furthermore, its function in cotton leaf senescence was further studied through transforming GhNAC7 gene into Arabidopsis. 【Method】Based on the cotton senescentleaves cDNA library, which was built by State Key Laboratory of Cotton Biology of Institute of Cotton Research of CAAS, this gene was cloned from upland cotton using an expressed sequence tag (EST) containing NAM domain after designing primer using Oligo 6.71. Gene Structure Display Sever was conducted to analyze its structure, PlantCARE was used on-line to study its promoter sequence, and GenScan was simultaneously performed to translate amino acid on-line. Meanwhile, NAC family genes with higher scores were chosen after aligning sequence from Arabidopsis in TAIR. Afterwards MEGA 6.06 was used to display evolutionary relationships of the gene and GeneDOC was conducted to perform sequence alignment of amino acids. Via constructing 35S::GhNAC7-GFP fusion expression vector with XbaⅠand SacⅠrestriction sites, subcellular localization of GhNAC7 was studied by transient expression analysis of onion epidermal cells. Expression profiles of GhNAC7 in various tissues, in response to 200 μmol·L-1ABA treatment and developmental leaf senescence stages were investigated through quantitative real-time PCR (qRT-PCR). Its promoter specificity was conducted by transforming into Arabidopsis thaliana after constructing pGhNAC7-GUS fusion expression vector. Simultaneously, using pBI101 and pBI121 with EcoRⅠand SalⅠrestriction sites were used to construct fusion expression vectors, and then over-expression analysis was performed by transforming GhNAC7 into Arabidopsis thaliana.【Result】In this study, a novel gene GhNAC7 was successfully cloned from upland cotton (Gossypium hirsutum L.). Its full-length was 1 064 bp with three exons and two introns. Results of bioinformatics analysis exhibited that its open reading frame (ORF) was 834 bp, which encoding 277 amino acids. The molecular weights of GhNAC7 encoding protein were 31.35 kD and isoelectric point was 9.22. Domain analysis of GhNAC7 showed that it belongs to the NAM subgroup of NAC family, moreover phylogenetic tree analysis showed that GhNAC7 has the closest genetic relationship with ANAC041 and ANAC083, and GhNAC7 displayed the same domain positions with ANAC083 at 17-58 aa. Its core promoter elements were also predicted, which contained a series of aging, hormone, stress-related cis-acting elements. Subcellular localization elucidated the protein of GhNAC7 encoding is a nucleoprotein. Tissue-specific analysis showed that this gene was significantly expressed in true leaves, cotyledons, flowers, anthers and senescent leaves, but displayed the highest expression in senescent leaves. And in promoter specificity analysis, it was also exhibited the strongest GUS activity in senescent leaf. Over-expression in transgenic Arabidopsis verified evident symptoms of aging compared to the wild type. Fluorescence quantitative PCR analysis showed that the gene expression was significantly up-regulated after 6 h of ABA treatment and displayed the highest at 48 h, so it was hypothesized that ABA could regulate GhNAC7 gene expression to mediate cotton leaf senescence.【Conclusion】It was concluded that GhNAC7 gene could promote cotton leaf senescence and be regulated by ABA.
upland cotton; NAM domain; leaf senescence; GhNAC7; over-expression
2016-08-12;接受日期:2016-10-14
国家棉花产业技术体系建设专项(CARS-18)
联系方式:郑学伟,Tel:15664065727;E-mail:xiangvswei418529@sina.com。通信作者庞朝友,Tel:0372-2562269;E-mail:chypang@163.com。
李鸿彬,Tel:13579760761;E-mail:lihb@shzu.edu.cn
