固定化酶新技术
——酿酒酵母孢子微胶囊固定化酶技术
2017-02-15孔军李子杰中西秀树高晓冬
孔军,李子杰,中西秀树,高晓冬
(江南大学 生物工程学院糖化学与生物技术教育部重点实验室,江苏 无锡,214122)
固定化酶新技术
——酿酒酵母孢子微胶囊固定化酶技术
孔军,李子杰,中西秀树,高晓冬*
(江南大学 生物工程学院糖化学与生物技术教育部重点实验室,江苏 无锡,214122)
随着固定化酶技术的日新月异,微胶囊技术得到了长足的发展。与传统微胶囊生产技术不同,文章介绍了一种“天然的”微胶囊固定化酶生产新技术——酿酒酵母孢子微胶囊固定化酶技术,详述了该固定化技术原理及实例。该技术优点在于:固定化酶的组装“自然”完成,具有天然抗逆性,大小均一,可重复使用,绿色环保,可实现多种酶的共同固定。最后,对其应用前景进行了展望。
固定化酶;微胶囊技术;酿酒酵母孢子固定化酶;“一孢多酶”法
固定化酶技术已成为酶工程研究的重点和热点之一。与游离酶相比,固定化酶提高了酶的稳定性,并使酶能够反复回收利用。微胶囊技术推动了固定化酶的广泛应用。微胶囊固定化酶采用高分子材料将酶包裹在介质周围形成微小颗粒,被包裹的物质称为囊心或芯材,壁壳称为囊壁或壁材[1]。微胶囊固定化酶能够提供一种特定的反应微环境,使反应物或产物选择性进入或释出[2]。微胶囊固定化酶最大的特色在于其能将囊内和囊外空间隔离,提高了酶的稳定性,并且可对囊内外的物理化学性质进行有效调控。如今该技术已广泛应用于食品、医药、化妆品、化工、纺织、生物、农业等领域[3]。然而传统的微胶囊固定化酶技术不足之处在于:纯酶和载体的准备以及对组装设备的依赖增加了生产成本,尤其是对产量要求不大,但仍需要昂贵设备进行加工的酶微胶囊,其成本更高;微胶囊颗粒均一性欠佳,产品性能差异较大[4];固定化过程产生有害物质[5],给人身安全、环境等带来隐患;等等。因而研究探索新的酶固定化技术,开发稳定、高效、生产便利的生产方式,改善微胶囊固定化酶产品性能、降低生产成本、减少生产过程中的资源消耗和环境污染等,是微胶囊固定化酶技术的主要研究内容,符合当今工业绿色环保、可持续发展的要求[6-7]。
对于酿酒酵母孢子的研究很早之前就有报道,但是将孢子应用于酶的固定化却鲜有报道。近些年来,随着对酿酒酵母孢子的深入研究[8-9],孢子的应用领域逐步得到拓宽,酿酒酵母孢子微胶囊固定化酶(Saccharomycescerevisiaespore microencapsulated enzyme)技术应运而生。该技术是利用基因工程技术,将可溶性分泌蛋白(即目标酶类)基因在孢子中进行表达,在产孢过程中实现对该蛋白的微胶囊固定化。该技术生产平台仅需发酵设备,生产规模可根据产品需求量灵活调节。基于对酿酒酵母孢子壁结构的研究,本研究室已经成功实现了多种目标酶类的酵母孢子微胶囊固定化[10-12]。本综述对酿酒酵母孢子微胶囊固定化酶(以下简称孢子固定化酶)的原理、孢子的组装过程以及孢子固定化酶的优势进行详细阐述,并对其应用前景进行展望。
酵母孢子固定化酶的方法主要有以下两种:生物学方法和化学方法。壳聚糖[13]是固定化酶的常用载体,也是野生型酵母孢子壁的重要组成成分,化学方法即通过交联剂将目标酶固定在“壳聚糖球”上,与目前常用的壳聚糖固定化酶操作基本相似;不同之处首先是利用孢子形成大小均一、性状稳定的天然的“壳聚糖球”,然后将目标酶通过理化处理固定在“壳聚糖球”上。因此,本文对化学法孢子固定化酶的原理不做详细阐述,仅举例说明。生物学方法是通过基因工程技术,将需要表达的可溶性分泌类酶在孢子形成过程中直接固定在孢子壁中,形成天然的孢子固定化酶微胶囊。该技术的重要物质基础是孢子壁中的壳聚糖层和二酪氨酸层。以下,将对孢子固定化酶技术的生物学原理及实例进行详细阐述。
1 酿酒酵母孢子微胶囊固定化酶原理
酿酒酵母二倍体细胞在营养丰富的条件下通常进行有丝分裂产生子代个体。在受到环境胁迫时,比如氮源缺乏,会迫使酵母营养细胞从有丝分裂进入减数分裂[14-18],启动产孢程序并最终形成一个含有4个单倍体细胞的子囊,每个单倍体细胞即为孢子。在产孢过程中,形成了前孢子膜这个特殊的膜结构,胞内的分泌系统通过重构,将可溶性分泌蛋白转运至前孢子膜内;在前孢子膜内形成孢子壁时,可溶性分泌蛋白被保留在孢子壁内部。虽然,前孢子膜的外膜在孢子壁的组装过程中消失,但是,成熟的孢子壁仍然能够将可溶性分泌蛋白截留固定在壁内,这主要是由于壳聚糖层和二酪氨酸层的存在。SUDA等[19]在孢子壁完整的野生型酿酒酵母孢子内表达分泌型绿色荧光蛋白GFP时发现,其能被固定在孢子壁内;但是缺少了二酪氨酸层的dit1Δ孢子却不能。因此,在孢子中,二酪氨酸层能够阻挡分泌型蛋白的扩散。
酿酒酵母孢子壁包含有四种形态学上不同的结构层次,从内侧到外侧依次为甘露糖层(Mannan)、β-葡聚糖层(β-glucan)、壳聚糖层(Chitosan)和二酪氨酸层(Dityrosine)[9]。这4层结构的合成是以从内到外的顺序依次进行的。比如,最外层二酪氨酸层的合成只有在次外层壳聚糖层合成完毕后才开始合成[20]。对于壳聚糖层缺失或其合成有严重缺陷的菌株,二酪氨酸层将不能合成。源于孢子壁特有的结构层次,酵母孢子具有高度的抗逆性,比如抗蛋白酶、抗有机试剂、抗热等[21-22],因此,在孢子将可溶性分泌蛋白固定在孢子壁的同时也将抗逆性赋予了固定在孢子壁内部的蛋白。酵母孢子产孢过程中可溶性蛋白的分泌表达机制及孢子壁的特殊结构层次,尤其是壳聚糖层和二酪氨酸层的存在是实现孢子固定化酶技术的基础;相比于二酪氨酸层,壳聚糖层对于酶的固定更加必不可少。而酿酒酵母营养细胞细胞壁疏松,结构简单,从内而外依次由β-葡聚糖层(β-glucan)和甘露糖层(Mannan)组成,壳聚糖含量很少,不具备二酪氨酸层,因此对可溶性分泌蛋白的固定比孢子要少很多[10]。酿酒酵母孢子壁和营养细胞细胞壁结构如图1所示。
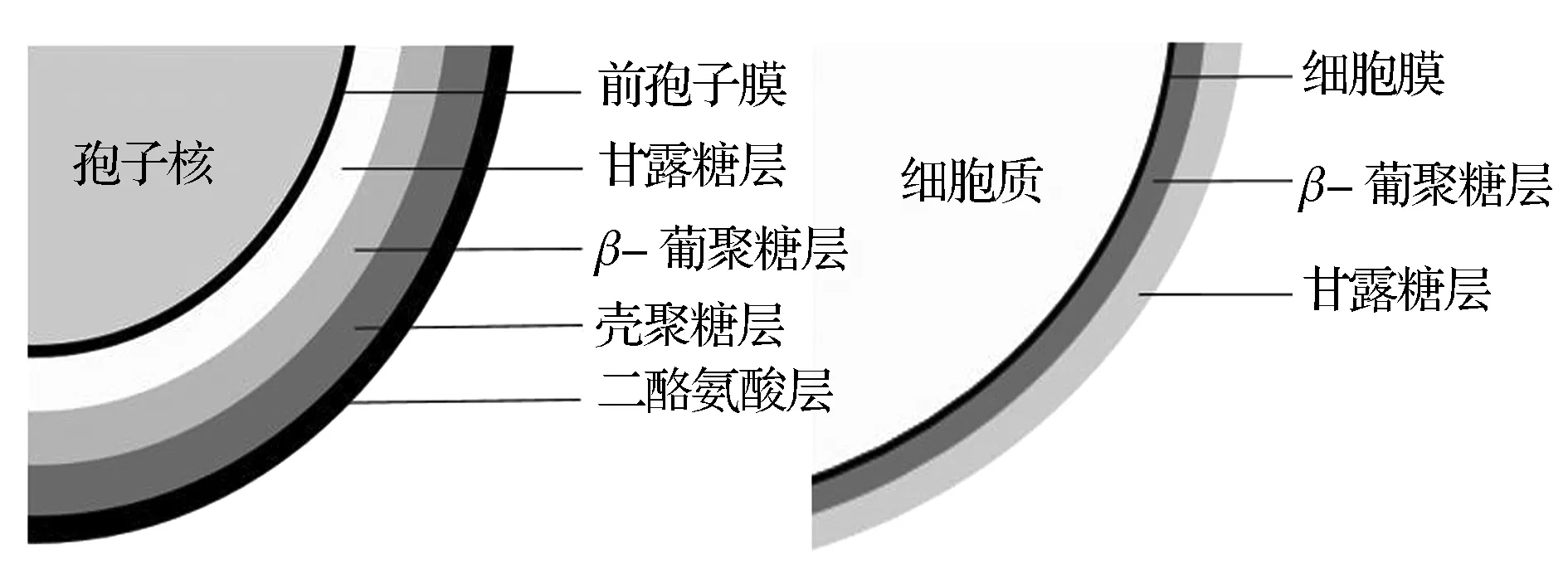
图1 酿酒酵母孢子壁和营养细胞细胞壁结构Fig.1 Saccharomyces cerevisiae spore wall and vegetative cell wall structure
酿酒酵母孢子微胶囊固定化酶技术指:通过分子生物学手段在孢子中表达的可溶性分泌蛋白,在被转运至孢子外部时经过孢子壁,被孢子壁的某些层次所截留:一部分镶嵌在壳聚糖层中,一部分被截留在葡聚糖层和二酪氨酸层之间的空间中;随着孢子的成熟,形成了天然、均一、稳定的孢子固定化酶。同时,由于孢子壁最外层二酪氨酸层是网状半透性膜,大分子物质如蛋白质等不能通过,而小分子物质如底物和产物等可以自由通过,底物与固定在孢子壁内部的酶蛋白结合,从而实现孢子固定化酶对底物的酶解。
2 孢子的组装过程
在孢子的形成过程中,孢子质膜和孢子壁都是在母体细胞的细胞质以从头合成的方式进行的[8]。孢子的形成需要2个重要的步骤:第一是前孢子膜的形成并包裹减数分裂Ⅱ期的子细胞核形成前孢子;第二是孢子壁的组装。
2.1 前孢子膜及前孢子的形成
如图2所示,在减数分裂Ⅱ期,在核被膜的外侧对称形成了4个纺锤极体SPB(spindle pole bodies)[23-24]。囊泡开始形成并定位在SPB的外侧,在可溶性N-乙基马来酰亚胺敏感蛋白受体SNARE(soluble N-ethylmaleimide- sensitive protein receptor)[25]帮助下逐渐融合,形成扁平的双层膜结构,这个结构便是前孢子膜形成的开端。随着减数分裂Ⅱ期核分裂的进行,囊泡不断地融入双层膜结构,最终扩大并包裹各自的子细胞核。核分裂完成后,双层膜最终将各自的细胞核和部分母细胞中的细胞质封闭在内部。这样在原来的母细胞中,最终形成含有四个前孢子的子囊。在每个核外部形成的双层膜结构称之为前孢子膜[23-24, 26]。

图2 前孢子膜的形成Fig.2 Formation of prosproe membrane
2.2 孢子壁的组装
孢子壁的组装即在前孢子膜双层膜之间的内腔填充孢子壁材料,该过程的起始发生在双成膜结构的内腔[27]。在填充孢子壁的过程中,前孢子膜包裹的部分细胞质进行蛋白质的合成和分泌,可溶性分泌蛋白经过前孢子膜分泌到孢子壁外侧。在反馈机制的协调作用下,孢子壁从内而外依层组装,只有靠内的一层完成组装后,靠外的一层才开始组装(图3):首先是最内层甘露糖层,其次为β-葡聚糖层,再次为壳聚糖层,最后为二酪氨酸层[20]。一旦组装完成,孢子壁便赋予孢子一系列抗逆性能[28]。
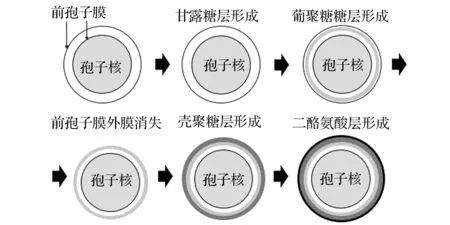
图3 孢子壁的组装Fig.3 Assembly of yeast spore wall
2.2.1 孢子壁合成的起始
孢子壁合成起始所必需的基因一般被认为是GIP1基因,同时有学者发现AMA1基因也是孢子壁合成起始所必需的[29-31]。和GIP1的突变相似,AMA1突变后将导致孢子壁无法合成[32-33]。研究表明,AMA1可能在孢子壁形成的起始和减数第二次分裂完成的衔接中起协调作用。
2.2.2 甘露糖层和β-葡聚糖层的形成
前孢子膜闭合之后,分泌性囊泡将甘露糖蛋白运送至前孢子膜双层膜内腔并大量积累以形成甘露糖层。
β-1,3-葡聚糖层的形成需要β-葡聚糖合成酶的参与,该酶在孢子质膜上合成β-葡聚糖链后将其释放入前孢子膜的内腔,然后这些糖链进一步连接组装形成β-葡聚糖层。在酿酒酵母中,该β-葡聚糖合成酶由3个亚基组成,其中,FKS1基因编码的亚基主要在营养生长时起作用,GSC2/FKS2基因编码的亚基主要在孢子壁组装中起作用,Fks2是负责该层合成的主要蛋白[34]。FKS3基因编码的亚基对孢子组装起一定作用[35],而fks3Δ缺陷菌株展现出孢子壁缺陷的性状[19]。还有研究表明,多个基因对β-葡聚糖层的正常组装都起到重要作用,如SPO77,SPS2,SPS22,CRR1,SSP2[36-39]。其中许多基因是序列簇产孢特有成员,例如,SPS2,SPS22分别与ECM33,PST1相关,CRR1与葡糖水解酶基因CRH1和CRH2相关,GAS与营养细胞壁蛋白合成基因PIR相关[40]。这些成孢特有蛋白在组装甘露糖层和β-葡聚糖层时发挥的作用等同于相应基因在营养细胞壁构建时发挥的作用。在β-葡聚糖层组装完毕后,前孢子膜外膜消失;虽然其具体机制尚不清楚;但是该膜的消失可以使某些孢子壁组装因子如Osw1进入,从而形成孢子外壁[36, 39];同时孢子装配好甘露糖层和β-葡聚糖层后可捕获折射光,使得孢子在光学显微镜下可见。
2.2.3 壳聚糖层的形成
β-葡聚糖层组装完成后,孢子进行壳聚糖(脱乙酰几丁质)层的合成。壳聚糖聚合物的自组装与β-葡聚糖类似,首先几丁质合酶CSⅢ催化亚基Chs3p将几丁质(β-1,4-N-乙酰氨基葡萄糖单体)两两结合形成聚合物,输送至孢子质膜外;此时存在于孢子壁中的特异性脱乙酰酶Cda1p和Cda2p将其脱乙酰化,形成壳聚糖(β-1,4-氨基葡萄糖)[41-43]。营养细胞中变构调节蛋白Chs4p作为几丁质合酶的正向调控蛋白,在产孢过程中其功能被同源的Shc1所取代[44]。一旦壳聚糖聚合物形成,则被精确定位到孢子壁中,定位相关基因至少包括MUM3和OSW1;Mum3蛋白与酰基转移酶同源,这可能与孢子壁组装的催化机制一致[45],然而Mum3蛋白的功能尚不明确;Osw1蛋白定位在孢子壁中,并可能直接参与壳聚糖层的组装[36, 39]。同时由于壳聚糖在包裹孢子的过程中形成分支后与相邻孢子的壳聚糖连接,因此在壳聚糖层形成的过程中,孢子桥连形成[32];即使将子囊膜去除,由于这种桥连的存在,4个孢子仍然连接在一起。
2.2.4 二酪氨酸层的形成
该层既不是由糖类组成,也不是由蛋白质组成,而是由含量大约为50%的非肽类N,N-二甲酰基二酪氨酸组成[46-47];然而其他成分以及二酪氨酸单体如何定位在孢子壁上仍然未知。该层的形成分为两步:首先是二酪氨酸的形成,其次是二酪氨酸的转运及定位。位于孢子基质中的DIT1和DIT2基因参与二酪氨酸的形成[48]。首先具有甲酰基转移酶活性的Dit1p,将甲基转移至L-酪氨酸上;作为细胞色素P450家族成员的Dit2p将2个N-甲基-酪氨酸的苯环连接在一起[48]形成二酪氨酸。新合成的二酪氨酸在ATP依赖的、位于前孢子膜上的转运蛋白Dtr1p的作用下由孢子胞质运送至孢子壁进行组装[49]。像前几层的合成一样,二酪氨酸需要在胞外酶的作用下组装为聚合体的形式,但是对这些酶的具体功能还有待研究。
形成的二酪氨酸层网状孔径大小、松散程度可以通过某些基因的突变进行调节,如OSW2基因的敲除,使得二酪氨酸层孔径增大。对于缺失了二酪氨酸层的dit1Δ孢子和同时缺失了二酪氨酸层与壳聚糖层的chs3Δ孢子,可溶性蛋白则渗漏到孢子壁外部的子囊胞质中[19]。以上突变,对孢子固定化酶酶学性质改进提供了物质基础。
3 酿酒酵母孢子微胶囊固定化酶实例
根据不同的实验要求,本研究室已利用酵母孢子固定化酶技术成功地固定了多种可溶性酶,下面的实例中将分别予以介绍。
3.1 酵母孢子固定化酶的成功先例——酿酒酵母孢子微胶囊固定化α-半乳糖苷酶
施李兵等[10]为了研究二酪氨酸层对于α-半乳糖苷酶的固定是否必需及其在孢子中的定位情况,将α-半乳糖苷酶(MEL1基因编码)与红色荧光蛋白RFP在孢子中进行融合表达。在此之前,研究者首先将RFP连接在信号肽C端构建spRFP融合蛋白,并在不同孢子突变体上进行表达(孢子固定化酶生物法):酿酒酵母野生型孢子(孢子壁完整)、osw2Δ孢子(二酪氨酸层疏松)和dit1Δ孢子(不含有二酪氨酸层)。通过荧光共定位发现:野生型孢子由于二酪氨酸层的存在,在孢子壁边缘有清晰的红色荧光轮廓;osw2Δ孢子的二酪氨酸层虽然有一定程度的缺陷,但孢子壁周围仍具有较强的清晰红色荧光;dit1Δ缺陷型孢子由于不含有二酪氨酸层,RFP不能被固定在孢子壁上,因此弥散在子囊胞质中,从而证实二酪氨酸层能够作为“扩散屏障”阻止可溶性蛋白的扩散。
随后,研究者为了证明壳聚糖层在孢子固定化α-半乳糖苷酶中的作用,将α-半乳糖苷酶与RFP在孢子中进行融合表达。与单独表达RFP的结果一致,野生型孢子和osw2Δ孢子均能够将融合蛋白表达定位在孢子壁上;但值得注意的是,与单独表达RFP不同,dit1Δ孢子表达的α-半乳糖苷酶-RFP融合蛋白也能够定位在孢子壁上;而chs3Δ孢子(不含有二酪氨酸层和壳聚糖层)表达的融合蛋白不能表达定位在孢子壁上。说明对于α-半乳糖苷酶,不需要二酪氨酸层的存在,壳聚糖层就可以将该酶表达定位在孢子壁上。
研究者为了确定最优的表达固定体系,对不同突变株固定的α-半乳糖苷酶的活性进行检测。同野生型孢子和osw2Δ孢子相比,dit1Δ孢子表达的α-半乳糖苷酶的活性最高,可能的原因是由于“扩散屏障”二酪氨酸层的缺失,使得底物更易于与酶接触发生反应;osw2Δ孢子表达的α-半乳糖苷酶的活性比野生型高,可能的原因是osw2Δ孢子的二酪氨酸层较野生型松散、孔径更大,底物更容易穿过二酪氨酸层与酶接触发生反应。
虽然dit1Δ孢子表达的α-半乳糖苷酶的活性最高,但由于“扩散屏障”二酪氨酸层的缺失,导致dit1Δ孢子表达固定的α-半乳糖苷酶更易释放。为了验证这种可能性,对各种突变体孢子用含有去污剂的高盐溶液(0.6 mol/L NaCl和0.1% Triton X-100)进行洗涤,然后测定洗涤后的活性。结果显示:同野生型孢子和osw2Δ孢子相比,dit1Δ孢子表达的酶活下降得最为明显;又经过4次洗涤,dit1Δ孢子表达的酶活下降了约50%,wt孢子和osw2Δ孢子表达的酶活下降了约25%。由此可以说明,二酪氨酸层的存在对于阻止α-半乳糖苷酶从孢子壁上的释放起到重要作用。
为了研究孢子固定化α-半乳糖苷酶对蛋白酶的抗逆性,使用蛋白酶K对不同孢子壁缺陷型孢子固定化α-半乳糖苷酶进行处理,并以酿酒酵母营养细胞分泌到培养基中并浓缩的α-半乳糖苷酶作为对照。结果显示:野生型孢子和osw2Δ孢子固定的α-半乳糖苷酶对蛋白酶K展现出很强的抗逆性。这是由于:二酪氨酸层作为“扩散屏障”只能允许小分子物质(如底物和产物)通过,不允许大分子物质(如蛋白酶K、固定在孢子壁中的酶)通过。蛋白酶K与α-半乳糖苷酶隔离,从而展现出抗蛋白酶K降解的能力;dit1Δ孢子固定的α-半乳糖苷酶经蛋白酶K处理后,虽然酶活下降较野生型和osw2Δ多,但仍有很高的相对酶活,说明壳聚糖层对α-半乳糖苷酶也有一定的保护作用;而游离的α-半乳糖苷酶由于不受到任何保护作用,经蛋白酶K处理后,相对酶活仅为35%左右。此外还发现孢子固定的α-半乳糖苷酶具有比游离酶具有更高的温度耐受性;同时,经多次洗涤后,孢子固定化酶残留酶活仍然很高。
3.2 酿酒酵母孢子微胶囊固定化酶在稀少糖合成中的应用
以葡萄糖为底物,在木糖异构酶(XI)和D-阿洛酮糖-3-差向异构酶(DPE)的联合作用下合成具有广泛应用前景的D-阿洛酮糖(Psicose)是目前稀少糖研究的重点。为避免直接使用游离酶,本研究室李子杰和李毅[12]利用生物法和化学法两种方法对木糖异构酶和D-阿洛酮糖-3-差向异构酶进行了孢子固定化。在生物法的研究中,将XI与GFP在孢子中进行融合表达,通过GFP的荧光定位来确定XI在孢子中的定位。对于野生型孢子和osw2Δ孢子,能够清晰地看到GFP绿色荧光信号完好地包裹在孢子壁上,说明XI成功地固定在了孢子壁上。相对于野生型孢子和osw2Δ孢子,dit1Δ孢子由于二酪氨酸层的缺失导致GFP信号强度稍弱,但仍可以观察到融合蛋白表达定位到孢子壁上。为了确定XI在孢子上是否具有催化活性,通过HPLC检测固定化XI将D-葡萄糖转化为D-果糖的能力。结果显示,底物D-葡萄糖在孢子固定化酶的催化作用下可以转化为D-果糖。之后对不同孢子壁缺陷型孢子固定化XI进行了酶活比较、重复利用率、最适温度和最适pH等实验,结果与施李兵研究相似,osw2Δ孢子是较野生型和dit1Δ孢子最适合用于固定化酶的载体。值得注意的是,野生型和osw2Δ孢子固定化酶在95℃高温下的相对酶活都能达到最高酶活的60%。综合考虑,孢子壁中二酪氨酸层的存在,一方面能有效地阻止酶的释放,另一方面提高了酶对高温的耐受性。
在化学法固定化酶的研究中,李毅[50]用缺少了二酪氨酸层的壳聚糖球-dit1Δ孢子作为固定化载体,对D-阿洛酮糖-3-差向异构酶进行了固定化研究。结果显示,孢子壳聚糖球固定化的D-阿洛酮糖-3-差向异构酶展现出较游离酶更广阔的温度和pH耐受区间。在重复利用8次后,相对酶活仍然高达80%。最后,又利用一锅两酶法进行了实验:首先利用osw2Δ孢子固定的木糖异构酶将D-葡萄糖转化成D-果糖,然后利用dit1Δ孢子固定的D-阿洛酮糖-3-差向异构酶将D-果糖转化成D-阿洛酮糖。通过工艺的改进,最终使得阿洛酮糖的转化率达到12%,比利用游离酶高出2倍。至此,利用酵母孢子固定化酶并进行生产已经在实验室水平取得成功。
在以上2个实例中,研究者均以营养细胞为对照,通过检测带有RFP或GFP标签的目的蛋白在孢子中的定位,证实了酵母孢子可以将可溶性分泌蛋白表达固定在孢子壁中;有学者利用酵母营养细胞进行酶的固定化[51],但相对于细胞壁简单的营养型细胞,孢子固定的酶量更多[10]。同时,为了比较不同孢子壁缺陷型孢子固定化酶量高低,将带有3×HA标签的目的蛋白在相应孢子中表达。野生型孢子和osw2Δ孢子表现出相对于dit1Δ更高的酶固定量,这与在dit1Δ孢子中表达的MEL1-RFP或GFP-XI融合蛋白荧光信号较弱相一致,说明二酪氨酸层的存在一定程度上阻止了酶的泄漏。二酪氨酸层作为保护屏障,可以保护固定在孢子壁中的酶免受蛋白酶的降解,在研究中也得到了证实[10]。之后对固定在孢子壁内部的酶的催化活性进行了确认,并对不同孢子壁缺陷菌株固定化酶的酶学性质进行了研究。总之,以上两个实验从各个方面证明了酵母孢子微胶囊固定化酶的可行性。
3.3 酵母孢子固定化酶的优点
通过酵母孢子固定化原理及已有实验结果可以看出,该方法的优点在于酶的固定化“自然”完成,具有天然抗逆性,大小均一,可重复使用,绿色环保,可实现1个孢子对多个酶共同固定:
(1)成本低廉:酵母孢子固定化酶随着孢子的形成而“自然”完成,只要在孢子内部表达所需固定化酶的基因,便可得到孢子固定化酶微胶囊;无需考虑芯材和壁材的兼容性,避免了对纯酶、固定化材料和组装设备的依赖,降低了生产成本;不需要理化过程介入,避免了理化处理对产品性能的影响;生产平台仅需发酵设备,生产规模可根据产量要求灵活调节;
(2)抗逆性强:由于酵母孢子壁的特殊结构层次,其较强的天然抗逆性可以保护固定在其中的酶抵抗外界不利条件的影响;
(3)产品质量均一:酵母产孢为严格的自然生理过程,因此,孢子固定化酶微胶囊大小一致,性状稳定;
(4)生产简便:孢子一经产出无需纯化,便于产品与酶的分离,可重复使用;
(5)安全:酵母产孢过程为自然生理过程,生产过程能耗低、污染少,产品作为生物质可自然降解,绿色环保。
(6)“一孢多酶”体系初步建立:笔者已成功将肌酐降解酶系中的2种酶共表达、固定在同一孢子内部。实验证实,“一孢两酶”法是可行的,较分别固定后再联合使用表现出更优越的酶学性能。下一步,笔者将对“一孢三酶”法进行研究。
“一孢多酶法”是本研究室目前正在进行的酵母孢子固定化酶研究项目之一,目的是实现多酶体系的孢子微胶囊固定化,即在一个孢子内部同时表达、固定两种或两种以上的酶。对于共表达双酶体系的优点,有报道[51-52]称:2种酶的共表达在空间上十分接近,形成的微环境利于第二种酶与第一种酶解产物的迅速结合,节省了第一种酶酶解产物扩散到第二种酶的时间,并为第二种酶解反应提供持续的底物浓度。有学者实现了D-葡糖异构酶和D-阿洛酮糖异构酶在大肠杆菌中的共表达[53],且产率达到16%。在肌酐检测中,肌酐经肌酐酶水解为肌酸,肌酸被肌酸酶水解为肌氨酸和尿素,肌氨酸被肌氨酸氧化酶水解产生双氧水,通过检测双氧水的生成量来确定肌酐的浓度[54-55]。目前,笔者已将“一孢两酶法”成功应用到肌酸酶和肌酐酶在孢子壁中的共表达、固定,这为“一孢多酶法”的研究和肌酐检测试剂盒的研发打下理论基础。届时,酵母孢子对多酶体系的微胶囊固定化将成为另一优势。
4 讨论与展望
截止目前,本研究室已经成功地将多种固定在酿酒酵母孢子中,为孢子微胶囊固定化酶的应用奠定了坚实的理论和实践基础。综合考察酵母孢子对不同酶的固定化效果后不难发现:各突变株孢子中,dit1Δ孢子固定化酶活性最高,但是经反复使用后其酶活下降较快,不宜重复使用;这是由于dit1Δ的孢子壁缺失最外层二酪氨酸层,导致一部分固定不牢固的酶失去扩散屏障而损失,同时抗逆性能减弱。而osw2Δ的酶活虽然比dit1Δ略低,但其经过反复使用后仍有较高的酶活;而且,osw2Δ比dit1Δ抗逆性更强,比如经蛋白酶K、SDS处理后残留酶活更高,对温度、pH的耐受性更强;这是由于该突变株受到二酪氨酸层保护,同时osw2Δ孢子壁的二酪氨酸层网孔较野生型松散,增加了底物与固定化酶的结合机会。因此,osw2Δ孢子是比较理想的固定化酶载体。因此,通过调节二酪氨酸层孔径大小,开发固定化酶性能优于osw2Δ的酵母孢子突变株是该研究的下一个目标。
目前,本研究室利用酵母孢子固定化技术正在进行的研究项目有:肌酸酶和肌酐酶的固定化,以期开发肌酐检测用试剂盒;固定化稀有糖酶,用于功能性稀有糖的生产;其他研究项目,如肠胃道给药载体的研究。虽然人的肠胃道环境和果蝇不同,但是孢子的抗逆性使其可以穿过果蝇肠胃道[56],这为利用孢子的强抗逆性开发一般药物难以到达的肠胃道中末端药物提供了思路。因此,将孢子微胶囊固定化酶用于药用试剂盒和功能性药物的研发具有极大的潜力。
在研究酵母孢子固定化酶的同时,我们发现:由于壳聚糖具有吸附性的特点,作为壳聚糖球的dit1Δ孢子可以吸附金属离子和负电性化合物。例如,本研究室张海妮[11]将二酪氨酸层缺失的dit1Δ孢子用作壳聚糖球,用来吸附Cu2+,并与孢子壁完整的野生型和同时缺失二酪氨酸层和壳聚糖层的chs3Δ孢子进行比较。结果显示,作为壳聚糖球的dit1Δ孢子,吸附Cu2+能力最强;其次是孢子壁完整的野生型;而缺失了二酪氨酸层和壳聚糖层的chs3Δ孢子吸附Cu2+能力最弱。之后,又对孢子吸附Cr3+、Ni2+、Zn2+、Cd2+、Pb2+等离子的能力进行了研究。同样,作为壳聚糖球的dit1Δ孢子,对各种离子的吸附量是所有细胞类型中最高的,而营养细胞是最低的;即便是缺失了最外2层的chs3Δ孢子,其吸附量也是营养细胞的2倍多。由此说明,缺失了二酪氨酸层的dit1Δ孢子作为壳聚糖球用于重金属离子的吸附是最佳选择。同时,由于壳聚糖带正电,又对作为壳聚糖球的dit1Δ孢子吸附带有负电荷的牛磺胆酸能力进行研究。与吸附金属离子结果相似,dit1Δ孢子吸附牛黄胆酸能力优于野生型孢子和chs3Δ孢子。上述两个实验说明dit1Δ孢子作为壳聚糖球对重金属及某些负电荷分子的吸附是可行的。该研究将酿酒酵母孢子的应用从固定化酶领域进一步拓展到了环保领域,为酿酒酵母孢子的应用开拓了道路。
综上所述,除了对孢子吸附能力的研究外,本实验室对于酵母孢子固定化酶的研究是酵母孢子应用研究的重点。随着微胶囊固定化酶技术的发展,酿酒酵母孢子固定化酶作为微胶囊固定化酶技术的新成员,以其独特的优点首次展现在大家面前:固定化酶的组装“自然”完成,具有天然抗逆性,大小均一,可重复使用,绿色环保,“一孢多酶体系”已经初步建立,等等。基于以上优点及研究结果,相信,酵母孢子固定化酶将会为酶的微胶囊固定化增光添彩!
[1] BENITA S. Microencapsulation: methods and industrial applications [M]. CRC Press, 2005.
[2] PEYRATOUT C S, DAEHNE L. Tailor-made polyelectrolyte microcapsules: from multilayers to smart containers [J]. Angewandte Chemie International Edition, 2004, 43(29): 3 762-3 783.
[3] MEIER W. Polymer nanocapsules [J]. Chemical Society Reviews, 2000, 29(5): 295-303.
[4] 姚善泾, 梅乐和. 一种新型生物微胶囊体系及其在生物质固定化过程中的应用 [J]. 膜科学与技术, 1999, 19(1): 19-23.
[5] HANEFELD U, GARDOSSI L, MAGNER E. Understanding enzyme immobilisation [J]. Chemical Society Reviews, 2009, 38(2): 453-468.
[6] 陈诗一. 能源消耗, 二氧化碳排放与中国工业的可持续发展 [J]. 经济研究, 2009(4):41-55.
[7] 付允, 马永欢, 刘怡君, 等. 低碳经济的发展模式研究 [J]. 中国人口 资源与环境, 2008, 18(3): 14-19.
[8] NEIMAN A M. Ascospore formation in the yeastSaccharomycescerevisiae[J]. Microbiology and Molecular Biology Reviews, 2005, 69(4): 565-584.
[9] NEIMAN A M. Sporulation in the budding yeastSaccharomycescerevisiae[J]. Genetics, 2011, 189(3): 737-765.
[10] SHI L, LI Z, TACHIKAWA H, et al. Use of yeast spores for microencapsulation of enzymes [J]. Applied and environmental microbiology, 2014, 80(15): 4 502-4 510.
[11] ZHANG H, TACHIKAWA H, GAO X-D, et al. Applied usage of yeast spores as chitosan beads [J]. Applied and environmental microbiology, 2014, 80(16): 5 098-5 105.
[12] LI Z, LI Y, DUAN S, et al. Bioconversion of d-glucose to d-psicose with immobilized d-xylose isomerase and d-psicose 3-epimerase onSaccharomycescerevisiaespores [J]. Journal of industrial microbiology & biotechnology, 2015, 42(8): 1 117-1 128.
[13] CHEN S-C, DUAN K-J. Production of Galactooligosaccharides Using β-Galactosidase Immobilized on Chitosan-Coated Magnetic Nanoparticles with Tris (hydroxymethyl) phosphine as an Optional Coupling Agent [J]. International journal of molecular sciences, 2015, 16(6): 12 499-12 512.
[14] HONIGBERG S M. Ime2p and Cdc28p: Co-pilots driving meiotic development [J]. Journal of cellular biochemistry, 2004, 92(5): 1 025-1 033.
[15] LEE B, AMON A. Meiosis: how to create a specialized cell cycle [J]. Current opinion in cell biology, 2001, 13(6): 770-777.
[16] MITCHELL A P. Control of meiotic gene expression inSaccharomycescerevisiae[J]. Microbiological reviews, 1994, 58(1): 56-70.
[17] ROEDER G S. Sex and the single cell: meiosis in yeast [J]. Proceedings of the National Academy of Sciences, 1995, 92(23): 10 450-10 456.
[18] STERN B M. FEARless in meiosis [J]. Molecular cell, 2003, 11(5): 1 123-1 125.
[19] SUDA Y, RODRIGUEZ R K, COLUCCIO A E, et al. A screen for spore wall permeability mutants identifies a secreted protease required for proper spore wall assembly [J]. PLoS One, 2009, 4(9): e7184.
[20] TACHIKAWA H, BLOECHER A, TATCHELL K, et al. A Gip1p-Glc7p phosphatase complex regulates septin organization and spore wall formation [J]. The Journal of cell biology, 2001, 155(5): 797-808.
[21] EGEL R. Selective spore survival during replica-plating of fission yeast [J]. Archives of microbiology, 1977, 112(1): 109-110.
[22] KUPIEC M, BYERS B, ESPOSITO R E, et al. 11 Meiosis and Sporulation inSaccharomycescerevisiae[J]. Cold Spring Harbor Monograph Archive, 1997, 21:889-1 036.
[23] BYERS B. Cytology of the yeast life cycle [J]. Cold Spring Harbor Monograph Archive, 1981, 11:59-96.
[24] STRATHERN J N, JONES E W, BROACH J R. Molecular biology of the yeast saccharomyces, life cycle and inheritance [M]. Cold Spring Harbor Laboratory, 1981.
[25] NAKANISHI H, MORISHITA M, SCHWARTZ C L, et al. Phospholipase D and the SNARE Sso1p are necessary for vesicle fusion during sporulation in yeast [J]. J Cell Sci, 2006, 119(7): 1 406-1 415.
[26] MOENS P B. Fine structure of ascospore development in the yeastSaccharomycescerevisiae[J]. Canadian Journal of Microbiology, 1971, 17(4): 507-510.
[27] LYNN R R, MAGEE P T. Development of the spore wall during ascospore formation inSaccharomycescerevisiae[J]. The Journal of cell biology, 1970, 44(3): 688-692.
[28] SMITS G J, VAN DEN ENDE H, KLIS F M. Differential regulation of cell wall biogenesis during growth and development in yeast [J]. Microbiology, 2001, 147(4): 781-794.
[29] COOPER K F, MALLORY M J, EGELAND D B, et al. Ama1p is a meiosis-specific regulator of the anaphase promoting complex/cyclosome in yeast [J]. Proceedings of the National Academy of Sciences, 2000, 97(26): 14 548-14 553.
[30] OELSCHLAEGEL T, SCHWICKART M, MATOS J, et al. The yeast APC/C subunit Mnd2 prevents premature sister chromatid separation triggered by the meiosis-specific APC/C-Ama1 [J]. Cell, 2005, 120(6): 773-788.
[31] PENKNER A M, PRINZ S, FERSCHA S, et al. Mnd2, an essential antagonist of the anaphase-promoting complex during meiotic prophase [J]. Cell, 2005, 120(6): 789-801.
[32] COLUCCIO A, NEIMAN A M. Interspore bridges: a new feature of theSaccharomycescerevisiaespore wall [J]. Microbiology, 2004, 150(10): 3 189-3 196.
[33] DIAMOND A E, PARK J-S, INOUE I, et al. The anaphase promoting complex targeting subunit Ama1 links meiotic exit to cytokinesis during sporulation inSaccharomycescerevisiae[J]. Molecular biology of the cell, 2009, 20(1): 134-145.
[34] HIMURO Y, TAKAI M, ISHIHARA K. Poly (vinylferrocene-co-2-hydroxyethyl methacrylate) mediator as immobilized enzyme membrane for the fabrication of amperometric glucose sensor [J]. Sensors and Actuators B: Chemical, 2009, 136(1): 122-127.
[35] DEUTSCHBAUER A M, WILLIAMS R M, CHU A M, et al. Parallel phenotypic analysis of sporulation and postgermination growth inSaccharomycescerevisiae[J]. Proceedings of the National Academy of Sciences, 2002, 99(24): 15 530-15 535.
[36] COLUCCIO A, BOGENGRUBER E, CONRAD M N, et al. Morphogenetic pathway of spore wall assembly in Saccharomyces cerevisiae [J]. Eukaryotic Cell, 2004, 3(6): 1 464-1 475.
[37] GOMEZ-ESQUER F, RODRGUEZ-PEA J M, DAZ G, et al. CRR1, a gene encoding a putative transglycosidase, is required for proper spore wall assembly inSaccharomycescerevisiae[J]. Microbiology, 2004, 150(10): 3 269-3 280.
[38] SARKAR P, FLORCZYK M, MCDONOUGH K, et al. SSP2, a sporulation-specific gene necessary for outer spore wall assembly in the yeastSaccharomycescerevisiae[J]. Molecular Genetics and Genomics, 2002, 267(3): 348-358.
[39] LI J, AGARWAL S, ROEDER G S. SSP2 and OSW1, two sporulation-specific genes involved in spore morphogenesis inSaccharomycescerevisiae[J]. Genetics, 2007, 175(1): 143-154.
[40] CHU S, DERISI J, EISEN M, et al. The transcriptional program of sporulation in budding yeast [J]. Science, 1998, 282(5389): 699-705.
[41] CHRISTODOULIDOU A, BOURIOTIS V, THIREOS G. Two sporulation-specific chitin deacetylase- encoding genes are required for the ascospore wall rigidity ofSaccharomycescerevisiae[J]. Journal of Biological Chemistry, 1996, 271(49): 31 420-31 425.
[42] MISHRA C, SEMINO C E, MCCREATH K J, et al. Cloning and expression of two chitin deacetylase genes ofSaccharomycescerevisiae[J]. Yeast, 1997, 13(4): 327-336.
[43] PAMMER M, BRIZA P, ELLINGER A, et al. DIT101 (CSD2, CAL1), a cell cycle‐regulated yeast gene required for synthesis of chitin in cell walls and chitosan in spore walls [J]. Yeast, 1992, 8(12): 1 089-1099.
[44] SANZ M, TRILLA O A, DURAN N, et al. Control of chitin synthesis through Shc1p, a functional homologue of Chs4p specifically induced during sporulation [J]. Molecular microbiology, 2002, 43(5): 1 183-1 195.
[45] NEUWALD A F. Barth syndrome may be due to an acyltransferase deficiency [J]. Current Biology, 1997, 7(8): R462-R6.
[46] BRIZA P, ELLINGER A, WINKLER G, et al. Characterization of a DL-dityrosine-containing macromolecule from yeast ascospore walls [J]. Journal of Biological Chemistry, 1990, 265(25): 15 118-15 123.
[47] BRIZA P, KALCHHAUSER H, PITTENAUER E, et al. N, N′ Bisformyl Dityrosine is an in vivo Precursor of the Yeast Ascospore Wall [J]. European journal of biochemistry, 1996, 239(1): 124-131.
[48] BRIZA P, ECKERSTORFER M, BREITENBACH M. The sporulation-specific enzymes encoded by the DIT1 and DIT2 genes catalyze a two-step reaction leading to a soluble LL-dityrosine-containing precursor of the yeast spore wall [J]. Proceedings of the National Academy of Sciences, 1994, 91(10): 4 524-4 528.
[49] FELDER T, BOGENGRUBER E, TENREIRO S, et al. Dtr1p, a multidrug resistance transporter of the major facilitator superfamily, plays an essential role in spore wall maturation inSaccharomycescerevisiae[J]. Eukaryotic cell, 2002, 1(5): 799-810.
[50] 李毅. 酿酒酵母孢子固定化酶催化D-葡萄糖合成D-阿洛酮糖的研究 [J]. 2015.
[51] CHOW C K, PALECEK S P. Enzyme encapsulation in permeabilizedSaccharomycescerevisiaecells [J]. Biotechnology progress, 2004, 20(2): 449-456.
[52] ROMERO C, SANCHEZ S, MANJON S, et al. Optimization of the pectinesterase/endo-D-polygalacturonase coimmobilization process [J]. Enzyme and microbial technology, 1989, 11(12): 837-843.
[53] MEN Y, ZHU Y, ZENG Y, et al. Co-expression of d-glucose isomerase and d-psicose 3-epimerase: development of an efficient one-step production of d-psicose [J]. Enzyme and microbial technology, 2014, 64:1-5.
[54] MASARU S, MITSUTAKA Y. A new enzymatic serum creatinine measurement based on an endogenous creatine-eliminating system [J]. Clinica chimica acta, 1984, 143(2): 147-155.
[55] FOSSATI P, PRENCIPE L, BERTI G. Enzymic creatinine assay: a new colorimetric method based on hydrogen peroxide measurement [J]. Clinical chemistry, 1983, 29(8): 1 494-1 496.
[56] COLUCCIO A E, RODRIGUEZ R K, KERNAN M J, et al. The yeast spore wall enables spores to survive passage through the digestive tract of Drosophila [J]. PLoS One, 2008, 3(8): e2873.
A novel way for enzyme immobilization——Saccharomycescerevisiaespore microencapsulated enzyme
KONG Jun, LI Zi-jie, Nakanishi Hideki, GAO Xiao-dong
(Key Laboratory of Carbohydrate Chemistry & Biotechnology Ministry of Education,Jiangnan University, Wuxi 214122, China)
The technologies for enzyme microencapsulation are making great progress due to development of enzyme immobilization. A novel way encapsulating enzymes “naturally” is introduced herein, i.e.,Saccharomycescerevisiaespore microencapsulated enzyme. Principles and example of this method s are exposed to readers. There are many advantages such as unnecessary preparation of core materials and wall materials, natural assembly of spore microcapsule is, without physicochemical treatment, no rely on expensive production equipments, strong stress resistance, easily manufacture, uniform and stable size. Finally, the application the yeast spore encapsulated enzyme in future was prospected herein.
immobilized enzyme; microencapsulation;Saccharomycescerevisiaespore encapsulated enzyme; “colocalization of multiple enzymes in one spore” skill
10.13995/j.cnki.11-1802/ts.201701043
博士研究生(高晓冬教授为通讯作者,E-mail: xdgao@jiangnan.edu.cn)。
2015年国家自然科学基金(21576118)
2016-05-31,改回日期:2016-06-27
