三重荧光定量PCR检测水产动物源细菌磺胺类耐药基因
2017-02-15黄国秋童桂香韦信贤陈静吴明媛黎小正
黄国秋,童桂香,韦信贤,陈静,吴明媛,黎小正
(广西水产科学研究院,广西 南宁 530021)
三重荧光定量PCR检测水产动物源细菌磺胺类耐药基因
黄国秋,童桂香,韦信贤,陈静,吴明媛,黎小正*
(广西水产科学研究院,广西 南宁 530021)
根据GenBank中细菌磺胺类耐药基因Sul1、Sul2和Sul3的保守序列设计3对特异性引物及相应TaqMan探针,以重组质粒标准品为模板对反应条件进行优化,建立一种可同时检测上述3种耐药基因的三重荧光定量PCR方法,并对该方法进行敏感性、特异性、重复性及临床菌株检测试验。结果表明:该方法定量范围为1×101~1×108拷贝/反应时,其标准曲线呈良好的线性关系;该方法灵敏度高(3个基因的最低质粒检测限均可达到10拷贝/反应)、特异性强(与其他病原菌及病毒无交叉反应)、重复性好(变异系数均小于2%);对临床菌株的检测结果与药敏试验结果的符合率达94.2%,且整个检测过程可在2 h内完成。所建立的可同时检测Sul1、Sul2和Sul3基因的三重荧光定量PCR方法可用于临床检测细菌的磺胺类耐药基因。
水产动物源细菌;磺胺类耐药基因;三重荧光定量PCR
细菌性疾病是中国水产养殖动物的主要病害,其造成水产养殖动物的损失占全部病害损失的50%以上[1–2]。在水产养殖过程中,使用各种抗菌素类药物抑制或者杀灭致病菌是防治水产养殖动物细菌性疾病最有效的手段。磺胺类药物作为主要的渔用抗菌药具有抗菌谱广、药效显著、价格低、易储存等优点,是目前各水产养殖场的必备药物。国内大量研究结果表明,水产动物源细菌对磺胺类药物已经产生了广泛的耐药性[3–5]。临床上细菌对磺胺类药物的抗性主要是由质粒上对磺胺类药物亲和力更低的新二氢叶酸合成酶(即具有抗性的DHPS)所介导,且耐药质粒可通过转化、易位、接合等方式在细菌之间进行转移,使 R–细菌从 R+细菌中获得这种抗性[6]。对磺胺类药物具有抗性的DHPS共有3种,其对应的编码基因分别被命名为Sul1、Sul2和Sul3[7]。对这3个基因的检测能反映细菌对磺胺类药物的耐药情况。荧光定量PCR具有灵敏度高、简便快速、可定量等优点,用于对耐药基因进行检测,能定量监测到细菌耐药性的强弱(已有对临床菌株耐药基因进行检测的研究[8])。目前,有关磺胺类药物耐药基因荧光定量 PCR检测方法的报道尚少。笔者结合多重PCR技术,针对磺胺类药物3个耐药基因Sul1、Sul2和Sul3分别设计引物及相应TaqMan探针,建立快速、简便、准确的三重荧光定量PCR检测方法,旨在为磺胺类药物耐药性的快速检测和耐药基因的调查提供一种全新的技术手段。
1 材料与方法
1.1 材料与试剂
待检菌株共72株,2013—2015年从广西水产动物或水产品中分离得到,其中沙门氏菌 13株,志贺氏菌7株,嗜水气单胞菌36株,温和气单胞菌6株,爱德华氏菌8株,肺炎克雷伯菌2株。
Sul1、Sul2和Sul3基因阳性菌株为肺炎克雷伯菌GX120222分离株,经PCR鉴定携带Sul1、Sul2和Sul3基因。
草鱼呼肠孤病毒(GCRV)、白斑综合征病毒(WSSV)、传染性皮下与造血组织坏死病毒(IHHNV)均由广西渔业病害防治环境监测和质量检验中心鉴定与保存。
磺胺嘧啶(Sulfadiazine,SD)和磺胺甲基异恶唑(Sulfamethoxazole,SMZ) 购于中国兽药生物制品监察所,含量均达99.5%以上;营养琼脂、营养肉汤培养基购自杭州滨和微生物试剂有限责任公司;2×Taq PCR MasterMix(含Taq DNA聚合酶、dNTPs、MgCl2、反应缓冲液)、琼脂糖凝胶回收试剂盒和质粒小量提取试剂盒购自北京艾德莱生物科技有限公司;DL1 000 DNA Marker、pMD18– T、DH5α感受态细胞、Premix Ex Taq(Probe qPCR)均购自宝生物工程(大连)有限公司;氨苄青霉素购自 Promega公司;琼脂糖购自英韦创津生物科技有限公司;其他试剂均为国产、分析纯。
1.2 方法
1.2.1 水产动物源临床菌株对磺胺类药物的敏感性测定
采用琼脂平板纸片扩散法(K–B法)分别测定72株水产动物源临床菌株对SD和SMZ的抑菌圈直径。药敏纸片的制备参见文献[9],药敏结果判读标准见文献[10]。
1.2.2 引物及探针设计
用 DNAStar软件对多种属细菌的 Sul1、Sul2和Sul3基因进行比较,采用Primer Express 3.0和Oligo 6.0软件,在保守序列区域按照多重荧光定量PCR引物设计原则,共设计合成3套引物及探针,其中探针5′端依次标记荧光报告基团FAM、HEX和CY5,3′端均标记荧光淬灭基团 BHQ1,引物和探针均由上海基康生物科技有限公司合成。各引物及探针序列见表1。

表1 引物及探针信息Table 1 Information about the primers and probes
1.2.3 细菌DNA的提取及重组质粒标准品的制备
将试验菌株的单菌落分别接种于营养肉汤中,36 ℃培养18 h;取2 mL菌悬液,采用煮沸法[11]提取细菌总DNA,于-20 ℃保存备用。
以菌株GX120222的DNA为模板,用Sul1、Sul2和Sul3基因引物扩增各自特异性目的片段,用DNA回收试剂盒按说明书分别纯化回收相应目的片段,然后将回收的目的片段与 pMD18–T载体连接。连接产物分别转化 E.coli DH5α感受态细胞,通过菌落PCR筛选阳性克隆,并测序鉴定。取测序结果正确的阳性克隆菌液5 mL,按照质粒小量提取试剂盒说明书提取质粒,并将提取的质粒分别命名为pMD18–T– Sul1、pMD18–T–Sul2和pMD18–T–Sul3。用GeneQuant核酸蛋白分析仪分别测定重组质粒的浓度。根据阿弗加德罗常数将浓度转换为拷贝数。稀释质粒DNA为1.0×101~1.0×108拷贝/μL。以该8个梯度质粒DNA为标准品,-20 ℃保存,备用。
1.2.4 三重荧光定量PCR反应条件的优化
用1.0×106拷贝/μL的标准品DNA作为模板,将引物终浓度0.1~0.8 μmol/L和探针终浓度0.1~0.5 μmol/L的不同组合(采用矩阵法进行组合)进行荧光定量 PCR,获得最低临界循环数(Ct)和较高相对荧光强度增加值(ΔRn)时的引物和探针浓度,作为优化单个靶基因的检测体系。在此基础上,以1.0×106拷贝/μL的3种标准品等比例混合液为模板进行三重荧光定量 PCR,逐步改变各引物(单个靶基因最佳引物浓度±0.2 μmol/L)与探针(单个靶基因最佳探针浓度±0.2 μmol/L)的浓度以达到最佳的扩增效果。
退火温度的优化:参考各引物的Tm值,在58~62 ℃以梯度1 ℃进行多重荧光定量PCR扩增,根据Ct值及扩增曲线形状确定最佳退火温度。
Premix Ex Taq Mix的优化:在反应体系中使Premix Ex Taq Mix添加量为0.8×、0.9×、1×、1.1×、1.2×、1.3×、1.4×、1.5×,根据Ct值及扩增曲线形状确定最佳Premix Ex Taq Mix反应浓度。
1.2.5 标准曲线的建立及敏感性、特异性、重复性试验
以10倍梯度稀释的Sul1、Sul2和Sul3基因标准品等比例混合 DNA(1.0×101~1.0×108拷贝/μL)为模板,运用优化后的多重荧光定量PCR方法进行扩增,建立关于模板起始拷贝数对数和Ct值的标准方程,并根据Ct值来判断该方法的敏感性。
以阳性菌株及部分磺胺类药物敏感菌、GCRV、WSSV和IHHNV的核酸样品为模板,用优化后的三重荧光定量PCR方法进行检测,观察结果。
取108、107、106拷贝/μL 3个梯度的各标准品DNA进行三重荧光定量PCR反应,每个浓度3次重复,计算Ct值的变异系数。
1.3 三重荧光定量PCR方法的临床应用
用三重荧光定量PCR方法检测72株临床菌株的Sul1、Sul2和Sul3基因,将出现扩增曲线的基因判为阳性,并将扩增结果与药敏试验结果进行比较。
2 结果与分析
2.1 药敏试验结果
药敏试验结果表明,72株水产动物源临床菌株对 SD和 SMZ的耐药率分别为 68.1%(49/72)和62.5% (45/72),对2种磺胺类药物的交叉耐药率为80.8%(42/72)。72株细菌中有16株对SD或SMZ中敏,仅4株对SD或SMZ敏感,这表明水产动物源细菌对磺胺类药物的耐药现象非常普遍。
2.2 重组质粒标准品的制备结果
Sul1、Sul2和Sul3基因扩增产物经琼脂糖凝胶电泳(图1),扩增出的条带大小分别为67、76、83 bp,均与预期大小一致。目的片段回收纯化后克隆入pMD18–T载体,重组质粒经PCR测序鉴定,所得序列与GenBank公布的同种细菌磺胺类药物耐药基因Sul1、Sul2和Sul3的同源性均为100%。取阳性克隆菌液提取质粒,测定其浓度并转换为拷贝数,可得pMD18–T–Sul1、pMD18–T–Sul2和pMD18–T–Sul3的浓度分别为3.72×109、4.56×109、6.32×109拷贝/μL,均能满足标准品的要求。
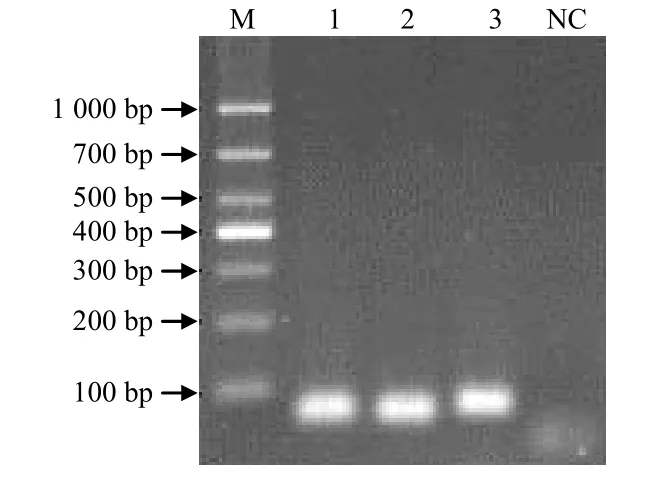
图1 Sul1和Sul2及Sul3基因的PCR扩增结果Fig.1 PCR amplifcation of gene Sul1, Sul2 and Sul3 from sulfonamides–resistance bacteria
2.3 三重荧光定量PCR反应条件的优化结果
对三重荧光定量PCR各条件进行优化后,最终确定反应体系为:Premix Ex Taq (2×) 18 μL,ROX Reference Dye 0.5Ⅱ μL,Sul1上、下游引物(20 μmol/L)均为0.9 μL,Sul1探针用量(10 μmol/L)为0.9 μL,Sul2和Sul3的上、下游引物(20 μmol/L)均为0.6 μL,Sul2和Sul3探针用量(10 μmol/L)均为0.6 μL,模板3 μL,用灭菌ddH2O补足至30 μL。反应条件:95 ℃预变性30 s;95 ℃变性5 s,60 ℃退火并延伸45 s,扩增40个循环;在每个循环60 ℃结束时收集荧光信号。
2.4 标准曲线的建立
以10倍梯度稀释的各标准品等比例混合DNA (1×101~1×108拷贝/μL)为模板,使用优化后的三重荧光定量PCR进行扩增,扩增曲线均呈明显的“S”型,而阴性对照和空白对照均没有出现任何扩增(图2)。利用仪器自带的数据分析软件进行分析,横坐标(X) 代表起始模板拷贝数的对数,纵坐标(Y) 代表Ct值,得到3条标准曲线(图3)。
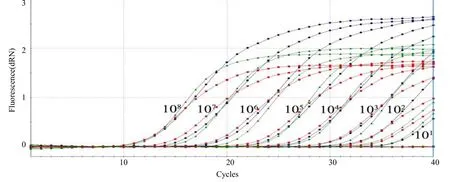
图2 Sul1和Sul2及Sul3基因的三重荧光定量PCR扩增曲线Fig.2 Amplification plot of the triple real–time qPCR from Sul1, Sul2 and Sul3
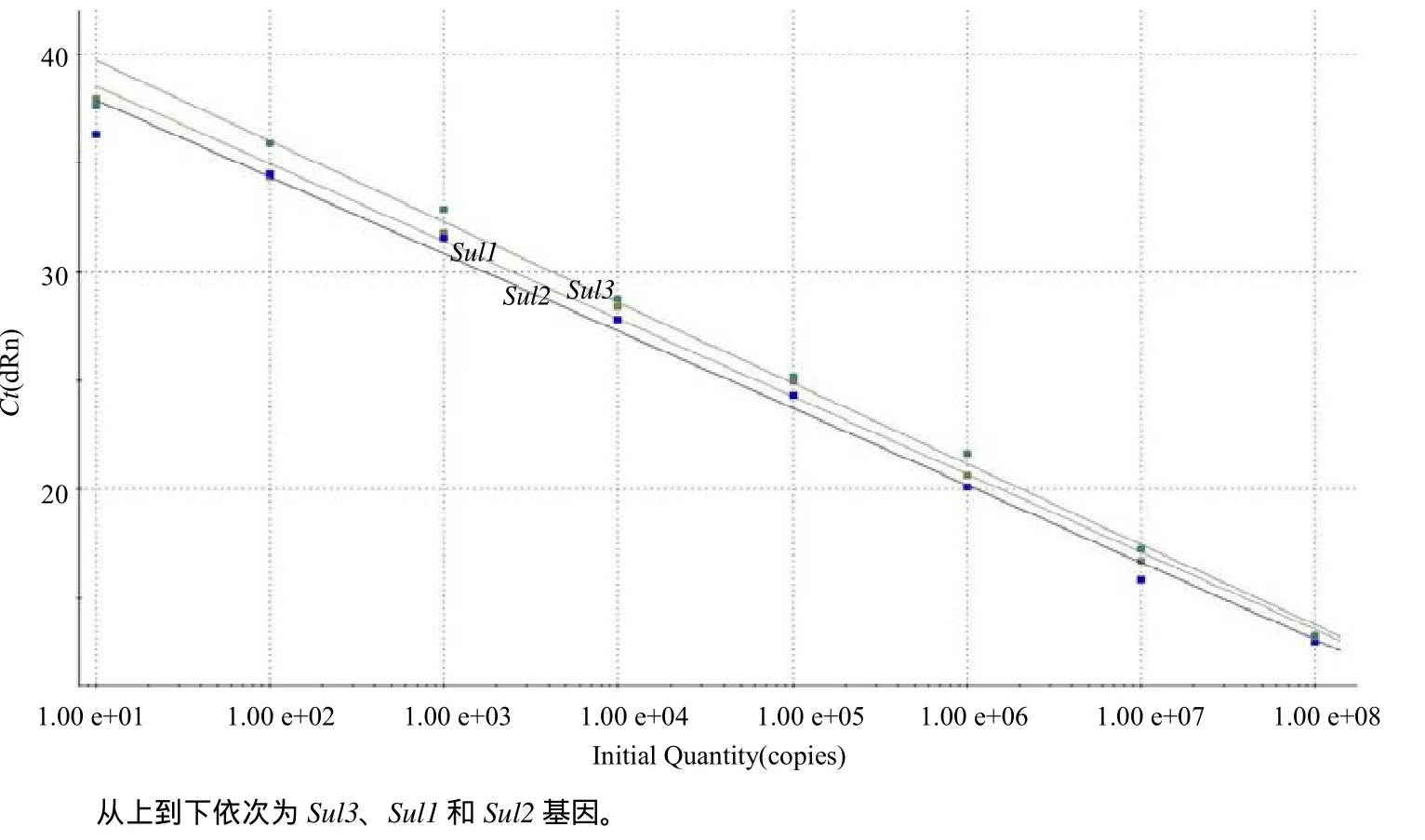
图3 三重荧光定量PCR检测Sul1和Sul2及Sul3基因的标准曲线Fig.3 Standard curves of the triple real–time qPCR from Sul1, Sul2 and Sul3
Sul1的标准曲线方程为Y= –3.572lg X+42.12,扩增效率(Eff.)和相关系数方值 (RSq)分别为 90.5%和0.996;Sul2的标准曲线方程为Y= –3.551lg X+ 41.49,Eff.和RSq分别为91.3%和0.993;Sul3的标准曲线方程为Y = –3.712 lg X+3.46,Eff.和RSq分别为86.0%和0.994。由以上标准曲线方程可见,起始模板拷贝数的对数与 Ct值均呈良好的线性关系,能够准确地反映目的产物的扩增。
2.5 三重荧光定量PCR的敏感性和特异性及重复性
2.5.1 敏感性
经检测,重组质粒 pMD18–T–Sul1模板为 10拷贝/反应时仍可检测到荧光信号值,3个重复的Ct值分别为 37.66、37.80、38.22;重组质粒pMD18–T–Sul2模板为10拷贝数/反应时也可检测到荧光信号值,3个重复的 Ct值分别为 37.34、37.54、38.06;重组质粒pMD18–T–Sul3模板为10拷贝数/反应时也可检测到荧光信号值,3个重复的Ct值分别为36.32、36.33和36.38。以上结果表明,本研究中建立的三重荧光定量PCR检测Sul1、Sul2和Sul3基因的灵敏度均可达到10拷贝/反应,与常规PCR检测灵敏度104拷贝/反应相比[12],敏感性显著提高(图4~6)。
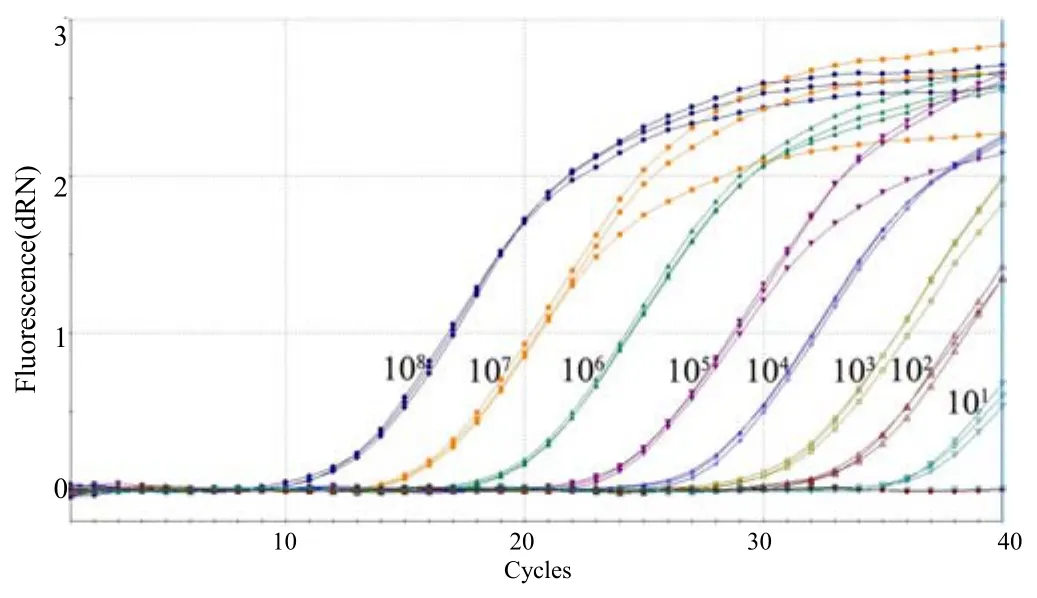
图4 Sul1基因敏感性的三重荧光定量PCR检测结果Fig.4 Sensitivity of the triple real–time qPCR in Sul1detection
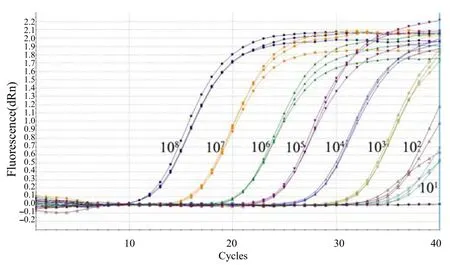
图5 Sul2基因敏感性的三重荧光定量PCR检测结果Fig.5 Sensitivity of the triple real–time qPCR in Sul2 detection
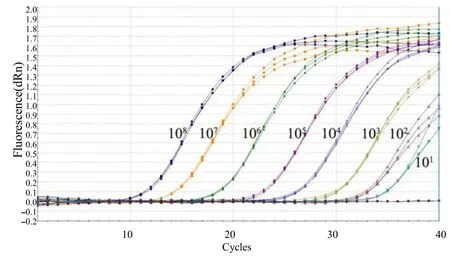
图6 Sul3基因敏感性的三重荧光定量PCR检测结果Fig.6 Sensitivity of the triple real–time qPCR in Sul3 detection
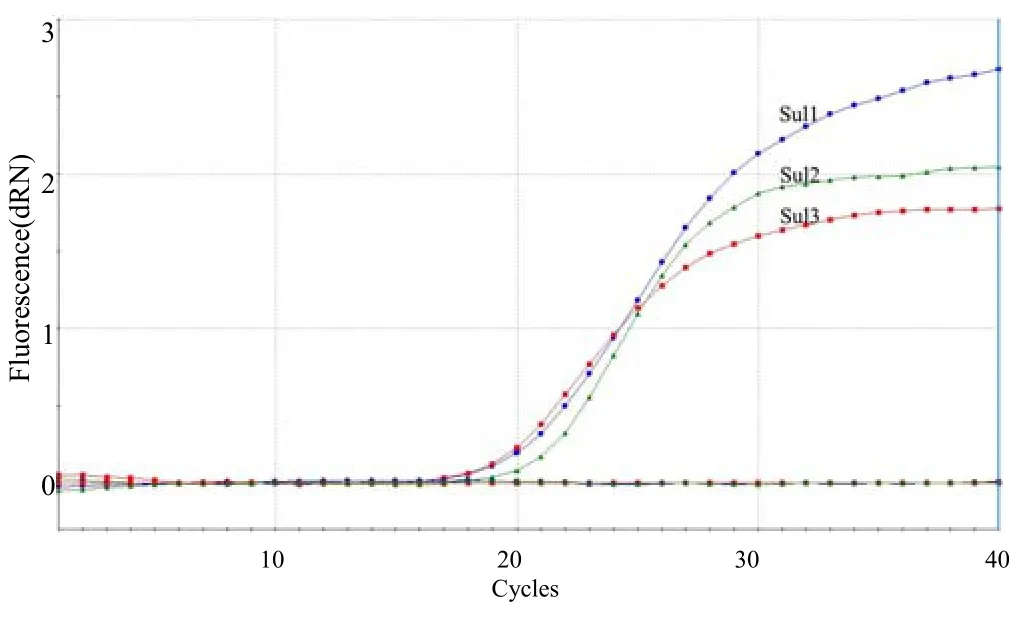
图7 三重荧光定量PCR的特异性检测结果Fig.7 Specificity evaluation for the triple real–time qPCR
2.5.2 特异性
用建立的三重荧光定量 PCR对阳性菌株及磺胺类药物敏感菌、GCRV、WSSV和IHHNV的核酸进行检测的结果(图7)表明,只有阳性菌株出现扩增曲线,其他菌株及病毒均未出现扩增曲线,均为阴性,表明本研究中建立的三重荧光定量 PCR对Sul1、Sul2和Sul3基因的检测具有很好的特异性。
2.5.3 重复性
108、107、106拷贝/μL各标准品DNA三重荧光定量PCR重复性试验Ct值的变异系数(CV)见表2。结果表明,Ct值的变异系数为0.30%~1.01%,说明建立的三重荧光定量PCR具有良好的重复性。
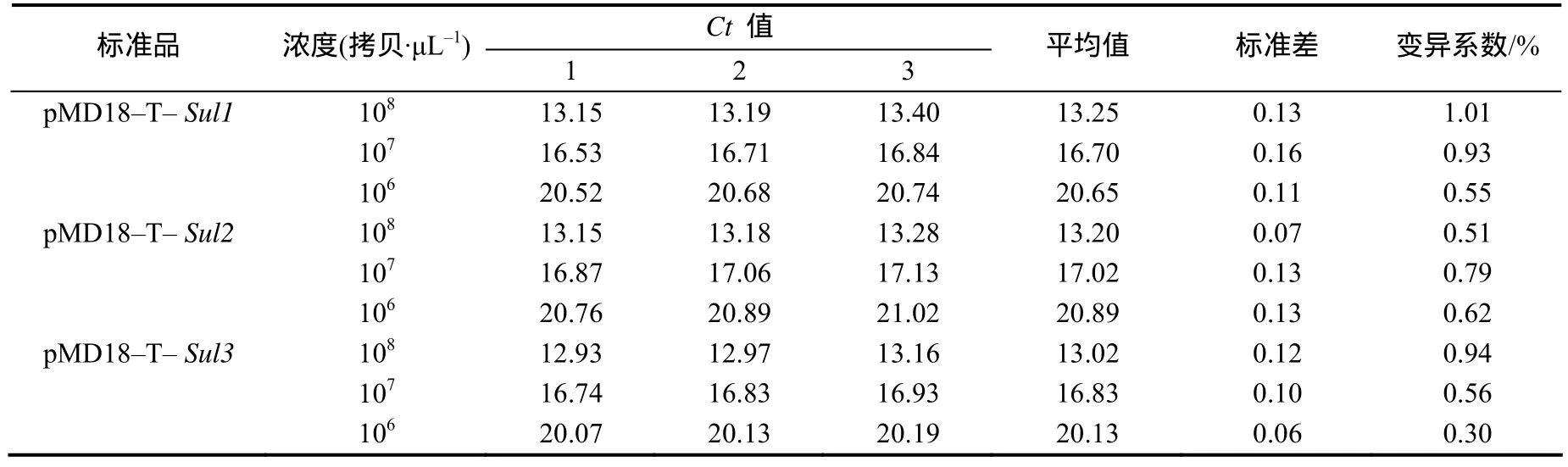
表2 三重荧光定量PCR重复试验结果Table 2 Results of repeatability test for triple real–time qPCR
2.6 三重荧光定量PCR对临床菌株的检测结果
用所建立的三重荧光定量PCR方法对72株临床菌株进行检测,结果表明,耐SD或SMZ的52株菌株中有49株检测出了Sul1或Sul2或Sul3基因,与药敏试验相比,耐药基因的符合率为94.2%,其中,有43株含有Sul1基因,有26株含Sul2基因,有4株含有Sul3基因,有23株含有2种或2种以上的基因。16株中度敏感菌中有13株检出耐药基因,其中,有11株含有Sul1基因,有7株含Sul2基因,有1株含有Sul3基因,有6株含有2种或2种以上的基因。在4株敏感菌中没有检测出任何耐药基因,与药敏试验相比符合率为 100%。检出耐药基因的菌株,其菌液耐药基因浓度为 109~106拷贝/mL。
3 结论与讨论
72株水产动物源临床菌株对磺胺类药敏感性的测定结果表明,72株菌株对SD和SMZ的耐药率分别为68.1%和62.5%,对2种磺胺类药物的交叉耐药率为80.8%,仅有16株细菌对SD或SMZ中敏,有4株对SD或SMZ敏感,其耐药率比广西罗非鱼源致病菌对磺胺类的耐药率(53.3%)[13]高,说明广西水产动物源菌株对磺胺类药物的耐药现象非常严重,且有不断加重的趋势。本试验中的耐药率与其他畜禽动物源菌株对磺胺类的耐药率[14]相近,说明磺胺类药物在使用过程中极易产生耐药性,这与用药频率和用药强度有关,还与细菌对磺胺类药产生耐药性的分子机制密切相关,因为细菌对磺胺类药的耐药基因大部分位于质粒或转座子上,可以容易地在同种属或不同种属的细菌间从一个质粒转移到另一个质粒或染色体上[15–16]。病原菌对磺胺类药物容易产生交叉耐药性[17]。本试验结果印证了这一结论。
中国目前临床上对细菌磺胺类药物耐药性的检测主要采用传统的药敏试验方法。这些方法耗时长,准确性差,灵敏度低,稳定性不强,已不能满足临床上快速、准确筛选敏感药物的需要。鉴于此,国内已有学者将 PCR技术应用于磺胺类药耐药性的快速检测和耐药基因的分子流行病学调查[7,12]。实时荧光定量PCR不仅有常规PCR技术扩增效率高的特点,还具有探针的高度特异性、光谱技术的高敏感性和精确性等特点[18],且能定量,不易受到污染,被广泛运用于医疗、药物研究及病原菌–检测[19–22]。多重PCR技术能够在一个PCR体系中同时检测多个目标基因,其快速、高效及低成本等特点具有十分广泛的应用前景。本研究中根据GenBank中Sul1、Sul2和Sul3基因的保守序列设计特异性引物及相应TaqMan探针,并对引物探针浓度配比、反应体系及反应条件等进行优化,建立了可同时检测上述3种耐药基因的三重荧光定量PCR方法。敏感性、特异性和重复性测试试验结果表明,该方法定量范围宽,检测模板范围为1×101~1×108拷贝/反应时,其标准曲线呈良好的线性关系;该方法灵敏度高,Sul1、Sul2和Sul3的最低质粒检测限均可达到10拷贝/反应;该方法特异性强,与其他病原菌及病毒无交叉反应;该方法重复性好,变异系数均小于2%;整个检测过程可在2 h内完成,符合临床上对菌株进行磺胺类药物耐药性快速检测的需求,具有较高的应用价值。
用本研究中建立的三重荧光定量PCR方法检测72株水产动物源临床菌株中的耐药基因Sul1、Sul2和Sul3,结果表明,耐SD或SMZ的52株菌株中有49株检测出了Sul1或Sul2或Sul3基因,与药敏试验结果的符合率为94.2%,具有较高的准确性;在52株耐SD或SMZ的菌株中,有45株(占86.5%)检测到了Sul1或(和)Sul2基因,仅有4株(占7.7%)检测到了Sul3基因,说明水产动物源菌株以Sul1和Sul2基因为主(91.8%),与畜禽动物源菌株(2/3的Sul1和Sul2基因、1/3的Sul3基因)相差较大[23]。本研究中检出耐药基因的菌株,其耐药基因的拷贝数都比较高,菌液浓度均为109~106拷贝/mL(约108个细菌),说明耐药基因在单个细菌中可单拷贝/多拷贝存在,但耐药基因拷贝数与耐药程度并不一定呈正相关,这可能是因为该耐药基因本身变异或缺失引起功能减弱或者功能丧失所致,也可能是该耐药基因在菌株中受到抑制而没有得到表达所致。可见,细菌耐药基因表达与耐药表型和耐药程度的关系值得进一步研究。此外,本研究中建立的方法可检出包括沙门氏菌、志贺氏菌、嗜水气单胞菌、爱德华氏菌和肺炎克雷伯菌等多种属细菌的磺胺类药耐药基因,说明该方法可广泛应用,同时说明Sul1、Sul2和Sul3基因在不同种属的细菌中非常保守。
总之,本研究中建立的同时检测Sul1、Sul2和Sul3基因的三重荧光定量PCR方法不仅具有快速、简便、灵敏、准确、定量、重复性好等优点,还能区分不同的耐药基因,可为临床细菌磺胺类药耐药基因的快速检测和流行病学调查提供快速简便、准确高效的检测手段。
[1]李立华.中国水产养殖病害控制技术现状与发展趋势[J].黑龙江科技信息,2016(8):274.DOI:10.3969/j.issn.1673–1328.2016.08.254.
[2]吴淑勤,王亚军.中国水产养殖病害控制技术现状与发展趋势[J].中国水产,2010(8):9–10.DOI:10.3969/j.issn.1002–6681.2010.08.003.
[3]王小亮,殷守仁,赵文,等.鱼类病原菌耐药性研究进展[J].北京农业,2011(27):35–37.DOI:10.3969/j.issn.1000–6966.2011.27.019.
[4]王玉堂,陈昌福,吕永辉.水产养殖动物致病菌耐药性检测数据实际作用[J].中国水产,2013(10):57–61.DOI:10.3969/j.issn.1002–6681.2013.10.029.
[5]乔毅.江苏省沿海地区水产养殖主要致病菌耐药性研究[D].上海:上海海洋大学,2015.
[6]DAVIES J,SMITH D I.Plasmid–determined resistance to antimicrobial agents[J].Annu Rev Microbiol,1978,32:469–518.DOI:10.1146/annurev.mi.32.100178.002345.
[7]周万蓉.细菌对磺胺类药物耐药基因三重PCR检测试剂盒研制与应用[D].成都:四川农业大学,2007.
[8]吕虹,刘志红,刘琪琦,等.复合探针实时荧光 PCR检测细菌耐药基因 blaNDM–1方法的建立[J].生物技术通讯,2012,23(3):411–414.DOI:10.3969/j.issn.1009–0002.2012.03.024.
[9]戴自英.实用抗菌药物学[M].上海:上海科学技术出版社,1998.
[10]Clinical and Laboratory Standards Institute (CLSI).Performance standards for antimicrobial susceptibility testing; twentith informational supplement[S].M100–S25,USA,2015.
[11]冯广达,陈美标,羊宋贞,等.用于PCR扩增的细菌DNA提取方法比较[J].华南农业大学学报,2013,34(3):439–442.
[12]羊云飞,王红宁,谭雪梅,等.二重PCR检测猪、鸡源致病性大肠杆菌、沙门氏菌磺胺类耐药基因(Sul1、Sul2、Sul3)的研究[J].畜牧兽医学报,2007,38(10):1088–1092.DOI:10.3321/j.issn:0366–6964.2007.10.014.
[13]黎小正,童桂香,吴祥庆,等.2011—2012年广西罗非鱼食源性致病菌及其药敏特性调查[J].南方农业学报,2013,44(11):1914–1918.DOI:10.3969/j:issn.2095–1191.2013.11.1914.
[14]陈杖榴,吴聪明,蒋红霞,等.兽用抗菌药物耐药性研究[J].四川生理科学杂志,2003(25):120–123.
[15]BOYD D,CLOECKAERT A,CHASLUS–DANCLA E,et al.Characterization of variant Salmonella genomic island 1 multidrug resistance regions from serovars Typhimurium DT104 and Agona[J].Antimicrob Agents Chemother,2002,46(6): 1714–1722.DOI:10.1128/aac.46.6.1714– 1722.2002.
[16]SUNDSTRÖM L,RÅDSTRÖM P,SWEDBERG G,et al.Site–specific recombination promotes linkage between trimethoprim–and sulfonamide resistance genes: sequence characterization of dhfrV and sulI and a recombination active locus of Tn21[J].Mol Gen Genet,1988,213(2/3):191–201.DOI:10.1007/bf00339581.
[17]金有豫.药理学[M].北京:人民卫生出版社,2001.
[18]周晓丽,朱国坡,李雪华,等.实时荧光定量PCR技术原理与应用[J].中国畜牧兽医,2010,37(2):87–89.
[19]周成江,周立社,吴刚,等.实时荧光定量PCR的研究进展及其应用[J].包头医学院学报,2007,23(2):204–207.DOI:10.3969/j.issn.1006–740X.2007.02.067.
[20]曾东,肖拉,倪学勤.建鲤IL–1β和IL–8基因实时荧光定量RT–PCR检测方法的建立[J].湖南农业大学学报(自然科学版),2014,40(6):627–632.DOI:10.13331/j.cnki.jhau.2014.06.013.
[21]韩熹,朱莉莎,姚建垣.实时荧光定量PCR分析技术的应用[J].生命科学仪器,2005,3(2):51–52.DOI:10.3969/j.issn.1671–7929.2005.02.016.
[22]宋琼,任佐华,杨华,等.应用实时荧光定量PCR技术检测土壤中根肿病菌休眠孢子[J].湖南农业大学学报(自然科学版),2015,41(6):631–635.DOI:10.13331/j.cnki.jhau.2015.06.010.
[23]PERRETEN V,BOERLIN P.A new sulfonamide resistance gene (sul3) in Escherichia coli is widespread in the pig population of Switzerland[J].Antimicrob Agents Chemother,2003,47(3):1169–1172.DOI:10.1128/aac.47.3.1169–1172.2003.
责任编辑:王赛群
英文编辑:王 库
Detection sulfonamides–resistance genes of pathogenic bacteria derived from aquatic animals using triple real–time PCR
HUANG Guoqiu, TONG Guixiang, WEI Xinxian, CHEN Jing, WU Mingyuan, LI Xiaozheng*
(Guangxi Academy of Fishery Science, Nanning 530021, China)
Triple TaqMan real–time PCR method was developed to simultaneously detect sulfonamides–resistance genes Sul1, Sul2 and Sul3, the three pathogenic bacteria in aquatic animals in the study.Three pairs of specific primers and fluorogenic–labeled probes were designed and synthesized in accordance with the above target genes using software Primer Express 3.0.The reaction system and procedure were optimized, as well as the detection sensitivity, specificity, repeatability and clinical application of the method.Results showed that the method had a wide quantitative range from 1×101to 1×108copies per reaction, which presented a good linear relationship in its standard curve.The triple real–time PCR method had a high specificity in detecting mixed DNA of Sul1, Sul2 and Sul3, but not those DNA from other bacteria and viruses; it also had a high sensitivity to the detection limit low to 10 copies per reaction for the purifed recombinant plasmids of Sul1, Sul2 and Sul3.The variation coeffcients of the established method were less than 2%.Sulfonamides–resistance genes of 72 clinical isolates were analyzed using triple real–time PCR method and they were compared to drug sensitive test, the coincidence rate could reach to 94.2%.The entire detection could be completed within 2 h.In conclusion, the triple TaqMan real–time PCR method developed in the present study could be applied to the rapid detection and molecular epidemiology survey in sulfonamides–resistance genes Sul1, Sul2 and Sul3 of clinical pathogenic strains isolated from aquatic animals.
pathogenic bacteria from aquatic animals; sulfonamides resistant genes; triple TaqMan real–time PCR
S917.1
:A
:1007-1032(2017)01-0064-07
2016–04–22
2016–12–06
广西水产畜牧科技项目(桂渔牧科201528044);广西科技攻关项目(桂科攻1355010–3)
黄国秋(1981—),女,广西南宁人,工程师,主要从事水产品微生物检验研究,33958119@qq.com;*通信作者,黎小正,研究员,主要从事水产品检验检疫和渔业环境保护等方面的研究,lixz@tom.com
