溃疡性结肠炎发病、疾病活动和生物治疗疗效相关基因CRELD2的生物信息学分析*
2017-02-11陶文惠马敏星
陶文惠 王 帆 林 雪 马敏星 赵 秋 李 瑾
武汉大学中南医院消化内科 湖北省肠病医学临床研究中心/肠病湖北省重点实验室(430071)
·论 著·
溃疡性结肠炎发病、疾病活动和生物治疗疗效相关基因CRELD2的生物信息学分析*
陶文惠 王 帆 林 雪 马敏星 赵 秋 李 瑾#
武汉大学中南医院消化内科 湖北省肠病医学临床研究中心/肠病湖北省重点实验室(430071)
背景:应用生物信息学方法能有效挖掘基因芯片数据,并对单基因功能进行预测分析。目的:对与溃疡性结肠炎(UC)发病、疾病活动和生物治疗疗效相关的CRELD2基因进行生物信息学分析,为深入研究该基因的生物学功能及其参与UC发生、发展的分子机制提供理论依据。方法:从基因表达综合数据库(GEO数据库)中下载UC发病、疾病活动和生物治疗疗效相关基因芯片数据,应用BRB-ArrayTools、ProtParam、ELM、SignalP 4.1、PBIL-IBCP Lyon Gerland、GO、STRING等生物信息学工具进行数据挖掘和分析。结果:交叉分析显示,在UC发病、疾病活动中进行性表达增高,同时在英夫利昔单抗治疗有效后表达明显降低的基因共有4个,分别为CDC25B、CRELD2、IL1RN、PITPNC1,其中CRELD2基因功能未知。生物信息学分析显示,人CRELD2基因位于染色体22q13.33,编码由402个氨基酸残基组成的分泌型蛋白。CRELD2蛋白含有多个表皮生长因子(EGF)样结构域,主要分布于高尔基体、内质网和细胞外,主要参与钙离子结合和蛋白结合,与CHRNA4、CHRNB2、RHBDD3等蛋白之间存在相互作用。结论:CRELD2可能具有类似EGF的功能,并可能通过直接或间接参与免疫细胞的调控对UC发病、疾病活动产生影响,有望成为UC诊断、疾病活动性评估、疗效评估的新指标和新的治疗靶点。
CRELD2基因; 结肠炎,溃疡性; 生物信息学; 微阵列分析
溃疡性结肠炎(ulcerative colitis, UC)是一种慢性非特异性肠道炎症性疾病,主要症状为持续或反复发作的腹泻、黏液脓血便伴腹痛、里急后重感。5-氨基水杨酸、糖皮质激素、免疫抑制剂是UC的主要治疗药物,但长期使用需关注药物不良反应,且部分患者疗效欠佳[1]。近年来,随着英夫利昔单抗(infliximab, IFX)、阿达木单抗(adalimumab)等肿瘤坏死因子抗体类生物制剂的应用,UC治疗的临床疗效明显改善。
UC的发病机制目前仍不清楚,可能是遗传易感人群中环境因素、微生物因素和肠道免疫系统间相互作用的结果,体内多种免疫相关基因异常参与了这一过程。寻找与UC发病、疾病活动和生物治疗疗效均相关的基因,对揭示UC发病机制以及寻找新的药物治疗靶点具有重要意义。基因芯片作为一种高效、大规模获取生物学信息的技术,能鉴别不同组织间的差异表达基因[2]。生物信息学(bioinformatics)是在生命科学研究中,以计算机为工具对生物学信息进行储存、检索和分析的科学。应用生物信息学方法能有效挖掘基因芯片数据,获取生物内在信息,并对单基因功能进行预测分析。本研究利用基因表达综合数据库(Gene Expression Omnibus, GEO数据库)中的基因芯片原始数据进行差异表达基因分析,获取同时与UC发病、疾病活动和生物治疗疗效相关的共性基因,并对其中功能未知的基因CRELD2进行初步生物信息学分析,以期为深入研究该基因的生物学功能及其参与UC发生、发展的分子机制提供理论依据。
资料与方法
一、主要资料
正常人、UC患者、UC患者IFX治疗前后的结肠黏膜表达谱芯片数据来源于NCBI GEO数据库(https://www.ncbi.nlm.nih.gov/gds/),登录号为GSE38713和GSE16879,分别由Planell等和Arijs等提交。GSE38713中包括13例正常人结肠黏膜、15例活动期UC炎症肠黏膜和7例活动期UC正常肠黏膜;GSE16879中包括24例活动期UC IFX治疗前后的肠黏膜,其中8例治疗有效,16例治疗无效。两项研究中的结肠黏膜标本均于结肠镜下取材,经RNA 提取、逆转录、荧光标记等步骤处理后,与Affymetrix人类基因组U133 Plus 2.0芯片(GPL570芯片平台)杂交,经图像扫描、数据标准化等处理后以CEL格式上传至GEO数据库。
二、方法
1. 数据过滤和标准化:从GEO数据库中下载GSE38713和GSE16879的原始数据。为使数据便于分析,同时获得质量高、表达信号强的基因,本研究采用justRMA算法对芯片数据进行预处理,以中位数的方法对数据进行过滤和标准化。基因过滤要求:①两组样本的基因表达中位数发生2倍及以上变化,且20%及以上样本可见此种变化;②基因表达数据缺失的样本不超过50%。
2. 差异表达基因筛选:应用BRB-ArrayTools软件(https://brb.nci.nih.gov/BRB-ArrayTools)[3]中的 Class Comparison工具,分别对GSE38713和GSE16879中的各组数据进行比较,IFX治疗前后数据的比较采用配对t检验,其他组间比较采用两独立样本t检验,P<0.001判定为差异表达基因。最后以Excel软件筛选出在UC发病、疾病活动中进行性表达增高(或降低),同时在IFX治疗有效后表达明显降低(或增高)的基因,并对其中功能未知的基因进行初步生物信息学分析。
3. 基因定位和序列分析:在NCBI Gene数据库(https://www.ncbi.nlm.nih.gov/gene/)中查找人CRELD2基因,获得其基因组定位、基因序列、mRNA序列和编码蛋白氨基酸序列。
4. 基因编码蛋白的理化性质:应用ProtParam工具(http://web.expasy.org/protparam/)分析CRELD2基因编码蛋白的氨基酸数量、相对分子质量、理论等电点、疏水性等理化性质。
5. 一级结构预测分析:应用ELM工具(http://elm.eu.org/)预测CRELD2蛋白一级结构和结构域中的修饰位点和模体(蛋白质中具有特定空间构象和特定功能的二级结构)。
6. 信号肽、跨膜区域和磷酸化位点预测分析:应用SignalP 4.1软件(http://www.cbs.dtu.dk/services/SignalP/)对CRELD2蛋白序列进行信号肽分析,应用TMHMM 2.0软件(http://www.cbs.dtu.dk/services/TMHMM/)预测蛋白跨膜区域,应用NetPhos 2.0软件(http://www.cbs.dtu.dk/services/NetPhos/)预测蛋白磷酸化位点。
7. 二级结构预测分析:应用PBIL-IBCP Lyon Gerland信息库中的HNN二级结构预测方法(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_hnn.html)对CRELD2蛋白序列进行二级结构预测。
8. 基因本体和通路分析:应用GO工具(Gene Ontology Consortium, http://www.geneontology.org/)对CRELD2基因进行分子功能、细胞内定位和生物学过程分析,应用KEGG工具(Kyoto Encyclo-pedia of Genes and Genomes, http://www.genome.jp/kegg/)对其进行信号通路分析。
9. 蛋白-蛋白相互作用网络分析:应用STRING软件(http://string-db.org/)对CRELD2蛋白进行蛋白-蛋白相互作用网络分析。
结 果
一、差异表达基因筛选
经数据过滤和标准化处理后,GSE38713和GSE16879中分别有2 012个和2 134个基因通过过滤标准。剔除重复和非特异性探针后发现,与正常人结肠黏膜相比,活动期UC炎症肠黏膜中有860个差异表达基因(563个上调,297个下调),活动期UC正常肠黏膜中有103个差异表达基因(32个上调,71个下调)。活动期UC炎症肠黏膜与正常肠黏膜的比较显示,614个基因表达存在差异,其中炎症部位498个基因表达上调,116个下调。IFX治疗有效的活动期UC,治疗后57个基因表达上调,176个下调;与IFX治疗无效者相比,治疗有效者77个基因表达上调,155个下调。交叉分析显示,在UC发病、疾病活动中进行性表达增高,同时在IFX治疗有 效后表达明显降低的基因共有4个,分别为CDC25B、CRELD2、IL1RN、PITPNC1(表1)。其中CDC25B、IL1RN、PITPNC1基因功能及其与UC的关系已比较明确,CRELD2基因功能则未知,与UC的关系亦不明,故进一步对该基因进行生物信息学分析。

表1 正常人和不同组别UC患者间显著差异表达基因*
*只列出上调或下调幅度最大的前15个基因和4个共表达基因
二、基因定位和序列分析
人CRELD2基因位于染色体22q13.33,全长9 374 bp,可转录为4个mRNA(NM_001135101.2、NM_001284317.1、NM_001284318.1、NM_024324.4),分别对应4个编码蛋白(NP_001128753.1、NP_001271246.1、NP_001271247.1、NP_077300.3),其中NM_001135101.2长1 596 bp,有11个外显子和10个内含子,编码含402个氨基酸残基的蛋白质NP_001128753.1。
三、基因编码蛋白的理化性质
CRELD2蛋白由402个氨基酸残基组成,相对分子质量为43 578.7 Da(1 Da=0.992 1 u),理论等电点为pH 4.71,半衰期在哺乳动物网织红细胞中为30 h;不稳定指数为48.98(>40考虑不稳定),分类为不稳定蛋白质;脂肪系数为55.85,平均疏水值为-0.450,为亲水性蛋白质。
四、一级结构预测分析
CRELD2蛋白一级结构和结构域中包含多个酶切位点(CLV)、配体位点(LIG)和蛋白修饰位点(MOD)。该蛋白包含6个重要结构域,分别为1个表皮生长因子(EGF)样结构域(EGF-like domain)、2个 富含半胱氨酸的Furin样重复序列(Furin-like repeats)、2个EGF结构域(EGF domain, unclassified subfamily)和1个钙结合EGF样结构域(calcium-binding EGF-like domain)(图1)。
五、信号肽、跨膜区域和磷酸化位点预测分析
CRELD2蛋白氨基酸序列中存在信号肽,为分泌型蛋白,剪切位点在第24~25 aa之间(图2);整条肽链均位于胞膜外,不存在跨膜结构域(图3);氨基酸序列包含24个可能的磷酸化位点,其中9个位于丝氨酸,10个位于苏氨酸,5个位于酪氨酸。
六、二级结构预测分析
CRELD2蛋白的拓扑结构和二级结构预测见图4,大写字母代表氨基酸,小写字母代表预测的二级结构,α螺旋(h)、β折叠(e)、无规则卷曲(c)的比率分别为21.39%、11.44%和67.16%。
七、基因本体和通路分析
GO分析显示CRELD2蛋白主要参与钙离子结合和蛋白结合,主要分布于高尔基体、内质网和细胞外,提示了其分泌型蛋白的特性;KEGG分析未发现其参与的信号通路。
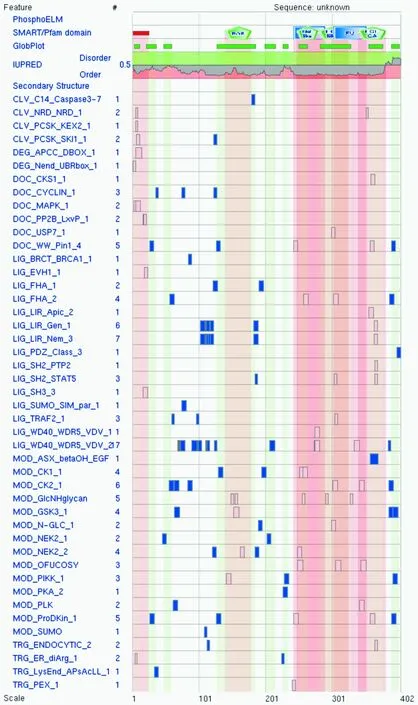
图1 CRELD2蛋白一级结构预测
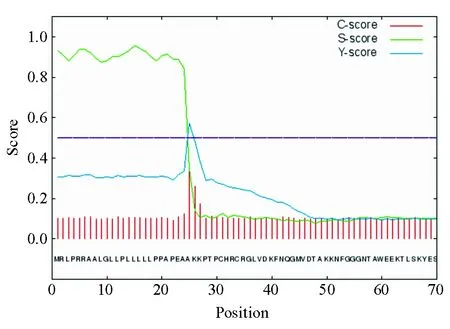
图2 CRELD2蛋白信号肽预测
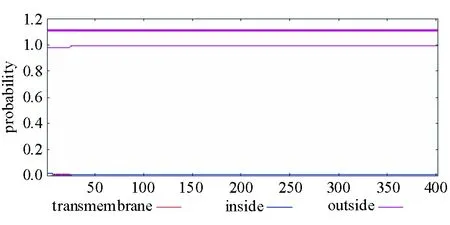
图3 CRELD2蛋白跨膜预测

图4 CRELD2蛋白二级结构预测
八、蛋白-蛋白相互作用网络分析
CRELD2蛋白的蛋白-蛋白相互作用网络见图5,该蛋白与TMEM50B、MANF、CHRNA4、GMPPB、CHRNB2、RHBDD3、ITFG1、PGAP2、ALG12、TMEM241蛋白之间存在相互作用。

图5 CRELD2蛋白与其他蛋白的相互作用网络
讨 论
本研究发现,与UC发病、疾病活动和生物治疗疗 效均相关的基因共有4个,分别为CDC25B、CRELD2、IL1RN、PITPNC1,这些基因在UC发病、疾病活动中进行性表达增高,同时在IFX治疗有效后表达明显降低。为验证这4个基因与UC疾病进程的相关性,本研究进一步分析了四者在IFX治疗前后、治疗有效与无效四组间的表达变化,结果显示治疗前、治疗后无效三组间(均处于疾病活动期)四者表达无明显变化,但与治疗后有效组相比其表达均明显增高,表明四者的表达变化与UC疾病活动性的相关性较高(图6)。
CDC25B(cell division cycle 25B)在细胞有丝分裂中发挥至关重要的作用,其表达增高可促进肿瘤生长和细胞恶性转化。CDC25B在多种恶性肿瘤中呈高表达,结直肠癌中其mRNA和蛋白高表达率分别为60%和43%[4],但CDC25B基因与炎症关系的报道尚少。持续炎症刺激可导致UC癌变,因此推测结肠炎症可能引起CDC25B表达增高。IL1RN(interleukin 1 receptor antagonist)基因主要编码白细胞介素-1受体拮抗剂(IL-1Ra)。IL-1β是与UC发病、疾病活动密切相关的促炎细胞因子,而IL-1Ra对其具有抑制作用。研究[5]发现与正常人肠黏膜相比,UC患者肠黏膜IL-1α、IL-1β、IL-1Ra mRNA表达均显著增高,提示IL1RN基因可能在UC中起重要调控作用。PITPNC1(phosphatidylinositol transfer protein, cytoplasmic 1)普遍存在于真核细胞内,能结合并转移磷脂酰肌醇和磷脂酸,对卵磷脂则无结合作用,结合有磷脂酸的PITPNC1可促进细胞膜组分间脂质的转运[6]。由此认为PITPNC1可能通过影响肠黏膜脂质代谢而在UC发病和疾病活动中发挥一定作用。
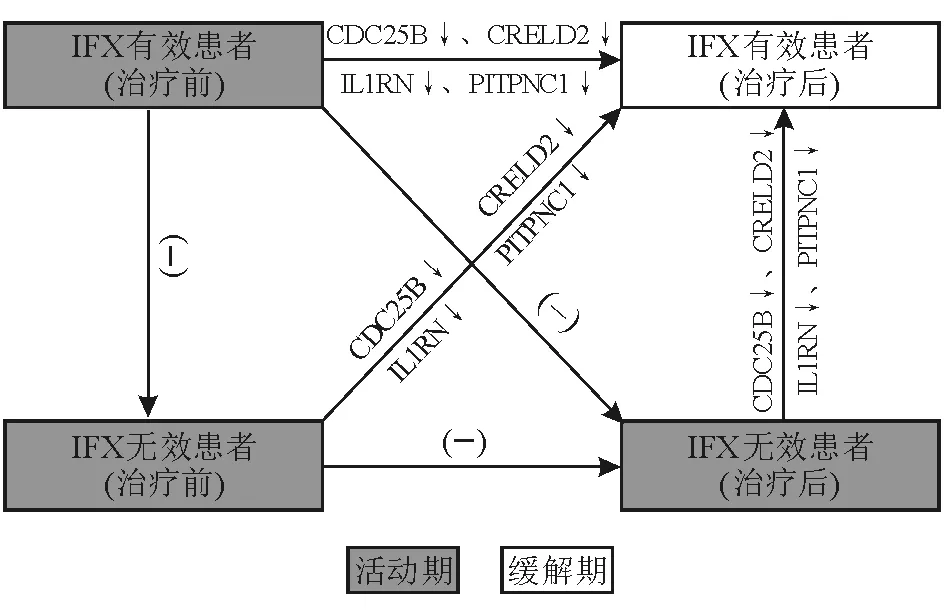
↓:表达明显降低,P<0.001;(-):表达无明显变化
图6 活动期UC IFX治疗前后CDC25B、CRELD2、IL1RN、PITPNC1基因表达变化
内质网是细胞内折叠、修饰蛋白质的重要细胞器,内质网应激所致的未折叠蛋白反应在IBD发病中发挥重要作用[7]。有研究[8]发现内质网应激可诱导CRELD2(cysteine-rich with EGF-like domains 2)基因表达,因此CRELD2基因可能参与了内质网应激相关疾病如UC发生、发展的调节过程。然而CRELD2基因的生物学功能及其在UC 发病、疾病活动中的作用机制均尚未明确。为此,本研究对CRELD2基因进行生物信息学分析,结果显示人CRELD2基因位于染色体22q13.33,编码由402个氨基酸残基组成的亲水性、分泌型蛋白。CRELD2蛋白含多个酶切位点、配体位点和蛋白修饰位点,且包含6个重要结构域,主要分布于高尔基体、内质网和细胞外。该蛋白功能活跃,主要参与钙离子结合和蛋白结合,与TMEM50B、MANF、CHRNA4、GMPPB、CHRNB2、RHBDD3、ITFG1、PGAP2、ALG12、TMEM241蛋白之间存在相互作用。
EGF作为一种生长因子,可通过激活其受体(EGFR)而发挥促进细胞增殖、分化、存活和损伤愈合作用,局部给予EGF可促进损伤黏膜愈合[9],诱导活动期UC缓解[10]。此外,EGF还可通过调节肠黏膜巨噬细胞的细胞因子产生发挥控制UC肠道炎症的作用[11]。CRELD2蛋白含有多个EGF样结构域,因此可能具有类似EGF的功能。经ToppGene工具(https://toppgene.cchmc.org/)分析后发现,CHRNA4、CHRNB2、RHBDD3基因与淋巴细胞激活和自然杀伤细胞抑制相关,而CRELD2蛋白与三者均存在相互作用,因此其间的相互作用可能影响淋巴细胞激活,CRELD2可能通过直接或间接参与免疫细胞的调控对UC发病、疾病活动产生影响。
本研究纳入NCBI GEO数据库中GSE38713和GSE16879两项研究的结肠黏膜表达谱芯片原始数据进行分析,分析时采取分别在单个研究中筛选差异表达基因,再提取两项研究共同差异表达基因的方法,避免了不同研究间芯片检测的批次效应。本研究首次发现了CRELD2基因与UC间的关系,因该基因功能未知,在UC中的作用机制亦不明,研究进一步对该基因进行生物信息学分析,为揭示UC发病机制提供了新思路,也为今后相关实验研究提供了有价值的信息。本研究的局限性主要在于基于基因芯片数据的差异表达基因分析以及基于数据库的单基因功能预测均偏于理论,需进一步应用分子生物学方法进行验证。
综上所述,本研究通过分析基因芯片原始数据,发现在UC发病、疾病活动中进行性表达增高,同时在IFX治疗有效后表达明显降低的4个基因CDC25B、CRELD2、IL1RN、PITPNC1,并对其中功能未知的CRELD2基因进行包括结构、功能、蛋白间相 互作用等项目的生物信息学分析,结果提示CRELD2基因可能与UC发生、发展相关,有望成为UC诊断、疾病活动性评估、疗效评估的新指标和新的治疗靶点,值得进一步深入研究。
1 Li J, Wang F, Zhang HJ, et al. Corticosteroid therapy in ulcerative colitis: Clinical response and predictors[J]. World J Gastroenterol, 2015, 21 (10): 3005-3015.
2 Gu C, Shen T. cDNA microarray and bioinformatic analysis for the identification of key genes in Alzheimer’s disease[J]. Int J Mol Med, 2014, 33 (2): 457-461.
3 Zhao Y, Simon R. BRB-ArrayTools Data Archive for human cancer gene expression: a unique and efficient data sharing resource[J]. Cancer Inform, 2008, 6: 9-15.
4 Takemasa I, Yamamoto H, Sekimoto M, et al. Overexpression of CDC25B phosphatase as a novel marker of poor prognosis of human colorectal carcinoma[J]. Cancer Res, 2000, 60 (11): 3043-3050.
5 Carter MJ, Jones S, Camp NJ, et al. Functional correlates of the interleukin-1 receptor antagonist gene polymorphism in the colonic mucosa in ulcerative colitis[J]. Genes Immun, 2004, 5 (1): 8-15.
6 Garner K, Hunt AN, Koster G, et al. Phosphatidylinositol transfer protein, cytoplasmic 1 (PITPNC1) binds and transfers phosphatidic acid[J]. J Biol Chem, 2012, 287 (38): 32263-32276.
7 Kaser A, Blumberg RS. Endoplasmic reticulum stress in the intestinal epithelium and inflammatory bowel disease[J]. Semin Immunol, 2009, 21 (3): 156-163.
8 Oh-hashi K, Koga H, Ikeda S, et al. CRELD2 is a novel endoplasmic reticulum stress-inducible gene[J]. Biochem Biophys Res Commun, 2009, 387 (3): 504-510.
9 Sakallioglu AE, Yagmurlu A, Dindar H, et al. Sustained local application of low-dose epidermal growth factor on steroid-inhibited colonic wound healing[J]. J Pediatr Surg, 2004, 39 (4): 591-595.
10 Sinha A, Nightingale J, West KP, et al. Epidermal growth factor enemas with oral mesalamine for mild-to-moderate left-sided ulcerative colitis or proctitis[J]. N Engl J Med, 2003, 349 (4): 350-357.
11 Lu N, Wang L, Cao H, et al. Activation of the epidermal growth factor receptor in macrophages regulates cytokine production and experimental colitis[J]. J Immunol, 2014, 192 (3): 1013-1023.
(2016-05-20收稿;2016-06-01修回)
Bioinformatics Analyses of Gene CRELD2 that Associated with Pathogenesis, Disease Activity and Efficacy of Biological Agents in Ulcerative Colitis
TAOWenhui,WANGFan,LINXue,MAMinxing,ZHAOQiu,LIJin.
DepartmentofGastroenterology,ZhongnanHospitalofWuhanUniversity;HubeiClinicalCenter&KeyLabofIntestinalandColorectalDiseases,Wuhan(430071)
LI Jin, Email: 18207118835@163.com
CRELD2 Gene; Colitis, Ulcerative; Bioinformatics; Microarray Analysis
10.3969/j.issn.1008-7125.2017.01.002
国家自然科学基金青年科学基金项目(81200283)
#本文通信作者,Email: 18207118835@163.com
Background: Bioinformatics is an effective technology for microarray data mining and gene function prediction. Aims: To analyze the gene CRELD2 that associated with pathogenesis, disease activity and efficacy of biological agents in ulcerative colitis (UC) by bioinformatics to provide a theoretical basis for subsequent studies on its biological function and molecular mechanism in the development and progress of UC. Methods: The microarray data associated with pathogenesis, disease activity and efficacy of biological agents in UC were downloaded from the Gene Expression Omnibus (GEO database); the data mining and analyses were conducted by using bioinformatics tools such as BRB-ArrayTools, ProtParam, ELM, SignalP 4.1, PBIL-IBCP Lyon Gerland, GO and STRING. Results: Cross-over analyses revealed that expressions of four genes (CDC25B, CRELD2, IL1RN, PITPNC1) were up-regulated in the order from colonic mucosa of healthy subjects, un-inflamed mucosa of active UC patients to inflamed mucosa of active UC patients, meanwhile these four genes were significantly down-regulated in infliximab responders after treatment when compared with that before treatment and infliximab non-responders. The function of CRELD2 gene was unknown. Bioinformatics analyses showed that CRELD2 gene was located on the long arm of chromosome 22 (22q13.33), and encoded a secreted protein composed of 402 amino acids. This protein contained several epidermal growth factor (EGF)-like domains, mainly distributed in Golgi apparatus, endoplasmic reticulum and extracellular site and had calcium- and protein-binding effect. Interactions existed between CRELD2 and CHRNA4, CHRNB2 and RHBDD3 proteins. Conclusions: Gene CRELD2 may have EGF-like biological function and via participating directly or indirectly the regulation of immunocytes to affect the pathogenesis and disease activity of UC. It might be used as a biomarker for diagnosis and assessment of disease activity and therapeutic efficacy of UC. Furthermore, it might be a potential target for treatment of UC.
