将分子动力学模拟方法引入体育领域研究的可行性分析
——以肌动蛋白、肌球蛋白和人类神经元钙传感蛋白为例
2017-02-11朱玉珍张庆文
朱玉珍,邹 昱,张庆文

将分子动力学模拟方法引入体育领域研究的可行性分析
——以肌动蛋白、肌球蛋白和人类神经元钙传感蛋白为例
朱玉珍1,邹 昱2,张庆文2
分子动力学模拟是基于牛顿运动方程来研究体系结构随时间变化的计算机模拟方法。该方法可应用于物理、化学、生物学及其交叉学科领域的相关问题研究,能够描述蛋白质等生物大分子在溶液中的运动随时间最详实的变化情况,从原子水平上解释实验结果,预测新的结构和现象,为实验提供参考和启示。鉴于分子动力学模拟方法的应用价值和国内鲜有分子动力学模拟方法应用于体育运动机制的相关研究,在系统考察分子动力学模拟方法及其应用的基础上,以肌动蛋白、肌球蛋白和人类神经元钙传感蛋白为例,说明分子动力学模拟方法在体育领域研究中的应用,阐述分子动力学模拟方法潜在的体育研究领域,并对其应用前景进行展望,以期为体育领域研究专业化探索一条创新的途径。
分子动力学模拟;体育;研究;生理机制;蛋白Abstract:Molecular dynamics simulation is the computer simulation method based on Newton’s equation of motion to study architecture with time variations.This method can be applied to physics,chemistry,biology and related issues in the field of cross-disciplinary research.It can describe the internal motions of protein and test the behavior of macromolecules in solution at the atomic scale to explain the experimental phenomena,to predict the new structure and phenomenonm and to provide the reference for experiment.In view of the value of molecular dynamics simulation method and few related research in the sports mechanism using molecular dynamics simulation method in China,this paper systematically described the molecular dynamics simulation method and its application in the sports research fields as actin,myosin and human neuronal calcium sensor-1 protein to explore an innovative way for the sports research fields.
1 前言
21世纪,创新作为世界经济发展的核心动力来源,越来越受到世界各国的重视。2004年,美国成立了未来创新委员会(The Task Force on the Future of American Innovation)。2005年和2006年11月16日,连续发表两份调查报告,指出美国创新优势受到削弱,并列出引起警觉的主要指标,这些数据引起美国政府的高度重视[3]。自2002年“教育创新”正式进入中国公众话语开始,2010年颁布的《国家中长期教育改革和发展规划纲要(2010—2020年)》又将创新人才培养和体制机制创新放在突出位置,并在全国范围内推行重大改革创新试点项目[2]。2015年10月,中国共产党第十八届中央委员会第五次全体会议通过了《中共中央关于制定国民经济和社会发展第十三个五年规划的建议》,第3部分明确提出“深入实施创新驱动发展战略”。纵观人类世界的发展历程,每一次革命性的飞跃,都必然伴随着方法的革新。在体育研究中,积极地引入新的研究方法,是每一位体育科研工作者的光荣使命,也是进一步提升体育研究专业化的重中之重。
分子动力学模拟具有严格的程序化的操作流程,能够从原子水平上观察并解释由于受到时间和空间分辨局限的实验现象,对实验上提出的理论假设和(或)机制进行验证,为实验研究提供新的视角和佐证;同时,也可以预测新的结构和现象,为实验上建立理论模型和作用机制提供参考和启示。将实验研究的手段和方法与分子动力学模拟方法密切结合起来,共同推动运动干预与机制研究的纵深发展,开辟新的研究领域,具有重要的理论和现实意义。纵观国内体育领域的研究,鲜见有采用分子动力学模拟方法的研究结果。在系统考察分子动力学模拟方法的基础上,本研究简要介绍分子动力学模拟方法的原理、功能、操作步骤以及国内外运用这一方法的研究现状,以肌动蛋白、肌球蛋白和人类神经元钙传感蛋白为例,阐述国际上运用分子动力学模拟方法在体育领域研究中的应用,对其应用前景进行了展望。
2 分子动力学模拟方法简介
分子动力学作为一种计算机模拟方法,主要依靠牛顿力学来模拟分子体系的运动,并从由分子体系的不同状态构成的系综中抽取样本,进而计算体系的构型积分,然后以构型积分的结果为基础,来计算体系的热力学量和其他宏观性质。分子动力学模拟最早始于1957年,Alder和Wainwright在硬球模型下[5],研究了气体和液体的状态方程,开创了分子动力学研究物质宏观和微观性质的先例。20世纪50年代末60年代初,Alder和Wainwright[4]以及Rahman[35]各自独立开发,计算若干液体的性质,体系粒子数也只有几十个;70年代末到90年代初,多位科学家从多个方面对分子动力学方法进行了完善,在运用时可以考虑恒压[31,34,40]、恒温[9,22,30]、非平衡[18]、粒子数可变[10,11]等情况。
2.1 分子动力学方法的原理
分子动力学的基本原理是利用牛顿运动定律,通过对牛顿第二定律积分而得到体系各原子的速度及坐标随时间的变化关系,从而得到体系随时间的状态演化。
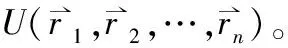
根据牛顿第二定律可得第i个粒子的加速度为:
对牛顿运动定律方程式进行时间积分,可得第i个粒子经过时间t后的速度和位置:
式中r和v分别为粒子的位置与速度,上标“0”为各物理量的初始值。
分子动力学的基本原理就是利用牛顿定律,首先由系统中各个粒子的位置计算体系势能,然后通过牛顿力学计算系统中各粒子的受力和加速度,再通过运动方程,令时间积分t=δt(δt表示非常短的时间间隔),则可以得到经过δt后体系各粒子的位置与速度。如此反复,可得到各时间下系统中粒子运动的位置、速度以及加速度等信息。
2.2 分子动力学方法的功能
通过不断地发展和完善,分子动力学模拟现已成为应用最为广泛的分子模拟方法之一。通过这种方法可以模拟数十个蛋白组成的复合体的构象运动,并且对于一定大小的蛋白,甚至可以描述其折叠过程,尤其在生物大分子的模拟方面,发挥着极其重要的作用[16,38,43]。分子动力学模拟体系的时间尺度和空间尺度不断得以突破,近年来已经可以模拟多达几十万甚至数百万个原子的体系,如核糖体[19]、病毒胞衣以及对载色体[29]等复杂结构。在理论上,分子动力学模拟体系的时间尺度和空间尺度是无限的,这主要取决于计算能力、计算资源和建模精度。在计算能力、计算资源和建模精度限定的情况下,空间尺度越大,时间尺度就越小;反之,时间尺度越小,空间尺度就可以越大。建模精度越小,模型越粗糙,则时间尺度和空间尺度都可以越大。Shaw D E于2010年在超级计算机Anton上进行了毫秒级别的蛋白质模拟,Anton拥有128个处理器,完成了一个包含23 558个原子的水化蛋白质体系17 μs的分子动力学模拟[36]。而后开发的Anton2已具有512个处理器,CPU核心数是一代的5倍,可以处理膜蛋白等超大体系[14,37]。2013年,日本名古屋大学Okazaki课题组使用当时最好的超级计算机K-computer,配合专门开发的软件MODYLAS,使用了65 536个节点,524 288个CPU核心,模拟了水溶液中完整的病毒分子,这个病毒分子包含240个蛋白质,超过10 000 000个原子,时间尺度达到了10 μs级别[6]。现今,分子动力学的发展仍然紧跟摩尔定律所预测的计算机发展趋势,未来20年内,在计算机上模拟一个完整的细菌,甚至一个完整的哺乳动物细胞都是极有可能的。分子动力学方法可以给出蛋白质、核酸等内部运动随时间最详实的变化,从而分析体系诸多的运动性质,以检测在实验上无法被直接观测到的生物分子在溶液中的行为,从原子水平上解释实验现象,为建立理论模型和作用机理提供依据;也可以预测新的结构和现象,对理论假设进行检验,为实验提供参考和启示。
2.3 分子动力学方法的操作步骤
分子动力学模拟有一套程序化的操作步骤,模拟对象和模拟软件有很多,此处分子动力学模拟的研究对象以蛋白为例,模拟软件以GROMACS为例进行说明,操作流程(图1)大体可以分为如下几步:
第1步:确定拟研究的问题。
第2步:确定起始构型。
蛋白分子的起始构型主要来自实验数据或量子化学计算,能量较低的起始构型是进行分子动力学模拟的基础。根据问题,确定研究对象,即确定进行分子动力学模拟的蛋白的起始构型,并从蛋白质数据库(protein data bank,PDB)中下载此蛋白结构,作为动力学模拟的起始结构。
第3步:起始构型模型化。
输入GROMACS 4.5.3程序包[20]中的命令,将下载蛋白的起始构型模型化,即转化为分子动力学模拟所需要的结构。在这一步骤中,涉及到分子动力学所用力场及对电荷的选择。
力场就是能量函数与其参数的结合,是计算机模拟的核心部分。分子动力学能够在众多领域得到广泛应用,关键在于有一套较为成熟的经验力场。常见的力场有AMBER、GROMACS、CHARMM、OPLS-AA,还有其他一些力场,如CVFF、ECEPP、ENCAD、MM4、MMFF、UFF等,具体选用哪种力场,应根据研究蛋白进行筛选;电荷的选择,主要根据研究蛋白所处的模拟环境(如酸性、中性还是碱性环境)进行选择。
第4步:为研究蛋白创设一个类似于真实生物环境的模拟环境。
根据生物体的环境或实验环境,采用GROMACS命令,对模型化了的研究蛋白所处的模拟环境进行设置。
采用GROMACS命令,首先根据模型化了的蛋白的形状和大小,选择一个适当形状和大小的盒子,然后分别采用相应的GROMACS命令,在盒子内加水、加离子,使模拟环境最大程度地接近生物体或实验的环境。
第5步:能量优化。
模拟中所有的宏观量,都是通过对分子的微观量进行统计平均之后得到的,这些热力学要基于平衡态的统计才是有意义的。能量优化就是使模拟环境的热力学基本处于平衡态,即对模拟环境进行能量最小化,使整个环境的能量最小。
常用的一阶能量优化的方法有最速下降法和共轭梯度法。最速下降法能够快速移除体系内的应力,但接近能量极小点时收敛较慢;共轭梯度法在能量极小点附近收敛效率相对高一些。 因此,做能量最小化时,通常先采用最速下降法进行优化,之后利用共轭梯度法对所得到的结构再优化一次,从而保证后续模拟的有效进行。但具体选用何种能量优化的方法,应视研究蛋白而有所不同。
第6步:约束分子动力学。
约束分子动力学方法是为了满足实验上已知的某一距离/角度约束或为了提高分子动力学模拟的抽样效率,而在体系的势能函数中加入一个简谐项,是分子动力学模拟中一种比较常用的方法。约束分子动力学是在保持蛋白结构不变的前提下,对模拟体系中的溶剂进行平衡,即限制蛋白分子的位置,允许溶剂分子运动,使溶剂浸入蛋白,从而使整个模拟体系达到平衡。
第7步:动力学模拟。
分子动力学技术已被公认为一个用来改善实验结构的可靠的工具。这一过程类似于一个高速摄像机,以ps(10-12s)为单位的速度记录蛋白在模拟体系中最详尽的运行和变化情况,从而分析蛋白诸多的运动性质,所用的数据正是从这个过程中获取的。
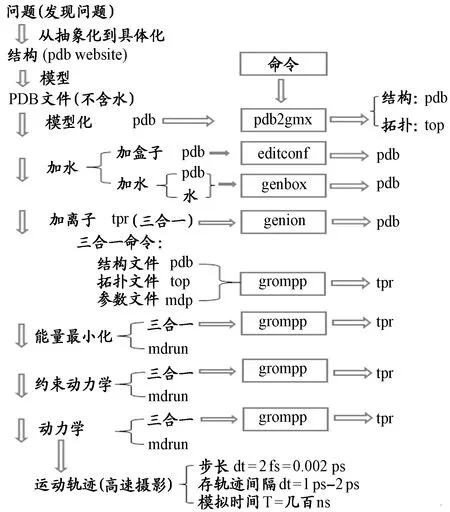
图 1 采用GROMACS程序包进行分子动力学模拟的操作流程Figure 1. Operation Procedure of Molecular Dynamics Simulations Using GROMACS Software
3 运用分子动力学模拟方法的研究现状
3.1 运用分子动力学模拟方法的发文量及其空间分布
为把握世界范围内分子动力学模拟的应用现状,在Web of scienceTM核心合集数据库中输入主题词“‘molecular dynamics simulations’OR ‘MD simulations’”作为核心检索词进行检索,文献格式为“Article”,语言类型为“English”,检索时间范围为2010—2014,检索下载日期为2015年11月16日,检索到原始文献24 369篇,由于检索主题词具体、明确,最终进入分析的文献24 369篇。将文献题目、作者、关键词、摘要、参考文献等信息全部下载,保存为纯文本形式,并对有关分子动力学模拟的文献进行统计分析。
3.1.1 2010—2014年间有关分子动力学模拟的文献发表数量呈递增趋势
近5年内,国际上发表有关分子动力学模拟的文献数量为24 369篇,年平均发文量为4 874篇(图2),呈直线上升的趋势。数据表明,在世界范围内,分子动力学模拟是一种应用极其广泛的研究方法。
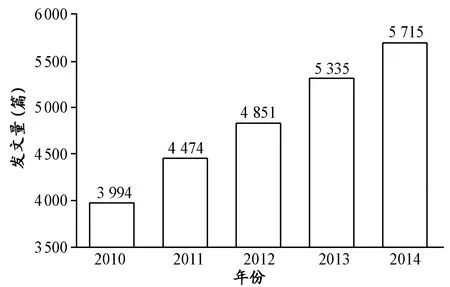
图 2 分子动力学模拟文献的年发文量Figure 2. Number of Literatures on Molecular Dynamics Simulations Each Year
3.1.2 中国是世界上采用分子动力学模拟方法进行研究的第二大国
分子动力学模拟文献每年的发表数量如此之大,应用之广泛,引导我们对世界各国在2010—2014年间采用分子动力学模拟方法进行研究的发文量进行统计,发现共有94个国家(地区)在采用这一研究方法进行科学研究,图3列出排名前10位国家的发文量。美国第一,中国遥遥领先于第3名发文量国家——德国,以相当的优势位于第2名,其他7个国家的发文量差别不大。在系统考察世界各国(地区)6 444个不同机构的发文量发现,中国科学院比其他各国(地区)机构的发文量都大,排名第一。数据表明,目前我国有很多专家学者在采用分子动力学模拟的方法进行研究,这能够为分子动力学模拟方法在体育领域进行相关研究提供前提条件和技术基础。
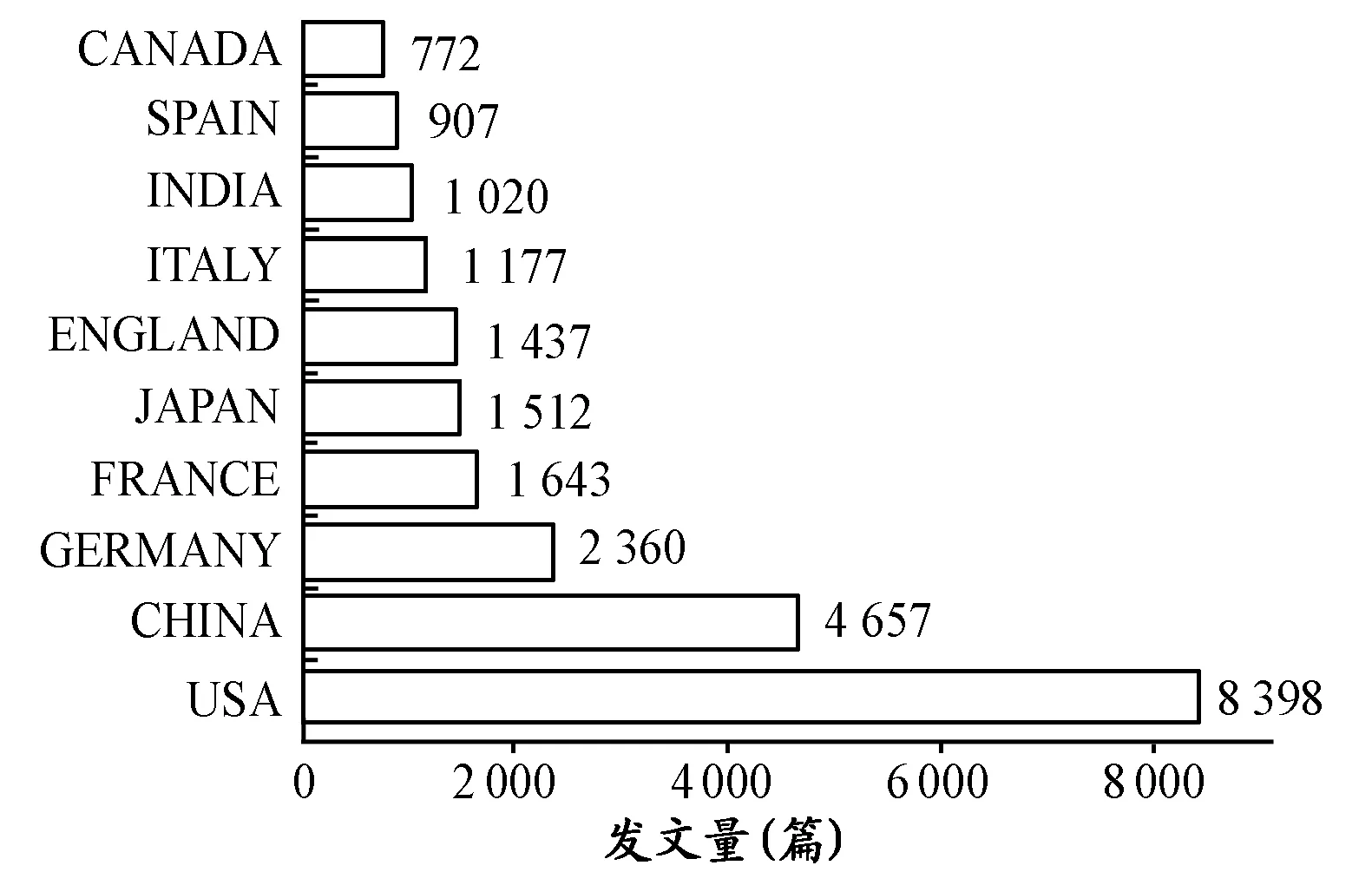
图 3 世界各国(地区)近5年有关分子动力学模拟文献的发文量Figure 3. Number of Literatures on Molecular Dynamics Simulations Worldwide for Lately Five Years
3.2 分子动力学模拟涉及的研究领域
随着计算机的飞速发展,分子动力学模拟越来越多地应用到不同领域。通过对相关文献的统计分析发现,目前这一方法所涉及的研究领域非常广泛,包括89个研究分支,主要应用于化学、物理、生物学(包括分子生物学、生物物理学、计算生物学、细胞生物学和生命科学)、材料科学、科学技术、工程学、计算机科学、药理学、力学、医学等领域的研究(图4)。尤其值得注意的是,在运用分子动力学模拟方法所涉及的众多研究方向中,生物学排名第3,仅次于化学和物理,是该方法主要研究的领域之一。
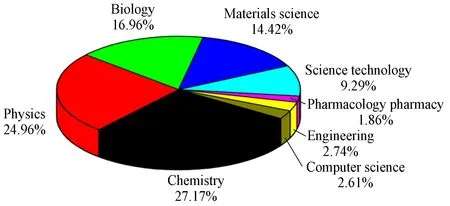
图 4 分子动力学模拟涉及的研究领域分布图Figure 4. Research Fields of Molecular Dynamics Simulations
3.3 分子动力学模拟的研究内容
关键词能够在一定程度上反映某一专题的研究内容。本文通过对在Web of science核心合集数据库中检索到的有关分子动力学模拟文献中的关键词进行分析,结果显示,24 369篇文献的关键词仅有63个,说明采用计算机模拟方法的研究内容相对集中。在关键词中包括与“molecular dynamics simulation(s)”“simulation(s)”或“molecular dynamics”等类似的关键词有12个;无实际意义的关键词有4个,如“model”“system(s)”“abinitio”“behavior”;将同义词或近义词合并,如“protein”和“proteins”“water”“liquid water”和“aqueous solution(s)”等,最后列出有意义的实词关键词及其频次(表1)。
在38个关键词中,大致可以分为以下6类:1)参数类,包括力场、温度、库伦类型;2)研究对象类,包括晶体结构、金属、碳纳米管、溶剂、薄膜、大肠杆菌、纳米粒子、核酸、细胞膜、复合物、石墨烯;3)数据分析类,包括能量、稳定性、自由能、密度、势能、自由能计算;4)研究结果类,包括结合、吸附、扩散、运输、转变、生长、变形、识别、聚集;5)方法类,包括光谱学、对接、蒙特卡洛;6)研究目的类,包括机制和机制特性。由关键词及其频次表明,采用分子动力学模拟方法进行研究的主要目的、研究对象、数据分析、研究结果以及与之相关的理论和方法都很清晰、具体,而且研究对象的范围非常广泛,2014年更是将研究对象扩展到细胞膜,出现频次为163次。
值得注意的是,关键词“温度”出现的频次为959,表明在分子动力学模拟中,温度的设置是一个十分重要而又变化的参数。温度在生物体中保持动态的平衡,而影响温度变化的因素有很多,如发热、疾病和运动等。在人体中,温度与体育运动密切相关,是一个对运动非常敏感并易于测量的指标。
关键词“机制”出现的频次为942,是分子动力学模拟的主要目的。在与人体有关的生命科学中,对于“机制”的探索始终是学者一直在努力并不断接近的目标。在体育运动中,很多有关机制的命题尚未完成,包括笼统意义上的机制,如运动促进脑认知的机制、运动促进健康的机制等;还包括具体的机制,如太极拳预防老年痴呆的机制、信号蛋白-神经元钙传感蛋白抑制运动的机制等。这些命题不仅是体育界面临的问题和挑战,更是整个生命科学领域正在挑战的难题。
表 1 分子动力学模拟的关键词频次
Table 1 Frequency of the Key Words on Molecular Dynamics Simulations
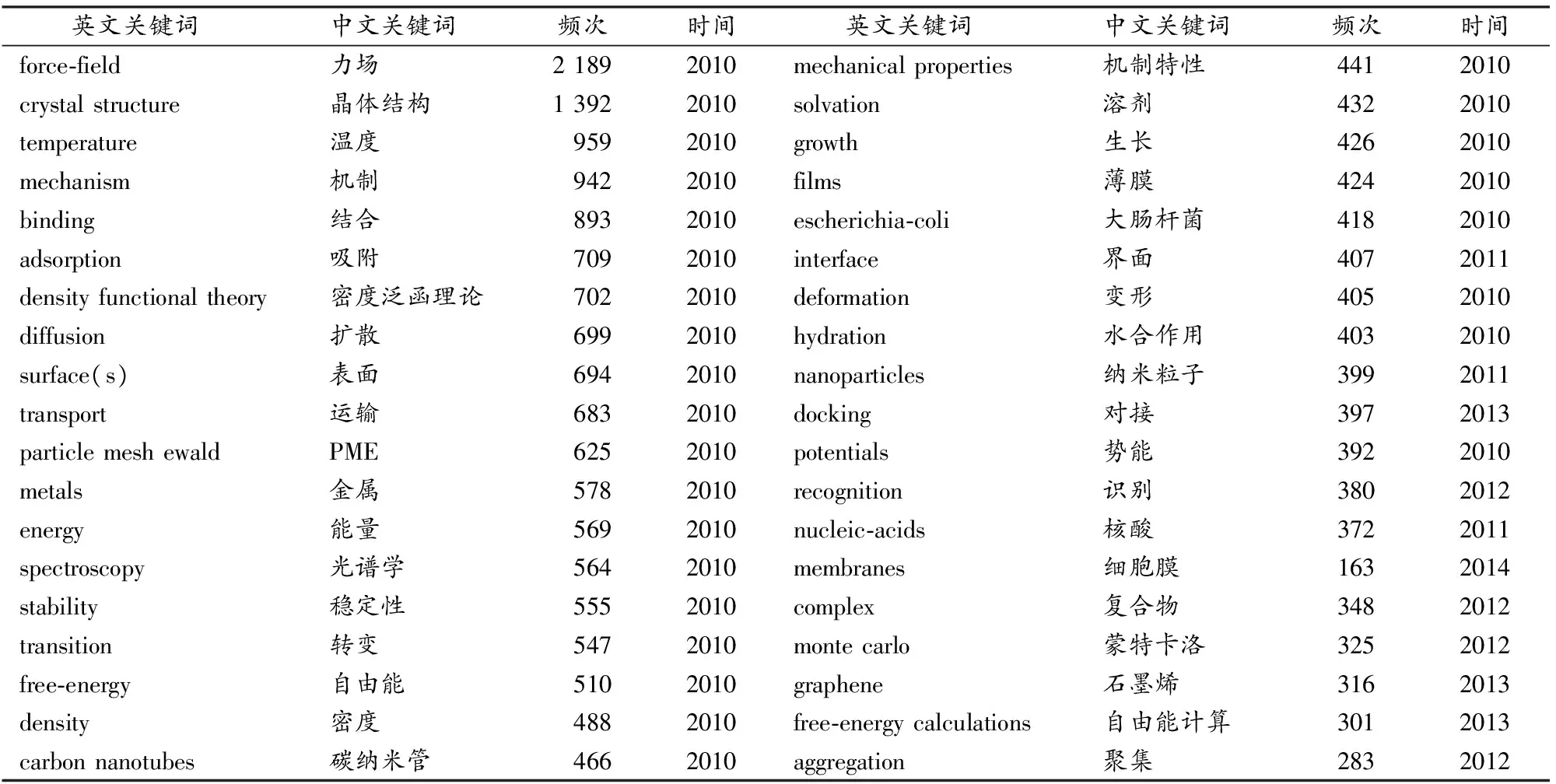
英文关键词中文关键词频次时间 英文关键词中文关键词频次时间force⁃field力场21892010mechanicalproperties机制特性4412010crystalstructure晶体结构13922010solvation溶剂4322010temperature温度9592010growth生长4262010mechanism机制9422010films薄膜4242010binding结合8932010escherichia⁃coli大肠杆菌4182010adsorption吸附7092010interface界面4072011densityfunctionaltheory密度泛函理论7022010deformation变形4052010diffusion扩散6992010hydration水合作用4032010surface(s)表面6942010nanoparticles纳米粒子3992011transport运输6832010docking对接3972013particlemeshewaldPME6252010potentials势能3922010metals金属5782010recognition识别3802012energy能量5692010nucleic⁃acids核酸3722011spectroscopy光谱学5642010membranes细胞膜1632014stability稳定性5552010complex复合物3482012transition转变5472010montecarlo蒙特卡洛3252012free⁃energy自由能5102010graphene石墨烯3162013density密度4882010free⁃energycalculations自由能计算3012013carbonnanotubes碳纳米管4662010aggregation聚集2832012
4 分子动力学模拟方法在体育领域研究中的应用
4.1 分子动力学模拟方法在功能性蛋白结构和功能研究中的应用——以肌动蛋白和肌球蛋白为例
4.1.1 分子动力学模拟方法在肌动蛋白研究中的应用
肌动蛋白(actin)由374~375个氨基酸组成,具有高度保守的基因编码。在运动过程中,肌动蛋白主要参与肌肉收缩。实验表明,肌动蛋白是一个高度保守的结构蛋白,在结构上多种多样,但在分子层面上单个和多个亚基接触动力学的异质性仍有待了解;同时,由于肌动蛋白结构难以用结晶的方法解析出来,导致肌动蛋白原子层面的结构尚不清楚。Wriggers等[42]首次采用分子动力学模拟方法研究G-肌动蛋白的结构特性,提出水分扩散机制对ATP水解作用的重要性;有学者采用分子动力学模拟的方法,比较了实验上给出的3种肌动蛋白纤维构型[21,26,32]的异同,研究了ADP和ATP影响不同蛋白构型的机制,指出第137号位点的谷氨酰胺对于ATP的水解起到非常重要的作用[39]。Chu J W等[12]对G-肌动蛋白、肌动蛋白三聚体以及全部的F-肌动蛋白进行了模拟,发现在ATP与ADP结合G-与F-肌动蛋白中可能存在的生物化学差别。运用全原子以及粗粒化等分子动力学模拟的方法,Fan J等[17]对肌动蛋白纤维进行了模拟,发现这些纤维呈现出无序的扭转角度,而且肌动蛋白结构的多样性对纤维特性的影响非常明显。这些研究结果从原子的层面对实验结果进行了补充和解释,为实验研究人员提供启示和参考。
4.1.2 分子动力学模拟方法在肌球蛋白研究中的应用
肌球蛋白是各类肌细胞中含量最多的结构蛋白和收缩蛋白,也是构成肌肉肌原纤维粗丝的基本组成蛋白;在结构方面,肌球蛋白是一个六聚体的蛋白质大分子,由两条肌球蛋白重链(Myosin heavy chain,MHC)和两对不同的肌球蛋白轻链(Myosin lightchain,MLC)组成[41]。肌球蛋白是典型的分子马达蛋白,它能把ATP水解作用的化学能转化为机械作用。对于肌肉收缩而言,其能量的直接来源是肌球蛋白对ATP的水解,因此,肌球蛋白也被称为肌球蛋白ATP酶,但其能量转化的机制尚不清楚。为了解释实验上观察到的分子马达的现象,Masuda T提出了一个“分离驱使”理论来解释肌球蛋白的工作原理,肌球蛋白动力冲程所需的能量来源是分离的肌球蛋白头部和肌动蛋白纤维之间的吸引力,并非直接由ATP供能。基于此理论,除ATP水解作用以外,肌球蛋白工作过程中的每一步都可以用分子动力学模拟来重现。2013年,Masuda T[27]模拟了肌球蛋白S1区域与肌动蛋白纤维对接的过程,30 ns的分子动力学模拟结果显示,肌球蛋白通过不断调整位置和方向,与两个肌动蛋白单体有了很紧密的接触,一旦这个对接产生,肌球蛋白和肌动蛋白之间的距离不再发生大的变化;如果对接没有成功,肌球蛋白将会在初始位置附近随机地移动或者远离肌动蛋白纤维。2015年,他又采用分子动力学模拟的方法,对肌球蛋白S1区域进行了64 ns的模拟,发现肌球蛋白S1区域形成了之前动力冲程状态的构型,颈部区域弯曲到头部和颈部区域的结合处,再次证明了他之前提出的分离驱使理论[28]。Masuda T将实验上目前还无法观测、解释的现象,通过分子动力学模拟的方法,从更加微观的视角进行观察,与实验结果相互结合,彼此印证并推动研究的纵深发展。
4.1.3 分子动力学模拟方法在肌动蛋白和肌球蛋白相互作用中的应用
肌球蛋白通过与肌动蛋白相互作用,水解ATP末端的磷酸基团与GTP、CTP等来发挥一系列的细胞功能。Voth G A等[8]运用全原子和粗粒化等分子动力学模拟的方法,对肌球蛋白II型S1区域如何作用于肌动蛋白进行了研究,他们在肌球蛋白和肌动蛋白的表面识别出一些起重要作用的盐桥和氢键,同时他们发现,这种结合增加了肌动蛋白纤维的扭转刚度,并从分子层面对这一机制进行了解释。Takano M等[33]运用粗粒化分子动力学模拟的方法对肌动蛋白和肌球蛋白的结合进行了模拟,解答了两种不同热力学结合机制共存的原因,并发现分子动力学模拟的结果与实验上观察到的不同盐浓度和温度对两者结合力的影响存在一致性,证实了实验上提出的结合机制。为在原子层面更好地了解肌肉收缩的机制,理解肌球蛋白如何以可逆的方式结合肌动蛋白,Lorenz M等[26]用分子动力学模拟的方法对肌动蛋白与肌球蛋白的复合体进行了研究,发现肌动蛋白与肌球蛋白头部S1区域有很多接触,两者的接触面也大大增加,S1能够结合2个肌动蛋白的单体,而且S1上很多无规则结构与肌动蛋白都参与到两者的接触面上。这些研究从原子-分子水平对肌动蛋白和肌球蛋白的相互作用进行了深入研究,一方面解释了实验上观察到现象的原因,验证了实验结果;另一方面,从更微观的视角对两者的结合以及肌肉收缩的机制进行了解释,提出新的观点,为实验研究提供新的视角和思路。
4.2 分子动力学模拟的参数改变在体育领域探索研究中的应用——以人类神经元钙传感蛋白为例
在分子动力学模拟的众多参数中,很多参数是根据研究对象的不同而进行相应的设置,如温度、离子及其浓度、pH值等。这些指标对运动非常敏感,并且简单、易测,因此在体育运动领域研究中经常作为常规指标进行测量。神经元钙传感蛋白能够通过运动干预调节特定的认知过程[15],是体育运动对认知功能产生积极影响的途径之一。这一部分以人类神经元钙传感蛋白为例,分别将温度、离子及其浓度作为自变量,在分子动力学模拟的参数中进行设置。
4.2.1 在分子动力学模拟参数设置中,温度作为自变量
正常情况下,人体温度控制在37℃左右,处于动态的平衡。但人体温度不是一成不变的,引起人体温度变化的原因有很多,如发热、疾病和运动等。研究显示,患有严重神经系统疾病的病人体温会上升[7,24];另外,在临床上,将达到43℃的过高热作为肿瘤和传染性疾病的治疗手段[13]。但同时,过高热也提高了脑损伤的风险,引起体内多个系统的功能障碍[13,24]。其中,通过运动能够使人类体温达到极限值。因此,将温度作为一个自变量,用分子动力学模拟的方法,探索了在正常体温37℃(相当于开氏温度310 K)和极限体温43℃(316 K)下,人类神经元钙传感蛋白构象动力学的变化,从原子-分子水平揭示了温度升高时神经元钙传感蛋白抑制运动的生理机制[44](图6),并将该研究结果与其NCS-1R102Q突变体[45]和去掉尾部NCS-1D176-V190[46]的结果进行比较发现,蛋白结构和动力学对温度升高的反应可能是人类神经元钙传感蛋白固有的功能特性。
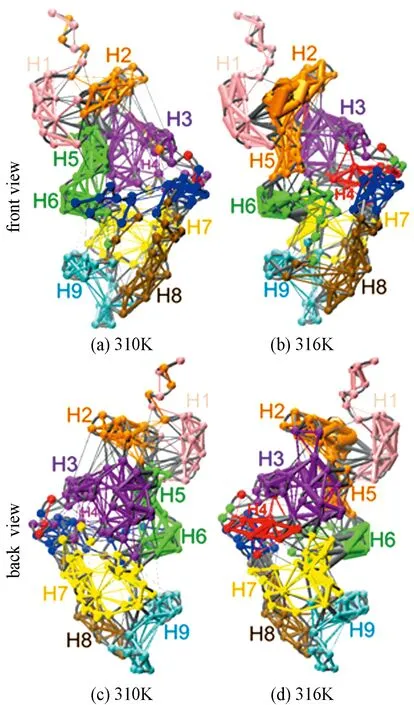
图 5 神经元钙传感蛋白抑制运动的生理机制示意图[44]Figure 5. Physiological Mechanism of Neuronal Calcium Sensor-1 Protein Inhibiting Motion
4.2.2 在分子动力学模拟参数设置中,离子及其浓度作为自变量
无机盐是人体的组成成分,虽然不能提供能量,但在维持机体完成各种生理活动中发挥重要作用。正常情况下,无机盐代谢处于平衡状态,运动时,尤其在热环境下,长时间的剧烈运动,通过汗液丢失大量无机盐,尤其是Na+,比其他元素丢失得都多[1],如得不到合理补充,将会出现无机盐失衡,影响运动能力。
在分子动力学模拟中,可以考虑加入的离子有很多,如Na+、K+、Mg2+、Ca2+、Cl-等,这些离子在运动中也简单易测,是生理生化测试指标中最常用的;而且不同强度的运动,其血清离子浓度不同。因此,在分子动力学模拟研究中,离子及其浓度改变对研究对象的影响也是一个非常重要的研究领域。有的研究为了专门观察NaCl浓度改变对肽、脂质膜结构的影响,将NaCl浓度分别设为0.15 M、1 M[25]及0 M、0.06 M、1 M[23]进行探索。另外,金属也是计算机模拟常用的对象,因此,运动中人体必需的微量元素铁、锌、铜等金属及其浓度也可以在参数中进行设置。目前,本文作者也尝试着采用分子动力学模拟的方法,观察Na+、Cl-离子浓度不同时,人类神经元钙传感蛋白结构的不同变化及对蛋白功能的影响。
另外,人体内的pH值也是一个对运动非常敏感的指标。人体适宜的酸碱度用动脉血pH值表示,范围为7.35~7.45,平均为7.40。但某些情况下可引起酸碱超负荷,严重不足或调节机能障碍,导致体内环境稳态破坏,形成酸碱平衡紊乱。尽管乳酸生成增多不是降低短时间最大强度运动能力的最主要因素,但肌细胞内酸度的改变会明显降低骨骼肌收缩能力。在分子动力学模拟中,pH值作为一个参数,既可以设定一个范围,也可以设置成具体数值,是易于操作的参数。但pH值的改变,对研究对象有一定要求,即研究对象中需含有对pH值敏感的组氨酸(His),pH值的改变才有意义。
5 将分子动力学模拟方法引入体育领域研究的前景
5.1 推动运动干预与机制研究的纵深发展
在体育领域研究中,对运动干预与机制的研究多采用实验方法,但实验方法和手段容易受到时间和空间分辨的局限以及研究对象复杂性的限制,导致实验上提出的很多运动干预理论假设和机制无法得到多方位的检验。分子动力学模拟方法能够从原子水平上观察并解释实验现象,对理论假设和(或)机制进行验证,为实验研究提供佐证;同时,也可以预测新的结构和现象,为建立新的运动干预理论模型和作用机制提供参考和启示。将实验研究的手段和方法与分子动力学模拟方法密切结合起来,拓展目前所进行研究的广度和深度,共同推动运动干预与机制研究的纵深发展,具有理论和现实意义。
5.2 开辟新的研究领域
采用分子动力学模拟方法进行体育领域的科学研究时,研究对象可以是功能性蛋白整体或者片段、蛋白与蛋白的相互作用、蛋白与小分子之间的相互作用、蛋白与细胞膜的相互作用、运动代谢产物的上游蛋白,也可以是在WOS数据库中检索出来的进行定量分析的分子动力学模拟关键词中出现频率较高的细胞膜、复合物、大肠杆菌、核酸、薄膜等,以探索关键词揭示的研究对象的结合、吸附、扩散、运输、转变、生长、变形、识别、聚集等方面的生理机制。另外,在确定研究问题和研究对象后,可以通过改变分子动力学模拟参数的设置而改变研究对象的周围环境,观察其在不同环境下生理结构和功能的变化,不断拓展新的研究视角和研究领域。
5.3 进一步提高体育领域研究的学科交叉
分子动力学模拟方法被广泛应用到化学、物理、生物学、材料科学、科学技术、工程学、计算机科学、药理学、力学、医学等各个领域(图4),研究对象极其广泛(表1),文献发表数目庞大(图2)。目前,体育领域的研究非常注重学科交叉,而学科交叉不仅体现在研究内容和研究对象上,更重要的是体现在研究方法和手段上,因此,有必要将分子动力学模拟的方法引入体育领域的研究中,实现与多学科的融合,为体育领域研究专业化探索一条创新的途径。
[1]郭浙斌.低氧下运动及补液对集体体液平衡和有氧运动能力的影响[D].广州:华南师范大学,2007:23-24.
[2]孟照海.中国教育创新话语的形成和重建[J].现代教育管理,2011,(2):1-4.
[3]周满生.国际竞争视角下中、印、美的教育创新与人才培养[J].北京大学教育评论,2007,(5):157-166,192.
[4]ALDER B J,WAINWRIGHT T E.Studies in molecular dynamics.I.General method[J].J Chem Phys,1959,31:459-466.
[5]ALDER B W.Phase transition for a hard sphere system[J].J Chem Phys,1957,27:1208.
[6]ANDOH Y,YOSHII N,FUJIMOTO K,etal.Modylas:A highly parallelized general-purpose molecular dynamics simulation program for large-scale systems with long-range forces calculated by fast multipole method(FMM) and highly scalable fine-grained new parallel processing algorithms[J].J Chem Theory Comput,2013,9(7):3201-3209.
[7]AXELROD Y K,DIRINGER M N.Temperature management in acute neurologic disorders[J].Neurol Clin,2008,26:585-603.
[8]BAKER J L,VOTH G A.Effects of ATP and actin-filament binding on the dynamics of the myosin II S1 domain[J].Biophys J,2013,105:1624-1634.
[9]BERENDSEN H J C,POSTMA J P M,VAN GUNSTEREN W F,etal.Molecular dynamics with coupling to an external bath[J].J Chem Phys,1984,81:3684-3690.
[10]CAGIN T,PETTITT B M.Molecular dynamics with a variable number of molecules[J].Mol Phys,1991,72:169-175.
[11]CAR R,PARRINELLO M.Unified approach for molecular dynamics and density-functional theory[J].Phys Rev Letters,1985,55:2471-2474.
[12]CHU J W,VOTH G A.Allostery of actin filaments:molecular dynamics simulations and coarse-grained analysis[J].Proc Natl Acad Sci U S A,2005,102:13111-13116.
[13]CREMER O L,KALKMAN C J.Cerebral pathophysiology and clinical neurology of hyperthermia in humans[J].Prog Brain Res,2007,162:153-169.
[14]DROR R O,ARLOW D H,MARAGAKIS P,etal.Activation mechanism of the β2-adrenergic receptor.[J].Proc Natl Acad Sci U S A,2011,108(46):18684-18689.
[15]DRUMOND L E,MOURAO F A,LEITE H R,etal.Differential effects of swimming training on neuronal calcium sensor-1 expression in rat hippocampus/cortex and in object recognition memory tasks[J].Brain Res Bull,2012,88:385-391.
[16]DUAN Y,KOLLMAN P A.Pathways to a protein folding intermediate observed in a 1-microsecond simulation in aqueous solution[J].Sci,1998,282:740-744.
[17]FAN J,SAUNDERS M G,VOTH G A.Coarse-graining provides insights on the essential nature of heterogeneity in actin filaments[J].Biophys J,2012,103:1334-1342.
[18]GILLAN M J,DIXON M.The calculation of thermal conductivities by perturbed molecular dynamics simulation[J].J Phys C:Solid State Phys,1983,16:869.
[19]GUMBART J,TRABUCO L G,SCHREINER E,etal.Regulation of the protein-conducting channel by a bound ribosome[J].Structure,2009,17(11):1453-1464.
[20]HESS B K,KUTZNER C,VAN DER SPOEL D,etal.Gromacs 4:Algorithms for highly efficient,load-balanced,and scalable molecular simulation[J].J Chem Theory Comput,2008,4:435-447.
[21]HOLMES K C,SCHRODER R R,SWEENEY H L,etal.The structure of the rigor complex and its implications for the power stroke[J].Philos Trans R Soc,B,2004,359:1819-1828.
[22]HOOVER W G.Canonical dynamics:Equilibrium phase-space distributions[J].Phys Rev A,1985,31:1695-1697.
[23]JARERATTANACHAT V,KARTTUNEN M,WONG EK-KABUT J.Molecular dynamics study of oxidized lipid bilayers in NaCl solution[J].J Phys Chem B,2013,117:8490-8501.
[24]LAUPLAND K B.Fever in the critically ill medical patient[J].Crit Care Med,2009,37:S273-S278.
[25]LEE H,KIM S M,JEON T J.Effects of imidazolium-based ionic liquids on the stability and dynamics of gramicidin A and lipid bilayers at different salt concentrations[J].J Mol Graph Model,2015,61:53-60.
[26]LORENZ M,HOLMES K C.The actin-myosin interface[J].Proc Natl Acad Sci U S A,2010,107:12529-12534.
[27]MASUDA T.Molecular dynamics simulation of a myosin subfragment-1 docking with an actin filament[J].Bio Systems,2013,113:144-148.
[28]MASUDA T.Molecular dynamics simulation for the reversed power stroke motion of a myosin subfragment-1[J].Bio Systems,2015,132-133:1-5.
[29]MEI C,SUN Y,ZHENG G,etal.Enabling and scaling biomolecular simulations of 100 million atoms on petascale machines with a multicore-optimized message-driven runtime[C]// International Conference for High Performance Computing,Networking,Storage and Analysis,ACM,2011:1-11.
[30]NOSE S.A molecular dynamics method for simulations in the canonical ensemble[J].Mol Phys,1984,52:255-268.
[31]NOSE S,KLEIN M L.Constant pressure molecular dynamics for molecular systems[J].Mol Phys,1983,50:1055-1076.
[32]ODA T,IWASA M,AIHARA T,etal.The nature of the globular- to fibrous-actin transition[J].Nature,2009,457:441-445.
[33]OKAZAKI K,SATO T,TAKANO M.Temperature-enhanced association of proteins due to electrostatic interaction:a coarse-grained simulation of actin-myosin binding[J].J Am Chem Soc,2012,134:8918-8925.
[34]PARRINELLO M,RAHMAN A.Polymorphic transitions in single crystals:A new molecular dynamics method[J].J Appl Phys,1981,52:7182-7190.
[35]RAHMAN A.Correlations in the motion of atoms in liquid argon[J].Phys Rev,1964,136:A405-A411.
[36]SHAW D E,MARAGAKIS P,LINDORFF-LARSEN K,etal.Atomic-level characterization of the structural dynamics of proteins[J].Sci,2010,330(6002):341-346.
[37]SHAW D E,GROSSMAN J P,BANK J A,etal.Anton 2:raising the bar for performance and programmability in a special-purpose molecular dynamics supercomputer[C]// High Performance Computing,Networking,Storage and Analysis,SC14:International Conference for IEEE,2014:41-53.
[38]SHRIVASTAVA I H,SANSOM M S.Simulations of ion permeation through a potassium channel:molecular dynamics of KcsA in a phospholipid bilayer[J].Biophys J,2000,78:557-570.
[39]SPLETTSTOESSER T,HOLMES K C,NOE F,etal.Structural modeling and molecular dynamics simulation of the actin filament[J].Proteins,2011,79:2033-2043.
[40]SWOPE W C,ANDERSEN H C,BERENS P H,etal.A computer simulation method for the calculation of equilibrium constants for the formation of physical clusters of molecules:Application to small water clusters[J].J Chem Phys,1982,76:637-649.
[41]WILBORN C D,WILLOUGHBY D S.The role of dietary protein intake and resistance training on Myosin heavy chain expression[J].J Int Soc Sports Nutr,2004,1:27-34.
[42]WRIGGERS W,SCHULTEN K.Stability and dynamics of G-actin:back-door water diffusion and behavior of a subdomain 3/4 loop[J].Biophys J,1997,73:624-639.[43]ZHONG Q,JIANG Q,MOORE P B,etal.Molecular dynamics simulation of a synthetic ion channel[J].Biophys J,1998,74:3-10.[44]ZHU Y Z,MA B Y,QI R X,etal.Temperature-dependent conformational properties of human neuronal calcium sensor-1 protein revealed by all-atom simulations[J].J Phys Chem B,2016,120:3551-3559.
[45]ZHU Y Z,WU Y,LUO Y,etal.R102Q mutation shifts the salt-bridge network and reduces the structural flexibility of human neuronal calcium sensor-1 protein[J].J Phys Chem B,2014,118:13112-13122.
[46]ZHU Y Z,YANG S,QI R X,etal.Effects of the C-terminal tail on the conformational dynamics of human neuronal calcium sensor-1 protein[J].J Phys Chem B,2015,119:14236-14244.
Feasibility of Molecular Dynamics Simulation Method in the Sports Research Fields—Taking Actin,Myosin and Human Neuronal Calcium Sensor-1 Protein as Example
ZHU Yu-zhen1,ZOU Yu2,ZHANG Qing-wen2
moleculardynamicssimulation;sports;research;physiologicalmechanism;protein
1000-677X(2017)01-0090-08
10.16469/j.css.201701009
体育科学
2016-10-19;
2017-01-04
上海体育学院研究生教育创新科研项目资助 (yjscx2015003)。
朱玉珍,女,讲师,博士,主要研究方向为运动相关蛋白结构与功能,E-mail:zhuyuzhen@shnu.edu.cn;邹昱,男,在读博士研究生,主要研究方向为运动相关蛋白结构与功能,E-mail:zouyu_1993@163.com;张庆文,男,教授,博士,博士研究生导师,主要研究方向为运动相关蛋白结构与功能,E-mail:zqw@sus.edu.cn。
1.上海师范大学 体育学院,上海 200234;2.上海体育学院 体育教育训练学院,上海 200438 1.Shanghai Normal University,Shanghai 200234,China;2.Shanghai University of Sport,Shanghai 200438,China.
G804.2
A
