广州地区猫泛白细胞减少症病毒分离鉴定及全基因组遗传分析
2017-02-09洪马林李守军
洪马林,付 橙,黄 三,周 沛,粟 硕,李守军
(华南农业大学 兽医学院/广东省兽医临床重大疾病综合防控重点实验室/广东省宠物工程技术研究中心,广东 广州 510642)
广州地区猫泛白细胞减少症病毒分离鉴定及全基因组遗传分析
洪马林,付 橙,黄 三,周 沛,粟 硕,李守军
(华南农业大学 兽医学院/广东省兽医临床重大疾病综合防控重点实验室/广东省宠物工程技术研究中心,广东 广州 510642)
【目的】分析广州地区猫泛白细胞减少症病毒(Feline panleukopenia virus, FPLV)的自然重组、跨宿主传播以及流行变异情况。【方法】采集疑似感染猫泛白细胞减少症病毒的猫粪便样品进行细胞分离,并对阳性病料提取基因组DNA,PCR扩增获得病毒的全基因组序列,并与GenBank中相关的参考毒株序列进行遗传进化分析,同时对VP2基因与NS1基因的主要氨基酸位点进行差异分析。【结果】成功获得2株猫泛白细胞减少症病毒及其全基因组序列。遗传进化分析显示,广州地区分离的猫泛白细胞减少症病毒FPLV-GZ01、FPLV-GZ02与我国其他分离毒株FPLV-XJ01、FPLV-HRB属同一分支,提示FPLV-GZ01、FPLV-GZ02由FPLV-XJ1进化而来;NS1基因系统进化树显示,FPLV存在与CPV-447重组的可能。主要氨基酸位点分析显示,FPLV在VP2基因中的主要氨基酸位点上的遗传变异比CPV保守;在NS1基因上发现FPLV-GZ01、FPLV-GZ02分别存在不同程度的氨基酸位点突变。【结论】广州地区分离的猫泛白细胞减少症病毒仍在不断地重组进化。
猫泛白细胞减少症病毒; 细小病毒; 分离鉴定; 全基因组; 遗传分析
细小病毒(Parvovirus)是一种单股负链的线状DNA病毒,属于细小病毒科Parvoviridae细小病毒属Parvovirus,病毒粒子在电子显微镜下呈圆形或六边形,无囊膜结构,核衣壳为轴对称的二十面体[1]。随着细小病毒的不断遗传进化,病毒基因组序列也在不断地发生突变,其宿主特异性、跨物种传播、自然重组等生物学特性存在改变的可能。细小病毒基因组含有2个开放阅读框(ORF):第1个ORF位于基因组的5′端,编码非结构蛋白NS1和NS2;第2个ORF位于基因组的3′端,编码结构蛋白VP1和VP2。2个ORFs分别含有各自独立的启动子,非结构蛋白和结构蛋白的mRNA终止于共同的Poly(A)信号,并且可以通过mRNA的可变剪接形成不同的翻译模板。VP2蛋白是衣壳蛋白的主要成分,是主要的抗原蛋白,具有凝血活性和介导细小病毒感染宿主细胞的作用。NS1蛋白是最主要的非结构蛋白,对细小病毒的复制具有重要的调节作用。病毒基因的中间部分,存在着500 bp的间隔区;而基因组的两侧,存在着与复制密切相关的发夹结构,并且高度磷酸化,这种结构可能与保护病毒基因组不被降解有关[2-3]。
猫泛白细胞减少症是由猫泛白细胞减少症病毒(Feline panleukopenia virus,FPLV)引起的一种高度接触性急性传染病,以高热、呕吐、白细胞严重减少和肠炎为特征,而犬细小病毒病是由犬细小病毒(Canine parvovirus,CPV) 引起的犬科动物的一种急性传染病,以剧烈呕吐、 出血性肠炎、白细胞显著减少及心肌炎为主要特征,幼犬较易发病。FPLV和CPV均属于细小病毒属成员,它们在宿主特异性上存在一定的差异。FPLV是目前细小病毒属中感染范围最宽、致病性最强的一种[4]。FPLV在自然条件下感染猫科和鼬科以及多种野生动物,如虎、豹、狮子和浣熊,但以体型较小的猫科动物最为易感。此外,在猴子中也分离出FPLV。CPV主要感染犬、猫[5-6]。随着宿主间的相互接触以及细小病毒的不断遗传变异,宿主范围也出现了一定的变化,CPV的宿主范围由最开始的单一犬群感染向犬、猫科动物等多种动物感染发展。在进化过程中,CPV出现了CPV-2、CPV-2a、new CPV-2a、CPV-2b、new CPV-2b、CPV-2c等基因型,而目前鲜见对FPLV进行基因分型,这可能与CPV的VP2基因核苷酸突变率远高于FPLV有关[7-8]。病毒VP2基因的一些突变使得病毒能够有效感染新的宿主,并在其间传播,适应性进化的逐渐积累导致了细小病毒的广泛流行[9]。此外,由于FPLV的基因组序列较小,因此可将FPLV的遗传进化过程作为研究细小病毒进化以及病毒跨种传播的重要模型[10]。
本试验通过对疑似感染猫泛白细胞减少症病毒的猫粪便样品进行细胞分离,将成功分离到猫泛白细胞减少症病毒的病料提取基因组DNA后,进行全基因组序列测定分析,以弄清猫泛白细胞减少症病毒主要基因VP2与NS1的突变情况,阐明广州地区猫泛白细胞减少症病毒在遗传进化方面的情况。
1 材料与方法
1.1 材料
2014年12月—2015年5月在华南农业大学动物医院采集疑似感染猫泛白细胞减少症病毒的病猫粪便样品11份。
F81细胞系由广东省兽医临床重大疾病综合防控重点实验室保存。
pZeroBack载体、Fast HiFidelity Polymerase高保真酶、DH5α感受态细胞、粪便基因组DNA提取试剂盒均为天根生化科技(北京)有限公司产品;DNA凝胶回收纯化试剂盒为Axygen公司产品;DMEM细胞培养液为Hyclone公司产品。
1.2 方法
1.2.1 猫泛白细胞减少症病毒的分离 称取猫粪便样品0.2 g,用DMEM细胞培养液混匀,制成质量比为1∶9的悬浮液,5 000 r·min-1离心15 min,上清液用0.22 μm的滤膜过滤,收集滤液,置于-20 ℃条件下保存备用。将处理好的上清液按照细胞培养液总体积的1/10接种于F81细胞系,置于37 ℃、CO2体积分数为5%的培养箱中培养,同时设置正常细胞作为对照,每日观察细胞病变(Cytopathic effect,CPE)情况。如未出现CPE,则待细胞长满后进行正常传代。若经过盲传5代后仍不产生CPE,则视为阴性;若CPE达到80%以上,则经-80 ℃/37 ℃反复冻融3次后收毒,置于-80 ℃条件下保存。
1.2.2 引物的设计与合成 根据GenBank上FPLV(登录号:M38246)的参考序列设计6对分段扩增FPLV全基因组序列的引物(表1),设计好的引物送广州华大基因科技股份有限公司进行合成。
表1 猫泛白细胞减少症病毒全基因分段引物
Tab.1 Segmented primers for the complete genome of FPLV

引物名称引物序列(5′→3′)FPLV⁃1F:GAATGATAGGCGGTTTGTGTGR:TCTTCTGCAATTTCTCTGAGCFPLV⁃2F:GCAGAAGATAGTGAATGGGTGR:TTGGTTGTGTATGTTCAGGTCFPLV⁃3F:TTATGACAACTAATGAAAATAR:TTTTAGTTGGTTTTGTTGGTCFPLV⁃4F:TGATACACCAGATCATCCATCR:TATTTGTTTGCCATGTATGTGFPLV⁃5F:GAAAATTCTGTGCCAGTACACR:TACCTTTCCACCAAAAATCTGFPLV⁃6F:AAGACTTCATGTAAATGCACCR:AGATTGATACTTATGGTAAGG
1.2.3 病毒基因组DNA提取、扩增、克隆及序列测定 粪便样品DNA提取参考天根粪便基因组DNA提取试剂盒操作手册,提取的DNA用无核酸酶的水溶解,置-20 ℃条件下保存。利用各分段PCR引物,以提取的基因组DNA为模板,分段扩增全基因组序列。50 μL反应体系:DNA模板2 μL,上、下游引物各1 μL,5×Fast HiFidelity PCR Buffer 10 μL,Fast HiFidelity Polymerase 1 μL,20×Fast PCR Enhancer 2.5 μL,ddH2O补足至50 μL。PCR产物以0.01 g·mL-1琼脂糖凝胶电泳分离,使用DNA凝胶回收试剂盒进行回收;纯化后的目的基因片段与pZeroBack载体连接、转化,将经PCR鉴定为阳性的待测菌液送广州华大基因科技股份有限公司进行序列测定,每个克隆送3个阳性菌液,并对测序正确的菌液提取质粒,-20 ℃条件下保存。
1.2.4 序列测定与分析 将测序获得的序列用DNAstar软件包中的Edit-seq和SeqMan程序进行整理,并拼接成线性的全基因组序列。使用Bioedit软件进行序列匹配和多重序列比对(Clustal W法);MEGA5软件用于遗传进化分析,并与从GenBank上收集的26个参考序列(表2)进行比较,采用Neighbor-joining (NJ,Replication=1 000)法绘制系统进化树。
表2 26株细小病毒参考序列
Tab.2 Reference sequences of 26 parvovirus strains
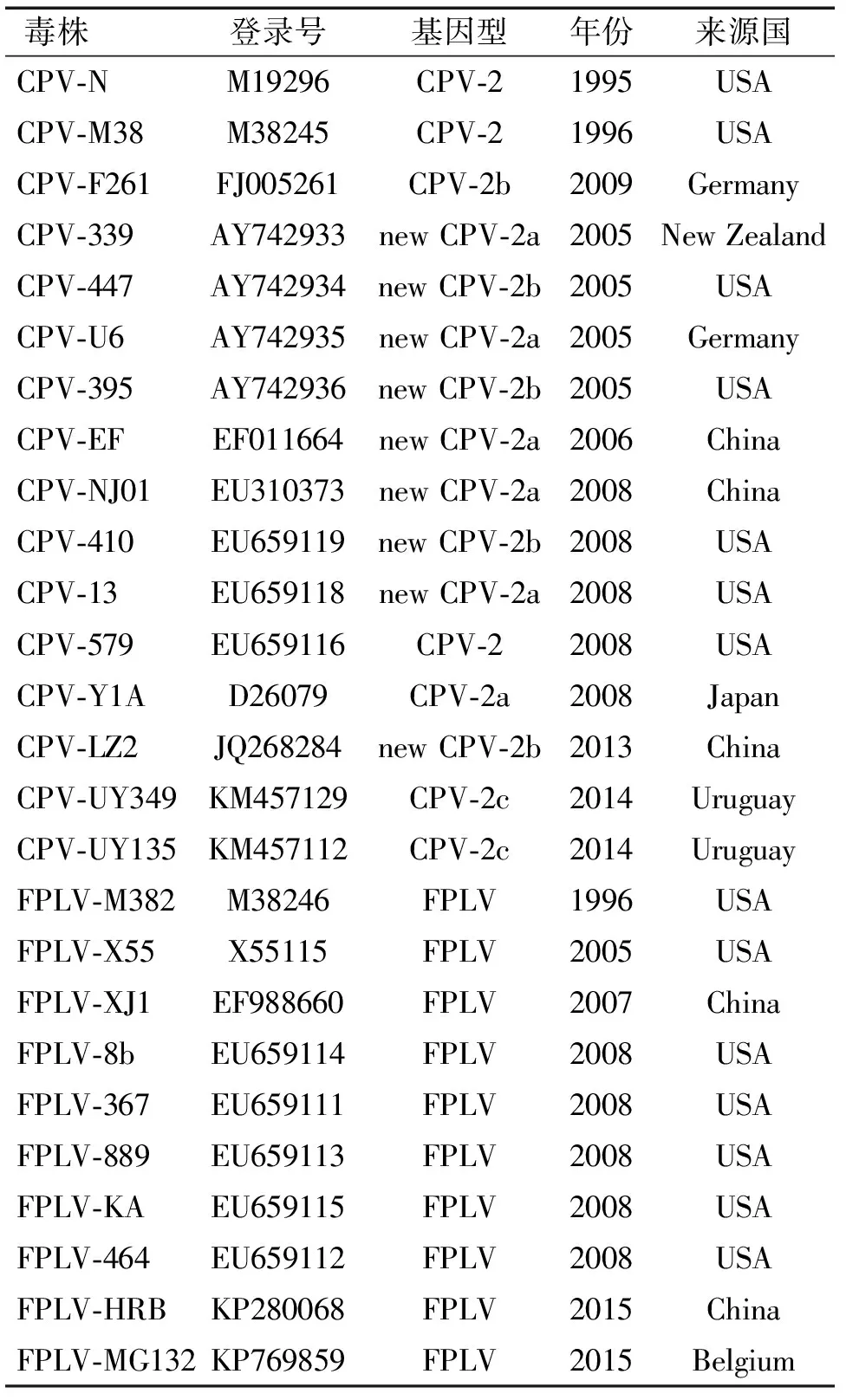
毒株登录号基因型年份来源国CPV⁃NM19296CPV⁃21995USACPV⁃M38M38245CPV⁃21996USACPV⁃F261FJ005261CPV⁃2b2009GermanyCPV⁃339AY742933newCPV⁃2a2005NewZealandCPV⁃447AY742934newCPV⁃2b2005USACPV⁃U6AY742935newCPV⁃2a2005GermanyCPV⁃395AY742936newCPV⁃2b2005USACPV⁃EFEF011664newCPV⁃2a2006ChinaCPV⁃NJ01EU310373newCPV⁃2a2008ChinaCPV⁃410EU659119newCPV⁃2b2008USACPV⁃13EU659118newCPV⁃2a2008USACPV⁃579EU659116CPV⁃22008USACPV⁃Y1AD26079CPV⁃2a2008JapanCPV⁃LZ2JQ268284newCPV⁃2b2013ChinaCPV⁃UY349KM457129CPV⁃2c2014UruguayCPV⁃UY135KM457112CPV⁃2c2014UruguayFPLV⁃M382M38246FPLV1996USAFPLV⁃X55X55115FPLV2005USAFPLV⁃XJ1EF988660FPLV2007ChinaFPLV⁃8bEU659114FPLV2008USAFPLV⁃367EU659111FPLV2008USAFPLV⁃889EU659113FPLV2008USAFPLV⁃KAEU659115FPLV2008USAFPLV⁃464EU659112FPLV2008USAFPLV⁃HRBKP280068FPLV2015ChinaFPLV⁃MG132KP769859FPLV2015Belgium
2 结果与分析
2.1 病毒分离结果
将病料上清液同步接种于F81细胞,并通过带毒传代的方法成功获得了2株FPLV,编号为FPLV-GZ01和FPLV-GZ02。对照组的F81细胞贴壁生长,呈不规则的细胞形态,连接紧密(图1A),而接种病料上清液的试验组则在连续传代3次后出现CPE,其表现为细胞生长缓慢、细胞圆缩、 胞间联系减少、细胞脱落等病变现象(图1B)。
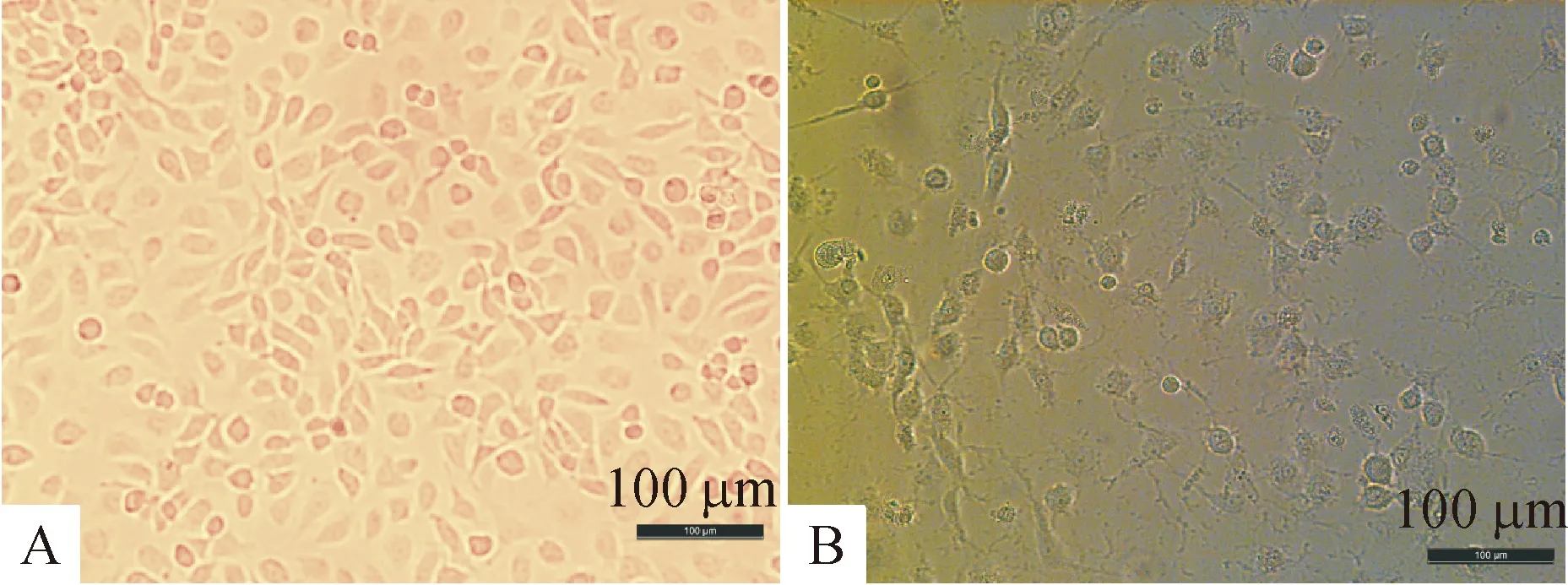
A:对照组的F81细胞;B:试验组的F81细胞。
2.2 序列测定结果及相似性分析
将实验室获得的2株FPLV的核苷酸、氨基酸序列与表2中的参考序列对比分析发现,FPLV-GZ01株NS1基因与其他毒株的核苷酸序列相似性为98.9%~99.6%,氨基酸序列相似性为98.7%~99.4%;VP2基因的核苷酸序列相似性为98.0%~99.5%,氨基酸序列相似性为97.1%~99.8%;全基因组的核苷酸序列相似性为87.6%~99.4%。FPLV-GZ02株NS1基因的核苷酸序列相似性为98.8%~99.8%,氨基酸序列相似性为98.8%~99.7%;VP2基因的核苷酸序列相似性为97.9%~99.8%,氨基酸序列相似性为96.9%~99.8%;全基因组的核苷酸序列相似性为82.4%~99.0%。表明分离毒株FPLV-GZ01、FPLV-GZ02与参考毒株之间的差异较小,相似性高,且NS1基因的核苷酸序列相似性高于VP2基因和全基因组的核苷酸序列相似性。
2.3 VP2、NS1基因及全基因组的核苷酸序列系统进化树分析
将实验室获得的2株FPLV序列与表2中的26株细小病毒序列进行核苷酸序列系统进化树分析(图2~图4)。结果表明,在VP2、NS1基因与全基因组的系统进化树上,分别形成FPLV与CPV 2个大的分支。FPLV-GZ01与FPLV-GZ02均处在FPLV分支上,属于FPLV,其中FPLV-GZ02与FPLV-HRB的亲缘关系最接近,且FPLV-GZ02与FPLV-HRB组成的分支与FPLV-GZ01属于同一分支。此外FPLV-GZ01、FPLV-GZ02与FPLV-HRB组成的分支与FPLV-XJ1接近。
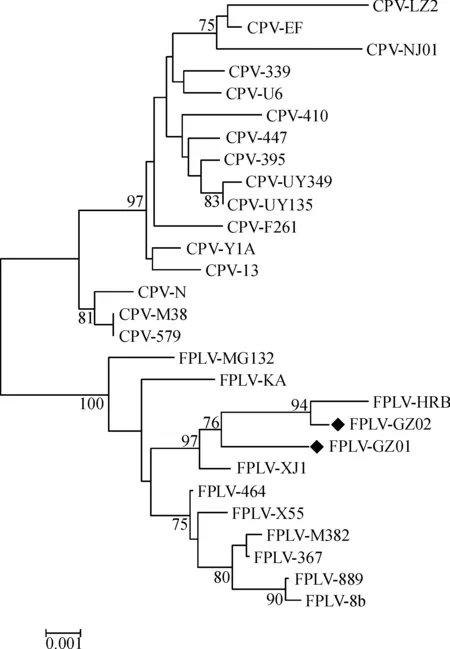
图2 细小病毒VP2基因的核苷酸序列系统进化树
Fig.2 The phylogenetic tree based on nucleotide sequences of parvovirusVP2 gene

图3 细小病毒NS1基因的核苷酸序列系统进化树
Fig.3 The phylogenetic tree based on nucleotide sequences of parvovirusNS1 gene

图4 细小病毒全基因组的核苷酸序列系统进化树
Fig.4 The phylogenetic tree based on nucleotide sequences of the complete genomes of parvovirus
2.4 主要氨基酸位点的分析
分别选取FPLV-M382、CPV-N、CPV-Y1A 、CPV-NJ01、CPV-F261、CPV-410和CPV-UY349作为FPLV、CPV-2、CPV-2a、new CPV-2a、CPV-2b、new CPV-2b和 CPV-2c的参考毒株,并与FPLV-GZ01、FPLV-GZ02进行NS1和VP2基因的主要氨基酸位点比对(表3、表4)。
在VP2基因中主要的氨基酸位点上,与FPLV-M382相比,FPLV-GZ01和FPLV-GZ02没有出现突变,提示FPLV VP2蛋白的功能在进化过程中较少发生改变;与CPV-N、CPV-Y1A、CPV-NJ01、CPV-F261、CPV-410和CPV-UY349相比,FPLV-GZ01、FPLV-GZ02、FPLV-M382在第80、93、103、323、564、568位点上出现氨基酸位点的宿主差异变化,表明VP2蛋白可能在不同宿主上表现出功能差异(表3)。在NS1基因中的部分氨基酸位点上,FPLV-GZ01分别在第546、584位点上出现新的氨基酸突变,FPLV-GZ02仅在第546位点上出现新的氨基酸突变,这些新的突变是否能引起NS1基因的功能甚至是细小病毒的生物学特性改变有待进一步研究(表4)。
表3VP2基因中主要的氨基酸位点分析
Tab.3 Analysis of major amino acid sites of theVP2 gene

毒株氨基酸位点808793103300305323426564568核酸类型FPLV⁃GZ01KMKVADDNNAFPLVFPLV⁃GZ02KMKVADDNNAFPLVFPLV⁃M382KMKVADDNNAFPLVCPV⁃NRMNAADNNSGCPV⁃2CPV⁃Y1ARLNAGYNNSGCPV⁃2aCPV⁃NJ01RLNAGYNNSGnewCPV⁃2aCPV⁃F261RLNAGYNDSGCPV⁃2bCPV⁃410RLNAGYNDSGnewCPV⁃2bCPV⁃UY349RLNAGYNESGCPV⁃2c
表4NS1基因中的部分氨基酸位点分析
Tab.4 Analysis of partial amino acid sites of theNS1 gene
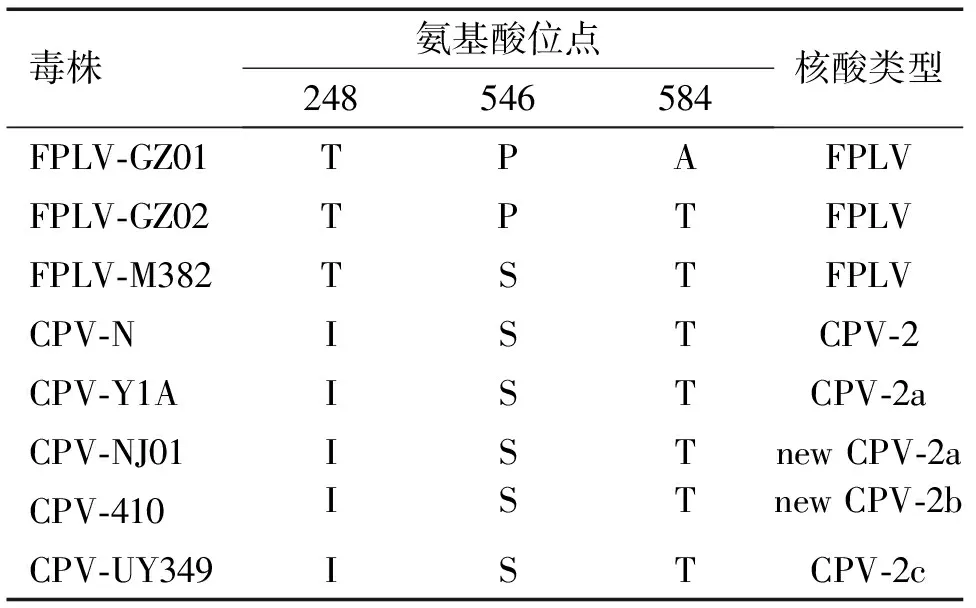
毒株氨基酸位点248546584核酸类型FPLV⁃GZ01TPAFPLVFPLV⁃GZ02TPTFPLVFPLV⁃M382TSTFPLVCPV⁃NISTCPV⁃2CPV⁃Y1AISTCPV⁃2aCPV⁃NJ01ISTnewCPV⁃2aCPV⁃410ISTnewCPV⁃2bCPV⁃UY349ISTCPV⁃2c
3 讨论与结论
本试验采用F81细胞对收集的疑似感染FPLV的猫粪便样品进行细胞分离。与对照组相比,试验组出现了细胞生长缓慢、细胞圆缩、胞间联系减少、细胞脱落等病变现象,与亢文华等[11]的报道相符。由于细小病毒的复制需要在宿主细胞分裂期才能满足宿主细胞为其提供复制所需的酶等条件,因此采取同步接毒的方法分离病毒。通过对FPLV-GZ01和FPLV-GZ02的序列相似性分析,发现在核苷酸相似性、氨基酸相似性分析上,VP2基因比NS1基因变化差异大,表明VP2基因作为细小病毒衣壳的主要结构蛋白在细小病毒分类、宿主特异性等方面具有重要作用。
系统进化树分析发现,广州地区猫泛白细胞减少症病毒分离株FPLV-GZ01、FPLV-GZ02与我国其他地区的猫泛白细胞减少症病毒分离株FPLV-XJ1、FPLV-HRB同在一个大分支上,且该大分支上又形成几个小的分支,显示FPLV-GZ01、FPLV-GZ02、FPLV-HRB毒株可能由早期的FPLV-XJ1毒株遗传进化而来,而FPLV-GZ01、FPLV-GZ02毒株处在FPLV-XJ1与FPLV-HRB毒株之间,是否表明FPLV-HRB毒株可能由广州地区分离株FPLV-GZ01、FPLV-GZ02进化而来,还需要进一步的研究。从宿主差异上来看,VP2、NS1基因与全基因组的系统进化树分析显示,总体上看猫泛白细胞减少症病毒与犬细小病毒单独形成2个大的分支,具有明显的宿主特异性,但NS1基因的系统进化树分析显示CPV-447与FPLV形成的大分支处在同一分支上,这可能是由于CPV-447的NS1基因存在与猫泛白细胞减少症病毒NS1基因重组的可能,这与Ohshima等[12]报道的FPLV-XJ1毒株出现CPV和FPLV的重组进化现象相符。为了进一步证明不同宿主细小病毒出现的重组现象,需要更深入的研究。
细小病毒中的VP2蛋白作为细小病毒衣壳的主要蛋白,在宿主差异方面起到关键作用,本研究选取了10个与宿主范围、抗原性和血凝性相关的VP2基因中的氨基酸位点进行分析,其中第80、564、568位点与FPLV能否在猫体内繁殖有关,而影响CPV在犬内繁殖的氨基酸位点为第93、103、323位点[13-14]。此外,有报道在VP2基因的第300位氨基酸位点上发现天冬氨酸(D)突变,此位点突变株首先报道于越南的猫体内,随后在犬体内也出现此氨基酸位点的突变株[15]。VP2基因的主要氨基酸位点分析结果显示,广州地区分离株FPLV-GZ01、FPLV-GZ02与参考毒株FPLV-M382相比,在这10个氨基酸位点上均一致,并没有出现氨基酸突变,表明FPLV-GZ01、FPLV-GZ02毒株仅在猫体内复制,尚不具备跨犬传播的可能,但VP2基因其他氨基酸位点、病毒其他基因或宿主基因的改变是否会引起细小病毒的跨宿主传播还有待更深入的研究。与CPV相比,FPLV在VP2基因的遗传进化上更为保守,说明猫泛白细胞减少症病毒在遗传进化上相对较慢。在NS1基因的序列比对中,与FPLV-M382相比,发现FPLV-GZ02在第546位点出现由丝氨酸(S)向脯氨酸(P)的突变,而FPLV-GZ01除了在546位点上具有相同的氨基酸突变外,同时还在第584位点上出现了由苏氨酸(T)向丙氨酸(A)的突变,NS1基因中第546、584位氨基酸位点的功能还有待于进一步研究,对于这2个位点的突变是否能引起猫泛白细胞减少症病毒宿主特异性改变、是否能引起与其他细小病毒发生基因重组等情况值得研究。
本研究通过收集疑似感染猫泛白细胞减少症病毒的猫粪便样品进行细胞分离,提取阳性病料的基因组DNA进行测序,对猫泛白细胞减少症病毒进行相似性、遗传进化、主要氨基酸位点的分析,表明广州地区的猫泛白细胞减少症病毒在不断地进化。为此,对猫泛白细胞减少症病毒进行监测,有利于掌握该地区猫泛白细胞减少症病毒的流行变异情况,不仅为该地区猫泛白细胞减少症病毒的预防和控制提供了一定的理论基础和参考依据,而且对研究细小病毒的生物学特性、评估疫苗保护水平等具有重要意义。
[1] REED A P, JONES E V, MILLER T J. Nucleotide sequence and genome organization of canine parvovirus[J]. J Virol,1988, 62(1): 266-276.
[2] AGBANDJE M, MCKENNA R, ROSSMANN M G, et al. Structure determination of feline panleukopenia virus empty particles[J]. Proteins,1993, 16(2): 155-171.
[3] PARRISH C R. Host range relationships and the evolution of canine parvovirus[J]. Vet Microbiol, 1999, 69(1/2): 29-40.
[4] 刘景华. 动物病毒学[M]. 北京:科学出版社, 1985.
[5] IKEDA Y, MOCHIZUKI M, NAITO R, et al. Predominance of canine parvovirus (CPV) in unvaccinated cat populations and emergence of new antigenic types of CPVs in cats[J]. Virology,2000, 278(1): 13-19.
[6] MARTELLA V, DECARO N, ELIA G, et al. Surveillance activity for canine parvovirus in Italy[J]. J Vet Med:Series B,2005, 52(7/8): 312-315.
[7] DECARO N, ELIA G, DESARIO C, et al. A minor groove binder probe real-time PCR assay for discrimination between type 2-based vaccines and field strains of canine parvovirus[J]. J Virol Methods,2006, 136(1/2): 65-70.
[8] HOELZER K, SHACKELTON L A, PARRISH C R, et al. Phylogenetic analysis reveals the emergence, evolution and dispersal of carnivore parvoviruses[J]. J Gen Virol,2008, 89(9): 2280-2289.
[9] STEINEL A, MUNSON L, VAN VUUREN M, et al. Genetic characterization of feline parvovirus sequences from various carnivores[J]. J Gen Virol, 2000, 81(2): 345-350.
[10]夏咸柱,高玉伟,王化磊,等. 略谈人兽共患病跨种传播与监测及免疫研究中的几个问题[J]. 中国病毒病杂志,2012,2(1): 4-10.
[11]亢文华,赵凤龙,郝霖雨,等. 猫泛白细胞减少症病毒的分离与鉴定[J]. 中国畜牧兽医,2008,35(10): 89-92.
[12]OHSHIMA T, MOCHIZUKI M. Evidence for recombination between feline panleukopenia virus and canine parvovirus type 2[J]. J Vet Med Sci,2009, 71(4): 403-408.
[13]LLAMAS-SAIZ A L, AGBANDJE-MCKENNA M, PARKER J S, et al. Structural analysis of a mutation in canine parvovirus which controls antigenicity and host range[J]. Virology,1996, 225(1): 65-71.
[14]TRUYEN U, AGBANDJE M, PARRISH C R. Characterization of the feline host range and a specific epitope of feline panleukopenia virus[J]. Virology,1994, 200(2): 494-503.
[15]KANG B K, SONG D S, LEE C S, et al. Prevalence and genetic characterization of canine parvoviruses in Korea[J]. Virus Genes,2008, 36(1): 127-133.
【责任编辑 李晓卉】
Isolation,identification and sequence analysis of complete genome of feline panleukopenia virus in Guangzhou area
HONG Malin,FU Cheng,HUANG San,ZHOU Pei,SU Shuo,LI Shoujun
( College of Veterinary Medicine,South China Agricultural University/Guangdong Provincial Key Laboratory of Prevention and Control for Severe Clinical Animal Diseases/Guangdong Technological Engineering Research Center for Pet,Guangzhou 510642,China)
【Objective】 To study the natural recombination,cross host transmission and prevalence of feline panleukopenia virus(FPLV)in Guangzhou area.【Method】Cells were isolated from stool samples of cats seemingly infected with FPLV,genome DNAs were extracted from positive samples,and complete virus genome sequences were amplified by PCR and were compared with relevant reference sequences from GenBank using genetic evolution analysis. The major amino acid sites were analyzed for differences betweenVP2 gene andNS1 gene. 【Result】Two FPLV strains were successfully isolated and the complete genome sequences of FPLV were obtained. The genetic evolution analysis indicated that FPLV-GZ01 and FPLV-GZ02 which were isolated from Guangzhou area belonged to the same branch with the other isolated strains FPLV-XJ1 and FPLV-HRB, and implied FPLV-GZ01 and FPLV-GZ02 evolved from FPLV-XJ01. TheNS1 gene phylogenetic analysis demonstrated that FPLV might have recombined with CPV-447. Analysis of major amino acid sites indicated that FPLV was more conservative than CPV in the genetic variation of theVP2 gene. FPLV-GZ01 and FPLV-GZ02 had different degrees of mutation in the amino acid sites of theNS1 gene. 【Conclusion】The feline panleukopenia viruses isolated from Guangzhou area have been still recombining and evolving.
feline panleukopenia virus; parvovirus; isolation and identification; complete genome; genetic analysis
2016- 03- 09优先出版时间:2016-12-28
洪马林(1989—),男,硕士研究生,E-mail:malinhongmark@foxmail.com; 通信作者:李守军(1968—),男,教授,博士,E-mail:shoujunli@scau.edu.cn
国家重点研发计划(2016YFD0501004);公益性行业(农业)科研专项(201303042)
S852.65
A
1001- 411X(2017)01- 0009- 06
优先出版网址:http://www.cnki.net/kcms/detail/44.1110.s.20161228.0937.026.html
洪马林,付 橙,黄 三,等.广州地区猫泛白细胞减少症病毒分离鉴定及全基因组遗传分析[J].华南农业大学学报,2017,38(1):9- 14.
