希金斯炭疽菌磷脂酰肌醇特异性磷脂酶C生物信息学分析
2017-02-05韩长志
doi:10.15889/j.issn.1002-1302.2016.10.046
摘要:希金斯炭疽菌侵染菜心、萝卜等十字花科植物引起炭疽病,给各国农业生产造成了巨大的经济损失。基于酿酒酵母典型的磷酸肌醇特异性磷脂酶C(PI-PLC)序列,利用Blastp以及关键词对该菌蛋白质数据库进行比对、搜索,以及通过SMART分析,明确该菌存在8个典型的PI-PLC序列;通过生物信息学分析,明确上述PI-PLC序列在蛋白质二级结构组成、信号肽等方面均具有一定差异。遗传关系比较分析明确该菌中的PI-PLC序列与禾谷炭疽菌(Colletorichum graminicola)、松针炭疽菌(C. fioriniae)等炭疽菌属中的病菌具有较近的亲缘关系。
关键词:希金斯炭疽菌;磷脂酰肌醇特异性磷脂酶C;亚细胞定位;二级结构;炭疽菌属
中图分类号:S432.4文献标志码: A文章编号:1002-1302(2016)10-0177-04
收稿日期:2015-08-28
基金项目:国家自然科学基金(编号:31560211);云南省森林灾害预警与控制重点实验室开放基金(编号:ZK150004);云南省优势特点重点学科生物学一级学科建设项目(编号:50097505);云南省高校林下生物资源保护及科用科技创新团队项目(编号:2014015);云南省教育厅科学研究基金(编号:2014Y330)。
作者简介:韩长志(1981—),男,河北石家庄人,博士,讲师,主要从事经济林木病害生物防治与真菌分子生物学研究。Tel:(0871)63862918;E-mail:hanchangzhi2010@163.com。植物与病原菌互作过程中,众多细胞信号转导途径参与其中,现已明确G蛋白信号途径及其下游的MAPK信号传导途径和cAMP信号传导途径具有重要的作用。因此,学术界关于真菌RGS的研究产生了较多成果[1],磷酸肌醇特异性磷脂酶C(PI-PLC)的一种异构体PI-PLC β为磷脂肌醇信号途径PKC系统中的重要效应物[2],可以将4,5-二磷酸磷脂酰肌醇水解为三磷酸肌醇和二酰甘油,而上述分子均作为细胞信号转导的第二信使,发挥着重要作用。
希金斯炭疽菌(Colletotrichum higginsanum Sacc.)可以侵染菜心、萝卜等十字花科蔬菜引起炭疽病,该菌现主要分布于美国、日本、印度等国家[3-5],在我国,由该菌侵染菜心引起的炭疽病成为影响菜心产量和品质的重要危害之一[6]。国内外对该菌的研究主要集中在病菌的生物学特性[7]、生防菌筛选[8]、遗传转化[9-10]以及不同营养元素(硅、氮、钾等)[11-14]、外源酚酸类物质pHBA[15]控制病害等方面,同时,随着该基因组序列的释放[16],林春花等对其开展了MAPK途径相关基因的找寻及信号通路简图的绘制工作[17],韩长志对其进行了RGS[18]、14-3-3[19]、septin[20]、AC、PITP等蛋白生物信息学分析。此外,对于该菌致病基因的研究,建立根癌农杆菌介导的转化技术及直接重复重组介导的基因打靶等技术[9-10]。华中农业大学黄俊斌教授实验室建立了希金斯炭疽菌转化子的突变体库,为进一步对这些转化子基因功能研究提供了技术支持[21-22]。然而,对于该菌中存在的PI-PLC数量、功能尚不清楚。本研究利用酿酒酵母S288c(Saccharomyces cerevisiae S288c)中已经报道的PI-PLC氨基酸序列,通过在炭疽菌属蛋白质数据库中进行Blastp比对分析[23],以及通过关键词搜索,获得与酿酒酵母PI-PLC同源的希金斯炭疽菌序列,并通过保守结构域分析、疏水性分析、二级结构预测位等生物信息学分析以及遗传关系分析,以期为进一步开展希金斯炭疽病菌的研究提供重要的指导。
1材料与方法
1.1材料
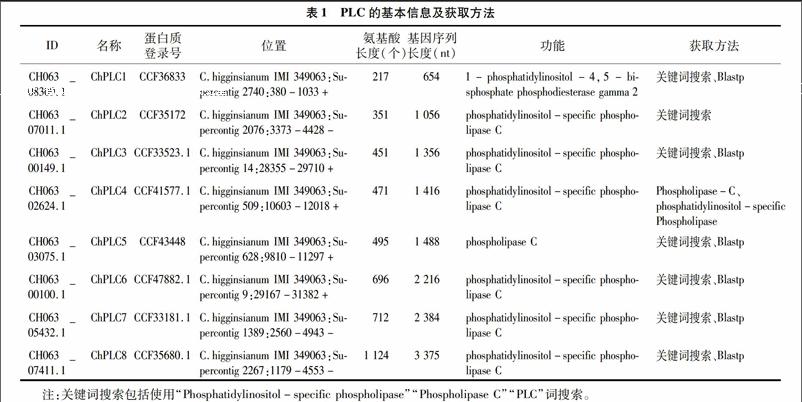
根据酿酒酵母S288c中含有的1个PI-PLC氨基酸序列,利用炭疽菌属蛋白质数据库在线Blastp比对,同时通过输入“phosphatidylinositol-specific Phospholipase”“Phospholipase C”“PLC”关键词在上述数据库中进行检索PLC(表1)。
1.2方法
1.2.1保守结构域预测利用SMART网站[24]在线分析 PI-PLC所具有的保守结构域特征。
1.2.2蛋白质疏水性预测利用Protscale程序[25]对PI-PLC进行疏水性测定。
1.2.3蛋白质转运肽及信号肽预测对蛋白质转运肽的预测利用TargetP 1.1 Server在线分析实现[26],信号肽的预测则是利用SignalP 3.0 Server[27]在线分析实现。
1.2.4蛋白质二级结构及跨膜区结构预测对蛋白质二级结构预测采用PHD[28]在线分析实现,利用TMHMM Server v. 2.0[27]实现PI-PLC的跨膜区结构预测。
1.2.5亚细胞定位分析利用ProtComp v 9.0实现PI-PLC的亚细胞定位分析[29]。
1.2.6系统进化树构建在NCBI中,以希金斯炭疽菌中所含PI-PLC氨基酸序列为基础,在线进行Blastp同源搜索,获得来自不同物种的同源蛋白质序列。对所获得的同源序列,利用ClustalX[30]进行多重比对分析,随后利用 MEGA 5.2.2 软件[31]构建系统进化树。
2结果与分析
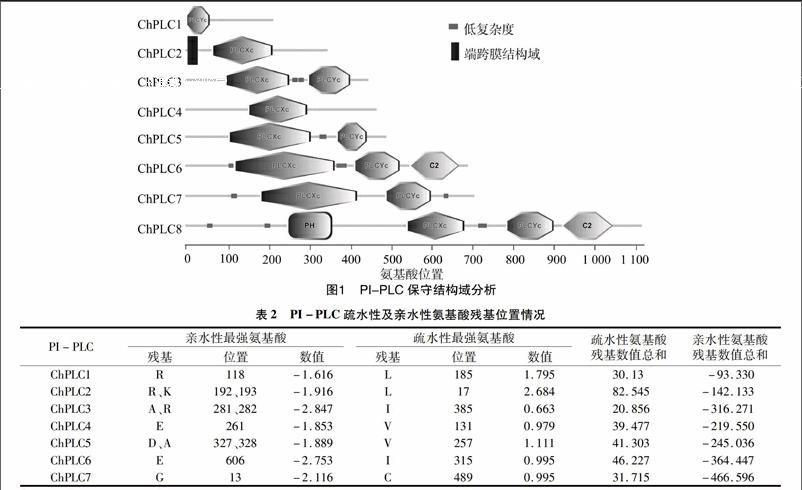
2.1希金斯炭疽菌中8个PI-PLC均具有典型的PLCXc或者PLCYc保守结构域
通过Blastp比对分析,共获得10个同源序列,同时,利用3个关键词搜索,分别获得8、9、7个蛋白序列,对上述蛋白进行整理、去除重复,共获得9个蛋白,其ID分别为CH063_08309.1、CH063_07411.1、CH063_07011.1、CH063_05432.1、CH063_04045.1、CH063_03075.1、CH063_02624.1、CH063_00149.1、CH063_00100.1(表1)。其中,发现一个蛋白序列(CH063_04045.1)仅可以通过Phospholipase-C搜索到,不能通过“phosphatidylinositol-specific Phospholipase”“PLC”搜索到,该蛋白通过Blastp也未能比对到,有待于进一步核实;随后,利用SMART在线分析,结果显示,仅有8条序列具有典型的PLCXc保守域结构,同时还具有诸如PLCYc、C2等保守结构域(图1)。
对CH063_04045.1进行深入分析,明确该蛋白为肌醇鞘磷脂磷脂酶C,并不是G蛋白下游的效应因子,同时,基于SMART分析,该蛋白并不含有磷脂酶所含有的保守PLCXc或者PLCYc结构域,因此将该蛋白排除。根据上述分析,共获得8条蛋白序列,依据其氨基酸大小进行排序,分别将其命名为ChPLC1、ChPLC2、ChPLC3、ChPLC4、ChPLC5、ChPLC6、ChPLC7、ChPLC8(表1)。
2.2ChPLC2具有典型的信号肽区域
通过SignalP分析,除ChPLC2在N端含有1个典型信号肽序列外,其他PI-PLC均不含有信号肽;同时,通过TMHMM分析,除ChPLC2在N端含有1个跨膜结构域外,其他PI-PLC均不含有跨膜结构域(图1),鉴于TMHMM不能将蛋白中所含的信号肽与跨膜结构域严格区分,因此,ChPLC2在N端含有的序列结构是信号肽,而不是由TMHMM、SMART程序所预测的跨膜结构域(图1)。
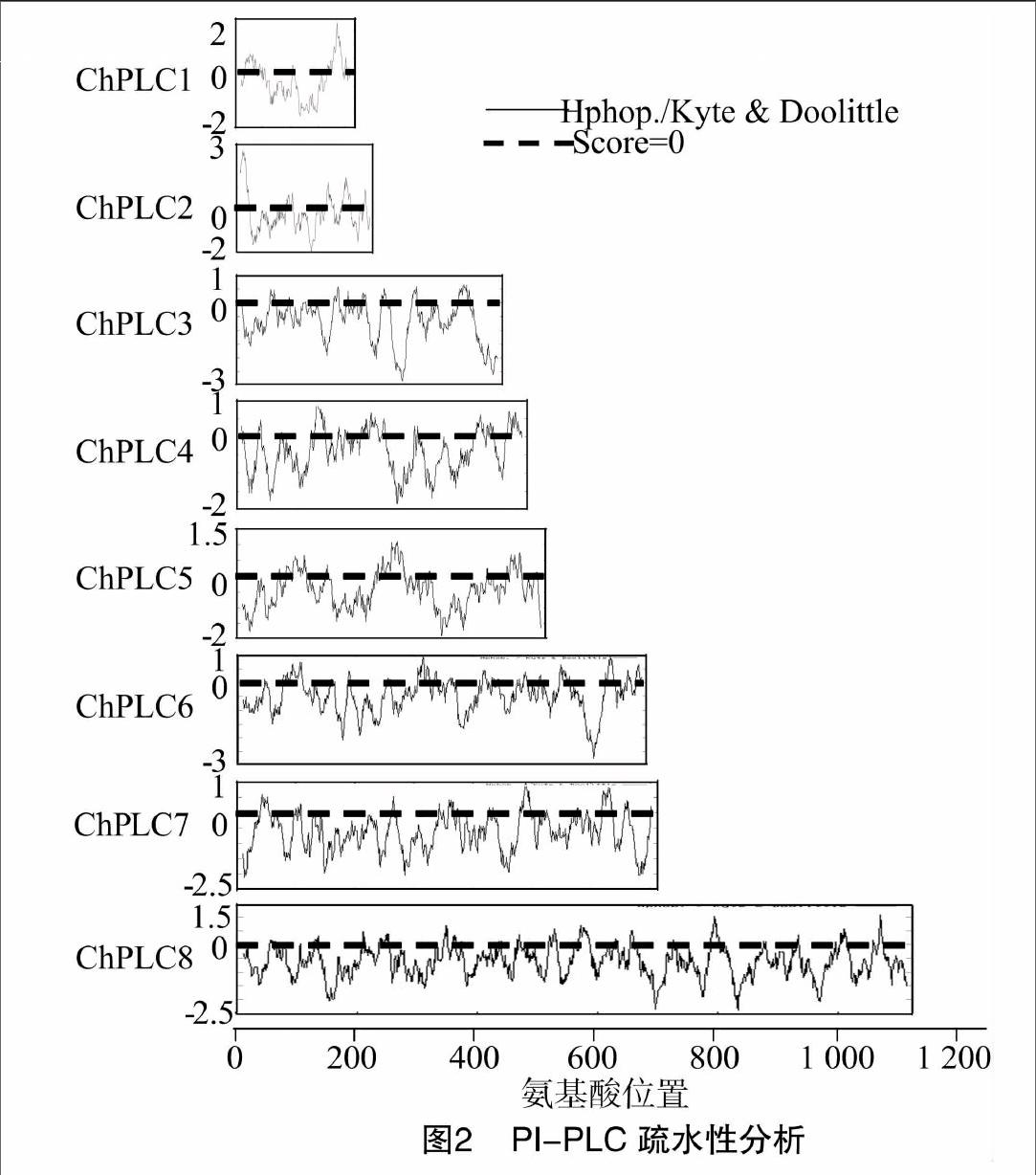
2.3希金斯炭疽菌PI-PLC均为亲水性蛋白
通过疏水性分析,明确ChPLC1和ChPLC2、ChPLC3和ChPLC6、ChPLC4和ChPLC5在疏水性最强氨基酸残基上相同,分别为L、I、V;ChPLC1和ChPLC8、ChPLC4和ChPLC6在亲水性最强氨基酸残基上相同,分别为R、E(表2)。尽管如此,其他PI-PLC彼此之间在亲(疏)水性最强氨基酸残基及位置方面仍然存在较大的差异(表2、图2),推测在细胞信号转导过程中,PI-PLC所具有的功能存在一定的差异性。
2.4二级结构分析
明确8个PI-PLC均含有无规卷曲、α-螺旋以及β-转角,所占比例不尽相同。就所含的无规卷曲比例而言,ChPLC2最低,为17%,ChPLC8最高,为45%,其他PI-PLC所含比例集中于20%~35%;就所含的α-螺旋而言,ChPLC1最低,为5%,ChPLC7最高,为41%,其他PI-PLC所含比例集中于20%~40%;就β-转角而言,ChPLC3最低,为5%,ChPLC1最高,为44%,其他PI-PLC所含比例集中于5%~25%(图3)。
2.5转运肽及亚细胞定位分析
转运肽分析结果显示,除ChPLC2明确定位于分泌途径,ChPLC1、ChPLC4、ChPLC7定位于线粒体,其他PI-PLC定位在任何位置(表3)。为了更好地明确PI-PLC的亚细胞定位,利用ProtComp进行分析,结果显示,ChPLC1、ChPLC7定位于线粒体,这与通过TargetP预测结果相同,ChPLC2定位于细胞膜,ChPLC3、ChPLC6定位于细胞核,ChPLC4、ChPLC5、ChPLC7定位于细胞质中(数据未显示)。上述定位预测结果说明希金斯炭疽菌中数量众多的PI-PLC定位情况不尽相同,在细胞信号转导过程中发挥着不同的功能。
2.6遗传关系分析
通过在NCBI中对PI-PLC进行Blastp比对,获得与希金斯炭疽菌PI-PLC的同源序列,选择炭疽菌属真菌其他序列进行遗传关系分析。结果显示,就物种之间关系而言,该菌中的PI-PLC与禾谷炭疽菌、松针炭疽菌亲缘关系较近;就该菌中的PI-PLC之间的关系而言,8个PI-PLC可以大致聚为五大类,具体而言,ChPLC6、ChPLC5、ChPLC7关系较近,为一类;ChPLC3与ChPLC1关系较近,为一类;ChPLC8、ChPLC4、ChPLC6各为一类。
3结论与讨论
随着希金斯炭疽菌全基因组序列的释放[16],为深入解析该菌致病因子研究提供了重要的数据支撑,一些涉及G蛋白信号转导途径上的重要效应物[18,32]以及MAPK途径上的重要蛋白得到进一步明确[17]。本研究基于前人研究成果,明确了该菌中存在7个典型的PI-PLC,初步明确了上述蛋白具有典型的PLCXc、PLCYc、C2等保守结构域、均为亲水性蛋白等特点。尽管如此,希金斯炭疽菌中8个PI-PLC彼此之间在亚细胞定位、信号肽、二级结构组成方面存在一定差异,推测上述蛋白在不同位置发挥不同作用,同时,对上述PI-PLC与其他物种进行遗传关系分析,明确希金斯炭疽菌与松针炭疽菌亲缘关系较近,为进一步深入研究其他炭疽菌属真菌PI-PLC打下坚实的理论基础。通过深入解析希金斯炭疽菌中G蛋白信号转导过程中的致病因子,有助于为开发用于以G蛋白信号通路为作用靶标的化学药剂提供重要的理论意义。
参考文献:
[1]赵勇,王云川,蒋德伟,等. 真菌G蛋白信号调控蛋白的功能研究进展[J]. 微生物学通报,2014,41(4):712-718.
[2]孙大业,崔素娟,孙颖. 细胞信号转导——基础篇[M].4版. 北京:科学出版社,2010.
[3]沈瑞清,张萍,郭成瑾,等. 宁夏炭疽菌属真菌资源研究[J]. 河南农业科学,2012,41(5):100-102,149.
[4]梁惠凌,唐辉. 广西常见花卉真菌性病害的防治[J]. 广西园艺,2002(2):18-19.
[5]Hyde K,Cai L,Cannon P,et al. Colletotrichum—names in current use[J]. Fungal Diversity,2009,39(1):147-182.
[6]卢博彬,杨暹. 菜心炭疽病研究进展[J]. 长江蔬菜,2009(24):1-5.
[7]周而勋,杨媚,张华,等. 菜心炭疽病菌菌丝生长、产孢和孢子萌发的影响因素[J]. 南京农业大学学报,2002,25(2):47-51.
[8]况福元,吴小丽,吕风青,等. 菜心炭疽病菌拮抗细菌的筛选及鉴定[J]. 微生物学通报,2009,36(9):1350-1355.
[9]Huser A,Takahara H,Schmalenbach W,et al. Discovery of pathogenicity genes in the crucifer anthracnose fungus Colletotrichum higginsianum,using random insertional mutagenesis[J]. Molecular Plant-Microbe Interactions,2009,22(2):143-156.
[10]Ushimaru T,Terada H,Tsuboi K,et al. Development of an efficient gene targeting system in Colletotrichum higginsianum using a non-homologous end-joining mutant and Agrobacterium tumefaciens-mediated gene transfer[J]. Molecular Genetics and Genomics,2010,284(5):357-371.
[11]杨暹,冯红贤,杨跃生. 硅对菜心炭疽病发生、菜薹形成及硅吸收沉积的影响[J]. 应用生态学报,2008,19(5):1006-1012.
[12]杨暹,陈晓燕,冯红贤. 氮营养对菜心炭疽病抗性生理的影响Ⅰ.氮营养对菜心炭疽病及细胞保护酶的影响[J]. 华南农业大学学报,2004,25(2):26-30.
[13]陈晓燕,杨暹,张璐璐. 氮营养对菜心炭疽病抗性生理的影响Ⅱ.氮营养对菜心炭疽病及膜脂过氧化作用的影响[J]. 华南农业大学学报,2004,25(3):1-5.
[14]郭巨先,刘玉涛,杨暹. 钾营养对菜薹(菜心)炭疽病发生和植株防御酶活性的影响[J]. 中国蔬菜,2012(14):86-89.
[15]康云艳,周小萌,杨暹,等. pHBA对菜心炭疽病的诱导抗性及植株生理特性的影响[J]. 植物病理学报,2014,44(4):393-404.
[16]OConnell R J,Thon M R,Hacquard S,et al. Lifestyle transitions in plant pathogenic Colletotrichum fungi deciphered by genome and transcriptome analyses[J]. Nature Genetics,2012,44(9):1060-1065.
[17]林春花,蔡志英,黄贵修. 全基因组法绘制禾谷炭疽菌和希金斯炭疽菌中MAPK级联信号途径简图[J]. 热带作物学报,2012,33(4):674-680.
[18]韩长志. 希金斯炭疽菌RGS蛋白生物信息学分析[J]. 生物技术,2014,24(1):36-41.
[19]韩长志. 希金斯炭疽菌14-3-3蛋白生物信息学分析[J]. 湖北农业科学,2014,3(15):3669-3672.
[20]韩长志. 希金斯炭疽菌中五个典型septin生物信息学分析[J]. 河南农业科学,2014,43(8):91-96.
[21]周鹏. 希金斯刺盘孢T-DNA插入体库的构建、筛选及相关突变体基因的克隆[D]. 武汉:华中农业大学,2011.
[22]赵典. 希金斯刺盘孢T-DNA插入体库的筛选及相关突变体基因的克隆[D]. 武汉:华中农业大学,2012.
[23]Altschul S F,Madden T L,Schaffer A A,et al. Gapped BLAST and PSI-BLAST:a new generation of protein database search programs[J]. Nucleic Acids Research,1997,25(17):3389-3402.
[24]Letunic I,Doerks T,Bork P. SMART 7:recent updates to the protein domain annotation resource[J]. Nucleic Acids Research,2012,40(D1):D302-D305.
[25]Gasteiger E,Hoogland C,Gattiker A,et al. Protein identification and analysis tools on the ExPASy server[M]. The proteomics protocols handbook:Springer,2005:571-607.
[26]Emanuelsson O,Brunak S,Heijne G,et al. Locating proteins in the cell using TargetP,SignalP and related tools[J]. Nature Protocols,2007,2(4):953-971.
[27]Bendtsen J D,Nielsen H,Heijne G,et al. Improved prediction of signal peptides:SignalP 3.0[J]. Journal of Molecular Biology,2004,340(4):783-795.
[28]Kelley L A,Sternberg M J. Protein structure prediction on the Web:a case study using the Phyre server[J]. Nat Protoc,2009,4(3):363-371.
[29]Emanuelsson O,Nielsen H,Brunak S,et al. Predicting subcellular localization of proteins based on their N-terminal amino acid sequence[J]. Journal of Molecular Biology,2000,300(4):1005-1016.
[30]Thompson J D,Gibson T J,Higgins D G. Multiple sequence alignment using ClustalW and ClustalX[M]//Current Protocols in Bioinformatics,2002.
[31]Tamura K,Peterson D,Peterson N,et al. MEGA5:molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J]. Molecular Biology Evolution,2011,28(10):2731-2739.
[32]韩长志. 禾谷炭疽菌RGS蛋白生物信息学分析[J]. 微生物学通报,2014,41(8):1582-1594.于居龙,缪康,赵来成,等. 氰氟虫腙与醚菊酯复配对稻飞虱的控制效果及其安全性评价[J]. 江苏农业科学,2016,44(10):181-185.
