不同粒型水稻品种遗传差异的SRAP分子标记分析
2017-01-20刘冠明陈兆贵
钟 丽,刘冠明,程 诚,陈兆贵
(1.惠州学院生命科学系,广东 惠州 516007;2.仲恺农业工程学院农学院,广东 广州 510550;3.惠州市世纪五丰农业科技股份有限公司,广东 惠州 516003)
不同粒型水稻品种遗传差异的SRAP分子标记分析
钟 丽1,刘冠明2,程 诚3,陈兆贵1
(1.惠州学院生命科学系,广东 惠州 516007;2.仲恺农业工程学院农学院,广东 广州 510550;3.惠州市世纪五丰农业科技股份有限公司,广东 惠州 516003)
研究利用SRAP标记方法对13种长粒水稻和9种短粒水稻材料进行遗传差异分析,以探讨不同粒型水稻品种的遗传差异。结果表明:共扩增了Me1~Me15和Em1~Em20两两配对形成的300对引物,其中带型清晰、有4条带以上的引物161对;161对引物组合共产生1 512条扩增带,1 244条呈多态性,多态性条带比率平均为81.9%,说明品种间存在着较丰富的遗传多态性。聚类分析结果显示,在遗传距离0.66处,可将供试材料分为3大类群。
水稻;粒型;分子标记;SRAP;遗传差异
水稻是人类最重要的粮食作物之一,全世界超过30亿人以稻米为主食。因此,水稻的安全生产受到国家的高度重视[1]。提高水稻产量仍是目前水稻育种研究的主要目标。水稻的产量与粒型有密切关系。水稻粒型性状的遗传比较复杂,研究表明,粒长受微效多基因控制[2]。石春海等[3]研究表明,粒长的表现在很大程度上是由遗传决定的,外界环境对其影响相当小。这一结论在籼稻稻米外观品质性状的遗传主效应和环境互作效应分析中可以体现出来。芮重庆等[4]研究表明,粒长的遗传虽然存在正向部分显性的现象,但是在很大程度上是由加性效应决定的;同时也可能存在细胞质与细胞核间的互作效应,但未观察到细胞质和上位效应的影响,其狭义遗传率高达95.3%。石春海等[5]在早籼稻谷粒性状遗传效应的分析研究中指出,加性效应是粒长的主要遗传规律,其贡献比率为50.6%~98.4%。
SRAP标记(sequence-based amplified polymorphism)是一种新型的分子标记技术,具有多态性高、重复性好、操作简单、易测序、引物通用等优点[6-7],广泛应用于植物的遗传基础研究和育种等领域,并在遗传多样性分析、遗传图谱构建、比较基因组学等多个领域都有所进展[8-12]。由于多年来育种家不断地对水稻资源材料进行定向选择,目前水稻资源已经形成了较多的衍生系。因此,在进行育种技术创新和优良种质资源收集筛选之前,要充分了解水稻资源的现状。试验采用SRAP分子标记技术对部分长粒水稻和短粒水稻进行亲缘关系和遗传差异分析,为进一步研究长粒基因的作用机理打下基础。
1 材料与方法
1.1 试验材料
长粒水稻品系:7-1、7-2、8、10、13、13-1、13-2、14-1、14-2、14-3、35、36、38;短粒水稻品系:1、15、HP11、HP25、HP26、HP27、HP41、HP63、HP85。全部水稻材料由惠州市世纪五丰水稻研究所提供。
水稻分蘖盛期在每个单株取上部1~3片长约5~10 cm的幼叶叶尖,每种材料取4株植株,即每种材料取4个样品。样品采集时,用消好毒的剪刀将水稻幼叶叶尖剪下装入已标号的密封袋中;样品采集完后,置于-80℃冰箱中保存。
1.2 试验方法
1.2.1 DNA提取采用DNA-TPS法提取水稻叶片的DNA[13],用琼脂糖凝胶电泳检测DNA的浓度,将提取的DNA置于4℃冰箱中冷藏待用。
1.2.2 PCR反应选取15条正向引物与20条反向引物(表1,所有引物由北京鼎国昌盛生物技术服务有限公司合成),两两配对,组成300对引物对进行PCR,以22种水稻材料的DNA为模板,进行PCR扩增。PCR反应体系(每个样品总体积20 µL): 10 mmol/ L的 dNTP 0.5 µL,含15 mmol/L MgCl2的10×PCR buffer 2 µL,2 µmol/L的引物M 1.5 µL,2 µmol/L的引物E 1.5 µL,5 U/µL的Taq酶0.1 µL,双蒸H2O 12.4 µL,DNA模板2 µL。混匀放入PCR仪中开始反应。PCR反应程序:94 ℃预变性5 min;94 ℃ 1 min,35℃ 1 min,72 ℃ 1 min,5个循环;94 ℃ 变性1 min,46 ℃退火1 min,72 ℃延伸1 min,35个循环;72 ℃延伸7 min;4 ℃保存。
1.2.3银染和结果记录电泳结束后,轻轻将玻璃板从电泳槽上拆下,做好标记,取出凝胶,在蒸馏水中漂洗2次;转移至0.1%AgNO3溶液中染色,在摇床上轻摇10 min;然后将凝胶转移至蒸馏水中漂洗2次;最后转入NaOH显色液中显色,着色后转入自来水中保存。记录带型结果或凝胶成像。
1.2.4数据统计与分析对SRAP-PCR试验电泳凝胶成像结果进行记录,采用人工读带的方法,在凝胶的某个相同迁移率位置上有DNA条带的记为“1”,无DNA条带记为“0”,缺失的记为“2”,获得每个引物的Excel 0-1矩阵。应用NTSYS-pc2.10软件处理并采用非加权类平均法(UPGMA)进行聚类分析,得到遗传相似系数,并根据遗传相似系数建立聚类图。

表1 试验所用的SRAP标记引物
2 结果与分析
2.1 DNA提取质量
将TPS法所提取的DNA在0.8 %琼脂糖凝胶上电泳分离,结果如图1。电泳显示的条带比较整齐明亮,说明DNA没有降解,纯度较好,所以TPS法提取的DNA可以用于SRAP分析。

图1 0.8%琼脂糖凝胶检测DNA纯度
2.2 引物的选择
PCR扩增结果表明,300对引物中,扩增出带型清晰、有4条带以上的引物对有161对,另外有57对引物对没有扩增出任何条带,有82对引物对扩增出的条带极少。
2.3 RAP标记的多态性分析
由表2可知,每对引物产生4~18条不等扩增带,共产生1 512条扩增带,其中1 244条呈多态性,平均每对引物组合产生9.4条扩增带和7.7条多态性条带,多态性条带比率平均为81.9%。不同引物扩增的多态性比率分布在20.00%~100%之间,而且绝大多数引物扩增的条带多态性较高,说明水稻的遗传多样性分析可以采用SRAP技术。图2是引物组合Me14/ Em11扩增的22种水稻材料的SRAP图谱。
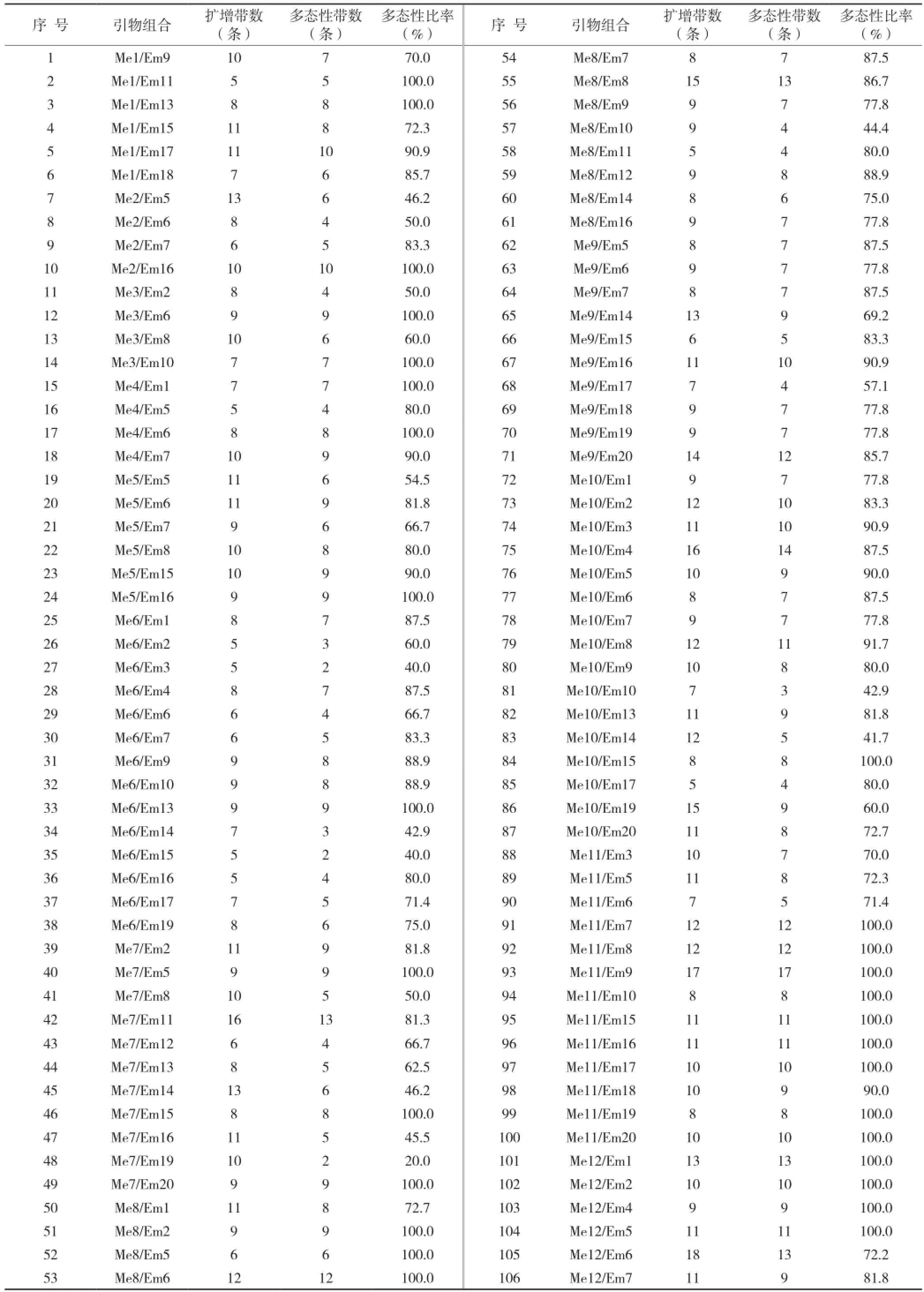
表2 SRAP引物组合、扩增带数及多态性带数

续表2

图2 由引物Me14/Em11扩增的22种水稻材料的SRAP图谱
2.4 聚类分析
根据UPGMA方法进行聚类分析,得到了22种水稻材料的SRAP聚类图(图3),从图3可知,在遗传距离0.66处,供试材料被分成了3大类群。第一类群包括13个材料,为7-1、7-2、8、10、14-1、14-3、36、38、14-2、35、13、13-1、13-2(图5中从上至下的顺序),包括全部的长粒水稻品种。第二类群有8个材料,为1、15、HP26、HP27、HP85、HP41、HP63、HP25,均为短粒水稻品种。第三类群只有1个材料,是HP11,为短粒水稻品种。

图3 22种水稻材料遗传多样性聚类图
3 讨 论
3.1 SRAP分子标记的可靠性
在作物育种工作中,对品种间的亲缘关系和遗传多样性的研究具有重要的理论与实践意义。SRAP分子标记技术之所以能够区分绝大多数的栽培品种,是因为其具有较高的鉴别能力和引物利用率[14],与RAPD技术相比,其引物数量虽然较少,但因为SRAP的正向引物和反向引物可以通用,即正向引物可以与任一反向引物配对,同时反向引物也可以与任一正向引物配对,这样就能在很大程度上提高引物的利用率。SRAP技术没有AFLP的酶切、连接和扩增杂交等繁琐步骤,同时该技术引物设计较容易,操作方便,与以往的一些分子标记技术相比,SRAP分子标记技术具有较大的优势[15-17]。因此,SRAP技术自产生以来就受到了大多数分子生物学家的青睐。对于扩增产物的检测,研究者可以根据实验室设备、药物以及其他实际情况来灵活选择,因为SRAP不但可以采用比较传统的琼脂糖凝胶电泳检测,还可以采用比较环保的聚丙烯酰胺凝胶电泳检测(该检测方法采用银染法染色)。
3.2 长粒和短粒水稻遗传背景差异
尽管关于水稻粒长的遗传机理众说纷纭,但近年来国内外多数研究结果倾向于粒长由多基因控制,并且在表达过程中存在着不完全显性效应[18]。石春海等[19]用3个长粒品种分别与3个短粒品种两两配对进行杂交,结果表明粒长性状是由多基因控制的数量性状,并且以加性效应为主。笔者利用SRAP分子标记技术扩增22种不同粒型水稻材料的DNA,发现SRAP扩增的多态性条带比率平均为81.9 %,多态性比率较高,说明品种间存在着较丰富的遗传多态性。
试验中,聚类分析结果显示,22种水稻材料在遗传距离0.66处,可分为3大类群,13种长粒水稻材料归为一类,8种短粒水稻材料归为另一类,此外,HP11单独聚为一类,说明HP11与其他品系之间遗传差异较大。鉴于HP11的独特性,在杂交育种工作中,选配亲本配制杂交组合时,可以考虑选用HP11与其他材料进行杂交组合,以便创造出更多杂交后代变异类型,提高育种工作效率。
[1] 程式华,胡培松. 中国水稻科技发展战略[J]. 中国水稻科学,2008,(3):223-226.
[2] 宫李辉,高振宇,马伯军,等. 水稻粒形遗传的研究进展[J]. 植物学报,2011,(6):597-605.
[3] 石春海,何慈信,朱 军,等. 籼稻稻米外观品质性状的遗传主效应和环境互作效应分析(英文)[J]. 中国水稻科学,1999,(3):179-182.
[4] 苪重庆,赵安常. 籼稻粒重及粒形性状F1遗传特性的双列分析[J].中国农业科学,1983,(5):14-20.
[5] 石春海,申宗坦. 早籼稻谷粒性状遗传效应的分析[J]. 浙江农业大学学报,1994,(4):405-410.
[6] Li G,Quiros C F. Sequence-related amplified polymorphism (SRAP),A new marker system based on a simple PCR reaction:its-application to mapping and gene tagging in Brassica [J]. Theor. Appl.Genet,2003,107(1):168-180.
[7] 邱文斌,孙伟生,窦美安. 基于PCR的新型分子标记SRAP研究进展[J]. 江西农业学报,2007,19(8):26-28.
[8] 文雁城,王汉中,沈金雄,等. 用SRAP标记分析中国甘蓝型油菜品种的遗传多样性和遗传基础[J]. 中国农业科学,2006,39(2):246-256.
[9] 何凤发,杨志平,张正圣,等. 马铃薯遗传资源多样性的SRAP分析[J]. 农业生物技术学报,2007,15(6):1001-1005.
[10] 林忠旭,张献龙,聂以春,等. 棉花SRAP遗传连锁图构建[J]. 科学通报,2003,48(15):1676-1679.
[11] 林忠旭,张献龙,聂以春. 新型标记SRAP在棉花F2分离群体及遗传多样性评价中的适用性分析[J]. 遗传学报,2004,31(6):622-626.
[12] 王 燕,龚义勤,赵统敏,等. 番茄SRAP-PCR体系优化与品种分子鉴定[J]. 南京农业大学学报,2007,(1):23-29.
[13] 赫 然,黄小乐,刘秋云,等. 快速制备水稻基因组DNA PCR模板的煮沸法[J]. 植物生理学通讯,2003,39(1):41-42.
[14] 李 严,张春庆. 西瓜杂交种遗传多态性的SRAP标记分析[J]. 园艺学报,2005,32(4):643-647.
[15] Budak H,Shearman R C,Parmaksiz I,et al. Comparative analysis of seeded and vegetative biotype buffalograsses based on phulogenetic relationship using ISSRs,SSRs,RAPDs and SRAPs[J]. Theor Appl Genet,2004,109:280-288.
[16] Ferriol M,Plco B,Nuez F. Genetic diversity of a gemplasm colletion of cucurbitapepo using SRAP and AFLP markers[J]. Theor Appl Genet,2003,107:271-282.
[17] Riaz A,Potter D,Stephen M. Genotyping of peach and nectarine cultivars with SSR and SRAP molecular markers[J]. Amer Soc Hort Sci,2004,129:204-211.
[18] 杨联松,白一松,许传万,等. 水稻粒形类型及其遗传的研究进展[J].安徽农业科学,2001,29(2):164-167.
[19] 石春海,申宗坦. 早籼粒形的遗传和改良[J]. 中国水稻科学,1995,(1):27-32.
(责任编辑:成 平)
Analysis on Genetic Differences in Rice by SRAP Molecular Markers
ZHONG Li1,LIU Guan-ming2,CHEN Cheng3,CHEN Zhao-gui1
(1. Department of Life Science, Huizhou University, Huizhou 516007, PRC; 2. Agronomy College, Zhongkai University of Agriculture and Engineering, Guangzhou 510550, PRC; 3. Huizhou Century Harvest Agricultural Science and Technology Inc, Huizhou 516003, PRC)
Taking thirteen kinds of long grain rice and nine kinds of short grain of rice as materials, the genetic differences between rice varieties were investigated by using SRAP markers method. The results showed that 300 pairs of primers Me1-Me15 and Em1-Em20 pairwise were formed, and 161 pairs of primers were amplifed clear, each of them got more than 4 bands. 161 pairs of primers amplifed 1 512 bands in total, and 1 244 bands were polymorphic bands, with an average rate of 81.9%. It revealed that there were abundant genetic polymorphisms between rice varieties. Cluster analysis indicated that these tested materials could be divided into three groups in the genetic distance of 0.66.
rice; grain type; molecular markers; SRAP; genetic differences
book=5,ebook=13
Q346+.5
A
1006-060X(2016)12-0005-05
10.16498/j.cnki.hnnykx.2016.012.002
2016-09-12
2012年广东省农业科技推广项目(201201162)
钟 丽(1994-),女,广东五华县人,本科生,生物科学专业。
陈兆贵
