利用CoMFA和CoMSIA对大肠杆菌DXR抑制剂进行3D-QSAR研究
2017-01-18李天恩覃初新
肖 玉,李天恩,覃初新,戴 康
(中南民族大学 药学院,武汉 430074)
利用CoMFA和CoMSIA对大肠杆菌DXR抑制剂进行3D-QSAR研究
肖 玉,李天恩,覃初新,戴 康
(中南民族大学 药学院,武汉 430074)
指出了1-脱氧-D-木酮糖醇-5-磷酸还原异构酶(DXR)是细菌细胞壁合成中的关键物质2C-甲基-D-赤藓糖醇-4-磷酸(MEP)合成途径中的限速酶,该酶作为新型的抗菌药物的靶点,被广泛的研究。利用CoMFA和CoMSIA方法建立了大肠杆菌DXR抑制剂的3D-QSAR模型,通过该模型主要分析了化合物的结构对活性的影响。结果表明:CoMFA模型预测值和实验值的交叉验证相关系数q2为0.671,回归相关系数R2为0.950;CoMSIA模型预测值和实验值的交叉验证相关系数q2=0.559,回归相关系数R2=0.973。说明所建立的模型具有较好的预测能力,能为DXR抑制剂的结构优化提供指导。
大肠杆菌;DXR抑制剂;CoMFA;CoMSIA;3D-QSAR模型
1 引言
类异戊二烯以其衍生物在细胞壁的合成和能量代谢过程中具有重要的作用[1]。在绝大多数细菌体内,类异戊二烯的前体异戊酰焦磷(IPP)或二甲烯丙基焦磷酸(DMAPP)只能通过2C-甲基-D-赤藓糖醇-4-磷酸(MEP)途径进行生物合成[2,3]。其中,1-脱氧-D-木酮糖醇-5-磷酸还原异构酶(DXR)催化MEP途径的第二步反应,并且是该途径中的限速酶.由于人体内不存在MEP途径和DXR类似酶[4,5],因此DXR可以作为潜在的靶点进行抗菌药物的研发。
研究发现膦胺霉素及其衍生物是DXR的有效抑制剂[6,7],对绝大多数革兰氏阴性和某些革兰氏阳性菌具有较强的抗菌活性[8],但是对大肠杆菌的抑菌作用较弱[9]。因此研究大肠杆菌DXR抑制剂的结构与生物活性之间的关系具有重要的临床意义和应用前景。以大肠杆菌DXR作为靶向蛋白,选择活性较高的化合物进行分子对接,以对接生成的构象作为模板建立CoMFA和CoMSIA模型,使模型的显著性强于经典的分析方法,从而为膦胺霉素类抑制剂的结构优化提供理论依据。
2 实验部分
2.1 分子库的准备
从相关文献[10~12]上获取39个大肠杆菌DXR抑制剂的分子结构和生物活性数据, 其中生物活性数据以pIC50表达。对化合物的结构进行初步分析,发现包含A和B两种母核结构.从RCSB PDB下载蛋白质(code:3ROI)的晶体结构,选择分子库中活性最高的化合物(Template,pIC50 = 7.553)与蛋白质进行分子对接。从剩下的38个化合物中随机的选择7个化合物作为测试集,其余的31个化合物作为训练集(图1)。

图1 A、B及Template的分子式
2.2 分子叠合
在3D-QSAR分析中,通常选取能量最小的活性构像作为分子排列的模版。本文选择活性最好的分子(Template)中对接打分最高的构象作为模板,利用Open3DALIGN软件进行分子叠合。将叠合生成的化合物构象输入新的数据库中并加载相对应的生物活性值。
2.3 CoMFA和CoMSIA分析
利用SYBYL-X软件进行CoMFA和CoMSIA分析,对训练集化合物加Gasteiger-Marsili电荷并计算CoMFA场参数。首先运用留一法(LOO)进行交叉验证分析,获得交叉验证相关系数q2和最佳主成分数(ONC)。然后利用获得的ONC做非交叉验证的PLS分析并利用Region Focusing对初始的CoMFA值和PLS分析结果进行优化。在CoMSIA分析中,还需考虑疏水场、氢键供体和氢键受体对化合物活性的影响,CoMSIA的PLS分析过程与CoMFA基本相同。
3 结果与讨论
3.1 PLS分析结果
如表1所示,利用训练集建立的CoMFA和CoMSIA模型的交叉验证相关系数q2值分别为0.671和0.559。当q2≥0.5时,表明由相应的变量建立的模型具有可靠的预测能力。这说明建立的CoMFA模型和CoMSIA模型可以对化合物的活性进行有效的预测.由于CoMFA模型的q2值明显大于CoMSIA,因此在理论上CoMFA模型的预测结果会更好。在CoMFA模型中,还获得非交叉验证相关系数(R2)为0.950,标准偏差(SEE)为0.229以及显著性因子(F)为95.309,立体场和静电场对模型的贡献值分别为58.1%和41.9%。在CoMSIA模型中,获得的非交叉验证相关系数(R2)值为0.973,立体场、静电场、疏水场、氢键供体、氢键受体的贡献值分别为19.1%、21.9%、24.8%、27.6%、6.6%。为了进一步检测模型的预测能力,对测试集的化合物活性进行预测。训练集和测试集化合物的结构及实验活性和预测活性见表2。通过对实验活性和预测活性做线性相关分析,发现测试集化合物的活性基本分布在回归直线的两侧(图1、2),这说明模型有较好的预测能力。

表1 CoMFA 和CoMSIA 模型的统计结果
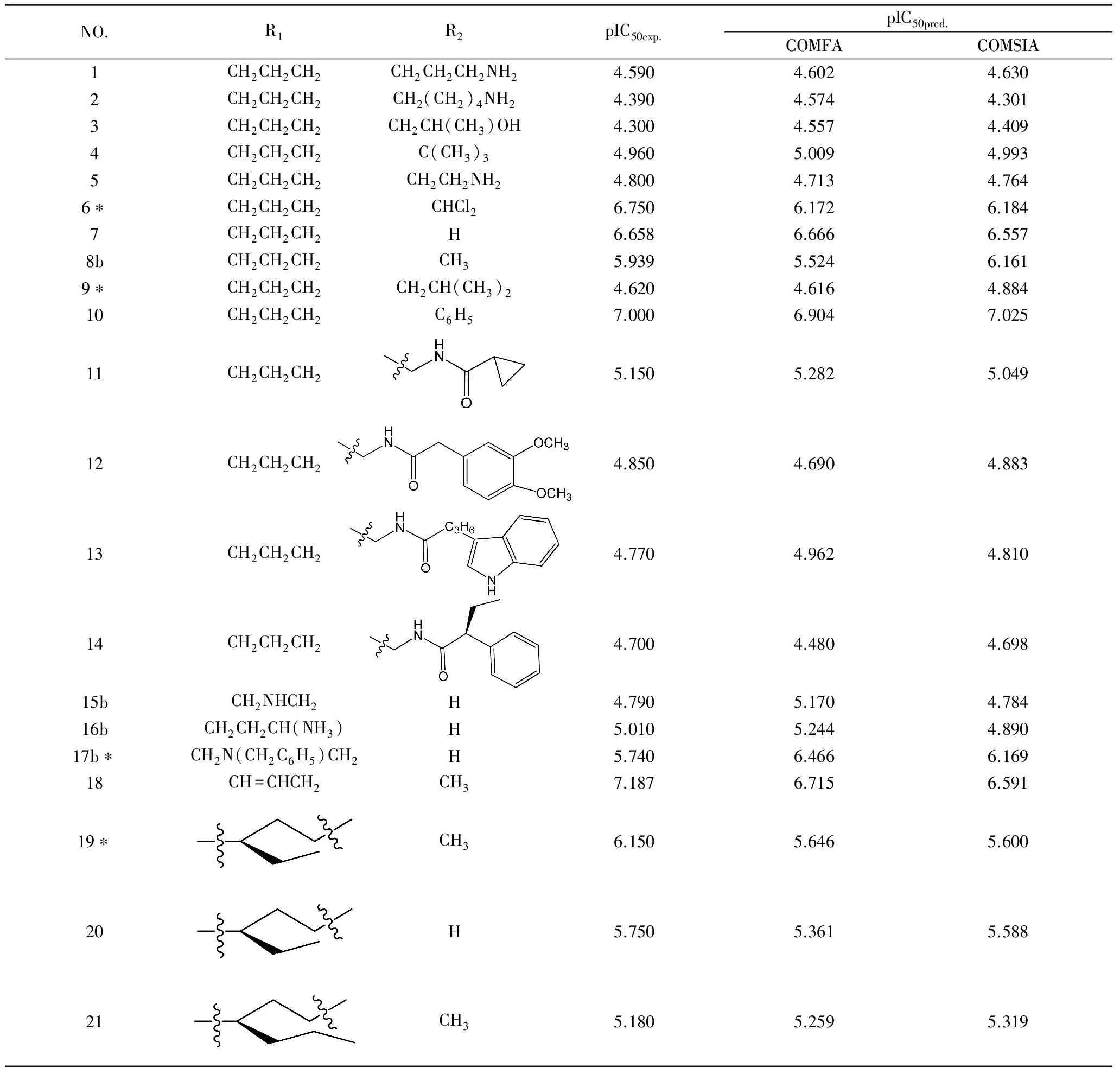
表2 38个化合物的结构和活性数据
续表2

22∗H5.0905.0115.30323H5.1805.0565.21324bCH34.9595.1395.08925CH36.1906.3736.36326CH36.7966.9126.68027H6.5056.3706.66028CH35.6305.8085.88129COOCH2CH34.8004.5184.72430H5.1905.0775.07631H7.2297.3527.31832∗CH37.0047.0527.06633CH36.9256.8386.89634H6.8076.9326.88535CH36.5506.4676.44736b∗CH36.5386.6577.14237CH36.4026.5256.52538CH36.3386.4456.314
注:其中R1的左侧与磷酸根相连;b表示化合物的母核结构为B类,其余为A类;*表示测试集

图2 CoMFA中实验活性和预测活性散点

图3 CoMSIA中实验活性和预测活性散点
3.2 三维等高线图
图4和图5分别显示的是CoMFA模型中立体场和静电场对化合物活性的影响。在立体场中,绿色区域表示增加取代基的体积有利于增加化合物的活性,黄色区域表示减小取代基的体积有利于增加化合物的活性。如图4所示,R1位于黄色区域和绿色区域之间,当R1体积较小时主要靠近黄色区域,此时减小R1体积可以提高化合物的活性。例如化合物26和28在R1处的取代基分别为环丙基和环戊基,由于环丙基的体积小于环戊基,所以化合物26的活性大于化合物28。化合物19和21的R1取代基分别为CH(CH2CH3)CH2CH2和 CH(CH2CH2CH3)CH2CH2,它们的pIC50值分别为6.150和5.180也验证了这一点。但是,当R1取代基上连有较大体积的支链取代基时,此时体积大的支链部分主要靠近绿色区域,所以增加支链的体积可以提高化合物的活性。例如化合物30和34,由于化合物34的苯环上连有-OCH3,所以化合物活性30<34。R2主要位于绿色区域,因此增加R2的体积可以提高化合物的活性。例如化合物19与20以及化合物26与27,由于CH3的体积大于H原子,所以它们的活性:19>20,26>27。

图4 CoMFA模型的立体场等值线
在静电场中,蓝色区域表示此区域范围内引入正电性基团会增加化合物的活性,红色区域表示此区域范围内引入负电性基团会增加化合物的活性。如图5所示,R1无论是体积较小的取代基还是连有较大体积支链的取代基,均位于红色区域附近,因此增加R1的电负性可以增加化合物的活性。例如化合物7和15,它们的R1分别为CH2CH2CH2和CH2NHCH2,所以化合物7的活性大于化合物15。比较化合物32、35和38的活性也可以验证这一点,由于吸电子的能力:Cl>H>OCH3,因此它们的活性大小为32>35>38。R2基团主要位于蓝色区域附近,因此增加正电性基团可以提高化合物的活性。比较化合物3和9的活性,由于供电子的能力CH2CH(CH3)OH 图5 CoMFA模型的静电场等值线 图6 CoMSIA模型的疏水场等值线 以CoMFA和CoMSIA方法为基础, 结合docking、Open3DALIGN进行综合分析,成功地建立了DXR抑制剂的3D-QSAR模型。不仅直观的解释了这些化合物的结构与活性之间的关系,为改进这些化合物的结构提供了理论依据和指导方向,而且利用建立的模型还可以对可能具有潜在抑制活性的化合物进行分析,从而发现一些新的高活性的大肠杆菌DXR抑制剂. [1]Barry C E, Crick D C, McNeil M R. Targeting the formation of the cell wall core of M. tuberculosis [J]. Infect Disord Drug Targets, 2007, 7(2): 182~202. [2] Hale I, O’Neill P, Berry N, et al. The MEP pathway and the development of inhibitors as potential anti-infective Agents [J]. Med Chem Commun, 2012,(3): 418~433. [3]Campos N, Rodriguez-Concepcion M, Sauret-Gueto S, et al. Escherichia coli engineered to synthesize isopentenyl diphosphate and dimethyallyl disphosphate from mevalonate: a novel system for the genetic analysis of the 2-C-methyl-d-erythritol 4-phosphate pathway for isoprenoid biosynthesis [J]. Biochem J, 2001(353):59~67. [4]Eoh H, Brennan P J, Crick D C. The Mycobacterium tuberculosis MEP (2C-methyl-d-erythritol 4-phosphate) pathway as a new drug target [J]. Tuberculosis (Edinb), 2009, 89(1): 1~11. [5]施文钧, 王洪海. MEP途径: 一个潜在的分子药靶 [J]. 中国抗生素杂志, 2008, 33(2): 65~68. [6]Jomaa H, Wiesner J, Sanderbrand S, et al. Inhibitors of the nonmevalonate pathway of isoprenoid biosynthesis as antimalarial drugs [J]. Science, 1999, 285(5433):1573~1576. [7]Yajima S, Hara K, Sanders JM, et al. Crystallographic structures of two bisphosphonate: 1-deoxyxylulose-5-phosphate reductoisomerase complexes [J]. J Am Chem Soc, 2004, 126(35): 10824~10825. [8]Okuhara M, Kuroda Y, Goto T, et al. Studies on new phosphonic acid antibiotics. III. Isolation and characterization of FR-31564, FR-32863 and FR-33289 [J]. J Antibiot, 1980, 33(1): 24~28. [9]陈习平, 谢丽萍, 胡又佳. 1-脱氧-D-木酮糖醇-5-磷酸还原异构酶(DXR)作为新的药物靶点的研究进展 [J]. 世界临床药物, 2014, 35(5):319~320. [10]Silber K, Heidler P, Kurz T, et al. AFMoC enhances predictivity of 3D QSAR: a case study with DOXP - reductoisomerase [J]. J Med Chem, 2005, 48(10): 3547~3563. [11]Haemers T,Wiesner J,Van Poecke S, et al. Synthesis of alpha-substituted fosmidomycin analogues as highly potent Plasmodium falciparum growth inhibitors [J]. Bioorg Med Chem Lett, 2006, 16(7): 1888~91. [12]Deng L, Endo K, Kato M, et al. Structures of 1-Deoxy-D-Xylulose-5-Phosphate Reductoisomerase/Lipophilic Phosphonate Complexes [J]. Med. Chem. Lett., 2011, 2 (2): 165~170. The 3D-QSAR Study on the E.coli DXR Inhibitors by CoMFA and CoMSIA Methods Xiao Yu, Li Tianen, Qin Chuxin, Dai Kang (CollegeofPharmacy,South-CentralUniversityforNationalities,Wuhan,Hubei430074,China) This paper pointed out that 1-deoxy-d-xylulose-5-phosphate reductoisomerase(DXR) is the rate-limiting enzyme in the synthesis of 2-methyl-D-erythritol-4-phosphate(MEP)which is essential for the formation of bacterial cell walls. This enzyme has been widely investigated and considered to be one of the most promising targets for screening novel antibiotics. In this paper, the CoMFA and CoMSIA methods were used to analyze the three-dimensional quantitative structure-activity relationship(3D-QSAR) for the inbititory activity against E.coli DXR. The results showed that the cross-validated correlated coefficient q2 was 0.671 and the conventional correlated coefficient R2 was 0.950 in the CoMFA model and the q2 was 0.559 and the R2 was 0.973 in the CoMSIA model, which illustrated that the predictability of the built models were reliable, and they could provide useful insights for designing novel and potent DXR inhibitors. E.coli;DXR inhibitors;CoMFA;CoMSIA;3D-QSAR model 2016-07-30 省部科研基金(编号:BZY14027) 肖 玉(1990—),男,中南民族大学药学院硕士研究生。 戴 康(1972—),男,副教授,主要从事药物合成及分子设计方面的研究工作。 R284 A 1674-9944(2016)18-0171-05

4 结论
