雷蒙德氏棉扩展蛋白基因家族的鉴定和特征分析
2017-01-17雷忠萍贺道华海江波邢宏宜赵俊兴程雪妮
雷忠萍,贺道华,海江波,邢宏宜,赵俊兴,程雪妮
(1.西北农林科技大学 农学院,陕西 杨凌 712100;2.西北农林科技大学 生命科学学院,陕西 杨凌 712100)
雷蒙德氏棉扩展蛋白基因家族的鉴定和特征分析
雷忠萍1,2,贺道华1,海江波1,邢宏宜1,赵俊兴1,程雪妮2
(1.西北农林科技大学 农学院,陕西 杨凌 712100;2.西北农林科技大学 生命科学学院,陕西 杨凌 712100)
扩展蛋白具有松驰细胞壁和增加细胞壁柔韧性的功能。为了弄清该基因家族在雷蒙德氏棉中的特征,利用隐马尔科夫模型(HMM)在雷蒙德氏棉基因组中进行扩展蛋白家族基因成员的鉴定,获得了39个扩展蛋白基因家族成员,分布在12条染色体上。系统发育分析将其归入EXPA、EXPB、EXLA和EXLB共4个亚家族,分别含有26,4,3,6个基因。各亚家族中扩展蛋白基因具有相对多样化的基因结构;扩展蛋白基因家族在进化中经历了3次扩张;染色体片段重复是该基因家族扩张的主要方式;扩张后纯化选择占主导地位。比较棉纤维发育不同时期及叶片、花瓣的深度测序和基因芯片表达谱,发现大部分扩展蛋白基因参与了雷蒙德氏棉纤维、叶片和花瓣的生长发育,在不同的时空范围均表现出表达多样性。GrEXLA1、GrEXLA2和GrEXPA16在花瓣中优势表达,GrEXPA23和GrEXPA24在叶片中表达水平较高,推测这些基因的表达分别参与了花瓣和叶片的发育进程;其余的基因,在纤维的不同发育阶段,表现出不同的表达丰度。研究结果有助于了解棉属扩展蛋白基因家族的特征及功能,为深入剖析棉纤维发育过程中的分子调控机理提供了基础。
雷蒙德氏棉;扩展蛋白;基因结构;系统发育分析;进化分析;表达模式
扩展蛋白(Expansin)是一类植物细胞壁伸展蛋白,是植物细胞壁的重要组分,广泛存在于各种植物细胞组织中。扩展蛋白一般由205~275个氨基酸组成,氨基末端含有1个长度为20~30个氨基酸的信号肽[1];中段含有1个类似于Glycoside hydrolase family 45的结构域DPBB_1,长度为120~135个氨基酸;羧基端含有结构域Pollen_allerg_1,长度为90~120个氨基酸。在植物细胞壁伸展研究中,Mcqueen-Mason等[2]首次从黄瓜胚轴中分离出扩展蛋白。近年来,由多基因家族编码的扩展蛋白在越来越多的物种中被鉴定,如葡萄(29个扩展蛋白家族成员[3])、苹果(41个[4])、杨树(36个[5])、玉米(88个[6])、大豆(75个[7])和大白菜(53个[8])。依据系统进化关系,扩展蛋白可以分为4个亚家族,分别是EXPA、EXPB、EXLA和EXLB(也称α、β、γ和δ亚家族[9])。其中,亚家族EXPA在大多数植物中是最大的1个亚家族,其次是亚家族EXPB[10]。
功能研究表明,扩展蛋白参与许多生长发育过程,如种子发芽[11]、根毛的起始和伸长[12-13]、木质部形成[14]、花粉管伸长[15-16]、水果成熟软化[17]和叶片脱落[18]等;在植物抗逆[19-20]中扩展蛋白也有重要作用。研究表明,扩展蛋白在特定的pH梯度下,以酶催化的作用方式,使细胞壁组分间疏松,细胞伸展,细胞的柔韧性增强,以此缓解各种环境因子对细胞的机械压力[2,10]。EXPA亚家族更多作用于植物的生长发育[12,21]和抗性[22],EXPB亚家族则趋向于在生殖系统[23-24]中起作用。然而,EXLA和EXLB两类亚家族仅仅知道其基因序列[1],尚没有试验证据发现其具有增加细胞壁柔韧性的功能[5]。
棉纤维是世界上重要的纺织原料之一,具有极其重要的经济价值。它是由胚珠外珠被表皮层的单细胞分化发育而成,大约历时45~50 d,包含着4个依次并部分重叠的发育时期:纤维原始细胞的分化和突起、初生细胞壁伸长、次生细胞壁加厚和脱水成熟。研究表明纤维的发育涉及大量基因的表达和蛋白相互作用[25]。因此,棉纤维是单细胞生长发育的典型模型[26]和最佳研究材料,剖析其发育过程的机理意义重大[27]。纤维的伸长与细胞壁的松弛密切相关,而扩展蛋白能够打断纤维素微丝间的氢键,从而使细胞壁延展疏松。EST数据库分析显示,在纤维细胞迅速伸长时期,4个编码扩展蛋白的基因转录水平较高,而在后续的次生壁合成期后这4个基因的转录量显著降低[28],暗示了它们主要在棉纤维细胞伸长期发挥功能。
目前,异源四倍体的陆地棉(Gossypiumhirsutum,染色体组AADD)是栽培面积及经济价值最大的棉种。普遍认为,在100~200万年前,雷蒙德氏棉(G.raimondii,染色体组DD)的祖先与亚洲棉(G.arboreum,染色体组AA)的祖先之间发生远缘杂交,杂交后代经染色体加倍然后进化成现今的陆地棉。雷蒙德氏棉基因组的测序工作已完成[29-30],为棉属基因组的研究开创了新局面。本研究利用生物信息学方法,对雷蒙德氏棉全基因组的扩展蛋白家族基因进行了预测和系统进化分析,并对其在棉纤维等组织发育过程中的表达模式进行分析,旨在为棉花优质、高产等重要农艺性状提供候选基因,为基因组水平上的棉花分子育种工作提供基础研究依据。
1 材料和方法
1.1 雷蒙德氏棉中搜索扩展蛋白基因家族成员
本试验的雷蒙德氏棉(G.raimondii)基因组及蛋白质序列均来自于DOE Joint Genome Institute(JGI)网站(ftp://ftp.jgipsf.org/pub/)。扩展蛋白家族成员序列信息下载于Daniel Cosgrove实验室的网站(http://www.personal.psu.edu/)。扩展蛋白特有的保守结构域DPBB_1(pfam03330)和Pollen_allerg_1(pfam01357)来自网站http://pfam.xfam.org/。利用基于隐马尔可夫模型的软件HMMER(http://hmmer.janelia.org/)和本地化的Blast 2.2.26(ftp://ncbi.nlm.nih.gov/blast/executables/)软件,在雷蒙德氏棉全蛋白质序列数据库中搜索扩展蛋白基因。
1.2 系统发育树的构建及基因命名
利用ClustalW对雷蒙德氏棉扩展蛋白家族的氨基酸序列(以拟南芥、水稻的扩展蛋白序列作为outgroup)进行序列比对,利用MEGA v5.1采用Neighbor-Joining算法构建系统发育树,从而将鉴定出来的雷蒙德氏棉扩展蛋白进行亚家族分类,在亚家族内根据其在染色体上的位置顺序进行命名。
1.3 扩展蛋白家族基因的结构、染色体物理定位和基因复制共线性分析
利用扩展蛋白基因的编码区序列(CDS)与全基因组序列比对,在网站http://bio.ieo.eu/fancygene/得到基因结构图。利用Blast工具将所获得的扩展蛋白基因序列,定位到基因组的各染色体上;同时,在Phytozome的数据库中对结果进行验证。利用MCScanX进行扩展蛋白家族基因成员倍增模式(基因复制共线性)分析,利用Circos绘制扩展蛋白基因在染色体上的物理位置及基因间的共线性关系。利用perl代码计算具有共线性关系的旁系同源基因对间的Ks、ω值(Ka/Ks)和4DTv。
1.4 扩展蛋白序列分析及理化性质分析
采用软件BioEdit v7.1对雷蒙德氏棉中扩展蛋白家族成员的mRNA和氨基酸序列进行一致性分析;使用DnaSP 5.10程序对雷蒙德氏棉中扩展蛋白密码子偏好性(Codon Usage Bias)进行统计;在http://www.cbs.dtu.dk/services/SignalP/分析信号肽;通过软件MEME 4.8.0以及在线软件ProtParam对雷蒙德氏棉中扩展蛋白的结构保守性、蛋白稳定性及相关的理化性质进行考察;利用Euk-mPLoc 2.0 (http://www.csbio.sjtu.edu.cn/bioinf/euk-multi-2/#)预测每个蛋白的亚细胞定位信息。
1.5 深度测序表达谱的获得与分析
雷蒙德氏棉的叶片、纤维和胚珠不同时期的深度测序和基因芯片表达谱数据下载于NCBI网站SRA数据库和GEO数据库。表达谱原始数据文件登录号见表1。参照He等[31]方法分析扩展蛋白基因家族每个基因的表达量,并识别差异表达基因。

表1 雷蒙德氏棉扩展蛋白家族基因表达谱分析数据Tab.1 Details of RNA-seq and microarray dataset mined for analysis of expansin expression profiles
2 结果与分析
2.1 雷蒙德氏棉扩展蛋白基因家族
根据扩展蛋白特有的保守结构域DPBB_1和Pollen_allerg_1,运用HMMER软件将多序列比对结果转化为扩展蛋白基因家族的HMM profile文件,在蛋白序列库搜索具有目标结构域的蛋白序列。在雷蒙德氏棉全基因组中共检索到39个扩展蛋白基因家族成员,经过Blast 和结构域分析,确定这39个基因均具有扩展蛋白家族的保守结构域。其中,26个基因属于EXPA亚家族,4个基因归为EXPB亚家族,3个基因归为EXLA亚家族,6个基因归为EXLB亚家族。按其在染色体上的位置分布,将这39个扩展蛋白基因依次命名为:GrEXPA01~GrEXPA26、GrEXPB1~GrEXPB4、GrEXLA1~GrEXLA3和GrEXLB1~GrEXLB6(表2)。与拟南芥(Arabidopsisthaliana)及杨树(Populustrichocarpa)基因组相比较,雷蒙德氏棉基因组中包含较低比例的EXPA亚家族基因,约占基因家族总成员数的67%。在已测序的同为双子叶植物的拟南芥和杨树基因组中,这一比例分别为72%和75%(表3)。而在已测序的单子叶植物的水稻(Oryzasativa)基因组中,EXPA亚家族扩展蛋白基因的比例为59%。比较而言,雷蒙德氏棉中EXLA亚家族扩展蛋白基因的比例较小,约为7.7%,同为双子叶植物的拟南芥和杨树中的这一比例约为8.3%和5.6%,而单子叶植物水稻中的EXLA亚家族扩展蛋白基因的比例为6.9%(表3)。
2.2 扩展蛋白基因在染色体上的分布及共线性分析
雷蒙德氏棉基因组包含13对染色体,根据基因组测序结果,将雷蒙德氏棉扩展蛋白基因家族的39个成员进行染色体定位。结果表明,除了染色体Gr10外,39个扩展蛋白基因分别位于雷蒙德氏棉的其余12条染色体上,并且扩展蛋白基因在染色体上的分布较为分散(图1)。染色体Gr03上仅1个扩展蛋白基因,染色体Gr04上多达7个扩展蛋白基因,且染色体Gr04上3个扩展蛋白基因紧密串联(成簇分布),4条染色体(Gr05、Gr07、Gr11和Gr12号)上均具有3个扩展蛋白基因。

表2 雷蒙德氏棉扩展蛋白基因家族成员鉴定Tab.2 Identification of expansin gene family members from G.raimondii
注:(+)和(-)分别表示该基因位于染色体正链和负链上。
Note:(+) and(-) indicated the forward and reverse orientation,respectively.
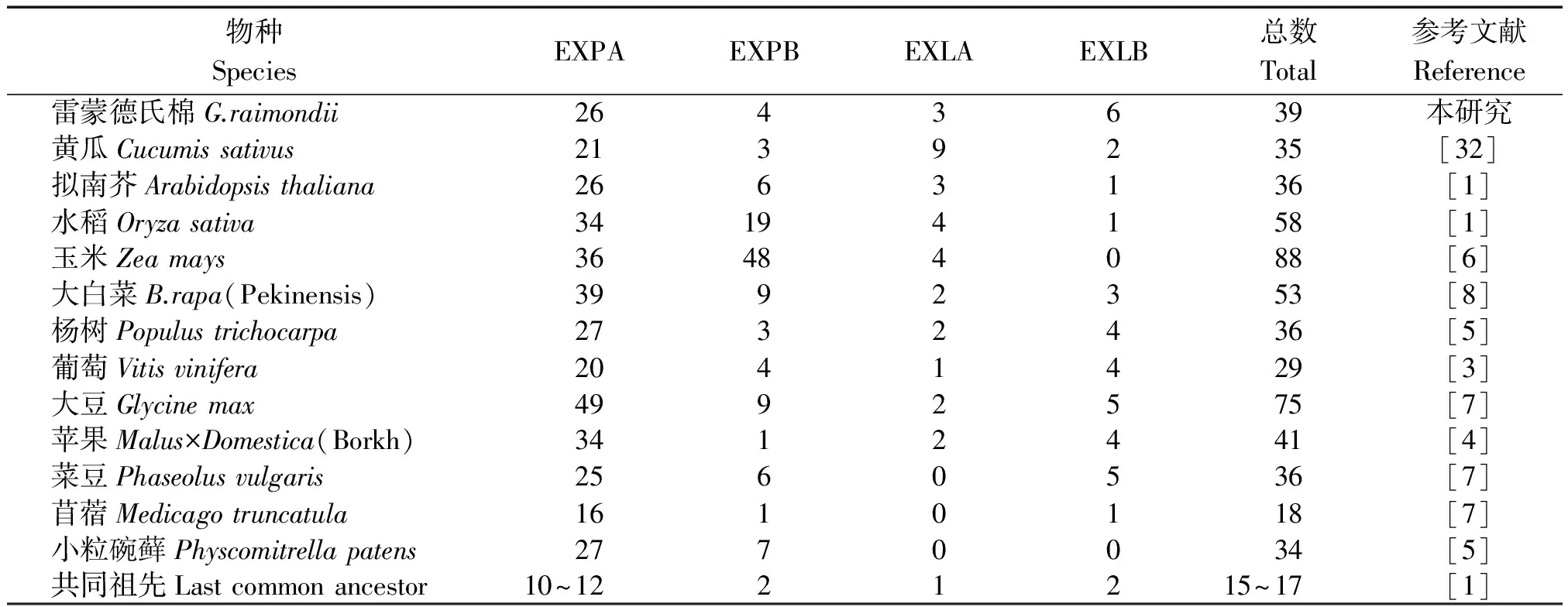
表3 不同植物扩展蛋白及其4类亚家族基因数目Tab.3 Number of expansin family members identified in various organisms
共线性分析显示,部分扩展蛋白基因之间存在着交叉的共线性关系,如GrEXPA01/GrEXPA04/GrEXPA10、GrEXPA05/GrEXPA08/GrEXPA09、GrEXPA16/GrEXPA20/GrEXPA21/GrEXPA23、GrEXLA1/GrEXLA2/GrEXLA3等(图1),表明这些扩展蛋白基因位于染色体倍增块(Duplicated block)上,即基因组进化过程中,含有这些扩展蛋白基因的染色体区段(大片段)发生了倍增(Duplication或Paleopolyploidy)。数据显示,共有30个扩展蛋白基因分属于11组染色体倍增块上(表4),其中5组倍增块由2个以上的染色体区段构成,说明这些扩展蛋白基因还存在着交叉匹配的现象。例如:GrEXPA01、GrEXPA04和GrEXPA10倍增块;GrEXPA16、GrEXPA20、GrEXPA21和GrEXPA23倍增块。这些倍增块的产生可能是由于染色体多次复制,从而使得倍增块内的数个基因间具有共线性的旁系同源或横向同源关系(Paralog)。这些信息显示了扩展蛋白基因家族在雷蒙德氏棉中的扩张历程。表4显示,倍增块内基因间的ω值(Ka/Ks)<1,显示在经历了古老的基因组加倍(Paleopolyploidy)后,扩展蛋白成员数增加,但主要经历着纯化选择(Purifying selection),所以倍增块内,基因间差异较小。Ks分布密度(图2)显示出3个峰值(0.516 9,1.275 3和1.685 4),表明了在17.2(Million years),98.1,129.6 Myr前扩展蛋白家族基因所在区段发生了3次扩张。数据显示,具有共线性的旁系同源基因对,其4DTv(4倍简并位点)值大部分(75%)为0.393 9~0.952 2,表明旁系同源基因对经历了很漫长的进化历程。

外层圆圈表示染色体Gr01-Gr13;圈内曲线表示基因间存在共线性。Chromosome(Gr) 01-13 are depicted as curve bars;Curve lines denote collinear regions containing expansin family genes.
2.3 雷蒙德氏棉扩展蛋白家族基因结构分析
对雷蒙德氏棉扩展蛋白基因序列统计分析显示,各扩展蛋白基因在基因组中的长度为981~2 922 bp (只有基因GrEXPA12,因为第3个内含子极长(图3),所以其核苷酸序列长达7 372 bp;黄瓜中也存在一个内含子极其长的EXPA基因[32]),而编码蛋白的外显子部分序列全长为735~870 bp,可编码244~289个氨基酸(但也存在特例,如:GrEXPB3短至176 aa,GrEXPA07长至303 aa)。植物扩展蛋白基因的内含子模式(Intron pattern:losses,presence and location(in the gene) of intron)具有较大的保守性,Sampedro等[33]对不同物种扩展蛋白基因内含子序列的统计分析发现,不同内含子可能具有不同的起源,并由此将其分为G、A、B、C、F 和H共6种不同的类型。在雷蒙德氏棉中,全部扩展蛋白基因均含有A类型内含子,82.1%(32/39)的成员含有B类型内含子,而含有G、C、F和H类型内含子的扩展蛋白基因比例分别为15.4%(6/39),25.6%(10/39),28.2%(11/39)和2.6%(1/39)。EXPA亚家族扩展蛋白基因含有4种内含子结构模式([A]、[AB]、[GAB]和[ABH]),EXPB亚家族成员的内含子结构模式为[AB]、[ACB]、[ABF],EXLA亚家族成员的内含子结构模式为[ACBF],而EXLB亚家族的结构模式则为[ACF]、[ACBF]和[GACF]。

表4 雷蒙德氏棉扩展蛋白基因倍增模式Tab.4 The segmental duplication events of expansin gene superfamily in G.raimondii
注:倍增基因对.具有共线性关系的基因集合。
Note:Set.Collinear gene set.

图2 具有共线性关系的expansin基因间Ks分布频率Fig.2 Frequency distributions of Ks of collinear paralogous expansin gene pairs in G.raimondii
2.4 序列一致性分析及系统进化树
扩展蛋白基因序列一致性比较发现,同一亚家族内成员间编码区核苷酸序列的一致性均在48.4%以上,氨基酸序列的一致性高于44.8%;而亚家族成员间,编码区核苷酸序列的一致性均低于45.28%,氨基酸序列的一致性则低于34.38%(表5)。相对而言,EXPA与EXPB亚家族之间亲缘关系较近,氨基酸序列一致性为15.0%~31.5%;EXLA与EXLB亚家族间亲缘关系更近,氨基酸序列一致性为29.5%~37.3%,而EXPA或EXPB与EXLA或EXLB间的氨基酸序列一致性相对较低(一般小于28.9%)。因此,基因编码区序列一致性比对可以作为扩展蛋白亚家族分型的重要依据。

表5 雷蒙德氏棉扩展蛋白家族成员间核苷酸及氨基酸序列一致性分析Tab.5 Identity of nucleotides(above) and amino acid(below) between two subfamilies of expansin genes in G.raimondii
依据扩展蛋白基因的氨基酸序列构建系统进化树显示,每一亚家族的成员往往聚集成群(图3)。依据内含子特点、相似程度的差异以及共线性分析[5,33],这些基因可进一步划分为15个大的分支(EXPA、EXPB、EXLA和EXLB亚家族分别包含10,2,1,2个分支),每1分支中的扩展蛋白基因之间,无论是氨基酸序列还是基因的结构组成都具有更大的一致性。如具有共线性关系的基因GrEXPA01/GrEXPA04/GrEXPA10、GrEXPA05/GrEXPA08/GrEXPA09、GrEXPA06/GrEXPA17/GrEXPA24、GrEXPA16/GrEXPA20/GrEXPA21/GrEXPA23、GrEXLA1/GrEXLA2/GrEXLA3分别位于同一分支上,其氨基酸序列的相似性分别达到79.0%,77.3%,81.0%,90.1%,80.0%,并且外显子数量、每个外显子大小也保持较高的一致性,预示其基因起源甚至功能方面有一定的关联,也暗示着高等植物进化过程中普遍存在三倍化(Triplicated)甚至四倍化(Tetraplicated)事件等。
2.5 雷蒙德氏棉扩展蛋白密码子偏好性分析
蛋白质中的氨基酸是由mRNA中三联体密码子决定,每种氨基酸至少对应着1个密码子,最多的对应着6个密码子(即同义密码子)。在蛋白质的合成过程中,同义密码子的使用概率并不相同,对雷蒙德氏棉扩展蛋白基因家族39个成员的10 041个密码子的使用偏好性进行统计发现,密码子有效数(Effective number of codons,ENC)为53.955,密码子偏爱指数(Codon bias index,CBI)为0.149。有些氨基酸密码子相对使用频率(Relative frequency of synonymous codon,RFSC)具有明显的密码子偏好性(表6),其中CCU(编码Pro)、GCU(编码Ala)、AGA 和AGG(均编码Arg)、UAA(编码终止子TER)分别为相应氨基酸的高频密码子(相对同义密码子使用频率单值超过60% 或超过该组同义密码子平均占有频率的1.5倍的密码子[34])。雷蒙德氏棉的4个高频密码子(CCU、GCU、AGA和AGG)在拟南芥、杨树等双子叶植物中同样为高频密码子。另外,在雷蒙德氏棉中,CCG(编码Pro)、ACG(编码Thr)、GCG(编码Ala)、CGG(编码Arg)和UGA(编码TER)属于稀有密码子。

图3 雷蒙德氏棉扩展蛋白基因的系统发生树(左)、基因结构(中)及模体结构(右)Fig.3 Phylogenetic tree,gene structure and motif analysis of the G.raimondii expansin
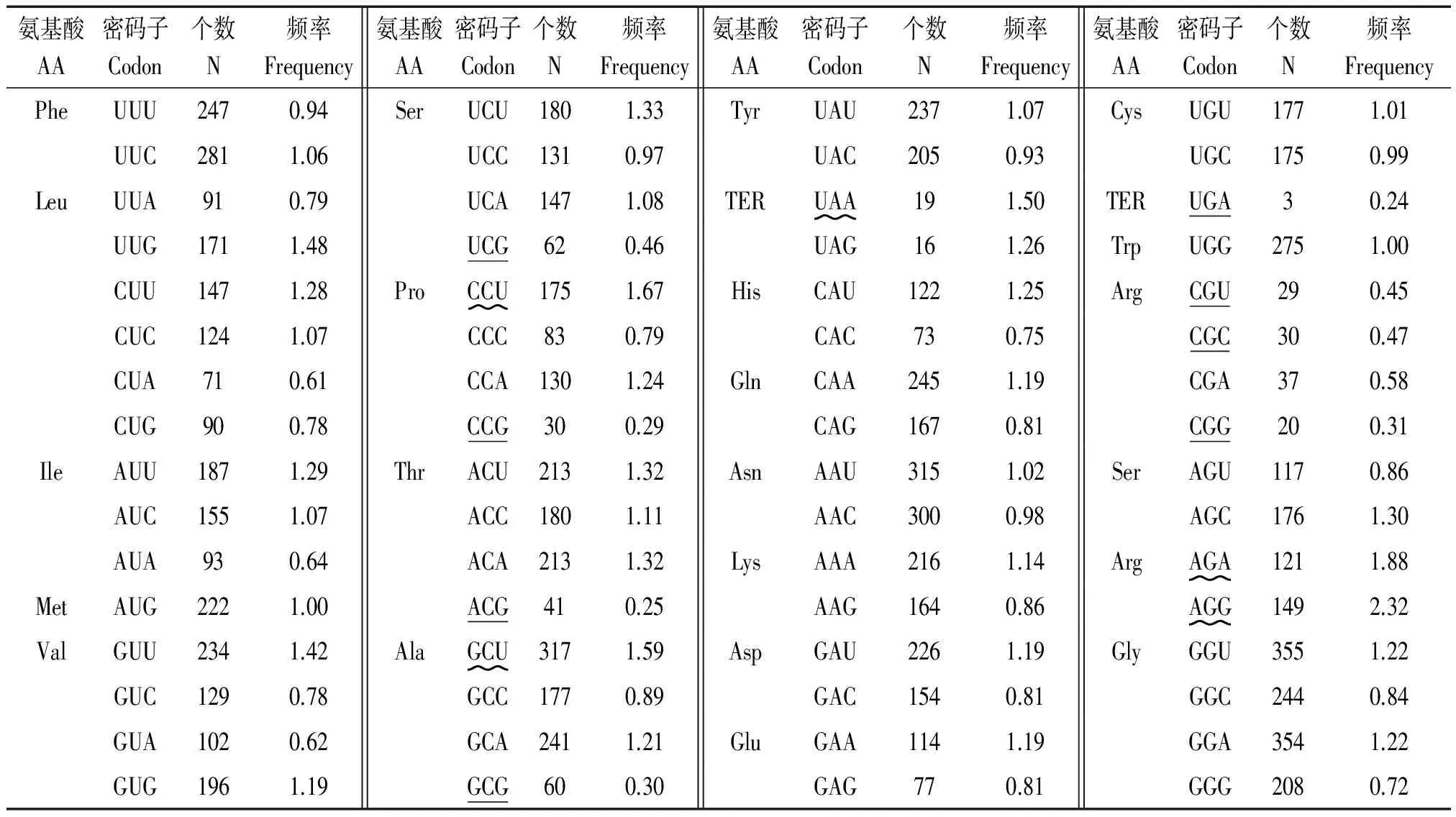
表6 雷蒙德氏棉扩展蛋白基因密码子的相对使用频率Tab.6 RFSC value of expansin genes in G.raimondii
注:下划横线和波浪线分别表示低频和高频密码子。
Note:Underline and wavy-underline indicated low-frequency and high-frequency codons.
2.6 雷蒙德氏棉扩展蛋白的一级结构
雷蒙德氏棉扩展蛋白和其他物种中的扩展蛋白具有相类似的一级结构,由信号肽、催化区(DPPB_1结构域)、结合区(Pollen_allerg_1结构域)3部分组成[1]。MEME模体分析结果如图(图3,4)所示。雷蒙德氏棉中,大部分(31/39)扩展蛋白的N端的信号肽区含有17~34个氨基酸,可以引导初生多肽透过内质网。MEME模体分析显示,信号肽区段包含Motif15(图4)。位于扩展蛋白序列中段的是催化区(Catalysis domain),其核心序列与内切葡聚糖酶GH45(纤维素酶系中的1个主要成分,可在葡聚糖链的随机位点降解底物产生寡糖)具有较高的一致性,并且含有丰富的保守性很高的半胱氨酸(Cys,即C,图4),推测与二硫键的形成有关[35]。MEME模体分析还显示,雷蒙德氏棉的EXPA亚家族的催化区由Motif2(r)+Motif6+Motif3组成;其他3个亚家族的催化区由Motif11(r)+Motif10+Motif8(l)组成。另外,在EXPA和EXPB亚家族的DPPB_1结构域中含有1个His-Phe-Asp(HFD)模体。位于扩展蛋白羧基端的部分是纤维素结合区(Cellulose-binding domain),也叫多糖结合区(Polysaccharide-binding domain),多由最后1个外显子编码,长度一般为90~120个残基,如EXPA亚家族基本上由101~104个氨基酸组成。该区段拥有丰富的色氨酸(Trp,即W),在4个亚家族之间也具有很高的保守性。有分析认为,色氨酸微环境对pH值变化非常敏感,降低pH值可以导致蛋白分子构象发生较大变化[36],并影响扩展蛋白的功能。MEME模体分析显示,EXPA亚家族的多糖结合区Motif1(r)+Motif4+Motif7(l);其他3个亚家族的催化区由Motif8(r)+Motif9+Motif12组成。
2.7 雷蒙德氏棉扩展蛋白理化特征分析
蛋白质的理化特性与其功能特征密切相关。雷蒙德氏棉39个扩展蛋白的分子量从18.48 kDa到33.77 kDa不等,平均值为27.87 kDa;其蛋白最短的有176个氨基酸残基,最长的有303个,平均值为256.46,跨度较大(表2)。根据扩展蛋白的氨基酸序列,利用软件Euk-mPLoc 2.0[37]进行亚细胞定位预测,发现雷蒙德氏棉的39个扩展蛋白全部定位于细胞外(Extracell)。

图4 雷蒙德氏棉扩展蛋白ExpA亚家族成员氨基酸序列保守性分析Fig.4 Conservation analysis of amino acid sequences among subfamily expansin A genes in G.raimondii genome
分析表明,家族39成员蛋白质的理论等电点(pI)为4.44~10.45,平均值达到8.213,表明多数雷蒙德氏棉扩展蛋白表现偏碱性,与杨树的扩展蛋白表现相近。对总平均疏水性(Grand average of hydropathicity,GRAVY)的计算发现,大部分(36/39)蛋白都属于亲水性的蛋白(-0.244~-0.015,正值表示疏水性,负值表示亲水性[38]),仅3个蛋白属于弱的疏水性蛋白。家族成员的蛋白不稳定性指数(Instability index,II)为16.96~45.39,平均值达到31.96;除GrEXPA07外,多数扩展蛋白结构稳定性较好(> 40则不稳定)。雷蒙德氏棉扩展蛋白的脂溶指数(Aliphatic index,AI)为59.64~84.98,平均值为71.17;相对较高的脂溶指数,保证该蛋白在各种环境中具有良好的稳定性,有利于其功能的正常发挥。GrEXLA1、GrEXLA2、GrEXLA3、GrEXLB2、GrEXLB4、GrEXLB5、GrEXLB6和GrEXPB3的脂溶指数甚至超过80,属于嗜热型蛋白[39]。
2.8 扩展蛋白家族成员在雷蒙德氏棉不同组织/器官、纤维发育不同时期中的表达特征分析
在雷蒙德氏棉3个不同组织/器官及纤维发育时期(成熟叶片、花瓣、0 d胚、2 d纤维、3 d胚、10 d种子、20 d种子、30 d种子和40 d种子)的转录组测序和芯片杂交数据中,7个基因表达试验项目(表1)共检测到34个扩展蛋白基因的表达(图5),其他5个基因(GrEXPA08、GrEXPA09、GrEXPA10、GrEXPB3和GrEXLB4)一直未检测到表达信号,推测这5个扩展蛋白基因为假基因。在花瓣、叶片和纤维中分别有15,10,6个基因没有表达信号。基于基因芯片的表达数据显示:19个基因未检测到表达;深度测序数据显示:6个基因未检测到表达。
基因芯片和深度测序显示:GrEXLA1、GrEXLA2和GrEXPA16在花瓣中表达量较高;在叶片中,GrEXPA23和GrEXPA24表达量较高,而GrEXLA1和GrEXPA02表达量较低。GrEXPA11、GrEXPA16、GrEXPA21和GrEXLB2在叶片和花瓣中活跃表达而在纤维中迟钝或沉默。这些结果表明,扩展蛋白基因的表达在不同的组织中具有特异性。
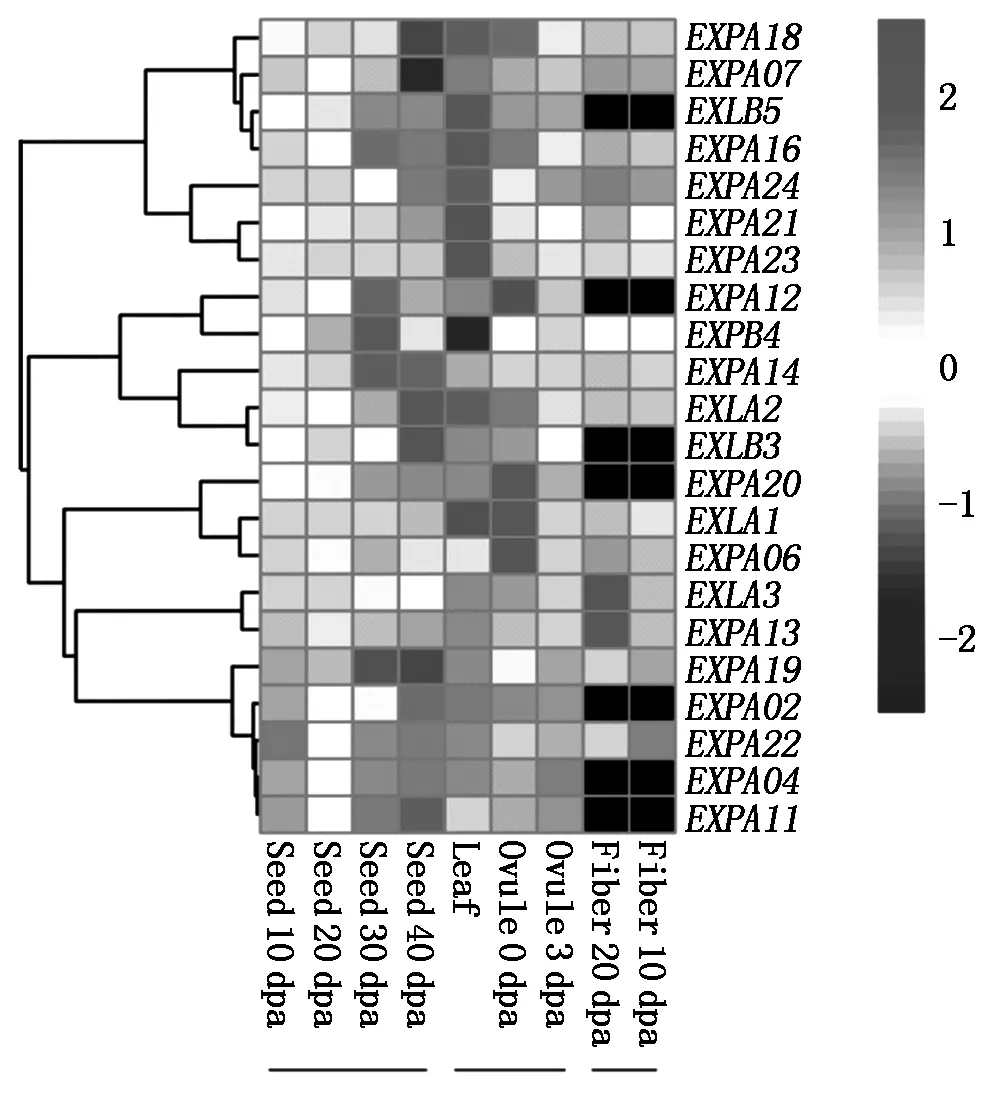
利用深度测序数据分析扩展蛋白基因在棉纤维发育阶段及成熟叶片中的表达谱。Levels of gene expression are depicted in different color on the right scale.
在纤维发育的起始阶段(0~8 d),GrEXPB1、GrEXPB2和GrEXLB2的表达量极低,说明这些基因可能不会促进胚珠的表皮细胞发育成纤维。GrEXPA22仅在起始期甚至伸长期2个时期表达,后期不再表达。GrEXLA3和GrEXLB5在伸长期的表达量显著高于纤维发育的其他时期。GrEXPA19前期(起始期、伸长期、次生壁合成期共3个时期)活跃,异常高表达,在0~20 d时间内表达量特高,进入成熟期(20 d以后)表达量迅速下降,接近为0。GrEXPA24在纤维的整个发育期均表达,但表达量持续地显著(P<0.05)下降。与此相反,GrEXPA02、GrEXPA12、GrEXPA14、GrEXPA15、GrEXPA17、GrEXPA18、GrEXLA2和GrEXLB3在次生壁合成期及成熟期高量表达。特别是GrEXPA17和GrEXPA23,在成熟期表达量特高。GrEXPA13在次生壁合成期表达量高。这些结果都显示,扩展蛋白基因的表达特征呈现多样化,表达在纤维发育的某一时期具有特异性。在3个基因表达研究项目(SRP017168、SRP009820和SRP001603)内,进行组织/时期间的比较分析(图5),发现18个扩展蛋白基因的表达丰度,在不同时期内存在显著差异(P<0.05),推测这18个基因在棉纤维发育各个过程中均起重要作用。
总的来看,亚家族内的不同基因有不同的表达特征,表明进化进程中,亚家族内同类基因其结构的趋异性和功能分化的多样性可能是相互关联的。
3 讨论与结论
本试验在全基因组水平上对雷蒙德氏棉扩展蛋白基因进行了鉴定和特征分析。雷蒙德氏棉扩展蛋白家族共有39个成员,其中包括26个EXPA、4个EXPB、3个EXLA和6个EXLB基因,与拟南芥、杨树、苹果、菜豆中该基因家族成员个数接近(表3)。Sampedro 等[1]认为,在植物界各物种分化前的共同祖先中,扩展蛋白家族大约含有15~17个成员。在后续的进化过程中,植物界各物种的基因组大多经历了多次的全基因组复制事件[30],这是基因家族成员数量扩张的重要原因。本试验表明,雷蒙德氏棉扩展蛋白家族成员数量相比于拟南芥、杨树、苹果、菜豆的成员数量,在进化过程中经历了相同次数的复制事件。Ks的分布频率曲线和成员数量均表明,进化中雷蒙德氏棉扩展蛋白大约经历了3次复制事件。将蛋白序列提交Euk-mPLoc网站,亚细胞定位预测显示39个成员全部定位于细胞外,表明该家族蛋白的亚细胞定位分化具有高度保守性。
雷蒙德氏棉的基因组经历了全基因组重复、染色体重排和串联重复等复杂的进化过程[40]。本试验结果表明,染色体大片段复制在扩展蛋白家族成员的扩增中起到了重要作用。在雷蒙德氏棉中共有30个扩展蛋白家族基因形成11个共线性支持的基因群(由同一基因祖先扩增而来),基因群内的基因在系统发育树中大多也处于同一分支内。根据系统发育树的分支进行分组,比较亚家族下的亚组(分支)内扩展蛋白家族基因的内含子、外显子排布情况、UTR长短、信号肽的有无及基序(结构域、信号肽)的分布情况,可以发现,同一亚组(分支)中各基因的基因结构具有一定的差异。但利用MEME进行模体分析,结果显示同一亚组(分支)中各基因翻译出的蛋白质具有类似(相对保守)的氨基酸结构。从不同时空的表达数据来看,这些由同一基因祖先扩增而来的基因群,并不具有相似的表达模式,说明在进化过程中其表达时空特性发生趋异分化现象,这可能与基因家族扩张后基因结构的趋异化有一定的联系。
雷蒙德氏棉是现今四倍体栽培棉物种的D亚组染色体的祖先的现代种,研究扩展蛋白基因的多次复制现象可以帮助人们更好地了解棉属基因组多倍化的形成过程。将棉花与水稻、拟南芥、杨树等12个物种的扩展蛋白基因家族进行比较,发现所列各物种的扩展蛋白家族成员个数并不与基因组大小完全成正比,说明扩展蛋白基因家族在各物种中的进化和复制具有多样性。众所周知,多倍体化(Paleopolyploidy)在陆生植物的进化过程中多次发生,并且这些多倍化事件对植物基因家族的扩张起到了重要作用,最终产生了基因冗余。冗余是生物为适应外界不利环境的一种对策,对生命系统有重要的意义[41]。雷蒙德氏棉扩展蛋白家族基因成员的冗余,也从侧面表明了该家族成员在其生长发育过程中的重要作用。该家族各亚族内成员之间在蛋白质水平有很强的保守性:MEME软件Motif分析的结果表明,各亚族的序列相似性很高;但从基因结构来看,各亚族的成员间基因结构存在一定差异,如:内含子模式存在多样性、UTR长短不一等,这可能与基因组进化过程中的串联重复、随机重复与插入等现象有关。据此推测,在基因冗余发生后,通过非编码区的趋异化,从而改变冗余基因间表达的时空依赖性,达到节约胞内资源,稳定细胞代谢,适应胞外环境的能力(Functional specialization)。深度测序和基因芯片的数据也表明,具有共线性关系的基因之间,其表达模式各不相同,其原因可能也在于此。扩展蛋白家族基因从植物界共同祖先的15~17个成员扩增到雷蒙德氏棉的39个成员,有可能经历了3次多倍化事件。在每次的多倍化事件后,因为基因的冗余,少数扩展蛋白基因可能经历了丢失(Selective loss of genes)或者去功能化(Dysfunctional或Neo-functionalization)或者转变(Gene conversion),剩余基因在纯化选择(Negative selection)压力(如ω值所示)的作用下进化成现今的39个成员。
纤维发育进程中涉及大量基因的表达,其中扩展蛋白基因在处于伸长期的纤维细胞中表达异常活跃[42-44],而在短绒或无纤维突变体中下调表达[45]。因此,扩展蛋白基因可能对纤维发育极其重要,对促进纤维(单)细胞的胞壁的伸长具有重要的作用,从而最终影响纤维的长度和强度。Harmer等[43]在纤维细胞中发现6个编码α扩展蛋白的cDNA(GhExp1~GhExp6),RT-PCR表达分析表明,GhExp1(属于扩展蛋白A亚家族)仅在伸长期的纤维中高量表达,GhExp2 (A亚家族)也是仅在伸长期的纤维中表达,表明这2个基因可能促进纤维的伸长。转基因过量表达GhEXPA1显示,GhEXPA1与GhRDL1互作,可以显著地增加单株铃数,从而提高纤维产量40%而对纤维品质无负面影响[46]。He等[47]通过关联作图发现,基因Exp2的序列多态性与纤维品质的变异显著相关。基因GbEXPA2在伸长阶段的纤维中显著地上调表达[48],深入的研究发现GbEXPA2的启动子主要在纤维中活跃表达[49]。基于陆地棉与海岛棉种间渗入系的基因表达分析显示,与纤维品质低劣的渗入系相比较,扩展蛋白基因(α-expansin 1、expansin-like B1等)在纤维品质优良的渗入系里表达更活跃,进一步试验发现,在纤维的伸长阶段扩展蛋白基因表达显著升高[50]。由此推测,扩展蛋白家族一些基因成员在棉纤维的发育过程中对纤维细胞的伸长起到调控作用,当这些基因表达下调时,会影响棉纤维的发育,导致纤维的重要品质指标(长度和比强度)下降。
本试验对雷蒙德氏棉扩展蛋白家族基因在不同时空范围的表达谱进行了分析发现,除了5个基因外,其余扩展蛋白基因广泛参与到棉纤维、叶片、花瓣等组织发育的复杂生理过程中。在纤维发育的不同时期,特异性表达的扩展蛋白基因各不相同,在纤维发育后期转录量上升的扩展蛋白基因,如GrEXPA17和GrEXPA23等,可能协助纤维细胞脱水与成熟;在中期转录量高的基因,如GrEXPA13等,可能协同其他基因一起促进纤维初生壁的延展和次生壁纤维素的沉积;而在分化时期转录水平显著较高的扩展蛋白基因,如GrEXPA22,可能参与胚珠表皮细胞的分化和突起过程。特别指出GrEXPA19在20 dpa前的胚珠中转录水平很高,推测其在纤维发育中起着重要作用。本试验利用RNAseq数据和芯片数据对扩展蛋白基因家族在棉花纤维等发育过程中的作用进行了初步探索,而其参与每一发育过程的分子调控机制还有待进一步的研究。
本试验利用雷蒙德氏棉基因组测序的数据库,首次对棉花中扩展蛋白家族基因进行了鉴定和生物信息学分析,鉴定出了39个扩展蛋白家族的成员,并对它们进行了进化分析、基因结构分析、蛋白特征分析以及时空表达模式分析,发现雷蒙德氏棉中扩展蛋白家族基因成员参与了纤维、叶片、花瓣等多种组织的发育过程。鉴定和研究雷蒙德氏棉扩展蛋白家族的特征可为进一步探讨陆地棉、海岛棉相关基因的功能提供依据,对创制优异的种质资源、培育优良的棉花新品种具有重要的意义。
[1] Sampedro J,Cosgrove D J.The expansin superfamily[J].Genome Biology,2005,6(12):242.
[2] Mcqueen-Mason S,Cosgrove D J.Disruption of hydrogen-bonding between plant-cell wall polymers by proteins that induce wall extension[J].Proceedings of the National Academy of Sciences of the United States of America,1994,91(14):6574-6578.
[3] Dal Santo S,Vannozzi A,Tornielli G B,et al.Genome-Wide analysis of the expansin gene superfamily reveals Grapevine-Specific structural and functional characteristics[J].PLoS One,2013,8(4):e62206.
[4] Zhang S Z,Xu R R,Gao Z,et al.A genome-wide analysis of the expansin genes inMalus×Domestica[J].Molecular Genetics and Genomics,2014,289(2):225-236.
[5] Sampedro J,Carey R E,Cosgrove D J.Genome histories clarify evolution of the expansin superfamily:New insights from the poplar genome and pine ESTs[J].Journal of Plant Research,2006,119(1):11-21.
[6] Zhang W,Yan H W,Chen W J,et al.Genome-wide identification and characterization of maize expansin genes expressed in endosperm [J].Mol Genet Genomics,2014,289:1061-1074.
[7] Zhu Y,Wu N N,Song W L,et al.Soybean(Glycinemax) expansin gene superfamily origins:segmental and tandem duplication events followed by divergent selection among subfamilies [J].Bmc Plant Biol,2014,14(1):1-19.
[8] Krishnamurthy P,Hong J K,Kim J A,et al.Genome-wide analysis of the expansin gene superfamily revealsBrassicarapa-specific evolutionary dynamics upon whole genome triplication[J].Molecular Genetics and Genomics,2015,290(2):521-530.
[9] Kende H,Bradford K,Brummell D,et al.Nomenclature for members of the expansin superfamily of genes and proteins[J].Plant Molecular Biology,2004,55(3):311-314.
[10] Cosgrove D J,Li L C,Cho H T,et al.The growing world of expansions[J].Plant & Cell Physiology,2002,43(12):1436-1444.
[11] Yan A,Wu M J,Yan L M,et al.AtEXP2 is involved in Seed germination and abiotic stress response inArabidopsis[J].PLoS One,2014,9(1):e85208.
[12] Yu Z M,Kang B,He X W,et al.Root hair-specific expansins modulate root hair elongation in rice[J].Plant Journal,2011,66(5):725-734.
[13] Guo W B,Zhao J,Li X X,et al.A soybean beta-expansin geneGmEXPB2 intrinsically involved in root system architecture responses to abiotic stresses[J].Plant Journal,2011,66(3):541-552.
[14] Gray-Mitsumune M,Mellerowicz E J,Abe H,et al.Expansins abundant in secondary xylem belong to subgroup a of the alpha-expansin gene family [J].Plant Physiology,2004,135(3):1552-1564.
[15] Cosgrove D J,Bedinger P,Durachko D M.Group I allergens of grass pollen as cell wall-loosening agents[J].Proceedings of the National Academy of Sciences of the United States of America,1997,94(12):6559-6564.
[16] Pezzotti M,Feron R,Mariani C.Pollination modulates expression of thePPALgene,a pistil-specific beta-expansin[J].Plant Molecular Biology,2002,49(2):187-197.
[17] Cosgrove D J.Expansive growth of plant cell walls[J].Plant Physiology and Biochemistry,2000,38(1/2):109-124.
[18] Belfield E J,Ruperti B,Roberts J A,et al.Changes in expansin activity and gene expression during ethylene-promoted leaflet abscission inSambucusnigra[J].Journal of Experimental Botany,2005,56(413):817-823.
[19] Han Y Y,Li A X,Li F,et al.Characterization of a wheat(TriticumaestivumL.) expansin gene,TaEXPB23,involved in the abiotic stress response and phytohormone regulation[J].Plant Physiology and Biochemistry,2012,54:49-58.
[20] Zhao M R,Han Y Y,Feng Y N,et al.Expansins are involved in cell growth mediated by abscisic acid and indole-3-acetic acid under drought stress in wheat[J].Plant Cell Reports,2012,31(4):671-685.
[21] Choi D,Lee Y,Cho H T,et al.Regulation of expansin gene expression affects growth and development in transgenic rice plants[J].The Plant Cell,2003,15(6):1386-1398.
[22] Ding X H,Cao Y L,Huang L L,et al.Activation of the indole-3-acetic acid-amido synthetase GH3-8 suppresses expansin expression and promotes salicylate-and jasmonate-independent basal immunity in rice[J].Plant Cell,2008,20(1):228-240.
[23] Russell S D,Bhalla P L,Singh M B.Transcriptome-based examination of putative pollen allergens of rice(Oryzasativassp. japonica) [J].Molecular Plant,2008,1(5):751-759.
[24] Tabuchi A,Li L C,Cosgrove D J.Matrix solubilization and cell wall weakening by beta-expansin(group-1 allergen) from maize pollen[J].Plant Journal,2011,68(3):546-559.
[25] Ferguson D L,Turley R B,Triplett B,et al.Comparison of protein profiles during cotton(GossypiumhirsutumL.)fiber cell development with partial sequences of two proteins[J].Food Chemistry,1996,44(12):4022-4027.
[26] Haigler C H,Betancur L,Stiff M R.Cotton fiber:a powerful single-cell model for cell wall and cellulose research[J].Frontiers in Plant Science,2012,3(4):104.
[27] Xu Z Y,Kohel R J,Song G L,et al.Gene-rich islands for fiber development in the cotton genome[J].Genomics,2008,92(3):173-183.
[28] Gou J Y,Wang L J,Chen S P,et al.Gene expression and metabolite profiles of cotton fiber during cell elongation and secondary cell wall synthesis [J].Cell Res,2007,17(5):422-434.
[29] Wang K,Wang Z,Li F,et al.The draft genome of a diploid cottonGossypiumraimondii[J].Nature Genetics,2012,44(10):1098-1103.
[30] Paterson A H,Wendel J F,Gundlach H A,et al.Repeated polyploidization ofGossypiumgenomesand the evolution of spinnable cotton fibres[J].Nature,2012,492(7429):423.
[31] He D H,Lei Z P,Tang B S,et al.Identification and analysis of theTIFYgene family inGossypiumraimondii[J].Genetics and Molecular Research,2015,14(3):10119-10138.
[32] 郝 西,理向阳,腊贵晓,等 黄瓜扩展蛋白基因家族的鉴定与生物信息学分析[J].分子植物育种,2015,13(10):2280-2899.
[33] Sampedro J,Lee Y,Carey R E,et al.Use of genomic history to improve phylogeny and understanding of births and deaths in a gene family[J].The Plant Journal:for Cell and Molecular Biology,2005,44(3):409-419.
[34] 林 涛,倪志华,沈明山,等.高频密码子分析法及其在烟草密码子分析中的应用[J].厦门大学学报:自然科学版,2002,41(5):551-554.
[35] 许成钢,范晓军,付月君,等.二硫键的形成与蛋白质的氧化折叠[J].中国生物工程杂志,2008,28(S1):259-264.
[36] 阎伯旭,曲音波,高培基,等.色氨酸残基在内切酶葡聚糖酶分子中的作用[J].中国生物化学与分子生物学报,1998,14(2):181-185.
[37] Chou K C,Shen H B.A new method for predicting the subcellular localization of eukaryotic proteins with both single and multiple sites:Euk-mPLoc 2.0[J].PLoS One,2010,5(3):e9931.
[38] 付海辉,辛培尧,许玉兰,等.几种经济植物UFGT基因的生物信息学分析[J].基因组学与应用生物学,2011,30(1):92-102.
[39] Ikai A.Thermostability and aliphatic index of globular-proteins[J].Journal of Biochemistry,1980,88(6):1895-1898.
[40] Lin L F,Gj P,Bowers J E,et al.A draft physical map of a D-genome cotton species(Gossypiumraimondii) [J].BMC Genomics,2010,11(1):395.
[41] Zheng X W,Zhu J H,Kapoor A,et al.Role ofArabidopsisAGO6 in siRNA accumulation,DNA methylation and transcriptional gene silencing[J].EMBO Journal,2007,26(6):1691-1701.
[42] Arpat A B,Waugh M,Sullivan J P,et al.Functional genomics of cell elongation in developing cotton fibers[J].Plant Molecular Biology,2004,54(6):911-929.
[43] Harmer S E,Orford S J,Timmis J N.Characterisation of six alpha-expansin genes inGossypiumhirsutum(upland cotton) [J].Mol Genet Genomics,2002,268:1-9.
[44] Lacape J M,Claverie M,Vidal R O,et al.Deep sequencing reveals differences in the transcriptional landscapes of fibers from two cultivated species of cotton[J].PLoS One,2012,7(11):e48855.
[45] Padmalatha K V,Patil D P,Kumar K,et al.Functional genomics of fuzzless-lintless mutant ofGossypiumhirsutumL.cv.MCU5 reveal key genes and pathways involved in cotton fibre initiation and elongation [J].Bmc Genomics,2012,13(1):1-15.
[46] Xu B,Gou J Y,Li F G,et al.A cotton BURP domain protein interacts with-Expansin and their Co-Expression promotes plant growth and fruit production[J].Molecular Plant,2013,6(3):945-958.
[47] He D,Lei Z,Xing H,et al.Exp2 polymorphisms associated with variation for fiber quality properties in cotton(Gossypiumspp.) [J].The Crop Journal,2014,2(5):315-328.
[48] Tu L L,Zhang X L,Liang S G,et al.Genes expression analyses of sea-island cotton(GossypiumbarbadenseL.)during fiber development[J].Plant Cell Reports,2007,26(8):1309-1320.
[49] Li Y,Tu L L,Ye Z X,et al.A cotton fiber-preferential promoter,PGbEXPA2,is regulated by GA and ABA inArabidopsis[J].Plant Cell Reports,2015,34(9):1539-1549.
[50] Fang L,Tian R P,Li X H,et al.Cotton fiber elongation network revealed by expression profiling of longer fiber lines introgressed with differentGossypiumbarbadensechromosome segments[J].BMC Genomics,2014,15(1):1-15.
Genome-wide Identification and Characterization of Expansin Gene Family in Gossypium raimondii
LEI Zhongping1,2,HE Daohua1,HAI Jiangbo1,XING Hongyi1,ZHAO Junxing1,CHENG Xueni2
(1.College of Agronomy,Northwest A&F University,Yangling 712100,China; 2.College of Life Sciences,Northwest A&F University,Yangling 712100,China)
Expansin can loosen the components of rigid plant cell walls and thereby allow cell expansion.To get insight into expansin genes inGossypiumraimondii,genome-wide exploration and comprehensive characterization of theG.raimondiiexpansin gene family members were conducted,and 39 expansin genes(including 26EXPA,4EXPB,3EXLAand 6EXLB,which was classified based on the phylogenetic tree) were identified.The results revealed that expansin family genes were located on 12 of 13G.raimondiichromosomes.And the gene structures were relatively diverse(not conserved) in each subgroup.Evolutionary analysis of expansins revealed that chromosome segmental duplications contributed mainly to the three evolutionary expansions of expansin family,which had experienced negative selection pressure.The expression pattern of expansin genes under series of fiber development stages,in leaf and petal indicated that most expansin genes(showing diverse and specific expression pattern) might participate in fiber development processes including fiber initiation and elongation,and in morphogenesis of leaf and petal.This identification and characterization provided the complete profiles ofGossypiumexpansin family genes for future study on their functions related to the molecular mechanisms of fiber and other tissues development.
Gossypiumraimondii;Expansin;Gene structure;Phylogenic analysis;Biological evolution;Expression pattern
2016-07-21
现代农业产业技术体系建设专项资金(CARS-18-45);转基因生物新品种培育科技重大专项(2014ZX08005-002)
雷忠萍(1979-),女,湖北保康人,实验师,在读硕士,主要从事棉花遗传学研究。
海江波(1966-),男,陕西扶风人,副教授,博士,主要从事高效耕作制度及农业生态研究。 贺道华(1975-),男,湖北随州人,讲师,博士,主要从事棉花生物技术育种研究。
Q78
A
1000-7091(2016)06-0044-12
10.7668/hbnxb.2016.06.008
