甘草SRAP-PCR正交试验设计优化及引物筛选
2017-01-17艾鹏飞靳占忠
艾鹏飞,苏 姗,靳占忠
(河北科技大学生物科学与工程学院,河北石家庄 050018)
甘草SRAP-PCR正交试验设计优化及引物筛选
艾鹏飞,苏 姗,靳占忠
(河北科技大学生物科学与工程学院,河北石家庄 050018)
为了建立甘草SRAP技术,采用四因素(Taq酶,Mg2+,dNTP,引物)四水平的正交试验设计[L16(44)]对甘草进行SRAP-PCR试验,电泳结果采用软件SPSS分析,退火温度和引物筛选采用单因子试验。结果发现,4种因素对甘草SRAP-PCR的影响依次为dNTP>Taq酶>Mg2+>引物;优化后的甘草SRAP-PCR体系(20 μL)为1×PCR缓冲液,引物0.6 μmol/L,Mg2+2.5 mmol/L,Taq酶 1.5 U,dNTP 0.3 mmol/L,模板DNA 40 ng;最佳的退火温度为50 ℃;81对引物中有22对扩增出明亮、清晰的谱带。该优化的SRAP-PCR反应体系为进行甘草资源遗传分析提供了技术支持。
植物遗传学;甘草;SRAP;PCR;正交试验设计;引物筛选
相关序列扩增多态性(sequence-relatedamplifiedpolymorphism,SRAP)是基于PCR的一种显性标记,它通过独特的双引物对基因的ORFs(openreadingframes,开放阅读框)特定区域进行扩增(上游引物17bp,扩增外显子区域;下游引物18bp,扩增内含子、启动子区域)。
由于不同物种、同物种的不同个体彼此之间的启动子、外显子、内含子和间隔区域长度不同,SRAP-PCR的结果表现出多态性。相较于其他的分子标记(如RAPD,SSR,ISSR,AFLP等),SRAP具有引物通用性强、操作简单、PCR产物稳定、多态性丰富等优点[1],目前广泛应用于物种鉴定[2]、遗传多样性分析[3-4]、遗传图谱构建[5]、比较基因组学[6]等方面。
本试验采用L16(44)正交试验设计[7],对甘草SRAP-PCR反应体系中的4个因素(Taq酶,Mg2+,dNTP,引物)在4个水平上进行试验,优化甘草的SRAP-PCR体系,为评价和利用其种质资源提供技术支撑。
1 材料和方法
1.1 试验材料
以本研究小组选育出来的甘草(Glycyrrhiza uralensisFisch.)优系JA-1(甘草酸质量分数为3.2%)为试材,SRAP引物序列(见表1)由北京赛百盛生物公司合成,dNTP和Taq酶等购自宝生物工程(大连)有限公司。
1.2 试验方法
1)DNA提取与电泳检测
CTAB法[8]提取甘草株系JA-1叶片基因组DNA,经1.2%(质量分数,下同)琼脂糖凝胶电泳检测,无菌水稀释至质量浓度为40 ng/μL,-20 ℃冰箱保存。
2)SRAP-PCR正交试验设计
采用引物组合M5-E5(表1)对影响SRAP-PCR反应的dNTP,Mg2+,引物,Taq酶进行四因素四水平的正交L16(44)试验设计(表2)。在20 μL的PCR反应体系中,含有1×PCR 缓冲液和40 ng模板DNA,其他成分含量按表2进行,共16个处理,3次重复。
PCR反应程序:94 ℃预变性5 min;94 ℃变性40 s,35 ℃复性45 s,72 ℃延伸1 min,5次循环;94 ℃变性40 s,50 ℃复性45 s,72 ℃延伸1 min,30次循环;72 ℃延伸8 min。1.2%琼脂糖凝胶(添加0.5 μg/mL EB)电泳扩增产物后,采用凝胶成像分析仪拍照。基于条带清晰和带型丰富程度,电泳图谱中最佳组合记为16分,其次记15分,依次类推,最差的记1分,统计结果采用软件SPSS V13.0分析。
3)退火温度优化
基于正交试验设计筛选出的最佳组合,采用单因子试验对退火温度进行优化。退火温度5个水平依次为46,48,50,52,54 ℃。
4)SRAP引物筛选
基于试验结果得出的SRAP-PCR最佳反应体系和反应程序,采用81对SRAP引物组合(见表1)进行稳定性验证和引物筛选。

表1 SRAP 引物序列
2 结果与分析
2.1 SRAP-PCR正交试验设计结果直观分析
由于Taq酶、引物、dNTP和Mg2+的浓度不同,SRAP-PCR扩增的谱带条数和清晰度也不同[8],正如图1所示的正交试验设计的SRAP-PCR电泳结果。正交试验设计L16(44)的统计结果见表2,极差分析表明4个因素对SRAP-PCR反应的影响大小依次为dNTP浓度、Taq聚合酶量、Mg2+浓度、引物浓度,方差分析(见表3)表明4个因素彼此之间的差异均表现出显著性水平(P<0.05)。因此,需要对4个因素的不同水平进行分析,其SRAP-PCR结果数值见图2。

表2 SRAP-PCR 正交试验设计L16(44)
注:K1—K4分别表示各因素1—4种水平的结果平均值;R表示平均值的极差值。

图1 正交试验设计SRAP-PCR电泳结果Fig.1 Electrophoresis profiles of SRAP-PCR by orthogonal test design
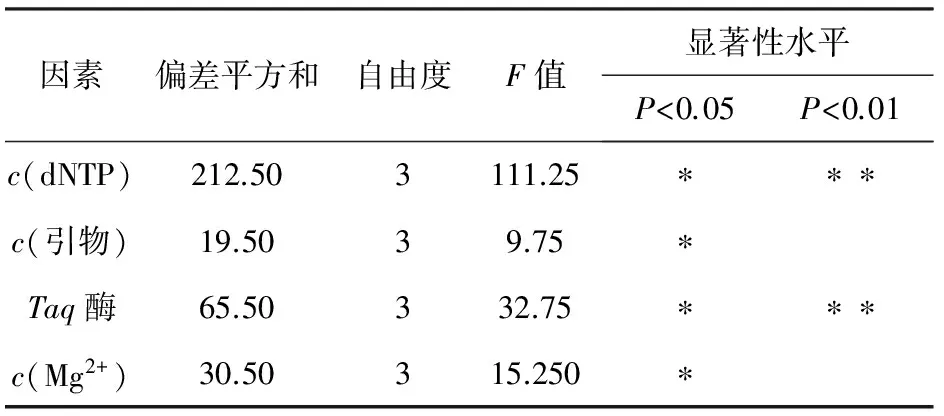
因素偏差平方和自由度F值显著性水平P<0.05P<0.01c(dNTP)212.503111.25∗∗∗c(引物)19.5039.75∗Taq酶65.50332.75∗∗∗c(Mg2+)30.50315.250∗
注:* 表示差异显著;**表示差异极显著。

图2 4个因素对SRAP-PCR 结果数值的影响Fig.2 Effects of four factors on the outcoming value in SRAP-PCR
2.2 dNTP浓度对SRAP-PCR结果数值的影响
dNTP是PCR反应的原料[9]。本试验中,dNTP浓度对SRAP-PCR结果的影响显示出极显著性水平P<0.01(见表3)。由图2可知,SRAP-PCR结果数值随着dNTP浓度0.10 mmol/L增至0.40 mmol/L时先增大后减小,且在0.30 mmol/L时结果数值最大,此时表现出带型清晰,主带明显(见图1)。应东山等[10]认为dNTP浓度过高时会结合Mg2+,使得反应体系中游离的Mg2+浓度降低。故dNTP最适浓度为0.30 mmol/L。
2.3Taq酶对SRAP-PCR结果数值的影响
表3表明,Taq酶对SRAP-PCR扩增结果的影响表现出差异极显著(P<0.01)。如图2所示,SRAP-PCR结果数值随着Taq酶用量的增加先增大后减小,且为1.5U时结果数值最大。图1中,Taq酶1.5U时谱带清晰、明亮、无杂带。因此。20μL体系中Taq酶为1.5U时较好。
2.4 Mg2+浓度对SRAP-PCR结果数值的影响
在PCR反应体系中,Mg2+是Taq酶的激活剂[10]。图2表明,Mg2+浓度为1.5 ~3.0 mmol/L时,SRAP-PCR扩增的结果数值先增大后减小,且在2.5 mmol/L时最大。其原因是Mg2+浓度过低时降低了Taq酶活性,减少了PCR产物量,过高时引起非特异性扩增[10]。故本试验最佳的Mg2+浓度为2.5 mmol/L。
2.5 引物浓度对SRAP-PCR结果数值的影响
引物浓度对SRAP-PCR结果数值的影响见图2,当引物浓度为0.2 ~0.8μmol/L时,SRAP-PCR的结果数值变化不大,其中引物浓度为0.6μmol/L时结果数值最大。图1也表明,引物浓度较低时扩增的谱带(非主带)较弱,浓度过高时谱带信号强,但同时背景加深。其原因是引物浓度过低时影响扩增效率,过高时引起与DNA模板链错配产生假阳性条带[11]。因此,引物浓度可选用0.6μmol/L。
2.6 退火温度对SRAP-PCR结果的影响
在PCR反应体系中,退火温度影响引物与DNA模板链的特异性结合[11]。由图3可知,退火温度过高或过低时扩增效果都不理想,当退火温度为50 ℃时,扩增出的谱带清晰、明亮,带型丰富。因此,适宜的退火温度为50 ℃。
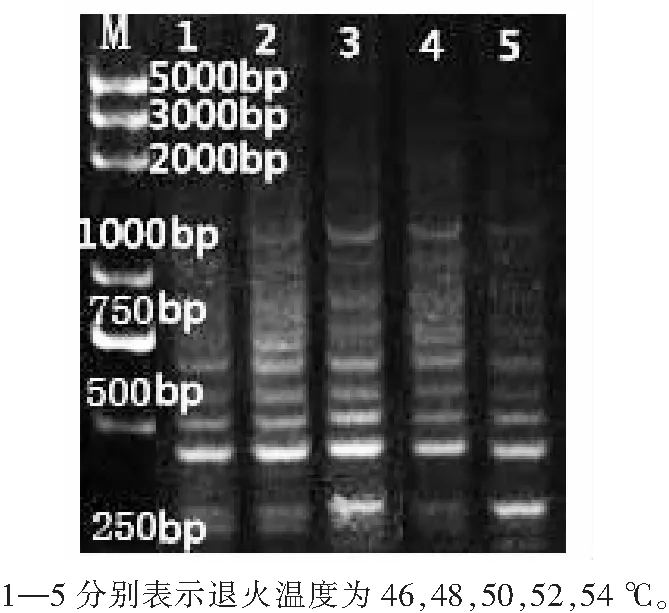
图3 不同退火温度的SRAP-PCR电泳结果Fig.3 Electrophoresis profiles of SRAP-PCR at various annealing temperatures
2.7 SRAP-PCR优化体系的验证与引物筛选
采用优化后的反应体系和反应程序,对81个SRAP引物组合(表1)进行PCR扩增,结果见图4,22对引物表现出了条带清晰、丰富,表明优化后的PCR体系适用于甘草的SRAP分析。筛选出的甘草SRAP引物组合22对(图4)依次为M1-E9,M2-E2,M2-E4,M3-E4,M3-Em8,Me4-E2,M4-E7,M4-E8,M5-E5,M5-E9,M6-E3,M6-E4,M6-E6,M7-E2,M7-E4,M7-E7,M8-E1,M8-E4,M8-E7,M9-E1,M9-E4,M9-E5。

图4 22对引物组合的SRAP-PCR电泳结果Fig.4 Electrophoresis profiles of SRAP-PCR with 22 primers combined
3 结 论
SRAP-PCR的扩增结果受到反应体系、扩增程序以及物种特性的影响[10-12]。因此,要获得条带清晰、稳定的电泳图谱,需要针对试验材料调整和优化反应体系中的不同组分和PCR程序等主要影响因子。
正交试验设计是基于统计学原理,从复因子试验中选出有代表性的水平组合进行试验,这样既减少了处理组合数,又统筹到各因素不同水平间的交互作用,相对于单因素试验和完全组合设计更加高效[7,13]。另外,在正交试验设计中,采用统计分析法可有效地考察出各组分不同水平下的差异显著性,以致试验结果分析得更加科学和完善[14]。当然,在该方法的直观分析中,对试验结果的赋值打分带有一定的主观成分,以致影响到最终各因素最佳水平确定的可靠性[15]。在具体试验中,如本研究中通过增加正交试验的重复次数和验证试验来减少直观因素带来的负面影响。
本研究基于正交试验设计优化了甘草SRAP-PCR反应体系,即在20 μL的反应液中,含dNTP 0.3 mmol/L,Taq酶1.5 U,Mg2+2.5 mmol/L,引物0.6 μmol/L,模板DNA 40 ng,1×PCR 缓冲液。通过单因素试验确定了最佳的退火温度为50 ℃。验证试验和引物筛选结果表明,该优化体系稳定、可靠,可以用于甘草的遗传分析和资源评价等研究中。
/
:
[1]LIG,QUIROSCF.Sequence-relatedamplifiedpolymorphism(SRAP),anewmarkersystembasedonasimplePCRreaction:Itsapplicationtomappingandgenetagginginbrassic[J].TAGTheoreticalandAppliedGenetics, 2001, 103 (2/3):455-461.
[2] AHMAD R, POTTER D, SOUTHWICK S M. Genotyping of peach and nectarine cultivars with SSR and SRAP molecular markers[J]. Journal-American Society for Horticultural Science, 2004, 129: 204-211.
[3] FERRIOL M, PIC B, NUEZ F. Genetic diversity of a germplasm collection of Cucurbita pepo using SRAP and AFLP markers[J].Theoretical and Applied Genetics, 2003, 107(2): 271-282.
[4] DINOLFO M I, CASTA ARES E, STENGLEIN S A. SRAP as an informative molecular marker to study the Fusarium poae genetic variability[J]. Journal of Phytopathology, 2015, 163(7/8): 657-663.
[5] LIN Zhongxu, ZHANG Xianlong, NIE Yichun, et al. Construction of a genetic linkage map for cotton based on SRAP[J]. Chinese Science Bulletin, 2003, 48(19): 2064-2068.
[6] LI G, GAO M, YANG B. Gene for gene alignment between the Brassica and Arabidopsis genomes by direct transcriptome mapping[J].Theoretical and Applied Genetics, 2003, 107(1): 168-180.
[7] 杨水云,李续娥,吴明宇,等.正交实验法在PCR反应条件优化中的应用[J].生物数学学报,2005,20(2):202-206. YANG Shuiyun, LI Xu’e, WU Mingyu, et al. Application of orthogonal design to optimize PCR conditions[J]. Journal of Biomathematics, 2005, 20(2): 202-206.
[8] 艾鹏飞,甄志军,方闪闪,等.仁用杏SRAP-PCR体系的正交设计优化[J].河北科技大学学报,2009,30(3):248-252. AI Pengfei, ZHEN Zhijun, FANG Shanshan, et al. Optimization of SRAP-PCR system for kernelled apricot by orthogonal design[J]. Journal of Hebei University of Science and Technology, 2009, 30(3): 248-252.
[9] 赵红燕,冯尚国,沈波,等.金钗石斛相关序列扩增多态性反应体系的正交优化研究[J].中草药,2010,41(8):1353-1358. ZHAO Hongyan, FENG Shangguo, SHEN Bo, et al. Optimization of sequence-related amplified polymorphism system in Dendrobium nobile based on orthogonal design[J]. Chinese Traditional and Herbal Drugs, 2010, 41(8): 1353-1358.
[10]应东山,罗海燕,王明,等.正交优化芒果SRAP扩增体系及引物筛选[J].热带作物学报,2014,35(4):700-705. YING Dongshan, LUO Haiyan, WANG Ming, et al. Optimized conditions of sequence-related amplified polymorphism(SRAP) system and primer screening for mango[J]. Chinese Journal of Tropical Crops, 2014, 35(4): 700-705.
[11]尹德洁,苏淑钗,刘肖,等.蓝莓SRAP-PCR反应体系的建立优化及引物筛选[J].东北林业大学学报,2013,41(2):35-39. YIN Dejie, SU Shuchai, LIU Xiao, et al. Establishment and optimization of SRAP-PCR system and primer screening for Vaccinium ssp[J]. Journal of Northeast Forestry University, 2013, 41(2): 35-39.
[12]王硕,何金宝,农民英,等.蓝薏苡种质资源的SRAP分子标记研究[J].中草药,2015,46(1):112-117. WANG Shuo, HE Jinbao, NONG Minying, et al. Research on SRAP molecular markers in germplasm resources of Coix lacryma-jobi[J]. Chinese Traditional and Herbal Drugs, 2015, 46(1): 112-117.
[13 ]张学宁,高志华,刘庆忠,等.蓝莓花色苷提取工艺的优化研究[J].河北工业科技,2013,30(2):66-72. ZHANG Xuening, GAO Zhihua, LIU Qingzhong, et al. Optimization of extraction procedure of anthocyanins from blueberry[J]. Hebei Journal of Industrial Science and Technology, 2013, 30(2): 66-72.
[14]周玉丽,崔广荣,胡能兵,等.甜叶菊甲基磺酸乙酯离体诱变及耐盐变异体的SRAP检测[J].中草药,2014,45(24):3612-3616. ZHOU Yuli, CUI Guangrong, HU Nengbing, et al. In vitro mutation induced by ethylmethane sulfonate in Stevia rebaudiana and SRAP identification of salt-tolerant mutants[J]. Chinese Traditional and Herbal Drugs, 2014, 45(24): 3612-3616.
[15]纪其雄,彭昕, 吴晓菁,等.三叶青相关序列扩增多态性聚合酶链式反应体系的建立与优化[J].中成药,2015,37(3):562-566. JI Qixiong, PENG Xin, WU Xiaojing, et al. Establishment and optimization of SRAP reaction system for Tetrastigma hemsleyanum[J]. Chinese Traditional Patent Medicine, 2015, 37(3): 562-566.
Optimization of SRAP-PCR system on Glycyrrhiza uralensis by orthogonal test design and selection of primers
AI Pengfei, SU Shan, JIN Zhanzhong
(School of Bioscience and Bioengineering, Hebei University of Science and Technology, Shijiazhuang, Hebei 050018, China)
In order to establish the SRAP-PCR technology forGlycyrrhizauralensis, an orthogonal design testing is used to optimize the reaction system of SRAP-PCR with 4 factors (Taqpolymerase, Mg2+, dNTP and primer) at 4 levels, and the electrophoresis profiles are analyzed by software SPSS. The annealing temperature and SRAP primer combinations are selected using the single factor experiment. The results show that effects of the 4 factors on the SRAP-PCR are dNTP,Taqpolymerase, Mg2+and primer in turn. The optimized SRAP-PCR reaction system (20 μL) forG.uralensisis constructed of 1×PCR buffer, 0.6 μmol/L primers, 2.5 mmol/L Mg2+, 1.5 UTaqpolymerase, 0.3 mmol/L dNTP and 40 ng DNA template. The optimized annealing temperature is 50 ℃. Using the optimized system, 22 primer combinations are selected among 81 primer combinations, which produced bright and clear bands. The optimized SRAP-PCR system can provide the technique support for the phylogenetic analysis ofG.uralensis.
plantgenetics;Glycyrrhiza uralensisFisch.;SRAP;PCR;orthogonaltestdesign;primerscreening
1008-1534(2017)01-0007-05
2016-06-05;
2016-11-21;责任编辑:王海云
河北省科技计划项目(14237503D-3)
艾鹏飞(1974—),男,湖北黄冈人,教授,博士,主要从事植物资源评价与应用方面的研究。
靳占忠教授。E-mail:1820482432@qq.com
Q37
A
10.7535/hbgykj.2017yx01002
艾鹏飞,苏 姗,靳占忠.甘草SRAP-PCR正交试验设计优化及引物筛选[J].河北工业科技,2017,34(1):7-11. AI Pengfei, SU Shan, JIN Zhanzhong.Optimization of SRAP-PCR system onGlycyrrhizauralensisby orthogonal test design and selection of primers[J].Hebei Journal of Industrial Science and Technology,2017,34(1):7-11.
