土壤微生物结构的T-RFLP及其法医学应用分析
2017-01-11王旭东张更谦张晓嘉王佳琦陈尚坤
王旭东, 张更谦, 张晓嘉, 王佳琦, 陈尚坤
(1.西南政法大学刑事侦查学院, 重庆 401120; 2.山西医科大学法医学院物证教研室, 山西太原 030001;3.吉林省警察学院 吉林长春 130117)
土壤微生物结构的T-RFLP及其法医学应用分析
王旭东1, 张更谦2, 张晓嘉2, 王佳琦2, 陈尚坤3
(1.西南政法大学刑事侦查学院, 重庆 401120; 2.山西医科大学法医学院物证教研室, 山西太原 030001;3.吉林省警察学院 吉林长春 130117)
目的 用末端标记限制性片段长度多态性方法(T-RFLP),观察不同地域土壤微生物构成的多态性,为法医学土壤成分分析和地域鉴别提供适当的分析方法。方法 采集吉林省和重庆市两地的土壤样本,用PowerSoil®试剂盒提取土壤细菌DNA,PCR扩增16S rRNA基因的多态性区域,引物的5’端采用FAM标记。扩增产物分别用MspⅠ、RsaⅠ内切酶消化后,用3130遗传分析仪进行电泳分离。采用主成分分析(PCA)法和主效可加护作用可乘模型(AMMI)进行统计分析。结果 采用MspⅠ酶切后,T-RFLP法可以明确分离重庆和吉林两地的土壤样本。RsaⅠ酶切后分离效果稍差。结论 T-RFLP法可以区分来自重庆、吉林两地域的土壤样本,为法医学土壤多样性分析提供了一种可靠的分析方法。
末端标记限制性片段长度多态性; 土壤细菌; 法医学
0 引言
法医学对土壤分析非常感兴趣,因为土壤很容易沾到人和物上,因此对案发现场的确定、犯罪嫌疑人的行动轨迹追踪很有意义,可为案件提供有价值的线索。以前在侦查破案中检测土壤主要是针对土壤的物理特征、化学构成,如土壤种类、颜色、颗粒大小、土壤酸碱度、主要元素、矿物和有机成分。但是,这些参数的多样性有限,不能充分地区分两种土壤样本。因此,分析土壤中微生物的差别来鉴别泥土的来源,无疑可成为提供侦查线索和证据的有效思路。环境微生物学的研究已充分证明,土壤中存在大量微生物,并且土壤中的微生物呈多样性分布,不同地区及同一地区不同地点所分布的微生物种类不同、菌株也不同。大量的微生物基因组测序显示,不同种类微生物的基因组序列存在差异,不同菌株的某些位置序列存在多态性[1-2]。
近年来发展起来的末端标记限制性片段长度多态性 (Terminal Restriction Fragment Length Polymorphism, T-RFLP技术)为这一区分策略提供了新的技术平台。该方法建立在PCR的基础上,不需要分离培养细菌,因而避免了培养方法的弊端。通过设计出细菌的通用引物,对所有细菌的16S rRNA 片段进行扩增,将扩增产物酶切,酶切后产生大小不同的片段,根据酶切产物片段图谱的差异,可明确微生物种群的差异,从而实现对检材来源的快速、准确的分析[3]。
1 材料
1.1 土壤采集
土壤样本共42个,分别来自吉林省和重庆市,具体见表1。
1.2 实验材料
PowerSoil®土壤微生物DNA提取试剂盒(MO BIO,美国);内切酶MspⅠ和RsaⅠ(New England Biolabs,英国);rTaq和dNTP(Takara,日本);引物序列为 8F: 5’FAM-AGAGTTTGATCCTGGCTCAG, 785R: 5’-ACTACCTGGGTATCTAATCC,扩增产物总长度约777 bp[4];引物合成和标记由生工生物工程(上海)股份公司完成。
2 方法
2.1 土壤DNA提取与扩增
采用MO BIO PowerSoil®土壤微生物DNA提取试剂盒,具体步骤按照试剂盒说明书进行。 简单步骤如下: 取250 mg的样品加入power Bead管中,加入C1溶液混匀,10 000 g离心10 min,将上清转移至新离心管中,加入C2溶液混匀,10 000 g离心1 min,将上清转移至新离心管中,加入C3溶液,10 000 g离心1 min,取上清转移至新离心管中,加C4溶液混匀,每次取600 μL加入过滤柱中,10 000 g离心1 min,重复3次,在过滤柱中加入C5洗涤,10 000 g离心1 min,将过滤柱换到新的离心管中,加入C6溶液,10 000 g离心1 min,收集离心管中的DNA。测定样本OD值并计算其浓度。

表1 土壤样本
用ABI PCR 9700扩增仪进行扩增。扩增条件如下:总体系50 μL,其中含 PCR缓冲液5 μL、dNTP 200 μM、引物浓度0.2 μM、rTaq 3U、DNA 100 ng。PCR反应条件:95 ℃ 5 min预变性;共36个循环:95 ℃ 10 s,55 ℃ 1 min,72 ℃ 30 s;72 ℃ 10 min,4 ℃保存。
2.2 酶切
扩增结束后,取扩增产物,分别用内切酶MspⅠ、RsaⅠ酶切。25 μL酶切体系含有缓冲液2.5 μL、内切酶(MspⅠ或者RsaⅠ)10U,扩增产物10 μL。放置于37 ℃水浴条件下过夜后煮沸灭活内切酶。
2.3 电泳和自动分析
电泳在ABI公司Genetic Analyzer 3130遗传分析仪上进行。具体步骤如下: 取去离子甲酰胺9.5 μL、0.2 μL内标(LIZ 500)和1.5 μL酶切产物混合后95 ℃ 3 min,立即冰浴,然后上样电泳。用GeneMapper 3.2分析电泳结果。显示每个实际位置,BIN设置为±0.5 bp,大于该值被认为是不同的OTU(Operational Taxonomic Units),即不同的峰。
2.4 统计分析
统计收集90~500 bp之间的所有峰高大于50的峰。记录峰位置、峰高和峰面积的大小,作为原始数据(Raw Data)。将Raw Data用T-REX软件进行在线匹配(http:∥trex.biohpc.org/),并生成矩阵数据。对所得数据分别采用IMMA分析和PCA分析。IMMA分析由T-REX在线完成,PCA分析由IBM SPSS23.0完成。
3 结果
3.1 不同地域的土壤的差别
MspⅠ酶切后T-RFLP图谱显示,吉林地区与重庆地区的样本T-RFLP差别较大。峰的大小和组合完全不同。两地区的土壤样本,其特征峰(箭头所示)的片段大小和高度以及丰度都相差较大,即拥有不同的特征峰型组合,可明确区别来自这两地区的土壤,如图1。而用RsaⅠ酶切后T-RFLP图谱,并不能将不同的地区的土壤分开(见图2)。

图1 不同地域土壤样本MspⅠ酶切后的T-RFLP图谱A:代表4号样本,B:代表9号样本,C:代表10号样本,D:代表15号样本,E: 代表16号样本,F: 代表17号样本。A、B、C来自重庆市,D、E、F来自吉林省。箭头指示特征峰。

图2 不同地域土壤样本RsaⅠ酶切后的T-RFLP图谱(a)图为根据土壤样本的峰高所做的PCA分析,(b)图为根据土壤样本的峰面积所做的PCA分析。
3.2 可重复性
重庆地区土壤样本4、样本9为在2 m2内采集的两份土壤,二者与样本10相距约50 m。用MspⅠ酶切所得T-RFLP图更为相似,其特征峰的片段大小高度相近,即采自于同一片区域的样本有一定的重复性(见图1)。
3.3 对MspⅠ酶切后T-RFLP图谱的分析
扩增产物用两种限制性内切酶MspⅠ和RsaⅠ进行酶切,对T-RFLP分别根据峰高和峰面积进行主成分分析(PCA)和主效可加护作用可乘模型(AMMI)分析。MspⅠ 酶切处理后,PCA分析可见重庆和吉林地区的样本大致分成两个群落,并且峰高和峰面积的分析结果一致。8号样本与其他样本多态性差别较大,显示出该地点土壤菌群的较高特异性(见图3)。

图3 MspⅠ酶切所得主成分分析(PCA)图(a)图为根据土壤样本的峰高所做的PCA分析,(b)图为根据土壤样本的峰面积所做的PCA分析。 可见重庆和吉林地区的样本大致分成两个群落。 8号样本与其他样本多态性差别较大,显示出该地点土壤的特异性。
用在线分析软件T-REX对吉林和重庆地区的土壤样本进行AMMI分析,分别以峰高、取样位置、取样高度作为环境因素和以峰面积、取样位置、取样高度作为环境因素。用MspⅠ酶切后,AMMI分析可以将吉林土和重庆地区土壤细菌分为两个群落。散点图中吉林地区土壤样本较为集中,重庆地区土壤样本较为离散,分析为重庆土壤取样位置、取样高度差异较大,从而导致了土壤中细菌种类和数量的较大差异,而吉林土壤取自同一高度及环境相似的农田,因而环境对细菌菌落影响较小。8号样本独立于吉林和重庆两地区土壤细菌群落外,影响因素除了位置,可能还有其较独特的环境因素。PCA 分析和AMMI分析均可较明确区分重庆与吉林地区的土壤。 但是AMMI分析后,吉林地区样本的结果显示更加集中(见图4)。

图4 MspⅠ酶切后进行T-REX在线AMMI分析数据图(a)图为根据土壤样本的峰高所得AMMI分析图,(b)图为根据土壤样本的峰面积所得AMMI分析图。吉林地区样本分布集中,重庆地区由于采样高度和采样位置的不同较为分散。其中两图中8号样本的土壤显示与其他地方土壤微生物的差异较大。
3.4 对RsaⅠ酶切后T-RFLP图谱的分析
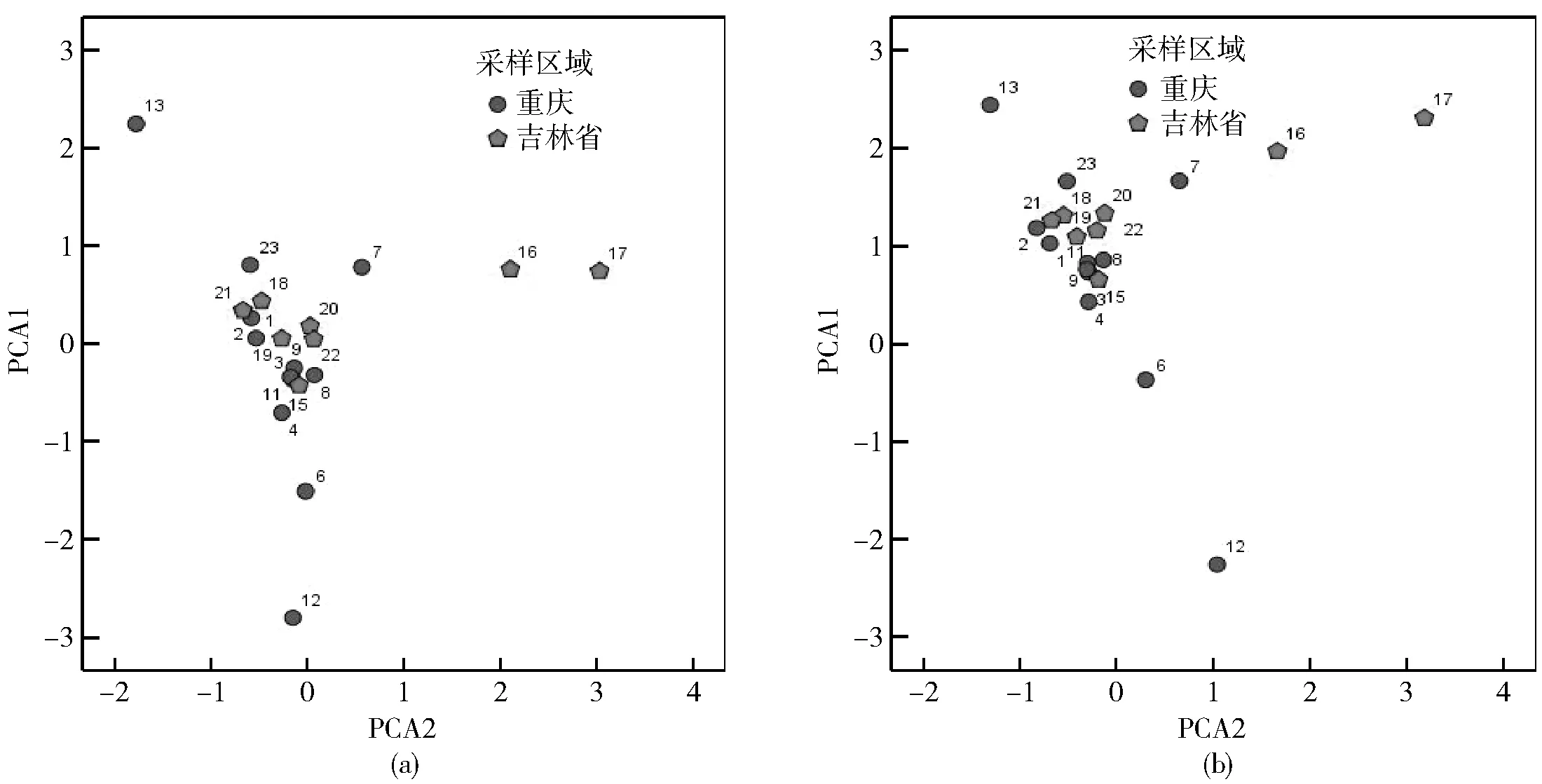
图5 RsaⅠ酶切后得到的主成分分析(PCA)分析图(a)图为根据土壤样本的峰高所做的PCA分析,(b)图为根据土壤样本的峰面积所做的PCA分析。根据分析图,RsaⅠ难以将重庆和吉林省两地的土壤样本明确区分,且多数样本较为集中。
对吉林和重庆地区的样本,经RsaⅠ酶切后的T-RFLP图谱,根据特征峰的峰高和峰面积进行PCA分析和AMMI分析,结果显示,两地区的点混杂在一起,无论用峰的有无还是峰面积均难以区分两地区土壤样本(见图5,图6)。
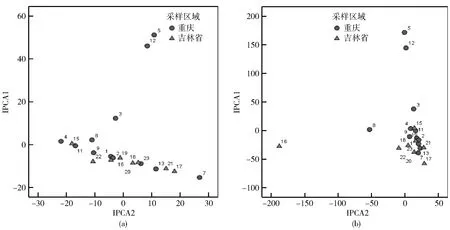
图6 RsaⅠ酶切后进行T-REX在线进行AMMI分析数据图(a)图为根据土壤样本的峰高所得AMMI分析图,(b)图为根据土壤样本的峰面积所得AMMI分析图。(a),(b)两图中样本5,样本12显示与其它区域的土壤差异较大。
4 讨论
构成土壤生态环境的因素众多,包括土壤的用途、植被、气候、光照、高度等,从而造成土壤微生物群落构成上的差别。而T-RFLP法正是以这种差别为理论基础区分各个地区土壤,即用内切酶切割后产生不同长度大小的DNA片段,通过末端标记的荧光PCR产物的电泳差异显示出来。凡有差异的土壤应该表现出不同的DNA图谱和特征峰[5-6]。
本文用T-RFLP分析不同地域土壤细菌16SrRNA基因的多态性。实验表明,采用该方法可以成功提取大部分土壤的细菌DNA并成功扩增和分析。本文中采自吉林、重庆有23份样本可成功分析,采自重庆的33份土壤样本中,有16份样本未能成功分析。吉林样本9份,一份样本未能分析。未能成功分析的样本,在DNA提取后,虽在琼脂糖凝胶电泳后观测到清晰的土壤细菌基因组DNA条带,但仍无法完成PCR过程,我们曾尝试多种方法,如更换DNA提取试剂盒,用乙醇沉淀后重新纯化DNA,改善扩增条件等方法仍无法获得有效的扩增产物。分析可能为该地区土壤腐殖酸等抑制扩增的物质含量过多,抑制PCR扩增[7]。随着模板量的增大,抑制扩增的物质也随之增加,一些样本在减少模板量的条件下可以扩增出产物。土壤成分复杂,如何有效减少扩增抑制物,这是在分析土壤时必需注意的。
两个地区的土壤细菌扩增产物采用内切酶MspⅠ处理后,可以将此二地区的土壤有效地分离开来,而内切酶RsaⅠ处理后,不能有效地将两地区土壤分开。推测是因为两种酶的酶切序列不同,MspⅠ的酶切序列为5’…C^CFF…, RsaⅠ的酶切序列为5’…GT^AC…。另一方面我们用的引物是8F-785R,能扩增出60%~90%的土壤细菌菌落DNA,但并不是全部细菌DNA,对于一些特异性菌落可能不能扩出,因此可以尝试其他引物结合不同的酶进一步改进。
我们用PCA和AMMI两种方法,通过对两种酶处理后的T-RFLP图谱的分析,结果显示,这两种方法结果一致。土壤样本距离越近,T-RFLP图谱越相似,应用此二方法所得出的位置也越接近,而且说明该两种分析方法均具有良好的可靠性和重复性。我们亦验证了AMMI相较于PCA能够发现更微小的差异,说明 AMMI模型能更透彻地分析交互作用(G×E)信息[8]。
内切酶MspⅠ处理后,用AMMI 模型分析吉林地区的土壤,结果显示分布非常集中,考虑为土壤样本来源的环境影响较为一致。来自重庆土壤样本分布较为离散,考虑可能为不同高度位置(山顶,山脚)和不同环境(水塘,路边干燥处)使细菌群落表现出了较大的差异。不同地点由近到远呈渐变趋势,如11号样本地处样本7和样本12之间,而在AMMI分析图上也位于两点之间。由于土壤的多样性,即使是取自同一地区的土壤,也有可能出现土壤菌落结构的差异。对于8号样本这样与其他菌群差异较大的样本,我们不能分析出其是哪一地区的土壤,但可以利用其特殊性,如沾在证据(如鞋、衣服)上的土壤和案发现场土壤直接进行比对,来推测其归属地的同一性。
直接提取土壤细菌全基因组DNA过程简单,扩增和结果分析易于自动化(仪器检测后以数字化的形式直接输出结果,减少了分析时人为误差),便于推广,在各地有DNA实验室的公安局即可自行检测;另外通过遗传分析仪对任何一种末端限制性片段的检测,均可以建立相应的地区土壤数据库,亦可与已知数据库的数据进行比较分析,确定样品所包含的微生物在系统中的分类地位,整个过程可在较短的时间内完成[9]。在本研究中选用16S rRNA保守序列与不同内切酶相结合的方法对土壤细菌群落进行研究,结果证明并不是所有的酶和引物都适合:16S rRNA序列与MspⅠ内切酶结合,在土壤多态性研究方面是一种有效的手段,重复性好,稳定性高,但16S rRNA序列结合RsaⅠ内切酶对不同地区土壤的鉴别能力较差。我们将进一步用不同引物和不同酶组合的方法检测土壤微生物的多样性,为法医微生物学研究土壤提供更有效的方法。
[1] LENZ E J,FORAN D R. Bacterial Profiling of Soil Using Genus-Specific Markers and Multidimensional Scaling[J]. Journal of Forensic Sciences,2010,55(6):1437-1442.
[2] LAZZARO A,HILFIKER D,ZEYER J. Structures of microbial communities in alpine soils:seasonal and elevational effects[J]. Frontiers in Microbiology,2015,6:1330.
[3] FINLEY S J,PECHAL J L,BENBOW M E,et al. Microbial Signatures of Cadaver Gravesoil During Decomposition[J]. Microbial Ecology,2016,71(3):524-529.
[4] FABRICE A,DIDIER R. Exploring Microbial Diversity Using 16S rRNA High-Throughput Methods[J]. Journal of Computer Science & Systems Biology,2009,2:69-92.
[5] HORSWELL J,CORDINER S J,Maas E W,et al. Forensic Comparison of Soils by Bacterial Community DNA Profiling[J]. Journal of Forensic Sciences,2002,47(2):350-353.
[6] MACDONALD L M,SINGH B K,THOMAS N,et al. Microbial DNA profiling by multiplex terminal restriction fragment length polymorphism for forensic comparison of soil and the influence of sample condition[J]. Journal of Applied Microbiology,2008,105(3):813-821.
[7] 葛芸英,陈松,胡兰,涂政. 土壤细菌DNA提取及多样性分析的T-RFLP方法[J]. 微生物学通报,2008(1):131-136.
[8] CULMAN S W,BUKOWSKI R,GAUCH H G,et al. T-REX:software for the processing and analysis of T-RFLP data[J]. Bmc Bioinformatics, 2009, 10(1):171.
[9] MACDONALD C A,ANG R,CORDINER S J,et al. Discrimination of Soils at Regional and Local Levels Using Bacterial and Fungal T-RFLP Profiling[J]. Journal of Forensic Sciences,2011,56(1):61-69.
(责任编辑 于瑞华)
吉林省科技发展计划重点项目“微生物16SrRNA的T-RFLP技术在法庭科学中的应用研究”(20110424)。
王旭东(1970—),男,山西平陆人,医学博士,副教授。研究方向为法医遗传学。
D919
