高山被孢霉ω-3脂肪酸脱饱和酶基因在麦胚无细胞蛋白质合成系统中的表达
2016-12-21陈海琴顾震南陈永泉
杨 芹, 陈海琴, 陈 思, 顾震南, 张 灏, 陈永泉, 陈 卫
江南大学食品学院, 江苏 无锡 214122
高山被孢霉ω-3脂肪酸脱饱和酶基因在麦胚无细胞蛋白质合成系统中的表达
杨 芹, 陈海琴*, 陈 思, 顾震南, 张 灏, 陈永泉, 陈 卫
江南大学食品学院, 江苏 无锡 214122
无细胞蛋白质合成系统具有快速、方便等特点,能表达对活细胞具有一定毒性的膜蛋白和抗菌肽,近年来被广泛应用。以来自产油丝状真菌高山被孢霉多不饱和脂肪酸合成途径中的关键膜结合酶——ω-3脂肪酸脱饱和酶为研究对象,构建了适宜体外表达ω-3脂肪酸脱饱和酶的表达载体pIVEX WG1.4-FADS15,并利用麦胚无细胞蛋白质合成系统实现了对该基因的高效表达。同时,通过在麦胚无细胞蛋白质合成过程中添加脂质体的方法,将所表达的膜蛋白正确定位至脂质体磷脂双分子层,以便目标蛋白质的正确折叠。研究结果显示,在此实验条件下目标蛋白质的表达量达1.8 mg/mL,经碘海醇密度梯度超速离心纯化后,该蛋白质的纯度可以达到90%以上。研究结果为后续对该酶进行催化特性研究和蛋白质晶体结构解析奠定了基础。
高山被孢霉;ω-3脂肪酸脱饱和酶;麦胚无细胞蛋白质合成系统;脂质体;蛋白纯化
在产油丝状真菌高山被孢霉(Mortierellaalpina)中,ω-3脂肪酸脱饱和酶是催化底物ω-6脂肪酸转化为ω-3脂肪酸的关键酶,高山被孢霉菌体中ω-6脂肪酸的含量可达到总脂的30%左右,而ω-3脂肪酸仅占总脂的3%[1]。因此,加大对ω-3脂肪酸脱饱和酶的研究是提高高山被孢霉中ω-3脂肪酸产量的必需途径。目前对ω-3脂肪酸脱饱和酶的研究主要集中在对其底物的催化特性上,有文献表明,不同来源的ω-3脂肪酸脱饱和酶序列高度保守,但其底物偏好性却存在着较大差异[2~10]。因此,全面考察ω-3脂肪酸脱饱和酶的催化特性并实现其蛋白质晶体结构的解析对提高ω-3脂肪酸的产量具有重要的指导意义。
为获得ω-3脂肪酸脱饱和酶的重组蛋白质,研究者们尝试了在不同的表达系统中重组表达不同菌株来源的ω-3脂肪酸脱饱和酶。2004年,Takahiro等[11]使用酿酒酵母异源表达了来自乳酸克鲁维酵母Saccharomyceskluyveri的ω-3脂肪酸脱饱和酶基因,发现其催化底物为十八碳和二十碳的长链脂肪酸。将串珠镰刀菌(Fusariummoniliforme)的ω-3脂肪酸脱饱和酶在大豆种子中进行表达,发现可以使其ω-3/ω-6(ALA/AA)比值提高45倍[12]。2013年,Xue等[13]在3株卵菌Oomycetes中发现了能在直链脂肪酸17位碳上进行脱饱和反应的ω-3脂肪酸脱饱和酶,并将其在耶氏解脂酵母(Yarrowialipolytica)中表达,结果显示它们可以同时利用酰基辅酶A(Acyl-CoA)形式和磷脂形式的C18和C20的长链脂肪酸底物。虽然上述报道均考察了不同来源的ω-3脂肪酸脱饱和酶的蛋白质功能,但其在细胞表达系统中表达量很低,无法进行定量检测,亦无蛋白纯化的研究报道,因此无法全面考察其酶学特性及催化活性。本实验室使用毕赤酵母表达系统(Pichiapink expression system)对来源于高山被孢霉的ω-3脂肪酸脱饱和酶进行了表达,虽对其实现了定量检测,但表达量仍停留在微克级[14]。
无细胞蛋白质合成系统作为一种开放的蛋白质表达方式,其蛋白质表达量可以达到毫克级别;而且由于该表达周期短、操作方便,近年来已被用来大量表达膜蛋白[15~17]。2005年,Ishihara等[18]使用自制的大肠杆菌无细胞抽提物合成了1 mg/mL的G蛋白耦联受体蛋白质;2008年,Goren等[19]首次使用麦胚无细胞蛋白质翻译系统成功表达了人类硬脂酰脂肪酸脱饱和酶,表达量达到了1.6 mg/mL;2009年,Shimono等[20]用无细胞蛋白质合成系统合成了跨膜蛋白质细菌视紫红素,表达量达5 mg/mL。
综上,本文以麦胚无细胞蛋白质转录翻译耦联系统为基础,结合蛋白质合成过程中脂质体的添加,成功表达了来自高山被孢霉的ω-3脂肪酸脱饱和酶基因,蛋白质表达量达1.8 mg/mL,整个反应过程操作简便、周期短。此方法为脂肪酸脱饱和酶类膜蛋白的表达提供了一种高效可行的方法,也为后续对该酶进行催化特性研究和蛋白质晶体结构解析奠定了基础。
1 材料与方法
1.1 菌株和质粒
麦胚无细胞蛋白质合成系统表达载体pIVEX WG1.4购自Roche公司;质粒pPinkα-FADS15由本实验室自行构建[14];克隆载体EscherichiacoliDH5α由本实验室提供。
1.2 主要试剂和仪器
限制性内切酶、T4 DNA连接酶、TaqDNA聚合酶、蛋白质分子量标准均购自TaKaRa公司;KOD Plus高保真DNA聚合酶购自ToYoBo公司;质粒抽提试剂盒购自天根生物科技有限公司;DNA胶回收试剂盒购自Fermentas公司;PCR产物纯化试剂盒购自上海生工生物工程技术服务有限公司;1 kb Plus DNA Ladder相对分子质量标准购自中科泰瑞生物科技有限公司;核酸染料(Goldview)购自赛百盛基因技术有限公司;麦胚无细胞蛋白质合成试剂盒购自Roche公司;大豆脂质提取物(soybean lipid extract)购自Avanti Polar Lipids公司;Accudenz购自Accurate Chemical and Scientific Corporation公司;引物由上海桑尼生物科技有限公司合成;DNA测序由北京华大基因有限公司完成。
PCR仪、电泳仪和凝胶成像系统均购自Bio-Rad公司;超速离心机购自贝克曼库尔特商贸有限公司。
1.3 方法
1.3.1 重组表达载体的构建 以本实验室构建保存的质粒pPinkα-FADS15为模板,利用引物:F:5′-ATAAGAATGCGGCCGCATGGCACCCCCTCACGTTGTC-3′和R:5′-GGCGAGCTCCTAATGCTTGTAGAACACTACGTCTCCC-3′(下划线为酶切位点)进行PCR扩增,克隆有NotⅠ和XhoⅠ的DNA片段连接至含有组氨酸亲和标签(His-tag)的表达载体pIVEX WG1.4中得到重组质粒pIVEX WG1.4-FADS15(图1,彩图见封三图版),转化至E.coliDH5α细胞,筛选阳性克隆,进行PCR验证、酶切验证及测序验证。

图1 重组质粒pIVEX WG1.4-FADS15的构建Fig.1 Schematic map of recombinant plasmid pIVEX WG1.4-FADS15. (彩图见封三图版)
1.3.2 麦胚无细胞蛋白质合成系统表达ω-3脂肪酸脱饱和酶 将重组菌株E.coliDH5α/pIVEX WG1.4-FADS15划线培养并挑取单菌落接种于50 mL LB培养基中(含100 μg/mL氨苄青霉素),37℃培养过夜后,提取质粒,并在麦胚无细胞蛋白质合成系统中表达,所用试剂盒为RTS 100 wheat germ CECF kit,各成分添加量见表1。
RTS 100 wheat germ CECF kit每个反应体积为50 μL,在900 r/min转速下24℃反应16 h,反应结束后蛋白质混合物储存于-20℃。
取5 μL反应混合物,用50 μL -20℃预冷的丙酮于4℃沉淀10 min,后4℃ 13 000 g离心30 min,待沉淀中丙酮完全挥发后,加入20 μL SDS-PAGE上样缓冲液(1×),100℃煮10 min,分别进行SDS-PAGE和Western Blot检测,具体操作参考《分子克隆实验指南》文献[21]。
表1 麦胚无细胞蛋白质合成系统(CECF)组成
Table 1 The composition of wheat-germ continuous exchange cell-free (CECF) system.

反应液体积(μL)补给液体积(μL)麦胚裂解液15氨基酸80反应混合物15甲硫氨酸20氨基酸4补给混合物900甲硫氨酸1表达载体(含2μgDNA)5~15不含DNA和RNA酶的水补充体积到50μL
1.3.3 添加脂质体条件下ω-3脂肪酸脱饱和酶的表达 取0.03 g大豆脂质提取物,溶于5 mL三氯甲烷后,真空旋转蒸发除去三氯甲烷,复溶于1 mL水合缓冲液(含有0.9% NaCl,5%葡萄糖,10%蔗糖)中,在55℃下孵育1 h,所得溶液于55℃超声5~10 min,待溶液变澄清即为浓度30 mg/mL脂质体,-80℃保存。
按照1.3.2的蛋白质表达方案,在反应10.5 h处添加上述制备的脂质体2 μL,继续以相同参数反应至16 h后结束,取样进行SDS-PAGE和Western Blot检测。
1.3.4 ω-3脂肪酸脱饱和酶重组蛋白质的纯化[22]取45 μL 1.3.3反应后混合液,加入含25 mmol/L Hepes、pH 7.4、100 mmol/L NaCl和30%(V/V)甘油的缓冲液至体积为75 μL,再加入等体积的80% Accudenz [Accudenz溶解于含有25 mmol/L Hepes,100 mmol/L NaCl,pH 7.4和10%(V/V)甘油的缓冲液中]混合,随后加入350 μL 30% Accudenz(同上),最后用100 μL缓冲液(25 mmol/L Hepes、100 mmol/L NaCl,pH 7.4)覆盖,17 000 g、4℃超速离心2 h,上清即为纯化蛋白质,取样进行SDS-PAGE和Western Blot检测。
2 结果与分析
2.1 重组表达载体的构建
重组质粒pIVEX WG1.4-FADS15在目的片段的5′和3′两端分别添加了翻译起始高效强化元件,使蛋白质表达效率提高;5′端的His-tag亲和标签可用作Western Blot检测。使用引物F和R对重组质粒进行PCR验证,得到1.2 kb单条带;NotⅠ和XhoⅠ进行双酶切验证,得到3.3 kb和1.2 kb的双条带,与预期结果一致(图2),经测序验证,目标片段序列正确。

图2 重组质粒pIVEX WG1.4-FADS15的酶切鉴定Fig.2 Restriction analysis of recombinant plasmid pIVEX WG1.4-FADS15. M:蛋白质分子量标准;1:质粒pIVEX WG1.4-FADS15;2:经NotⅠ/XhoⅠ双酶切后的pIVEX WG1.4-FADS15质粒片段;3:质粒pIVEX WG1.4-FADS15中目的基因FADS15的PCR扩增。
2.2 麦胚无细胞蛋白质合成系统表达ω-3脂肪酸脱饱和酶
麦胚无细胞蛋白质合成系统表达的ω-3脂肪酸脱饱和酶的SDS-PAGE及Western Blot结果如图3所示。Western Blot选用Anti-His Antibody作为一抗,Horseradish peroxidase (HRP)-conjugated anti-mouse IgG作为二抗。蛋白质样品预先通过丙酮沉淀以除去多余的杂质,使目的条带更清晰可见。
从图3中可以看出,相对于空白对照组,实验组在分子量45 kDa附近有一明显的条带,并与目的条带的理论分子质量相符[14];而且其表达量与葡萄糖醛酸酶(GUS)阳性对照(泳道1中66 kDa处)的表达量(>400 μg/mL)相当,由此确定麦胚无细胞蛋白质合成系统可以实现对ω-3脂肪酸脱饱和酶的表达。

图3 麦胚无细胞蛋白质合成系统表达的ω-3脂肪 酸脱饱和酶的SDS-PAGE(A)和Western Blot 分析(B).Fig.3 SDS-PAGE(A) and Western Blot analysis(B) of ω-3 fatty acid desaturase expressed in the wheat germ cell-free protein expression system.注:M:蛋白质分子量标准;1:葡萄糖醛酸酶阳性对照;2:FADS15;3:阴性对照。
2.3 添加脂质体条件下ω-3脂肪酸脱饱和酶的表达
与多数脂肪酸脱饱和酶类似,ω-3脂肪酸脱饱和酶是一种膜整合蛋白质,膜结构的存在能为其正确折叠提供合适的疏水环境[23]。本文尝试了在麦胚无细胞蛋白质合成系统合成蛋白质过程中添加脂质体以使目标蛋白质正确折叠。SDS-PAGE及Western Blot结果显示添加脂质体后目标蛋白质表达量与不添加时目标蛋白质表达量相当(图4)。以BSA为标准样品,经Bradford测定蛋白质浓度,并通过Quantity One软件进行灰度扫描,确定目标蛋白质浓度为1.8 mg/mL。说明该无细胞蛋白质合成系统在此条件下可高效合成ω-3脂肪酸脱饱和酶,脂质体的添加不会影响目标蛋白质的表达量。

图4 添加脂质体后ω-3脂肪酸脱饱和酶在麦胚无 细胞蛋白质合成系统中表达情况的SDS-PAGE(A) 和Western Blot分析(B)Fig.4 The SDS-PAGE(A) and Western Blot analysis(B) of ω-3 fatty acid desaturase expressed in the wheat germ cell-free protein expression system in the presence of liposome.注:M:蛋白质分子量标准; 1:ω-3脂肪酸脱饱和酶在不添加脂质体的麦胚无细胞蛋白质合成系统中的表达;2:ω-3脂肪酸脱饱和酶在添加脂质体的麦胚无细胞蛋白质合成系统中的表达。
2.4 ω-3脂肪酸脱饱和酶重组蛋白质的纯化
脂质体与翻译后的ω-3脂肪酸脱饱和酶结合形成脂质体蛋白质复合物(proteoliposome),经Accudenz密度梯度离心后,脂质体蛋白质复合物处于离心管上方约30%处,未结合脂质体的重组蛋白质则存在于离心管底部。SDS-PAGE和Western Blot结果分析显示经密度梯度离心纯化后在45 kDa处有一明显的目标蛋白质条带(图5),Quantity One软件分析表明纯化后的重组蛋白质FADS15纯度大于90%。
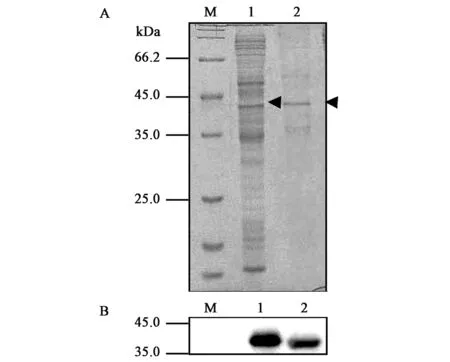
图5 重组蛋白质纯化后的SDS-PAGE(A)和 Western Blot分析(B)Fig.5 SDS-PAGE(A) and Western Blot analysis(B) of the recombinant protein after purification.注:M:蛋白质分子量标准;1:纯化前的ω-3脂肪酸脱饱和酶;2:纯化后的ω-3脂肪酸脱饱和酶。
3 讨论
ω-3脂肪酸脱饱和酶作为催化ω-6脂肪酸生成ω-3脂肪酸的关键酶,近年来获得较多关注。但由于在传统的表达系统中ω-3脂肪酸脱饱和酶的过表达会使宿主细胞生长缓慢,甚至死亡。前期我们尝试在毕赤酵母表达系统里表达ω-3脂肪酸脱饱和酶,实现了在 PichiaPinkTM酵母表达系统中pPinkα-HC-FADS15质粒的重组表达,FADS15基因能够整合到毕赤酵母基因组并进行稳定的遗传表达。重组表达过程中毕赤酵母首先使用 BMGY培养基扩大培养,后使用BMMY培养基诱导 ω-3脂肪酸脱饱和酶表达,中途两次添加0.5%甲醇诱导,48 h后收菌,得到目标蛋白质表达量为0.13 mg/mL。因此蛋白质表达量偏低,使得其相关研究主要局限在其底物偏好性等少数生理功能,无法全面考察其酶学特性及催化活性。本文使用麦胚无细胞蛋白质合成系统,解除了细胞膜对蛋白质合成的限制。膜结合蛋白质在无细胞蛋白质合成系统中以两种表达形式进行[24]。其一是在体系中不添加外源膜结构物质,使得表达的蛋白质以沉淀形式存在,随后将其溶解于合适的去垢剂或脂质体中形成可溶性的脂质体蛋白质复合物,进而实现目标蛋白的分离;另一种是在无细胞反应过程中添加脂质体,使翻译后的蛋白质直接嵌入到脂质体中重构得到脂质体蛋白质复合物。在本工作中我们同时考察了这两种表达形式。但是实验结果表明在不添加外源的脂质体时,麦胚无细胞蛋白质合成系统表达出来的以沉淀形式存在的ω-3脂肪酸脱饱和酶,无法在与脂质体或1%的Fos-Choline 16混合孵育之后被整合为可溶性蛋白质。采用脂质体或表面活性剂与反应后沉淀蛋白质混合的实验方案无法获得可溶蛋白。
因此在上述实验的基础上,我们采取第二种方案,在麦胚无细胞蛋白质合成体系中添加脂质体以使目标蛋白质正确折叠[25],获得可溶性目标膜蛋白。通过对脂质体添加浓度及时间的优化,实现了蛋白质的可溶性表达,并确定在反应进行10.5 h 时添加终浓度为1.2 mg/mL的脂质体为最佳条件。此时ω-3脂肪酸脱饱和酶表达量达到毫克级(1.8 mg/mL),可溶蛋白质表达量为0.23 mg/mL。经进一步纯化,每毫升反应液可得到57 μg纯度大于90%的重组蛋白质,这为ω-3脂肪酸脱饱和酶的催化特性研究和晶体结构分析提供了充足的原材料,同时也为脂肪酸脱饱和酶类膜蛋白的合成提供了一种简便高效的表达方法。
[1] Bajpai P K, Bajpai P, Ward O P. Arachidonic acid production by fungi[J]. Appl. Environ. Microbiol., 1991, 57(4): 1255-1258.
[2] López A D, Garcia-Maroto F, Rodriguez-Ruiz J,etal.. Evolution of the membrane-bound fatty acid desaturases[J]. Biochem. System. Ecol., 2003, 31(10): 1111-1124.
[3] Sakuradani E, Abe T, Iguchi K,etal.. A novel fungal omega3-desaturase with wide substrate specificity from arachidonic acid-producingMortierellaalpina1S-4[J]. Appl. Microbiol. Biotechnol., 2005, 66: 648-654.
[4] Sakamoto T, Los D A, Higashi S,etal.. Cloning of ω-3 desaturase from cyanobacteria and its use in altering the degree of membrane-lipid unsaturation[J]. Plant Mol. Biol., 1994, 26(1): 249-263.
[5] Pereira S L, Huang Y S, Bobik E G,etal.. A novel ω-3 fatty acid desaturase involved in the biosynthesis of eicosapentaenoic acid [J]. Biochem. J., 2004, 378: 665-671.
[6] Higashiyama K, Yaguchi T, Akimoto K,etal.. Enhancement of arachidonic acid production byMortierellaalpina1S-4[J]. J. Am. Oil Chemist. Soc., 1998, 75(11): 1501-1505.
[7] Zhang X, Li M, Wei D,etal.. Identification and characterization of a novel yeast omega3-fatty acid desaturase acting on long-chain n-6 fatty acid substrates fromPichiapastoris[J]. Yeast, 2008, 25(1):21-27.
[8] Ando A, Sumida Y, Negoro H,etal.. Establishment ofAgrobacteriumtumefaciens-mediated transformation of an oleaginous fungus,Mortierellaalpina1S-4, and its application for eicosapentaenoic acid producer breeding[J]. Appl. Environ. Microbiol., 2009, 75(17): 5529-5535.
[9] Tocher D R, Leaver M J, Hodgson P A. Recent advances in the biochemistry and molecular biology of fatty acyl desaturases [J]. Prog. Lipid Res., 1998, 37(2-3): 73-117.
[10] Suzette L, Pereir A, Amanda E,etal.. Identication of two novel microalgal enzymes involved in the conversion of the ω3-fatty acid, eicosapentaenoic acid, into docosahexaenoic acid[J]. Biochem. J., 2004, 384:357-366.
[11] Oura T, Kajiwara S.SaccharomyceskluyveriFAD3 encodes an ω3 fatty acid desaturase[J]. Microbiology, 2004, 150(6): 1983-1990.
[12] Damude H G, Zhang H, Farrall L,etal.. Identification of bifunctional delta12/omega3 fatty acid desaturases for improving the ratio of omega3 to omega6 fatty acids in microbes and plants[J]. Proc. Natl. Acad. Sci. USA, 2006,103(25):9446-9451.
[13] Xue Z, He H, Hollerbach D,etal.. Identification and characterization of new Δ-17 fatty acid desaturases[J]. Appl. Microbiol. Biotechnol., 2013, 97(5): 1973-1985.
[14] Chen H, Gu Z, Zhang H,etal.. Expression and purification of integral membrane fatty acid desaturases[J]. PLoS ONE, 2013, 8(3): e58139.
[15] Endo Y, Sawasaki T. High-throughput, genome-scale protein production method based on the wheat germ cell-free expression system[J]. Biotechnol. Adv., 2003, 21(8): 695-713.
[16] Liguori L, Marques B, Villegas-Méndez A,etal.. Production of membrane proteins using cell-free expression systems[J]. 2007, 8(19): 3933-3946.
[17] Nozawa A, Ogasawara T, Matsunaga S,etal.. Production and partial purification of membrane proteins using a liposome-supplemented wheat cell-free translation system[J]. BMC Biotechnol., 2011, 11(1): 35.
[18] Ishihara G, Goto M, Saeki M,etal.. Expression of G protein coupled receptors in a cell-free translational system using detergents and thioredoxin-fusion vectors[J]. Protein Express. Purif., 2005, 41(1): 27-37.
[19] Goren M A, Fox B G. Wheat germ cell-free translation, purification, and assembly of a functional human stearoyl-CoA desaturase complex[J]. Protein Express. Purif., 2008, 62(2): 171-178.
[20] Shimono K, Goto M, Kikukawa T,etal.. Production of functional bacteriorhodopsin by anEscherichiacolicell-free protein synthesis system supplemented with steroid detergent and lipid[J]. Protein Sci., 2009, 18(10): 2160-2171.
[21] 萨姆布鲁克,塞尔D W. 分子克隆实验指南(第三版)[M]. 北京:科学出版社,2002.
[22] Goren M A, Nozawa A, Makino S,etal.. Cell-free translation of integral membrane proteins into unilamelar liposomes[J]. Methods Enzymol., 2009, 463: 647-673.
[23] Klammt C, Schwarz D, Löhr F,etal.. Cell-free expression as an emerging technique for the large scale production of integral membrane protein[J]. FEBS J., 2006, 273(18): 4141-4153.
[24] Zhang X, Sheng J, Huang L,etal.. High-level soluble expression of one model olfactory receptor (ODR-10) inEscherichiacolicell-free system[J]. World J. Microbiol. Biotechnol., 2014,30(3): 893-901.
[25] Los D A, Murata N. Structure and expression of fatty acid desaturases[J]. Biochim. Biophys. Acta, 1998, 1394(1): 3-15.
Expression of the ω-3 Fatty Acid Desaturase fromMortierellaalpinain Wheat-germ Cell-free Protein Expression System
YANG Qin, CHEN Hai-qin*, CHEN Si, GU Zhen-nan, ZHANG Hao, CHEN Yong-quan, CHEN Wei
SchoolofFoodScienceandTechnology,JiangnanUniversity,JiangsuWuxi214122,China
Cell-free protein expression system as a tool of rapid and convenient protein expression has been widely used in producing membrane proteins and antibacterial peptides that have certain toxicity to living cells. In this paper, the membrane-bound ω-3 fatty acid desaturase gene, which encoding the key enzyme involving in the polyunsaturated fatty acid biosynthesis pathways in the oleaginous fungiMortierellaalpina, was cloned into the in vitro expression vector pIVEX WG1.4, and expressed in the wheat-germ cell-free protein expression system. To get the correctly folded ω-3 fatty acid desaturase FADS15, liposomes that was able to relocate the expressed membrane protein into the phospholipid bilayer were also added in the process of cell-free protein synthesis, and the production of FADS15 reached 1.8 mg/mL, after accudenz density gradient centrifugation, the purity of FADS15 was higher than 90%. This research work paved the way for the further study of catalytic property and crystal structure analysis of this enzyme.
Mortierellaalpina; ω-3 fatty acid desaturase; wheat-germ cell-free protein expression system; liposome; protein purification
2016-05-12; 接受日期:2016-06-03
国家自然科学基金项目(21276108)资助。
杨芹,讲师,主要从事脂质研究。E-mail:qyang@jiangnan.edu.cn。*通信作者:陈海琴,教授,研究方向为微生物与分子生物学。E-mail: haiqinchen@jiangnan.edu.cn
10.3969/j.issn.2095-2341.2016.05.07
