基于宏基因组技术的聚磷菌研究进展
2016-12-21陈禹保
徐 媛 , 陈禹保*
(1.北京市计算中心,北京 100094;2.北京市基因测序与功能分析工程技术研究中心,北京 100094)
基于宏基因组技术的聚磷菌研究进展
徐 媛1,2, 陈禹保1,2*
(1.北京市计算中心,北京 100094;2.北京市基因测序与功能分析工程技术研究中心,北京 100094)
聚磷菌是一类非常重要的工程菌,广泛应用于污水处理厂生物除磷过程。聚磷菌可以吸收超过自身所能利用的数倍的磷,在体内合成聚磷化合物从而达到生物除磷的目的。在过去的几年里,宏基因组学以及测序技术的发展大大推动了对聚磷菌物种组成及其磷代谢过程的认识。本文主要对宏基因组技术进行介绍并对近几年基于宏基因组技术深入研究聚磷菌的文章进行综述,以期对这类重要的微生物的生理功能、代谢途径和物种多样性进行全面的了解。
聚磷菌;宏基因组;代谢途径
聚磷菌是一类特殊微生物的统称,广泛存在于活性污泥中,是一类非常重要的工程菌。在好氧/厌氧交替的条件下可以吸收远远超过其自身所能利用的磷,并以聚磷物的形式存在于细胞内,达到生物除磷的目的。通过对厌氧装置和好氧装置中的化学元素进行测定[1],人们对活性污泥中微生物除磷代谢过程有了初步的认识。在厌氧阶段,聚磷菌吸收污水中的小分子有机物(主要是挥发性脂肪酸,VFA),将其转化为聚羟基烷酸(PHA)储存在细胞内,同时释放出磷,这个过程所需的能量来自于分子内聚磷和糖原的水解。好氧阶段,聚磷菌氧化其积累的大量PHA,释放能量,吸收超过自身所需的磷,并在体内合成聚磷和糖原,最后通过排放剩余污泥达到除磷的效果。尽管聚磷菌用于污水除磷已经有几十年的历史,但是应用聚磷菌进行污水去磷的系统还是不稳定,人们在应用的过程中还是会遇到一些不能预知和不能解决的问题。了解哪类微生物参与了污水除磷过程以及它们是如何工作的,可以为研制更好的除磷装置提供理论基础,也可以更有效地解决除磷过程中遇到的问题。聚磷菌的分离培养非常困难,至今为止还没有一株纯培养物,限制了人们对其生理过程的认识。宏基因组技术的发展,促进了人们对这类特殊微生物的认识。宏基因组技术主要以环境样品中的微生物群体基因组为研究对象,以测序分析为研究手段,研究环境中微生物多样性、种群结构和功能活性等。随着高通量测序技术的发展,宏基因组学已经成为微生物研究领域一项非常普遍的研究方法。目前有两种主要的研究策略,基于单一基因的靶向测序研究策略和基于全基因组的研究策略,前者只关注单一基因,目前应用最多的是16S rDNA基因,此基因进行高通量测序,可以对微生物种群结构进行分析,但是对其功能的分析仍欠缺。后者可以同时进行物种多样性和功能多样性的分析。本文主要就近年来基于宏基因组技术对聚磷微生物及其除磷过程的研究进行综述,以对其研究进展做全面了解。
1 基因组信息
Martín等[2]通过对强化生物除磷工艺(EBPR)污泥中的聚磷菌进行富集培养,分别得到了以乙酸和丙酸为碳源的聚磷菌的富集物,来自美国威斯康辛州的污泥富集物含有80%的聚磷菌,来自澳大利亚昆士兰的污泥富集物含有60%的聚磷菌,对两个富集物进行宏基因组测序,得到聚磷菌的基因组草图。尽管这两个活性污泥的培养方式和来源都不同,但是主要的聚磷菌菌株共同拥有95%的基因。通过后续的测序、拼接和注释,得到了威斯康辛富集物中聚磷菌的全基因组,命名为Candidatus Accumulibacter clade IIA str. UW-1。这株菌含有1个5.06 Mbp染色体和3个质粒,整个菌株基因组大小为5.31 Mbp,含有4 735个基因,3 000多个基因具有明确的功能注释信息,可以完成对整个代谢通路的重建。Flowers等[3]通过宏基因组测序得到了另一株聚磷菌的基因组草图,并把这株聚磷菌命名为Candidatus Accumulibacter Clade IA str. UW-2。尽管这两株菌的16S rRNA序列有98%的一致性,但是全基因组信息却有很大的差异,平均核苷酸同源性(ANI)只有78.3%。与UW-1不同的是,UW-2缺少氮固定和碳固定的基因,但是它们拥有共同的反硝化基因和聚磷代谢途径。Skennerton等[4]通过宏基因组深度测序的方法得到了8个不同聚磷菌的基因组信息,其中7个聚磷菌在之前并没有被测序过。通过比较基因组学的研究发现,这些聚磷菌除了拥有共同的碳代谢和磷代谢外,每个聚磷菌还拥有大约700个特有的基因,主要区别在于硝酸盐还原和反硝化过程等,作者认为这些代谢差异导致了聚磷菌的生态位分化并且为优化脱氮除磷过程提供了新的机会。通过这些基因组信息,研究人员对聚磷菌的代谢途径进行了深入研究。
1.1 碳代谢途径
乙酸是研究EBPR系统的主要碳源,通过基因预测发现乙酸通透酶(ActP)通过质子泵或者钠离子泵在细胞膜上转运乙酸。乙酸转运到细胞膜内后通过acs 或者AckA/pta 对其进行活化,acs 和AckA/pta 分别执行高亲和性和低亲和性的乙酸激活,都可以与Actp形成基因簇。通过对蛋白组进行分析,发现acs是主要的基因激活途径[5]。厌氧条件下,乙酸辅酶A利用糖原降解过程中提供的还原力NAD(P)H转化为PHAs。聚磷菌的糖原降解途径是EMP还是ED途径一直存在争论,通过对基因组信息的分析,发现聚磷菌缺少编码ED途径的基因,解决了这一问题。在转录水平对基因进行表达分析,也再一次证明了这个过程[6]。
好氧微生物通过TCA循环进行糖代谢产生能量,由基因组信息发现聚磷菌也具有TCA代谢过程,那么它们在厌氧条件下是否执行TCA循环以及它们是如何做到的?Burow等[7]研究发现,TCA循环中的关键酶SDH在厌氧条件下表达,并且当SDH被抑制后乙酸吸收和PHA产生的速率都会下降,暗示了在厌氧条件下TCA循环参与了NAD(P)H的产生。Zhou等[8]通过碳代谢通路测定实验,发现在厌氧条件下TCA循环依赖于糖原的浓度,说明完整的TCA循环参与了厌氧代谢过程中。
1.2 多聚磷酸盐代谢途径
基因分析发现2个磷酸盐透性酶参与了低亲和性的磷代谢系统(Pit),3个非常保守的基因簇参与了高亲和性的磷代谢系统(Pst)[2]。冗余的磷代谢系统保证了聚磷菌在高磷和低磷环境下都能高效地吸收磷。聚磷菌通过聚磷激酶ppk1以ATP为底物合成聚磷化合物,基因组分析发现,ppk1可以与外聚磷酸酶基因(ppx)和3′,5′二磷酸合成酶(RelA)形成ppx-ppk1-relA基因簇,共同调控聚磷化合物的合成。转磷酸酶(PAP)可以转移聚磷化合物的磷酸集团,使AMP磷酸化生成ADP,并进一步生成ATP。
1.3 硝酸盐还原途径
研究发现,聚磷菌在有硝酸盐的情况下可以硝酸盐为电子受体,而不需要氧气,以反硝化过程释放的能量进行聚磷。 但是,对得到的聚磷菌基因组进行分析,并没有发现反硝化基因[2]。通过室内培养试验发现了两类形态不同的聚磷菌[9-10],一类是以乙酸为底物富集的球形聚磷菌,它们不能以硝酸盐做为电子受体;一类是以丙酸为底物富集的杆状聚磷菌,它们可以硝酸盐和亚硝酸盐做为电子受体。Guisasola等[11]发现,以亚硝酸盐为电子受体的污泥中主要以球状聚磷菌为主,不能还原硝酸盐。研究发现它们依赖于周围的微生物为其提供亚硝酸盐。Clade IIA属于球状聚磷菌,不能还原硝酸盐,印证了基因组信息的发现。
1.4 聚磷菌的分子标签基因
微生物生态学领域应用最为广泛的分子标签基因是16S rRNA基因,但是对于聚磷菌的研究发现,16S rRNA基因的灵敏度并不够区分不同类型的聚磷菌[12]。16S~23S基因区间序列[12]、ppk1基因[12]、nirS基因[13]和phaC基因[14]都曾被用作分析标签来研究环境中聚磷菌的多样性分布。ppk1基因最终以稳定性和高灵敏性得到了认可并广泛应用于科研工作中。ppk1基因在聚磷菌中只有一个拷贝,它的进化速度比16S rRNA基因快4倍,可以做为一个很好的分子标签来区分聚磷菌[15]。通过对ppk1基因进行研究,McMahon 等[16]发现聚磷菌可以分为两种类型(类型I和类型II),并且ppk1基因的高分辨性可以使每个类型的聚磷菌分为多个亚型(类型IA~IE,类型IIA~IIF),说明聚磷菌的多样性远远大于之前发现的。通过16S rRNA基因和ppk1基因比较建树分析,发现它们对聚磷菌的分类具有高度的一致性[12]。通过ppk1基因对聚磷菌进行分类,可以解决很多以前未能解决的问题。例如,得到全基因组测序信息的聚磷菌UW-2[2],它缺少硝酸盐还原酶,属于clade IIA。通过一系列的研究发现,在环境中clade IA 的聚磷菌确实可以利用硝酸盐进行聚磷,而clade IIA的聚磷菌不可以利用硝酸盐。
2 聚磷菌的分布及影响因素
聚磷菌最初是在乙酸富集的生物反应器活性污泥中被发现[17],这个环境中有着丰富的碳源和磷源。之后在活性污泥系统中,聚磷菌被多次发现且其含量可达到40%~80%。最近的研究发现,在营养贫乏的淡水环境(湖泊、小河)中也有聚磷菌的分布。通过对Accumulibacter clade II A 基因组注释分析,发现聚磷菌有两套聚磷机制[2]:一个是低亲和性的聚磷途径,一个是高亲和性的聚磷途径。它们分别在高磷和低磷的环境中发挥作用,解释了为什么在营养贫乏的环境中也有聚磷菌存在。这个发现促使研究人员通过标签基因ppk1在自然的淡水环境中寻找更多的聚磷微生物[18]。研究发现聚磷菌在环境中的分布非常广泛,具有丰富的多样性,不同环境中的聚磷菌群落结构不同,且可分为两种类型,11个亚型(图1)[19]。
自从聚磷菌被发现后,大量的研究关注于环境因子对聚磷菌群落结构的影响以及它们对活性污泥系统除磷功能的影响。对5个不同的除磷系统进行研究后发现聚磷菌可以占到整个生物细胞的9%~24%,且80%的聚磷菌具有很强的聚磷功能[20]。另外一项研究发现,聚磷菌占到环境中细菌的5%~20%,且与环境中的COD含量和磷的比率有着很好的正相关关系[21]。但是,也在一些环境中发现聚磷菌并不是主要的除磷微生物[22],尽管在一些环境中它的含量并不低[23]。说明在研究聚磷微生物的贡献时需要考虑结合多种方法(如转录组、化学测定等),不能仅仅依靠数量来确定。
基因组水平的分析可以更好地解释聚磷菌对环境的适应性。He 等[6]根据聚磷菌基因组信息设计了关键基因的芯片,研究了实验室聚磷菌富集物在不同条件下聚磷菌基因的表达情况,研究发现当培养进入好氧阶段初期,参与基因转录、翻译和蛋白运输的基因显著上调,说明聚磷菌从厌氧阶段进入好氧阶段初期进行了快速的繁殖。同时发现在好氧初期,参与TCA循环和合成ATP酶的基因都显著上调,验证了聚磷菌在有氧条件下通过TCA循环氧化细胞内的有机化合物提供ATP供细胞生长。He等[19]根据Accumulibacter clade IIA的全基因组信息设计引物,在转录水平研究了参与乙酸代谢途径的酶以及聚磷酶在不同环境因子下的表达差异,阐明了碳源代谢与聚磷过程在不同环境因子下的相互关系。
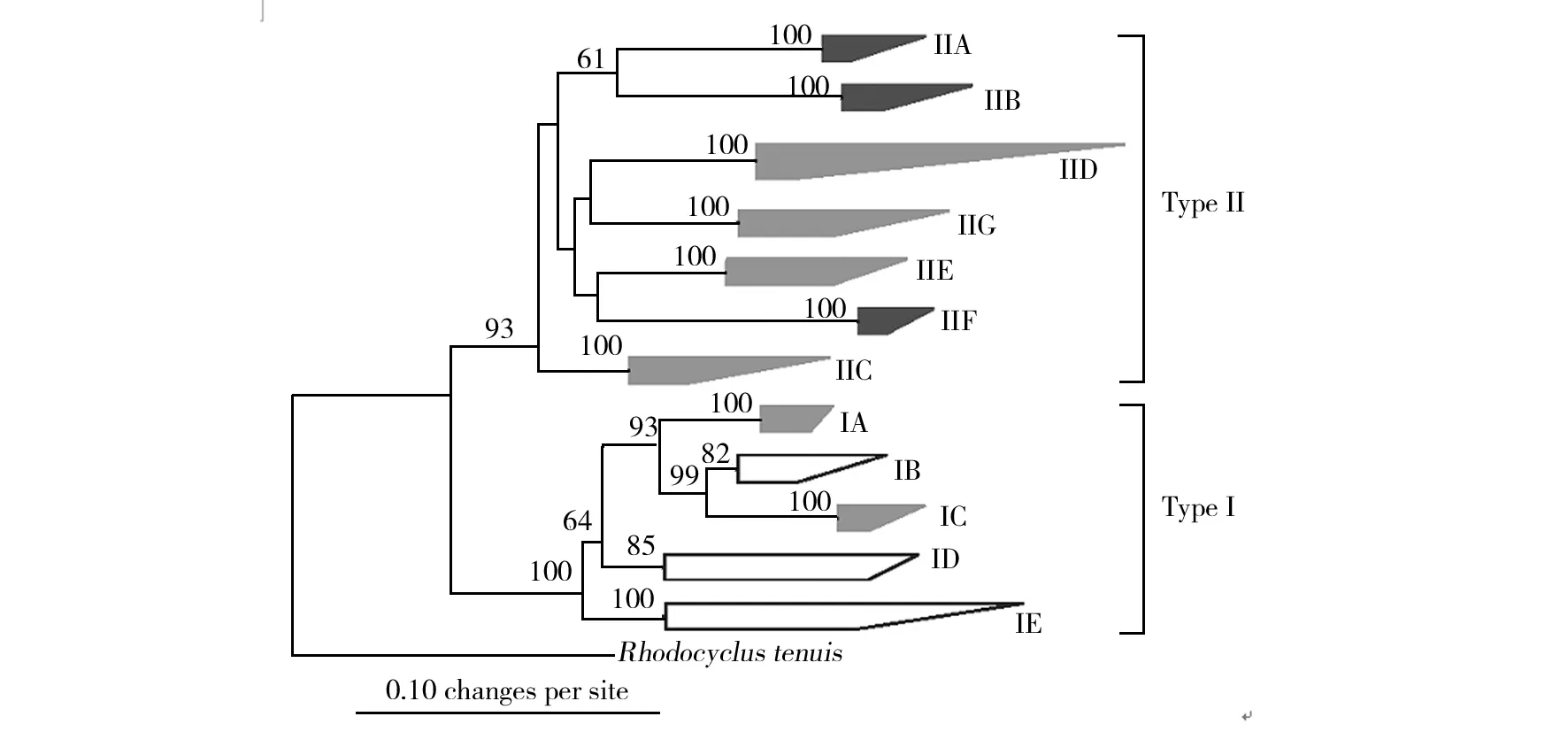
图1 聚磷菌的系统发育树[19]Fig.1 The phylogenetic tree of Accumulibacter[19]
3 展 望
宏基因组测序及对聚磷菌全基因组的组装促进了对聚磷菌的生理、物种及功能多样性的认识和研究,解决了很多以前不能解决的问题,同时也提出了更多新的研究视角。上述对于聚磷菌的研究建立在仅有的几个单一环境下富集培养的聚磷菌的参考基因组之上,而Albertsen 等[24]对EBPR体系进行宏基因组测序,发现拼接的很多聚磷菌的重叠群(contig)和参考菌株的相似性很小,说明环境中的聚磷菌是由多个菌株构成的,通过分子标签ppk1基因的研究也发现了类似的结果。仅以1株参考菌株对环境中的聚磷菌进行研究是不够的,将造成研究的偏差。首先,聚磷菌生活的环境具有非常丰富的多样性,不同环境下聚磷菌代表菌株的基因信息、代谢途径并不完全相同,仅用现有的基因信息进行研究,会造成重要信息的流失。其次,聚磷菌也有多态性,其代表菌株很有可能不止一种,研究这些聚磷菌代谢的多样性以及它们对除磷的贡献也有非常重要的研究与应用价值。但是,通过分离培养或者富集培养得到新的代表菌株困难重重,需要在复杂的环境中,不通过富集培养直接从宏基因组、宏转录组以及宏代谢组的角度,更加深入地研究聚磷菌,扩展对环境中聚磷微生物的认识。
[1] Oehmen A, Lemos PC, Carvalho G, et al. Advances in enhanced biological phosphorus removal: from micro to macro scale[J]. Water Research, 2007, 41(11): 2271-2300.
[2] Martín HG, Ivanova N, Kunin V,et al. Metagenomic analysis of two enhanced biological phosphorus removal (EBPR) sludge communities[J].Nature Biotechnology, 2006, 24(10): 1263-1269.
[3] Flowers JJ, He S, Malfatti S, et al. Comparative genomics of two ‘Candidatus Accumulibacter’ clades performing biological phosphorus removal[J]. The ISME Journal,2013,7(12):2301-2314.
[4] Skennerton CT, Barr JJ, Slater FR, et al. Expanding our view of genomic diversity in Candidatus Accumulibacter clades[J]. Environmental Microbiology, 2015, 17(5):1574-1585.
[5] Wilmes P, Andersson AF, Lefsrud MG, et al.Community proteogenomics highlights microbial strain-variant protein expression within activated sludge performing enhanced biological phosphorus removal[J].The ISME Journal, 2008, 2(8):853-864.
[6] He S, Kunin V, Haynes M, et al. Metatranscriptomic array analysis of ‘Candidatus Accumulibacter phosphatis’ enriched enhanced biological phosphorus removal sludge[J]. Environmental Microbiology, 2010, 12(5): 1205-1217.
[7] Burow LC, Mabbett AN, Blackall LL. Anaerobic glyoxylate cycle activity during simultaneous utilization of glycogen and acetate in uncultured Accumulibacter enriched in enhanced biological phosphorus removal communities[J]. The ISME Journal, 2008, 2(10): 1040-1051.
[8] Zhou Y, Pijuan M, Oehmen A, et al. The source of reducing power in the anaerobic metabolism of polyphosphate accumulating organisms (PAOs)-a mini-review[J]. Water Science & Technology, 2010, 61(7): 1653-1662.
[9] Carvalho G, Lemos PC, Oehmen A, et al. Denitrifying phosphorus removal: linking the process performance with the microbial community structure[J]. Water Research, 2007, 41(19): 4383-4396.
[10]Kim JM, Lee HJ, Lee DS, et al. Characterization of the denitrification-associated phosphorus uptake properties of "Candidatus Accumulibacter phosphatis" clades in sludge subjected to enhanced biological phosphorus removal[J]. Applied and Environmental Microbiology, 2013, 79(6): 1969-1979.
[11]Guisasola A, Qurie M, Vargas MM, et al. Failure of an enriched nitrite-DPAO population to use nitrate as an electron acceptor[J]. Process Biochemistry, 2009, 44(7): 689-695.
[12]He S, Gall DL, McMahon KD. "Candidatus Accumulibacter" population structure in enhanced biological phosphorus removal sludges as revealed by polyphosphate kinase genes[J]. Applied and Environmental Microbiology, 2007, 73(18): 5865-5874.
[13]Miyauchi R, Oki K, Aoi Y, et al. Diversity of nitrite reductase genes in ‘Candidatus Accumulibacter phosphatis’-dominated cultures enriched by flowcytometric sorting[J]. Applied Environment Microbiology,2007, 73(16): 5331-5337.
[14]Wang Q, Shao Y, Huong VTT, et al. Fine-scale population structure of Accumulibacter phosphatis in enhanced biological phosphorus removal sludge[J]. Journal of Microbiology Biotechnology,2008, 18(7):1290-1297.
[15]Kunin V, He S, Warnecke F, et al. A bacterial metapopulation adapts locally to phage predation despite global dispersal[J]. Genome Research, 2008, 18(2): 293-297.
[16]McMahon KD, Yilmaz S, He S, et al. Polyphosphate kinase genes from full-scale activated sludge plants[J].Applied Microbiology and Biotechnology,2007, 77(1): 167-173.
[17]Hesselmann RPX, Werlen C, Hahn D, et al. Enrichment, phylogenetic analysis and detection of a bacterium that performs enhanced biological phosphate removal in activated sludge[J]. Systematic and Applied Microbiology,1999, 22(3): 454-465.
[18]Peterson SB, Warnecke F, Madejska J, et al. Environmental distribution and population biology of Candidatus Accumulibacter, a primary agent of biological phosphorus removal[J]. Environmental Microbiology, 2008, 10(10): 2692-2703.
[19]He S, McMahon KD. Candidatus Accumulibacter’gene expression in response to dynamic EBPR Conditions[J]. The ISME Journal,2011,5(2): 329-340.
[20]He S, Gu AZ, McMahon KD. Progress toward understanding the distribution of Accumulibacter among full-scale enhanced biological phosphorus removal systems[J]. Microbial Ecology, 2008,55(2):229-236.
[21]Gu AZ, Saunders A, Neethling JB, et al. Functionally relevant microorganisms to enhanced biological phosphorus removal performance at full-scale wastewater treatment plants in the United States[J]. Water Environment Research, 2008, 80(8): 688-698.
[22]Beer M, Stratton HM, Griffiths PC, et al. Which are the polyphosphate accumulating organisms in full-scale activated sludge enhanced biological phosphate removal systems in Australia[J].Journal of Applied Microbiology,2006, 100(2):233-243.
[23]Wong MT, Mino T, Seviour RJ, et al. In situ identification and characterization of the microbial community structure of full-scale enhanced biological phosphorous removal plants in Japan[J]. Water Research, 2005, 39(13): 2901-2914.
[24]Albertsen M, Hansen LBS, Saunders AM, et al. A metagenome of a full-scale microbial community carrying out enhanced biological phosphorus removal[J]. The ISME Journal, 2012, 6(6): 1094-1106.
Advances in Accumulibacter Research Based on Macrogenomics Technique
XU Yuan1,2, CHEN Yu-bao1,2
(1.BeijingComputingCtr,Beijing100094; 2.BeijingEngin.Technol.Res.Ctr.ofGeneSequencing&GeneFunctionAnalysis,Beijing100094)
Accumulibacter is a number of important engineering bacteria broadly applied in sewage treatment plant for disposing inorganic phosphate (Pi). These bacteria can assimilate many folds of phosphate in excess of their own need and synthesize polyphosphate intracellularly and accordingly to remove phosphorus biologically. In the past few years, the development of macrogenomics and sequencing technology vastly motivate the understanding of the specific components of Accumulibacter and their phosphate metabolism processes. In this paper, technology of macrogenomics were introduced, articles reported recently based on intensive study on Accumulibacter macrogenomic technology were summarized so as to comprehensively understand the physiological functions, metabolic pathways, specific diversity of this number of the important microbes.
Accumulibacter; macrogenomics; metabolic pathway
北京市科学技术研究院创新团队项目(IG201406C1)
徐媛 女,博士后。研究方向为微生物生态学、宏基因组学。E-mail:xuyuan@bcc.ac.cn
* 通讯作者。男,副研究员。研究方向为生物信息与生物技术经济。Tel:010-59341760,E-mail:chenyb@bcc.ac.cn
2015-05-07;
2015-07-27
Q93-33
A
1005-7021(2016)02-0104-05
10.3969/j.issn.1005-7021.2016.02.018
