山楂PCR-SSCP反应体系与反应条件的优化
2016-12-20撒云俐那冬晨
撒云俐,那冬晨
(山西师范大学生命科学学院,山西临汾 041000)
山楂PCR-SSCP反应体系与反应条件的优化
撒云俐,那冬晨*
(山西师范大学生命科学学院,山西临汾 041000)
[目的]优化山楂PCR-SSCP反应体系与反应条件。[方法]以山楂为材料,采用改良的CTAB法分步提取叶绿体DNA和核DNA,对PCR-SSCP引物进行筛选,同时进行PCR-SSCP反应体系和反应条件的优化,琼脂糖凝胶电泳检测PCR扩增产物,非变性聚丙烯酰胺凝胶电泳检测变性后的PCR扩增产物。[结果] ropB、ropL、rpoC1、ITS2、psbA-trnH 5对引物适用于山楂PCR-SSCP分析。电泳时,采用6%聚丙烯酰胺凝胶(交联度29∶1),4 ℃恒温,200 V恒压,上样缓冲液(不含甘油)与PCR产物等量混合,98 ℃碱(1%NaOH)变性15 min,电泳缓冲液为0.5×TBE,电泳3~4 h可得到较好的山楂PCR-SSCP电泳图谱。[结论]建立了山楂PCR-SSCP最优反应体系与反应条件,为山楂SSCP分析奠定基础。
山楂;PCR-SSCP;引物;反应体系;反应条件
山楂(Crataegusspp.)隶属于蔷薇科(Rosaceae)苹果亚科山楂属,广泛分布于亚洲、欧洲、中北美洲及南美洲北部,是蔷薇科的一种重要植物,也是起源于我国的特产果树[1]。目前对山楂的研究主要集中在化学成分分析、药理、种质资源评价、育种和遗传多样性研究等方面。用于山楂果树研究的分子标记技术主要有同工酶技术、RAPD、ISSR、SSR、PCR-RFLP等[2-3]。山楂种质资源的研究仍以传统分类法为主,已无法满足对山楂种质创新和产品更新的需求[4]。因此,应用分子生物学技术,从分子水平上探讨山楂种质资源分类,为山楂种质资源保存、评价和鉴定提供依据[2]。
SSCP(Single Strand Conformation Ploymorphism)技术即DNA单链构象多态性技术,基本原理是DNA分子中单个碱基的变化,可造成DNA单链构象的巨大变化。DNA片段变性后产生sDNA,sDNA由于分子内氢键等非共价键的作用,形成二级结构,最终形成单链构象。相同长度不同构象sDNA的电泳速率不同,电泳后在凝胶中停留的位置不同,从而将在双链水平无法检测的DNA微小差异在单链水平上检测出来[5]。SSCP技术是随着对人类基因组的研究而发展起来的用于检测碱基突变的技术,同时也是分子遗传标记中最常用的方法之一[6]。
近年来,SSCP技术不断发展和完善,与PCR技术相结合建立的PCR-SSCP技术,具有快速、简便、灵敏度高、需要样品少和适于大样本筛选等优点,被广泛应用于分子生物学的各种领域,为开展群体遗传分析、基因分型、检测由基因突变引起的各种遗传病等研究提供了有力工具[6-7]。但仍存在一些缺陷,如对大于300 bp的DNA片段,随着DNA片段长度的增加,检测的敏感性逐渐降低[6]。同时,SSCP技术也易受上样缓冲液、变性时间及温度、电泳条件等因素的影响,目前在已有的报道中,SSCP分析所用的方法和试验条件由于研究对象的不同而存在较大差别。笔者对山楂SSCP分析的前期试验过程进行优化,以期为山楂SSCP分析奠定基础。
1 材料与方法
1.1 试验材料 山楂幼嫩叶片采于山西师范大学校园内。
1.2 试验方法
1.2.1 DNA的提取。采用改良的CTAB法进行叶绿体DNA和核DNA的分步提取[8-9],具体步骤:①采摘山楂新鲜幼嫩叶片,洗净,吸干水分,放入预冷的研钵中,加入适量预冷的0.14 mol/L NaCl溶液,快速充分研磨成浆。②吸取植物浆液1.0 mL放入1.5 mL离心管中。③4 ℃、1 000 r/min离心20 min,取上清液于另一离心管(标记为A)中,用于叶绿体DNA的提取。沉淀标记为B,用于核DNA的提取。④A管4 ℃、3 000 r/min离心20 min,弃上清。⑤在A、B管沉淀中均加入600 μL预热的2%CTAB溶液,迅速混匀,65 ℃水浴20 min。⑥冷却至室温,加入等体积的氯仿异戊醇(氯仿∶异戊醇=24∶1),作用15 min,12 000 r/min离心10 min,取上清。重复此步骤2次。⑦加入等体积预冷的异丙醇,轻柔颠倒混匀,室温沉淀5 min后,12 000 r/min离心8 min,弃上清,用70%乙醇洗涤沉淀2次,挥发乙醇至沉淀透明,用适量灭菌的去离子水溶解沉淀,4 ℃保存备用。⑧用1.0%的琼脂糖凝胶电泳检测。
1.2.2 引物筛选。选取psbA-trnH、ropB、ropL、rpoC1、ITS2、ACT2p、SOD、I1-5.8-I2作为候选引物,其序列见表1。
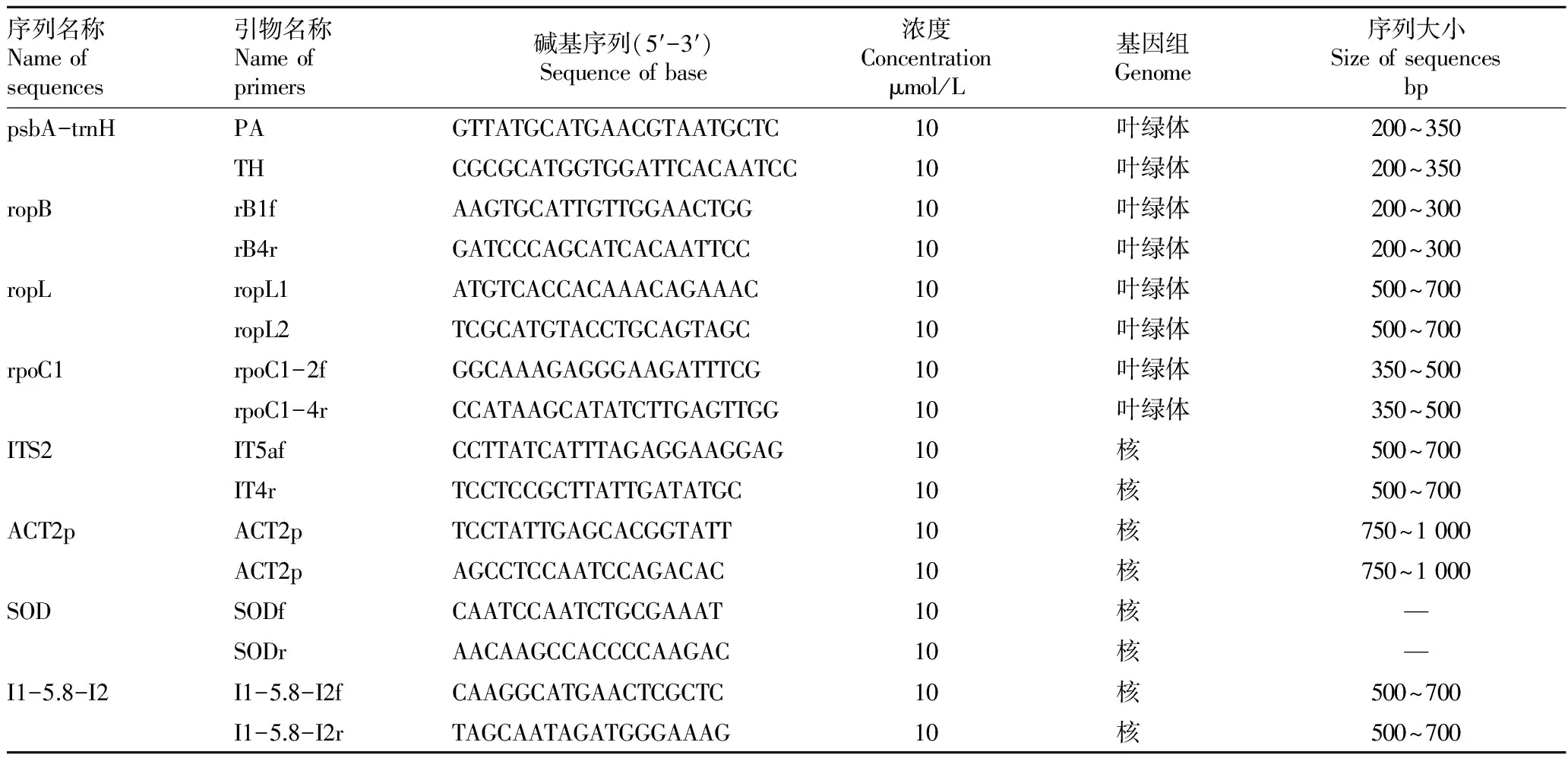
表1 候选引物序列
从上述8对候选引物中分别筛选出适合叶绿体DNA和核DNA进行PCR扩增的引物,同时进行PCR反应体系和反应条件的优化。每对引物对应的PCR反应体系见表2,反应条件见表3。

表2 PCR-SSCP反应体系

表3 PCR-SSCP反应条件
1.2.3 电泳检测。PCR扩增产物用1.0%琼脂糖凝胶电泳(85 V)检测,Gene Green核酸染料染色。
1.2.4 SSCP分析。6%非变性聚丙烯酰胺凝胶的配制参考鲍思元[10]方法,30%丙烯酰胺15.0 mL、10×TBE 2.5 mL、ddH2O 32.5 mL、10%过硫酸铵750.0 μL、TEMED 75.0 μL。上样缓冲液含有1%NaOH,不含甲酰胺,上样缓冲液量∶PCR产物=1∶1,98 ℃变性15 min后,立即置于冰上,取8 μL上样,电泳缓冲液为0.5×TBE。电泳温度4 ℃,电压为200 V,电泳完毕后进行银染,并观察照相。
2 结果与分析
2.1 山楂叶绿体DNA序列引物的筛选 PCR扩增产物的电泳结果表明,psbA-trnH、ropB、ropL、rpoC1 4对引物均可用于山楂PCR-SSCP分析。其中,rpoC1、ropL、ropB 3对引物扩增效果较好,扩增率可达100%,扩增片段长度分别约375、600、300 bp(图1)。psbA-trnH作为引物扩增出的产物具有2条亮带,扩增产物长度约220 bp,另一亮带出现的原因可能是引物二聚体(图2)。

注:M.DL 2000 DNA Marker,CK.对照,1.rpoC1,2.ropL,3.ropB。Note:M.DL 2000 DNA Marker, CK.control , 1.rpoC1, 2.ropL, 3.ropB.图1 rpoC1、ropL和ropB引物PCR-SSCP电泳图谱Fig.1 The PCR-SSCP results of rpoC1,ropL and ropB primers
2.2 山楂核DNA序列引物的筛选 PCR扩增产物的电泳结果表明,ITS2适合作为山楂核DNA的PCR-SSCP分析,扩增率达100%,扩增片段长度为550~600 bp(图2)。I1-5.8-I2、ACT2p、SOD 3对引物不适合作为山楂核DNA PCR-SSCP扩增。其中,I1-5.8-I2和SOD经多次优化,尚未得到稳定的PCR产物。ACT2p经过多次优化,均得到多个非特异性扩增条带。
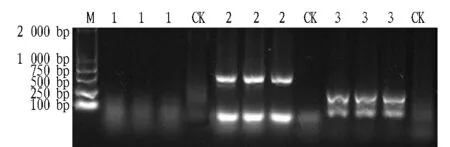
注:M.DL 2000 DNA Marker,CK.对照,1.I1-5.8-I2,2.ITS2,3.psbA-trnH。Note:M.DL 2000 DNA Marker,CK.control,1.I1-5.8-I2,2.ITS2,3.psbA-trnH.图2 ITS2、I1-5.8-I2和psbA-trnH引物PCR-SSCP电泳图谱Fig.2 The PCR-SSCP results of ITS2,I1-5.8-I2 and psbA-trnH primers
2.3 聚丙烯酰胺凝胶电泳 根据琼脂糖凝胶电泳扩增结果,选择6%的凝胶配比,对PCR扩增产物采用碱变性。恒压200 V、4 ℃条件下电泳3~4 h。电泳结束后,蒸馏水冲洗凝胶2~3次,0.2% AgNO3染色液中染色约8 min,再用蒸馏水漂洗2~3次,显色液(3%NaOH、1.5 mL甲醛)中显色约20 min,至出现清晰的条带(图3)。
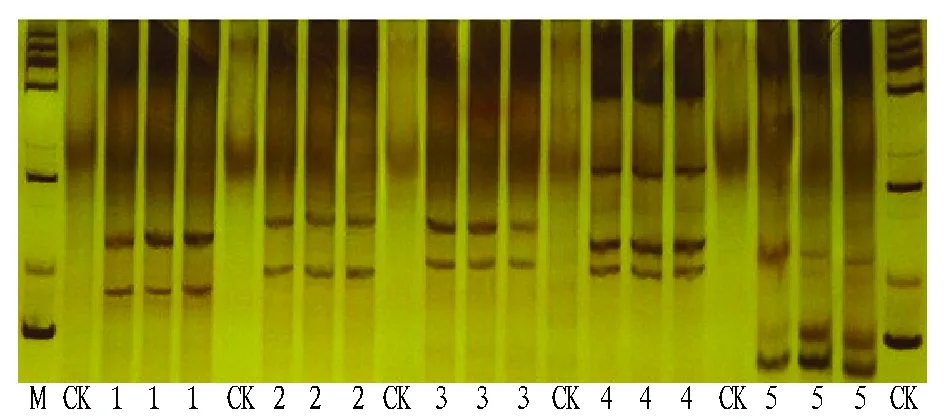
注:M.DL 2000 DNA Marker,CK.对照,1.ropB,2.ropL,3.rpoC1,4.psbA-trnH,5.ITS2。Note:M.DL 2000 DNA Marker,CK.control,1.ropB,2.ropL,3.rpoC1,4.psbA-trnH,5.ITS2。图3 山楂PCR-SSCP电泳图谱Fig.3 The PCR-SSCP results of Crataegus spp.
3 结论与讨论
通过反复试验从候选的8对引物中筛选出5对适于山楂叶绿体DNA(ropB、ropL、rpoC1、psbA-trnH)和核DNA(ITS2)PCR-SSCP分析的引物。在PCR产物进行琼脂糖凝胶电泳时发现凝胶浓度不能过高,上样量不能过多,否则,电泳图谱不整齐,条带拖尾现象较严重,以致于在确定片段大小时,会造成较大误差。
该试验对聚丙烯酰胺凝胶制备时过硫酸铵和TEMED的加入量进行了反复试验,参考鲍思元[10]的制胶方法,配制了6%的非变性聚丙烯酰胺凝胶,结果放置7 h也未能凝固。张俊等[11]认为在排除凝胶成分配比错误的前提下,胶未凝固大多是由于过硫酸铵溶液存放时间过长引起的。鲍思元[10]认为10%的过硫酸铵必须现配现用,4 ℃冰箱保存不超过48 h。根据查阅的信息,重新配制了过硫酸铵(10%),对凝胶成分配比进行了调整,重新制胶,结果凝胶时间不理想,约2 h后才能凝固,且所制的凝胶韧性不好,易碎,对于电泳后的染色、显色操作不利。根据胡建斌等[12]的结果,该研究加大了10%过硫酸铵和TEMED的量,50 mL凝胶中,加入1 000 μL 10%过硫酸铵和100 μL TEMED,发现胶凝固时间明显缩短,但所制的胶凝固不均匀,经过反复试验,最终50 mL凝胶加入750 μL 10%过硫酸铵和75 μL TEMED,约1 h胶即可凝固,且胶的韧性较好,电泳后条带也较整齐。该试验对PCR扩增产物的变性采用碱变性的方法,发现其效果与余桂红等[5]采用98%去离子甲酰胺的变性效果相差不大。
该试验并未对影响山楂SSCP分析的各种因素进行逐一研究,仅优化了山楂PCR-SSCP的反应体系和反应条件,最后对6%非变性聚丙烯酰胺凝胶的制备通过查阅文献资料并经过反复试验确定,电泳结果较好,条带较清晰。该试验结果对以后山楂分子生物学方面的研究以及聚丙烯酰胺凝胶的制备提供参考。参考文献
[1] 柳唐镜,张棵,刘国英,等.中国山楂属植物资源研究和利用现状[J].南方农业学报,2011,42(8):847-852.
[2] 杨明霞,温映红,崔克强,等.中国山楂育种现状及相关分子标记研究进展[J].中国农学通报,2015,31(13):90-94.
[3] 吴俊,魏钦平,史怀瑞,等.分子标记及其在果树种质资源研究中的应用(综述)[J].安徽农业大学学报,2002,29(2):158-162.
[4] 兰士波.中国山楂种质资源的研究现状及利用前景[J].安徽农业科学,2016,44(7):182-184.
[5] 余桂红,唐克轩,马鸿翔,等.小麦SSCP分子标记体系的优化[J].核农学报,2007,21(4):333-338.
[6] 李玉梅,姚纪元,吴静,等.PCR-SSCP技术的研究及应用进展[J].生物技术通报,2007(6):71-74.
[7] 董辉,柳晓利,钱海涛,等.赤眼蜂SSCP分子标记体系的优化[J].沈阳农业大学学报,2011,42(4):428-432.
[8] 郝海叶,张洋,那冬晨.景天属植物叶绿体DNA与核DNA分步提取方法研究[J].安徽农业科学,2013,41(25):10230-10231.
[9] 王文斗,段英俊,那冬晨.植物总DNA提取方法的改进[J].安徽农业科学,2013,41(24):9913-9923.
[10] 鲍思元.DNA变性聚丙烯酰胺凝胶电泳的实验教学改进[J].实验科学与技术,2015,13(2):122-124.
[11] 张俊,潘兆娥,张亚楠,等.棉花SSR-PCR过程中聚丙烯酰胺凝胶电泳操作常见问题的解决与优化[J].中国棉花,2016,43(2):10-13.
[12] 胡建斌,李静,袁鸣,等.PAGE凝胶制备方法和银染方法的改进[J].江西农业大学学报,2009,31(4):742-745.
Optimization of PCR-SSCP Reaction System and Reaction Condition inCrataegusspp.
SA Yun-li, NA Dong-chen*
(Shanxi Normal University School of Life Science, Linfen, Shanxi 041000)
[Objective] The aim was to optimize PCR-SSCP reaction system and reaction condition. [Method] The chloroplast DNA and nuclear DNA inCrataegusspp. were step-by-step extracted by improved CTAB method, the PCR-SSCP primers were selected, and the PCR-SSCP reaction system and reaction conditions were optimizated. The PCR-SSCP productions were tested by agarose gelelectrophoresis, the denatured PCR-SSCP products were analyzed by native polyacrylamide gel. [Result]The results showed that five pairs primers (psbA-trnH、ropB、ropL、rpoC1、ITS2) of all are suitale for PCR-SSCP inCrataegusspp.. The clear electrophoretogram of PCR-SSCP inCrataegusspp. were obtained through using 6% native polyacrylamide gel. (Crosslinking degree is 29∶1) at 4 ℃ and 200 V for 3-4 h, with the volume of the loading buffer (without glycerol) was same with the volume of PCR products, denaturation temperature was 98 ℃ under 1%NaOH, denaturetion time was 15 minutes, electrophoresis buffer was 0.5×TBE.[Conclusion]The study optimize PCR-SSCP reaction system and reaction condition, in order to lay the foundation for optimization of hawthorn SSCP analysis.
Crataegusspp;PCR-SSCP;Primers;Reaction system;Reaction conditions
撒云俐(1994- ),女,山西运城人,硕士研究生,研究方向:生物化学与分子生物学。*通讯作者,副教授,硕士,从事遗传学方面的研究。
2016-10-21
S 188
A
0517-6611(2016)33-0137-03
